九孔鲍MSTN基因cSNP多态性及与生长性状关联分析
艾加林, 栗志民, 2, 刘建勇, 申玉春, 2
九孔鲍基因cSNP多态性及与生长性状关联分析
艾加林1, 栗志民1, 2, 刘建勇1, 申玉春1, 2
1. 广东海洋大学水产学院, 广东 湛江 524088; 2. 湛江市海洋生态与养殖环境重点实验室, 广东 湛江 524088
九孔鲍()是中国南方具有较高经济价值的养殖贝类。肌肉生长抑制素(Myostatin, MSTN)是转化生长因子超家族(TGF-)中参与动物肌肉生长的重要调控因子, 因此基因是动物遗传改良的重要候选基因。为探究九孔鲍基因的多态性及其生长相关性, 实验采用PCR产物直接测序方法对九孔鲍基因进行单核苷酸多态性(SNP)筛选, 对基因SNP与体质量、壳长、壳宽进行关联性分析, 运用实时荧光定量PCR (qRT-PCR)技术检测九孔鲍基因不同基因型的表达水平。结果显示: 在九孔鲍基因中共筛选出9个SNP位点。SNP位点与生长性状关联分析发现, 九孔鲍基因编码区SNP位点g909C>T发生C/T同义突变与九孔鲍体质量、壳长、壳宽显著相关。TT基因型九孔鲍的体质量显著高于CC基因型和TC基因型个体; TT基因型的壳长、壳宽显著高于CC基因型(<0.05)。通过qRT-PCR检测显示, 在九孔鲍基因SNP位点g909C>T的3种基因型中, TC和CC基因型的基因表达量显著高于TT基因型(<0.05)。研究结果表明基因SNP位点g909C>T可作为九孔鲍标记辅助选择育种重要候选标记。
九孔鲍;; 单核苷酸多态性; 生长性状; 关联分析
肌肉生长抑制素(Myostatin, MSTN)又称生长分化因子8 (growth differentiation factor 8, GDF-8), 属于转化生长因子超家族(transforming growth factor, TGF-)成员。肌肉生长抑制素基因是1997年在小鼠中发现的具有抑制肌细胞增殖功能的基因, 为肌肉生长发育的负调控因子(McPherron et al, 1997)。研究表明基因在各物种间的功能高度保守, 例如牛的基因突变, 产生具有双肌表型的比利时蓝牛和肌肉含量比普通牛增加20%的皮埃蒙特牛(Kambadur et al, 1997)。通过基因编辑技术或利用RNA干扰使基因失去抑制作用, 也可使青鳉()(Chiang et al, 2016)、斑马鱼()(Acosta et al, 2005; Lee et al, 2009)等动物的肌肉含量增加。由于基因突变失去抑制肌细胞生长的功能, 使肌肉含量增加(Gao et al, 2014), 进而提高肉产量和经济效益,基因已在哺乳动物(Hill et al, 2010)、鱼类(Gabillard et al, 2013)等脊椎动物, 虾类(Zhuo et al, 2017)、贝类(Fan et al, 2017)等无脊椎动物中开展了相关研究, 且被选作经济物种遗传改良的重要候选基因(Wang et al, 2010; Sánchez-Ramos et al, 2012)。
由于单核苷酸多态性(single nucleotide polymorphism, SNP)位点具有多态性高、分布广和遗传稳定性强等特点, 被广泛运用于标记辅助选择、基因定位及动植物的遗传图谱构建等研究(Brookes, 1999; Wenne, 2018)。在育种中, 通过候选基因中的SNP位点与目标性状进行关联分析, 找到与目标性状相关的位点有助于对个体进行有效筛选(Hemmer-Hansen et al, 2011)。
九孔鲍()属于软体动物门(Mollusca), 腹足纲(Gastropoda), 前鳃亚纲(Prosobranchia), 原始腹足目(Archaeogastropoda), 鲍科(Haliotidae), 主要分布于日本、韩国及中国南方各省(高绪生等, 2000), 具有较高的营养和经济价值。然而由于病害频发, 种质退化, 九孔鲍产量逐年下降, 限制了九孔鲍产业的发展(Cai et al, 2006)。鉴于基因在肌细胞生长过程中的重要作用, 实验采用PCR产物直接测序法对九孔鲍基因进行SNP筛选, 并对九孔鲍基因SNP与九孔鲍的体质量、壳长和壳宽进行关联分析, 运用qRT-PCR技术检测基因在不同基因型中的表达水平, 为九孔鲍标记辅助选择育种提供参考。
1 材料与方法
1.1 材料
实验所用九孔鲍采自深圳大鹏新区某养殖场, 随机选择在相同条件下养殖的1年龄九孔鲍105个, 分别测量壳长、壳宽和体质量, 取右侧壳肌保存于RNA保存液RNAhold (全式金, 北京)中, 4℃保存过夜后转移到-20℃短期保存。
1.2 九孔鲍MSTN基因SNP筛选
按照TransZol Up Plus RNA Kit (全式金, 北京)试剂盒使用说明书提取九孔鲍右侧壳肌总RNA, 1%琼脂糖凝胶电泳检测RNA完整性, 并用核酸微量定量仪(Nanodrop 2000 Thermo Scientific, 美国)检测RNA浓度, 保证A260/280在1.8~2.0之间。根据TransScript one-step gDNA Removal and cDNA Synthesis supermix (全式金, 北京)反转录试剂盒说明书, 利用检测合格的总RNA (1μg)和Oligo(dT) Primer合成cDNA 20μL, 反应程序为: 42℃ 30min, 85℃ 5s,-20℃保存。
利用实验室已获得的九孔鲍基因(登录号: MG870196) cDNA序列设计引物, 随机选择20个九孔鲍, 采用2×EasyTaq® PCR Super Mix (全式金, 北京)进行扩增, 0.5μL模板, SNP-F/R各1μL, 12.5μL 2×EasyTaq® PCR SuperMix, 补水至25μL, PCR反应程序为: 94℃预变性4min; 94℃ 30s, 57℃ 30s, 72℃ 1min, 40个循环, 72℃ 10min, PCR产物经1%琼脂糖凝胶电泳检测, 合格PCR产物送上海生工生物工程(上海)股份有限公司测序, 测序后通过DNAMAN6.0进行序列比对, 筛选出潜在的SNP位点, 并利用chromas软件进行峰图确定SNP位点, 获得潜在的SNP位点和引物。
1.3 九孔鲍MSTN基因多态性及与生长性状关联分析
利用筛选出的SNP引物SNP-1F/R和SNP-2F/R (表1), 对105个九孔鲍进行扩增, 0.5μL模板, SNP- F/R (10μM)各1μL, 12.5μL 2×EasyTaq® PCR SuperMix, 补水至25μL, PCR反应程序为: 94℃预变性4min; 94℃ 30s, 57℃ 30s, 72℃ 1min, 40个循环, 72℃ 10min, PCR产物经1%琼脂糖凝胶电泳检测, 合格PCR产物送上海生工生物工程(上海)股份有限公司测序。通过DNAMAN6.0进行序列比对后, 利用chromas软件对SNP位点进行统计, 某个位点的突变频率大于或等于10%记作一个SNP位点, 其他舍弃。

表1 实验中所用的引物序列
通过序列比对后, 利用chromas软件对SNP位点进行统计、基因分型, 采用SPSS 19.0单因素方差分析(one way ANOVA)对九孔鲍的生长性状与基因SNP位点进行关联分析。此外通过Popgene软件计算各SNP位点的基因型频率、有效基因数(e)、期望杂合度(e)、观测杂合度(o), 并进行哈迪温伯格平衡(HWE)检验。利用PIC_CALC软件计算群体中SNP位点的多态性息含量(PIC)分析。
1.4 九孔鲍MSTN基因SNP g909C>T与表达水平关联分析
在经过SNP g909C>T分型的九孔鲍中, 每种基因型随机选取5个九孔鲍, 取右侧壳肌用于荧光定量分析, 总RNA提取按照TransZol Up Plus RNA Kit (全式金, 北京) 试剂盒使用说明书进行, 根据TransScript one-step gDNA Removal and cDNA Synthesis supermix (全式金, 北京)反转录试剂盒说明书, 利用检测合格的总RNA (1μg)和Oligo(dT) Primer合成cDNA 20μL, 反应程序为: 42℃ 15min, 85℃ 5s,-20℃保存。采用Roche荧光定量PCR, 以-actin为内参基因, 利用基因序列设计荧光定量引物MSTN-RF、MSTN-RR (表1)分析九孔鲍不同基因型基因表达水平, 每份组织样品5个重复。反应体系为: PowerUp SYBR Green Master Mix(2x) (TaKaRa, 大连) 5.0μL, 引物各1.5μL (1μM), cDNA 模板2.0μL (1ng×μL-1), 反应程序: 50℃ 2min; 95℃ 2min, 95℃ 15s, 60℃ 1min, 40个循环, 95℃ 15s, 60℃ 15s。
以基因型中基因表达量最低的为基准, 采用2-ΔΔCT法计算基因相对表达量, 数据采用平均值±标准差(means±SD), 通过SPSS 19.0单因素方差分析(one way ANOVA)进行统计检验, 差异显著性<0.05。
2 结果与分析
2.1 九孔鲍MSTN基因SNP筛选
利用九孔鲍基因设计引物对20个九孔鲍进行扩增、直接测序及序列比对分析后, 获得候选的SNP位点, 使用105个九孔鲍对筛选出的SNP位点进行扩增、直接测序验证, 去除测序失败的个体。序列比对分析结果显示: 在九孔鲍基因中共获得9个SNP位点, 4个转换(A/G、C/T), 5个颠换(A/C、A/T、C/G), 以基因序列第一个碱基为起始位置命名SNP位点。5¢非编码区(5¢-UTR)有1个SNP位点, 为g252G>A; 开放阅读框(ORF)有4个SNP位点, 分别为g414A>C、g558C>A、g909C>T和g960A>T; 3¢非编码区(3¢-UTR)有4个SNP位点, 分别为g2148G>A、g2164A>G、g2420C>G和g2520C>G (表2), 9个SNP位点均检测到3种基因型(图1)。其中开放阅读框中的4个SNP位点突变均不改变所编码的氨基酸, 为同义突变。
2.2 九孔鲍MSTN基因SNP多态性分析
通过Popgene软件计算九孔鲍MSTN基因9个SNP位点的有效基因数(e)、期望杂合度(e)、观测杂合度(o), 并进行哈迪-温伯格平衡(HWE)检验。同时利用PIC_CALC软件计算群体中SNP位点的多态性息含量(PIC)。结果显示: 九孔鲍MSTN基因9个SNP位点的期望杂合度(e)和观测杂合度(o)分别在0.2155~0.4778和0.2195~0.4471间, 多态信息含量(PIC)的范围为0.1913~0.3621 (表2), 除g2164A>G为低度多态性外, 其他均为中度多态性。哈迪-温伯格平衡(HWE)检验结果表明: 除g2420C>G位点外, 其他8个SNP位点均符合哈迪-温伯格平衡(表2)。
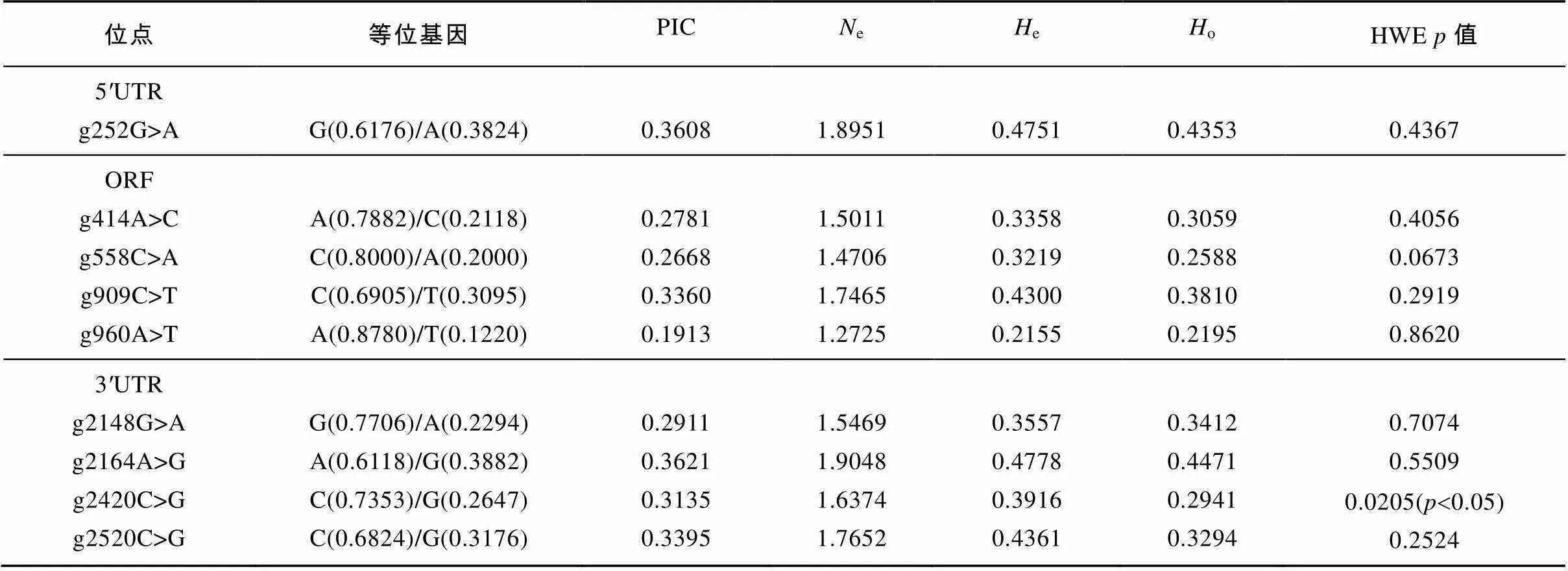
表2 九孔鲍MSTN基因9个SNP位点多态性分析
注: PIC: 多态信息含量;e: 有效等位基因数;e: 期望杂合度;o: 观测杂合度; HWE: 哈迪-温伯格平衡
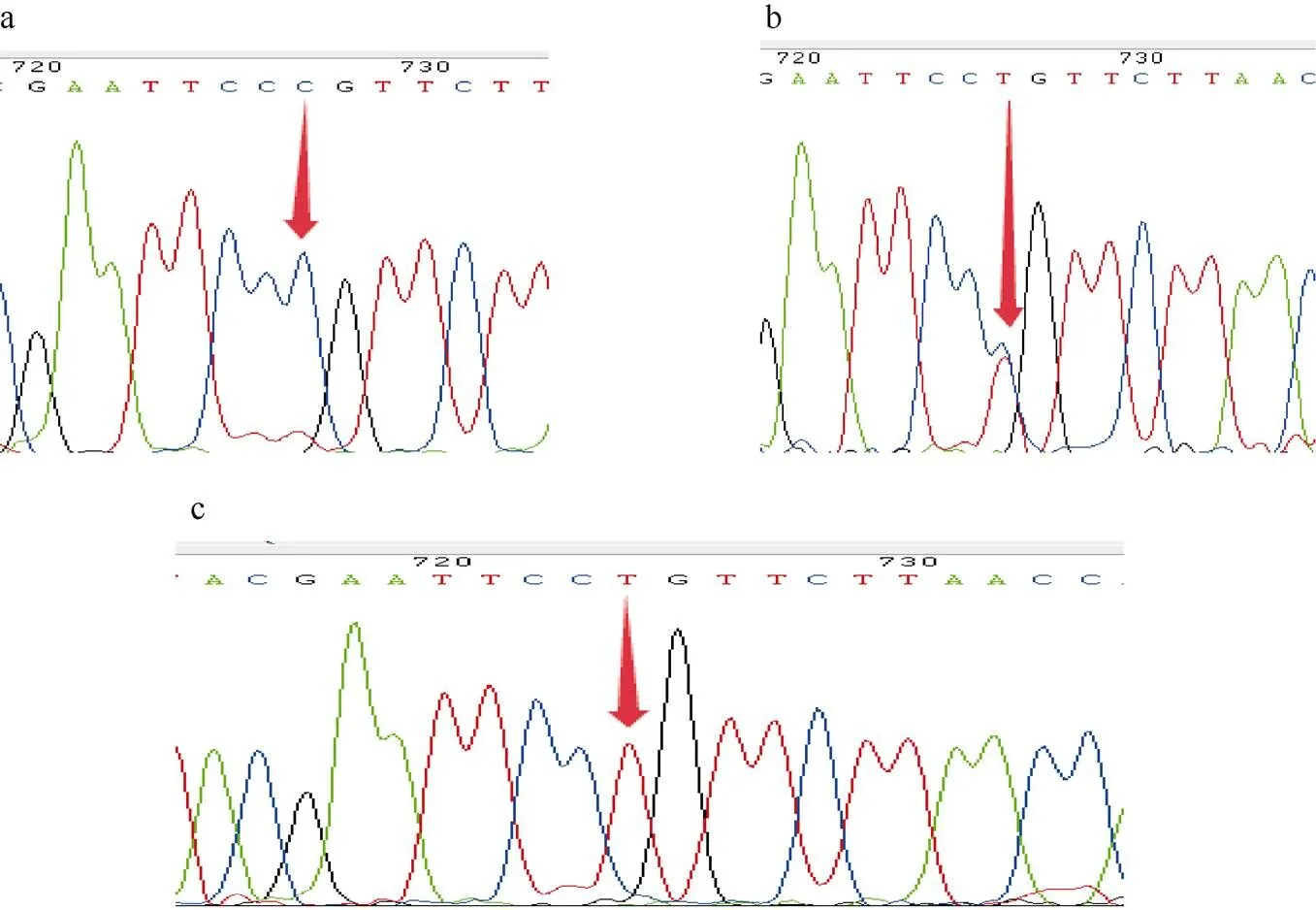
图1 九孔鲍MSTN基因SNP峰图
a. CC型; b. TC型; c. TT型。红色箭头表示突变位置, 数字表示基因序列突变位点的位置
Fig. 1 The peak chart of SNP ofgene in. (a), (b), and (c) represent the chromatograms of CC, TC, TT three genotypes. The arrows represent the mutation position
对9个SNP位点进行基因分型后, 采用SPSS 19.0单因素方差分析(one way ANOVA)对九孔鲍的体质量、壳长和壳宽与基因SNP位点进行关联分析, 结果显示:基因SNP位点g909C>T与九孔鲍3个生长性状(体质量、壳长、壳宽)显著相关, 其中TT基因型九孔鲍的体质量显著高于CC基因型和TC基因型的个体, TT基因型九孔鲍的壳长、壳宽显著高于CC基因型(<0.05)。TC基因型的九孔鲍处于中间值, 与TT、CC基因型差异不显著(>0.05)。其他8个SNP位点的基因型与生长性状的相关性未达到显著水平(表3)。
2.3 九孔鲍MSTN基因SNP g909C>T与表达水平关联分析
为进一步分析九孔鲍基因SNP g909C>T位点与生长性状的相关性, 采用qRT-PCR检测了SNP g909C>T 3种基因型基因的表达水平。结果显示: 九孔鲍基因SNP g909C>T位点的 3种基因型的基因表达水平存在显著差异(<0.05), TC和CC基因型九孔鲍的MSTN基因的表达量显著高于TT基因型(<0.05), TC和CC基因型的九孔鲍基因表达量差异不显著(>0.05)(图2)。
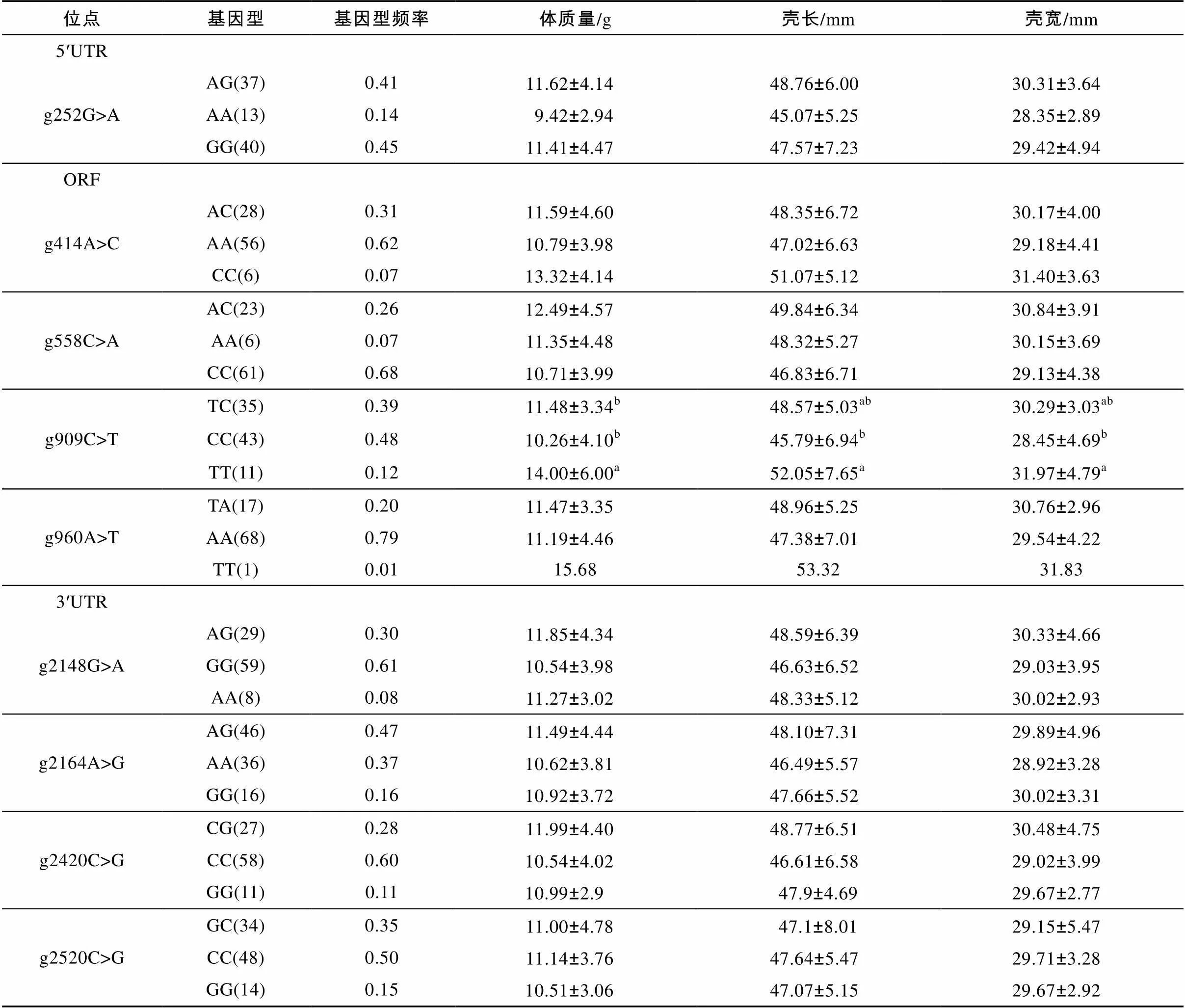
表3 九孔鲍MSTN基因SNP位点及与生长性状关联分析
注: 不同字母表示差异显著(0.05)
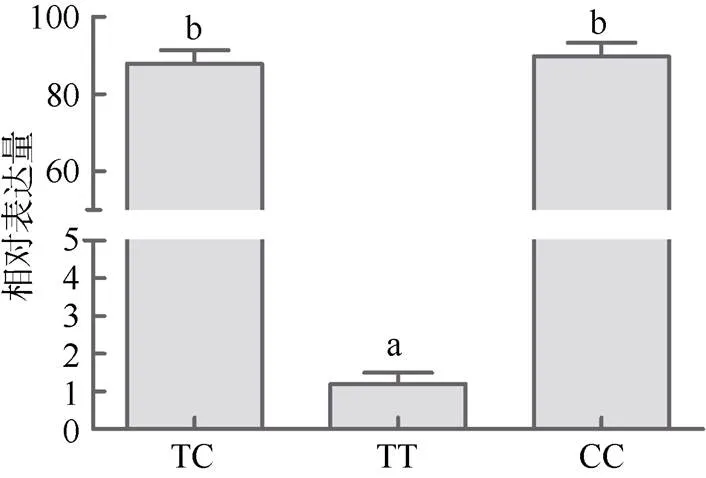
图2 九孔鲍SNP g909C>T不同基因型MSTN表达情况
不同字母表示差异显著(0.05)
Fig. 2 Relative expression ofin the addutormusle ofwith different genotypes at SNP g909C>T. Different letters indicate significant differences (0.05)
3 讨论
肌肉生长抑制素(MSTN)是转化生长因子超家族(TGF-)的重要成员, 主要功能是抑制肌细胞增殖。肌肉生长抑制素基因突变会使MSTN蛋白失去抑制肌细胞生长的功能, 从而使肌肉含量增加(Gao et al, 2014)。SNP是重要的分子标记, 广泛分布生物基因组中, 通过候选基因中的SNP位点与目标性状进行关联分析, 找到与目标性状相关的位点有助于对个体进行有效筛选(Hemmer-Hansen et al, 2011)。基因中的SNP位点已被用于经济物种肌肉重、出生重和腹部脂肪量等经济性状的关联分析研究中(Grisolia et al, 2009)。
本文利用PCR产物直接测序法从九孔鲍基因中成功筛选出了9个SNP位点。贝类基因具有较高多态性, 在合浦珠母贝()中SNP的发生频率为0.8SNP×100bp-1(Li et al, 2014), 在贻贝() SNP的发生频率为1SNP×100bp-1(Núñez-Acuña et al, 2014), 九孔鲍的SNP发生频率为0.2SNP×100bp-1, 多态性较低, 表明九孔鲍基因序列保守。基因编码区的突变, 可发生非同义突变导致基因的功能改变, 发生同义突变则可能通过调控基因表达或改变蛋白质结构影响基因功能(Collins et al, 1998)。发生在非编码区的突变, 可通过调控mRNA的结合部位影响基因功能(Jiang et al, 2001; Miretti et al, 2013)。基因具有N端信号肽、前肽区域和C末端成熟肽区域等形成具有生物活性蛋白所必需的TGF-超家族保守结构, 这些保守的位点在蛋白折叠过程中具有重要作用(Kingsley, 1994; Shin et al, 2015)。因此基因中单个碱基的突变就可能改变基因功能, 提高肌肉含量, 如皮埃蒙特牛基因编码区中发生G/A非同义突变, 改变了所编码的氨基酸, 肌肉含量比普通牛增加20% (Kambadur et al, 1997); 羊基因3′非编码区发生C/A突变影响与microRNA结合, 抑制基因转录, 提高羊肌肉含量(Clop et al, 2006)。通过基因SNP位点与生长性状关联分析, 已筛选到许多与生长性状相关的SNP位点,基因中的这些SNP位点可被用于标记辅助选择育种中。如在肉牛基因中筛选到与肌肉重相关的SNP位点(Grisolia et al, 2009), 在黑尾近红鲌()基因外显子和内含子中筛选到与体重、体长相关的2个SNP位点(Sun et al, 2017); 在海参()基因外显子中筛选到与海参干重相关SNP位点, TC基因型海参的干重显著高于CC基因型(Li et al, 2016); 在缢蛏()基因开放阅读框中筛选到与壳长相关的SNP位点, CC、CG基因型缢蛏的壳长显著长于GG基因型(Niu et al, 2015); 在栉孔扇贝()基因5′非编码区筛选到与闭壳肌重、壳长、壳宽相关的SNP位点, GG、TT基因型扇贝的闭壳肌重显著高于GT基因型, TT基因型扇贝的壳长、壳宽显著高于GT基因型(Fu et al, 2016)。SNP位点与生长性状关联分析结果显示九孔鲍基因编码区SNP g909C>T位点与九孔鲍体质量、壳长、壳宽显著相关, TT基因型九孔鲍的体质量显著高于CC基因型和TC基因型的个体, TT基因型九孔鲍的壳长、壳宽显著高于CC基因型(<0.05), 非编码区的SNP位点与生长性状的相关性均未达到显著水平(>0.05)。九孔鲍MSTN基因4个位于编码区的SNP位点均为同义突变, 不改变所编码的氨基酸, SNP g909C>T位点的突变可能抑制基因转录, 使九孔鲍TT基因型的体质量增加。
为进一步分析九孔鲍基因SNP g909C>T位点与生长性状的相关性, 采用qRT-PCR检测了SNP g909C>T位点3种基因型基因的表达水平。结果显示九孔鲍基因TC和CC基因型的基因表达量显著高于TT基因型, 而TT基因型的九孔鲍体质量显著高于CC基因型和TC基因型的个体, TT基因型九孔鲍的壳长、壳宽显著高于CC基因型(<0.05)。基因是肌肉生长发育的负调控因子, 主要是通过控制肌细胞的数量、大小及增殖抑制肌肉的生长发育(Thomas et al, 2000)。在鱼类(Acosta et al, 2005)、甲壳动物(Zhuo et al, 2017)中通过RNAi抑制基因表达, 均可使肌肉含量增加。在墨吉明对虾()同龄和同家系的大中小3种规格的虾中,基因的表达水平存在显著差异, 小规格的虾中基因的表达量显著高于大规格的虾(Zhuo et al, 2017)。在栉孔扇贝基因与闭壳肌重、壳长相关的SNP位点中, 3种基因型基因的表达水平存在显著差异, GT基因型的基因表达量显著高于GG、TT基因型, 而GG、TT基因型扇贝的闭壳肌重显著高于GT基因型(Fu et al, 2016)。九孔鲍SNP g909C>T位点TT基因型基因表达量较低, 基因抑制肌细胞增殖作用较弱, 右侧壳肌细胞的数量增多, 体质量显著高于其他两种基因型。综上,基因SNP g909C>T可作为九孔鲍标记辅助选择育种的候选标记。
高绪生, 王琦, 王仁波, 等, 2000. 鲍鱼[M]. 沈阳: 辽宁科学技术出版社: 11–29. GAO XUSHENG, WANG QI, WANG RENBO, et al, 2000. Abalone[M]. Shenyang: Liaoning Science and Technology Press: 11–29 (in Chinese).
ACOSTA J, CARPIO Y, BORROTO I, et al, 2005. Myostatin gene silenced by RNAi Show a zebrafish giant phenotype[J]. Journal of Biotechnology, 119(4): 324–331.
BROOKES A J, 1999. The essence of SNPs[J]. Gene, 234(2): 177–186.
CAI JUNPENG, HAN YUN, WANG ZHI, 2006. Isolation offrom abalone (L.) postlarvae associated with mass mortalities[J]. Aquaculture, 257(1–4): 161–166.
CHIANG Y A, KINOSHITA M, MAEKAWA S, et al, 2016. TALENs-mediated gene disruption of myostatin produces a larger phenotype of medaka with an apparently compromised immune system[J]. Fish & Shellfish Immunology, 48: 212–220.
CLOP A, MARCQ F, TAKEDA H, et al, 2006. A mutation creating a potential illegitimate microRNA target site in the myostatin gene affects muscularity in sheep[J]. Nature Genetics, 38(7): 813–818.
COLLINS F S, BROOKS L D, CHAKRAVARTI A, 1998. A DNA polymorphism discovery resource for research on human genetic variation[J]. Genome Research, 8(12): 1229–1231.
FAN SIGANG, XU YOUHOU, LIU BAOSUO, et al, 2017. Molecular characterization and expression analysis of the myostatin gene and its association with growth traits in Noble scallop ()[J]. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 212: 24–31.
FU QIANG, GUO HUIHUI, FENG LIYING, et al, 2016. Association ofvariants with growth traits of Zhikong scallop ()[J]. Journal of Ocean University of China, 15(1): 145–151.
GABILLARD J-C, BIGA P R, RESCAN P-Y, et al, 2013. Revisiting the paradigm of myostatin in vertebrates: insights from fishes[J]. General and Comparative Endocrinology, 194: 45–54.
GAO FENG, SUN BOXING, XING SHENYANG, et al, 2014. The effect of leader peptide mutations on the biological function of bovine myostatin gene[J]. Gene, 540(2): 171–177.
GRISOLIA A B, D’ANGELO G T, PORTO NETO L R, et al, 2009. Myostatin (GDF8) single nucleotide polymorphisms in Nellore cattle[J]. Genetics and Molecular Research, 8(3): 822–830.
HEMMER-HANSEN J, NIELSEN E E, MELDRUP D, et al, 2011. Identification of single nucleotide polymorphisms in candidate genes for growth and reproduction in a nonmodel organism; the Atlantic cod,[J]. Molecular Ecology Resources, 111(S1): 71–80.
HILL E W, GU JINGJING, EIVERS S S, et al, 2010. A sequence polymorphism inpredicts sprinting ability and racing stamina in thoroughbred horses[J]. PLoS One, 5(1): e8645.
JIANG HONGLIN, LUCY M C, 2001. Variants of the 5¢-untranslated region of the bovine growth hormone receptor mRNA: isolation, expression and effects on translational efficiency[J]. Gene, 265(1–2): 45–53.
KAMBADUR R, SHARMA M, SMITH T P L, et al, 1997. Mutations in() in double-muscled Belgian blue and piedmontese cattle[J]. Genome Research, 7(9): 910–915.
KINGSLEY D M, 1994. The TGF-superfamily: new members, new receptors, and new genetic tests of function in different organisms[J]. Genes & Development, 8(2): 133–146.
LEE C-Y, HU SHAO-YANG, GONG HONGYI, et al, 2009. Suppression of myostatin with vector-based RNA interference causes a double-muscle effect in transgenic zebrafish[J]. Biochemical and Biophysical Research Communications, 387(4): 766–771.
LI QIN, LI YAOGUO, HUANG XIANDE, et al, 2014. Molecular cloning and single nucleotide polymorphisms identification of myostatin cDNA in the pearl oyster,[J]. Journal of the World Aquaculture Society, 45(6): 638–651.
LI SHILEI, ZHOU ZUNCHUN, DONG YING, et al, 2016. Molecular characterization, expression analysis of the myostatin gene and its association with growth traits in sea cucumber ()[J]. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 201: 12–20.
MCPHERRON A C, LAWLER A M, LEE S-J, 1997. Regulation of skeletal muscle mass in mice by a new TGF-superfamily member[J]. Nature, 387(6628): 83–90.
MIRETTI S, MARTIGNANI E, ACCORNERO P, et al, 2013. Functional effect of mir-27b on myostatin expression: a relationship in piedmontese cattle with double-muscled phenotype[J]. BMC Genomics, 14: 194.
NIU DONGHONG, WANG LIE, BAI ZHIYI, et al, 2015. Identification and expression characterization of the() gene and association analysis with growth traits in the razor clam[J]. Gene, 555(2): 297–304.
NÚÑEZ-ACUÑA G, GALLARDO-ESCÁRATE C, 2014. Thegene ofevidences a high level of polymorphism and ubiquitous transcript expression[J]. Gene, 536(1): 207–212.
SÁNCHEZ-RAMOS I, CROSS I, MÁCHA J, et al, 2012. Assessment of tools for marker-assisted selection in a marine commercial species: significant association between MSTN-1 gene polymorphism and growth traits[J]. The Scientific World Journal, 2012: 369802.
SHIN S, SONG YAN, AHN J, et al, 2015. A novel mechanism of myostatin regulation by its alternative splicing variant during myogenesis in avian species[J]. American Journal of Physiology - Cell Physiology, 309(10): C650–C659.
SUN YANHONG, LI QING, WANG GUIYING, et al, 2017. Polymorphisms in the myostatin-1 gene and their association with growth traits in[J]. Chinese Journal of Oceanology and Limnology, 35(3): 597–602.
THOMAS M, LANGLEY B, BERRY C, et al, 2000. Myostatin, a negative regulator of muscle growth, functions by inhibiting myoblast proliferation[J]. The Journal of Biological Chemistry, 275(51): 40235–40243.
WANG XIULI, MENG XIANGYING, SONG BIN, et al, 2010. SNPs in the myostatin gene of the mollusk: association with growth traits[J]. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 155(3): 327–330.
WENNE R, 2018. Single nucleotide polymorphism markers with applications in aquaculture and assessment of its impact on natural populations[J]. Aquatic Living Resources, 31(2): 17.
ZHUO RUIQUN, ZHOU TINGTING, YANG SHIPING, et al, 2017. Characterization of a molt-related myostatin gene () from the banana shrimp[J]. General and Comparative Endocrinology, 248: 55–68.
Single nucleotide polymorphisms of myostation gene and its association with growth traits in
AI Jialin1, LI Zhimin1, 2, LIU Jianyong1, SHEN Yuchun1, 2
1. Fisheries College Guangdong Ocean University, Zhanjiang 524088, China; 2. Marine Ecology and Aquaculture Environment of Zhanjiang, Zhanjiang 524088, China
Myostatin is an important member of the transforming growth factor (TGF) family that functions to regulate muscle development and growth in animals. It has become one of the most important target genes for genetic improvement of the production of farmed animals. The purpose of this study was to analyze correlation between single nucleotide polymorphisms of the myostatin gene and growth traits of the, which is an important aquaculture shellfish. The single nucleotide polymorphisms of the myostatin gene () ofwere identified by direct sequencing of PCR products, and association analysis of SNPs withgrowth traits andexpression level.Nine SNPs were identified in cDNA of, including four synonymous mutations.The associations of these SNPs were analyzed withgrowth traits, including shell length, shell height, and body weight. The g909C>T SNP locus was found to be associated with shell length, shell height, and body weight. The TT typeshowed significantly higher traits values than those of TC and CC types in body weight, and the TT typealsoshowed significantly higher traits values than CC type in shell length and shell height (<0.05). On the contrary,transcripts in the musle of those of TC and CC types significantly more than in that of TT type (<0.05).The results suggested thatmight regulate theaddutormusle growth negatively, and SNP g909C>T could be used as a candidate marker for slective breeding of
;; SNP;growth traits; association analysis
2018-05-16;
2018-10-27. Editor: YIN Bo
Development Special Fund of Shenzhen Dapeng New District (KY20170211); Special Fund of Zhanjiang (2015A06006)
LI Zhimin. E-mail: lizhimin811@163.com
2018-05-16;
2018-10-27。殷波编辑
深圳市大鹏新区产业发展专项资金科技研发项目(KY20170211); 湛江市财政资金科技专项(2015A06006)
艾加林(1989—),男, 硕士研究生,研究方向是贝类遗传育种。E-mail: 980158435@qq.com
栗志民(1972—),男, 博士, 教授。E-mail:lizhimin811@163.com
P735.542
A
1009-5470(2019)02-0078-08
10.11978/2018053

