东海产麻痹性贝毒链状亚历山大藻共附生菌群多样性研究
唐莹莹, 乔玉宝, 蒋志伟, 张 静, 张若男,田晓清,马丽艳, 张晓玲, 陆亚男, 樊成奇, 杨 桥
(1. 中国水产科学研究院东海水产研究所,上海 200090; 2. 上海海洋大学,上海 201306; 3. 浙江海洋大学海洋科学与技术学院,浙江舟山 316022; 4. 舟山出入境检验检疫局综合技术服务中心,浙江舟山 316021; 5. 哈尔滨商业大学生命科学与环境科学研究中心,黑龙江哈尔滨 150076)
赤潮(harmful algal blooms, HABs)是全球性的重大环境问题与生态灾害[1],其不仅严重破坏海洋生态系统平衡、危害海产品质量安全,其引发的赤潮毒素经食物链传递还严重威胁人类健康与生命安全[2]。麻痹性贝类毒素(paralytic shellfish poisoning,PSP)是一种代表性赤潮毒素,其分布广、毒性强、对人类生命健康威胁大[3]。但PSP来源问题仍悬而未决:PSP是由甲藻单独产生,还是由其共附生菌单独产生,或由藻菌两者相互作用共同产生,尚无定论[3-7]。藻菌相互作用关系(algae-bacterial interaction)是揭示PSP产生机制的关键,而甲藻共附生菌群多样性信息的解析是阐释藻菌相互作用关系的必要前提,但目前此方面研究尚无明确定论[6-11]。
链状亚历山大藻(Alexandriumcatenella)是一种全球性的典型赤潮甲藻[4],目前对其共附生菌群多样性研究鲜有报道。本文利用高通量测序及微生物纯培养技术,解析了东海链状亚历山大藻(LZ1706)共附生菌群的物种种类及相对丰度信息,并分离获得其可培养微生物菌株,以期为后续解析藻菌相互作用关系提供必须的微生物菌株及生物背景信息资料。
1 材料与方法
1.1 实验材料
链状亚历山大藻(AlexandriumcatenellaLZ1706)分离自东海舟山海域。
1.2 实验方法
1.2.1 藻培养
藻培养使用f/2培养基。首先用超纯水配制75.0 g·L-1NaNO3溶液、5.0 g·L-1NaH2PO4·H2O、L1 Trace Metal Solution灭菌后备用,再用高温灭菌后的超纯水配制Vitamin Solution作为储备液。f/2培养基配制过程如下:用超纯水将盐卤稀释至盐度为25‰,按照表1用量加入除Vitamin Solution以外各储备液,115 ℃高温灭菌30 min,冷却至室温后加入Vitamin Solution。藻培养条件为光照强度:4 500~5 000 lx,光照周期:12L/12D,培养温度:25 ℃。
1.2.2 高通量测序及数据分析
待藻细胞培养至对数生长期,取50 mL藻液于6 000 r·min-1离心收集藻细胞,使用50 mL PBS缓冲液(pH 7.2)洗涤细胞沉淀3次后备用[10-11]。
使用 Stool DNA Kit 试剂盒(美国OMEGA)提取藻样共附生菌总基因组DNA,具体操作参照试剂盒说明书进行[6-9]。PCR扩增反应使用细菌16S rRNA基因V3-V4区通用扩增引物338F及806R。引物序列分别为上游引物338F:5′-ACTCCTACGGGAGGCAGCAG-3′、下游引物806R:5′-GGACTACHVGGGTWTCTAAT-3′。PCR扩增反应体系如下:5×FastPfu 缓冲液:4 μL,2.5 mM dNTPs:2 μL,上游引物(5 μM):0.8 μL,下游引物(5 μM):0.8 μL,DNA多聚酶:0.4 μL,BSA:0.2 μL,模板DNA:10 ng,补去离子水至总体积50 μL。PCR反应条件:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,共27个循环,最后72 ℃下作用10 min进行延伸。使用2%琼脂糖凝胶电泳对PCR产物进行检测,使用AXYGEN DNA凝胶回收试剂盒(AP-GX-50)切胶回收PCR产物。
数据质控:回收后的PCR产物样品送样进行Illumina Miseq高通量测序(上海美吉生物公司)。数据质控参照文献[6-9]进行:根据PE读长之间相互重叠关系进行序列拼接,同时对读长质量和拼接效果进行质控分析。过滤读长尾端质量值小于20的碱基;最小重叠长度:10 bp;拼接序列的重叠区所允许的最大错配比率:0.2;根据序列两端DNA条形码及引物进行样品区分;使用软件:FLASH及Trimmomatic,应用FLASH软件制作矢量图,应用Trimmomatic软件处理Illumina测序数据中一些质量低或者带有adapters序列的raw reads。
数据分析:对获得的非重复序列进行(operational taxonomic units,OTU)聚类分析以获得OTU代表序列,从中选取与代表序列相似度大于97%的序列产生OUT数据表。通过贝叶斯分类算法(ribosomal database project,RDP)对OUT数据表进行分类学分析,分别在不同分类水平上进行样本群落组成统计分析[6-8]。选取Silva、RDP、Greengene为细菌16S比对数据库,使用Qiime平台与 RDP Classifier分别作为计算软件及算法,设定0.7为置信度阈值。群落丰富度指数(community richness)采用Chao指数和ACE指数;群落多样性指数(community diversity)采用Shannon指数与Simpson指数。利用不同测序深度时微生物多样性指数构建测序样品的稀释曲线(rarefaction curve)[9]。
1.2.3 可培养共附生菌株的分离及鉴定
取对数生长期藻液1 mL,于6 000 r·min-1离心收集藻细胞,用无菌水重悬细胞沉淀,冰浴下对藻细胞进行超声波处理5 min,加入2 mL无菌水重悬,对重悬液进行10倍梯度稀释后,取100 μL稀释液均匀涂布于2216E培养基平板,28 ℃培养2~3 d,待平板上生长出单菌落后,挑取单菌落进行进一步划线纯化后,挑取单菌落至固体斜面,置4 ℃进行菌株保存[10-11]。
16S rRNA测序及比对:将菌株斜面培养物接种到5 mL Zobell 2216E液体培养基中,置摇床28 ℃培养36 h,使用细菌基因组DNA提取试剂盒(美国Sigma)提取基因组DNA。16S rRNA基因PCR扩增通用引物27F及1492R,其序列分别为上游引物27F:5′-AGAGTTTGATCCTGGCTCAG-3′;下游引物1492R:5′-GGTTACCT TGTTACGACTT-3′。PCR扩增体系包括:2×TaqPCR MasterMix:25 μL,上/下游引物:各1 μL,模板DNA:3 μL,无菌水:20 μL。PCR扩增程序如下:94 ℃预变性5 min,94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸1 min,共35个循环,最后72 ℃延伸10 min。PCR产物进行1%琼脂糖凝胶电泳。送样至上海美吉生物进行基因测序,分析获得的16S rRNA基因序列通过GenBank及BLAST 软件进行序列同源性对比,使用MEGA软件(7.0)构建菌株的16S rRNA基因系统发育进化树[10-11]。
2 结果与分析
2.1 高通量测序数据筛选
链状亚历山大藻(LZ1706)共附生菌群的测序结果如表1所示,高通量测序共获得36 727条序列,长度分布在363~493。 441~460范围内的序列数量最多,为22 024条,其次是421~440范围,为14 700条。样品的Shannon-Wiener曲线(图1)表明本次所测样品的测序数据量合理,足以反映样品中的物种多样性。样品的生物多样性指数(alpha-diversity)统计分析结果(表1)表明,所测样品的微生物群落多样性较高。
2.2 链状亚历山大藻的共附生菌多样性分析
MiSeq高通量测序结果表明,链状亚历山大藻(LZ1706)共附生菌群包含43个OUT,包括6门,10纲,19目,25科及34属。其中优势门(>5%)3个,包括变形菌门(Proteobacteria,53.9%)、拟杆菌门(Bacteroidetes,27.5%)及蓝藻门(Cyanobacteria,16.8%);优势纲(>5%)4个,包括γ-变形菌纲(Gammaproteobacteria,28.4%)、黄杆菌纲(Flavobacteriia,24.3%)、α-变形菌纲(Alphaproteobacteria,21.4%)、蓝藻纲(Cyanobacteria,16.8%)(图2);优势属(>5%)5个,包括Cryomorphaceae科下未知属(22.8%)、Cyanobacteria纲下未知属(16.8%)、Saccharospirillum属(14.6%)、红细菌科(Rhodobacteraceae)下未分类属(7.9%)及Maricaulis属(5.6%)。

图1 链状亚历山大藻(LZ1706)共附生菌群高通量测序样品Shannon-Wiener 曲线Fig.1 Shannon-Wiener curve of the sequencing sample from A. catenella (LZ1706)
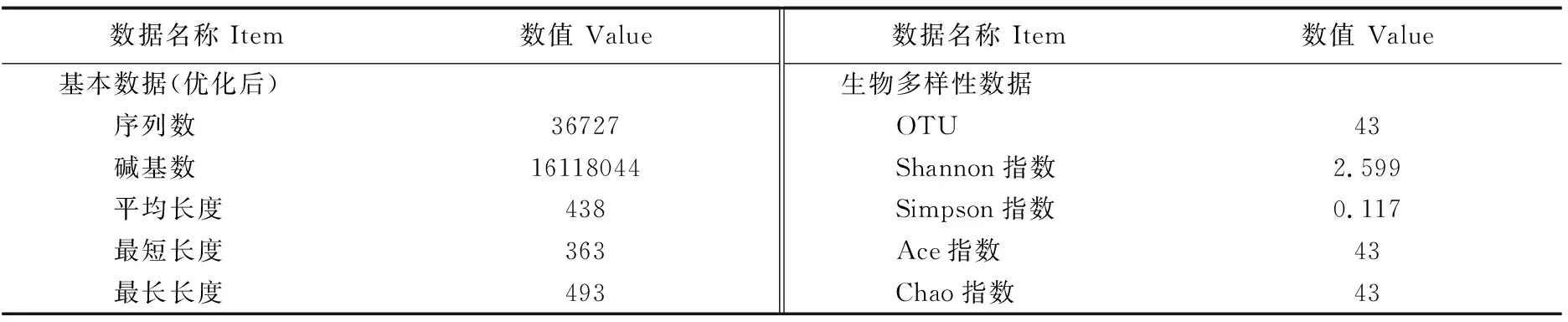
数据名称 Item数值 Value基本数据(优化后) 序列数36727 碱基数16118044 平均长度438 最短长度363 最长长度493数据名称 Item数值 Value生物多样性数据 OTU43 Shannon指数2.599 Simpson指数0.117 Ace指数43 Chao指数43

图2 基于OTU的链状亚历山大藻(LZ1706)共附生菌群系统发育进化树Fig.2 Phylogenetic tree of the bacterial community associated with A. catenella LZ1706 based on OTU

图3 链状亚历山大藻(LZ1706)共附生菌群属水平的相对丰度分布图Fig.3 Relative abundance at genus level of the bacterial community associated with A. catenella LZ1706
2.3 链状亚历山大藻可培养共附生菌的系统发育进化分析
从链状亚历山大藻(LZ1706)培养物中分离获得可培养微生物菌株12株,其16S rRNA基因序列测序及系统进化分析结果表明,这些菌株属于9个属,包括Marinobactersp.(2株)、Ponticoccussp. (2株)及Mameliellasp.、Pseudooceanicolasp.、Marivitasp.、Limnobactersp.、Saccharospirillumsp.、Maricaulissp.、Halieasp.、Maritimibactersp.各1株。其16S rRNA系统发育树如图4所示,其中菌株LZ-27与LZ-7与已知菌株的16S rRNA基因同源性最高值分别为96.0%及97.7%,表明其分别为Ponticoccus属及Pseudooceanicola属潜在新种,其多相分类学鉴定分析目前正在进行中。
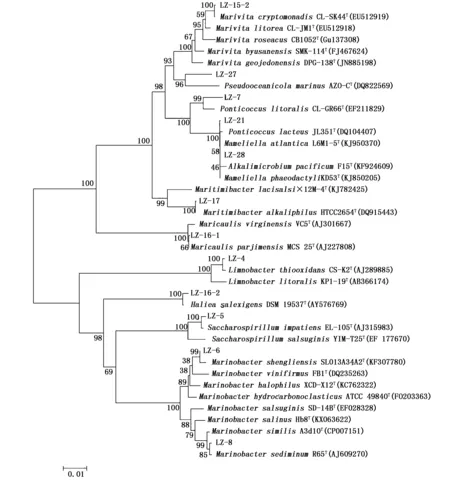
图4 链状亚历山大藻(LZ1706)可培养菌株的16S rRNA基因系统发育进化树Fig.4 Phylogenetic tree based on 16S rRNA gene sequences of the cultivable bacterial strains and related taxa
3 讨论
藻菌相互作用关系是揭示PSP产生机制的关键,可为赤潮的防治提供科学参考[12-16]。免培养(culture-independent)Illumina MiSeq高通量测序分析可解析复杂共生体系中环境微生物群落多样性信息,从而为优化甲藻共附生菌的分离并提高其可培养性提供有益参考[5,8-11]。本文利用MiSeq高通量测序首次对东海链状亚历山大藻(LZ1706)共附生菌群多样性进行了分析。从获得的43个OTU中归类出34个属,其优势属5个,其中糖螺菌属(Saccharospirillum)及Maricaulis属为首次在亚历山大藻中发现。此外,其未知属共附生菌群比例高达50.6%。表明链状亚历山大藻(LZ1706)蕴含潜在的海洋新种属微生物及发掘其未知生物学功能的潜力,值得进一步深入挖掘。
此外,基于微生物纯培养技术从链状亚历山大藻(LZ1706)中分离获得包括2株潜在新种在内的12株可培养菌株,但此数量远低于MiSeq高通量测序分析所呈现的藻共附生菌群的丰富多样性。表明海洋复杂共生体系内共附生菌群的低可培养性(cultivability)仍是海洋微生物研究领域的一个关键瓶颈,今后在提高赤潮藻共附生菌选择性分离方面的研究仍须继续加强。
4 小结
东海产PSP典型赤潮甲藻-链状亚历山大藻(LZ1706)共附生菌群种优势属有5个,包括Cryomorphaceae科未知属、Cyanobacteria纲未知属、红细菌科(Rhodobacteraceae)未知属、糖螺菌属(Saccharospirillum)及Maricaulis属。未知类群比例较高,其中蕴含潜在的新种属微生物。

