高山马铃薯种质资源遗传多样性的同工酶分析
洪森荣,张铭心,叶思雨,宁本松
(上饶师范学院 生命科学学院,江西 上饶 334001)
马铃薯为茄科茄属一年生草本植物,是世界性重要的粮菜兼用作物,在全球主要粮食作物中,排在水稻、小麦和玉米之后,位居第四[1]。高山马铃薯一般是指适合种植于高海拔山区的马铃薯品种,如云南的合作88号和威芋3号、湖北恩施的鄂马铃薯5号、鄂马铃薯11号和鄂马铃薯14号,以及怀玉山麻籽洋芋等。高山马铃薯具有高食用价值和高营养价值,成本和技术要求低,而且高山马铃薯的适应能力强,对土壤的肥力、酸碱度、坡度、湿度,以及光照等生存条件要求低,可在山区、丘陵缓坡大面积种植,其产业化潜力优势十分明显[2]。我国南方大部分地区属于高山地区,独特的气候条件非常适合高山马铃薯的生长。2014年,农业部提出马铃薯主粮化粮食安全战略,因此,发展高山马铃薯产业具有重要意义。
种质资源是遗传育种的物质基础,对种质资源遗传多样性和亲缘关系的正确评价是合理利用种质资源的前提,遗传多样性是确保物种延续和不断进化的关键[3]。目前,我国高山马铃薯的种植区域主要集中于湖北恩施、云南曲靖和德宏,以及江西怀玉山等地。开展高山马铃薯种质资源遗传多样性研究,明确其亲缘关系,对扩大高山马铃薯品种的遗传基础和选育新品种均有重要作用。目前,关于高山马铃薯遗传多样性研究尚未见报道。同工酶是基因表达的产物,其蛋白多肽链结构中的氨基酸排列顺序是由DNA上结构基因所携带的遗传信息所决定的,通过酶谱分析,就能识别控制这些谱带表达的基因[4]。同工酶的酶谱与等位基因之间有明确的对应关系,因此是一种十分有效的遗传多样性的检测标记[5]。本研究采用过氧化物酶(POD)、酯酶(EST)、多酚氧化酶(PPO)同工酶对6份高山马铃薯品种的遗传多样性进行检测,以期为进一步开展高山马铃薯品种遗传改良、杂交亲本选配及种质资源的利用提供依据。
1 材料与方法
1.1 材料
怀玉山高山马铃薯苗,麻籽洋芋,编号M1~M5,来源于江西怀玉山;怀玉山高山马铃薯块茎,麻籽洋芋,编号M6~M10,来源于江西怀玉山;云南高山马铃薯块茎,合作88号,编号M11~M14,来源于云南德宏盈江;云南高山马铃薯块茎,威芋3号,编号M15~M18,来源于云南曲靖;恩施高山马铃薯块茎,鄂马铃薯5号,编号M19~M22,来源于湖北巴东官渡口镇;恩施高山马铃薯块茎,鄂马铃薯11,编号M23~M26,来源于湖北巴东官渡口镇;恩施高山马铃薯块茎,鄂马铃薯14,编号M27~M30,来源于湖北巴东信陵镇。
1.2 方法
1.2.1 样品提取
将马铃薯块茎清洗干净,吸干水分,切取0.5 g左右材料(马铃薯苗取叶片0.5 g左右),加入预先冷却的研钵中,加液氮研磨成粉末。再加入2.5 mL左右酶提取液继续冰上研磨成浆状,12 000g4 ℃下离心10 min,收集上清即为同工酶粗提液,分装后于-80 ℃保存。粗酶提取液配方[6]:蔗糖11.98 g、Tris 0.606 g、抗坏血酸钠0.088 g、半胱氨酸0.030 g和氯化镁0.020 g溶于80 mL纯化水中,充分溶解后调节pH值到7.4,再定容到100 mL。
1.2.2 样品处理与电泳
所有样本用考马斯亮蓝法测定蛋白浓度,并用提取液调整蛋白浓度到5 μg·μL-1。取400 μL粗酶液加入100 μL 5×上样缓冲液(250 mmol·L-1pH 6.8 Tris-HCl,0.5%溴酚蓝,50%甘油),混匀后分装小份于-80 ℃保存。
按常规方法[6]配制非变性的分离胶和浓缩胶(配方中不加SDS),其中分离胶浓度为10%。非变性电泳缓冲液,配方为0.025 mol·L-1Tris,0.2 mol·L-1甘氨酸,pH为8.3。
电泳上样量为10~20 μL,冰水浴电泳。电泳条件为80 V 30 min,再120 V 2~3 h,以溴酚蓝离边缘1~2 cm为准。
1.2.3 同工酶染色
POD(过氧化物酶)同工酶。采用联苯胺染色法[6]。首先配制显色液:0.1 g联苯胺加5 mL无水乙醇,再加10 mL 1.5 mol·L-1乙酸钠及10 mL 1.5 mol·L-1乙酸,加蒸馏水75 mL,染色前加5~6滴H2O2原液。其次进行显带与固定:取出凝胶漂洗后,放入显色液中,稍加振动,片刻即显示出蓝色酶带,待酶带完全出现后,随即用水冲洗,照相或保存在7%乙酸中。
EST(酯酶)同工酶。参照文献[6]的方法配制显色液:7%醋酸-α-萘酯的丙酮溶液0.5 mL,坚牢蓝RR 12.5 mg,0.2 mol·L-1Tris-HCl pH 7.1缓冲液1 mL,水23.4 mL。随后,凝胶在0.2 mol·L-1Tris-HCl pH 7.1的缓冲液中预浸,然后放在显色液中,37 ℃保温30~60 min,至酶带清晰为止,水冲洗后照相或保存在7%乙酸中。
PPO(多酚氧化酶)同工酶。参照文献[6]的方法配制染色液:0.5 g对苯二酚,0.5 g邻苯二酚,0.02 g对苯二胺,溶解在少量无水己醇中,再用0.2 mol·L-1pH 6.8磷酸缓冲液定容到100 mL。将凝胶取下后,先用纯化水洗涤,再放入染色液中,室温染色20~30 min。
1.2.4 数据处理
凝胶上酶谱带颜色的深浅反映同工酶活性的强弱,根据电泳酶带图,将酶活性分为强、较强、弱三个等级,绘制酶谱模式图。分别计算酶带迁移率Rf和出现频率Af,Rf=酶带迁移距离/前沿溴酚蓝距离,Af=酶带出现次数/样本数。使用popgene软件计算各同工酶多态性百分率和等位基因数。使用NTsys2.1软件计算各同工酶酶谱带的遗传相似系数,并用非加权组平均法进行聚类分析,绘制相似系数树状聚类图。
2 结果与分析
2.1 同工酶电泳酶带图
不同同工酶用不同的染色方法,各样本间条带数目和强弱也有区别(图1)。
2.2 同工酶酶谱模式图
根据各同工酶的电泳酶带图,绘制出各个同工酶的酶谱模式图(图2),其中酶活性分为强、较强、弱3个等级。模式图左侧为各种同工酶的不同酶带命名,右侧为酶带迁移率Rf。POD同工酶共有10条酶带,分别命名为POD-01~POD-10,酶带迁移率Rf为0.034~0.638。EST同工酶共有7条酶带,分别命名为EST-01~EST-07,酶带迁移率Rf为0.102~0.492。PPO同工酶共有11条酶带,分别命名为PPO-01~PPO-11,酶带迁移率Rf为0.034~0.690。

编号1~5,怀玉山高山马铃薯苗(麻籽洋芋);编号6~10,怀玉山高山马铃薯块茎(麻籽洋芋);编号11~14,云南高山马铃薯块茎(合作88号);编号15~18,云南高山马铃薯块茎(威芋3号);编号19~22,恩施高山马铃薯块茎(鄂马铃薯5号);编号23~26,恩施高山马铃薯块茎(鄂马铃薯11);编号27~30,恩施高山马铃薯块茎(鄂马铃薯14)。No. 1-5, Huaiyushan alpine potato plantlets (Maziyangyu); No. 6-10, Huaiyushan alpine potato tuber (Maziyangyu); No. 11-14, Yunnan alpine potato tuber (Hezuo No. 88); No. 15-18, Yunnan alpine potato tuber (Weiyu No. 3); No. 19-22, Enshi alpine potato (Hubei potato No. 5); No. 23-26, Enshi alpine potato tuber (Hubei potato 11); No. 27-30, Enshi alpine potato tuber (Hubei potato 14).图1 POD(A)、EST(B)、PPO(C)同工酶Fig.1 POD(A), EST(B), PPO(C) isozyme
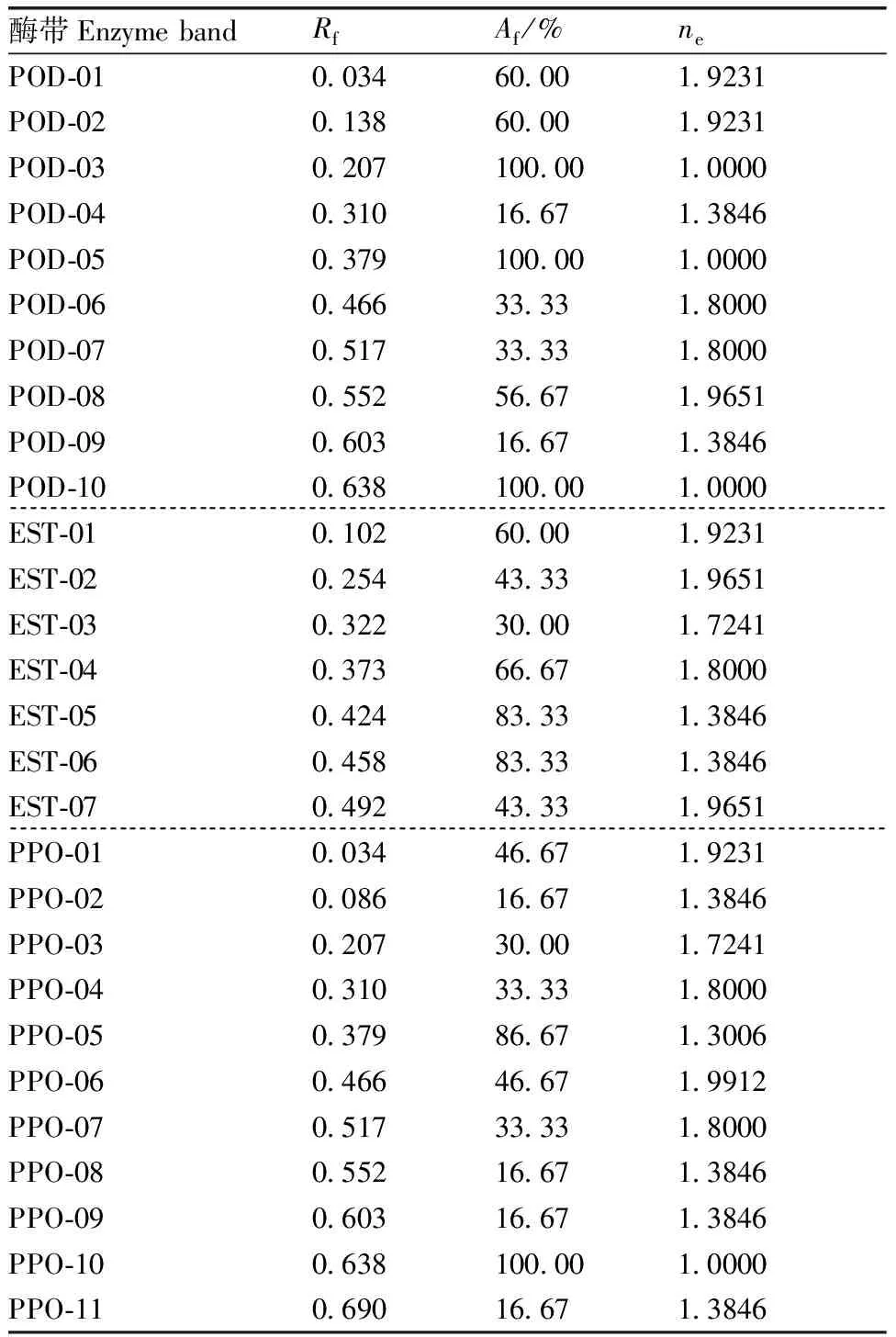
2.3 同工酶酶带迁移率(Rf)、出现频率(Af)、多态性百分率(P)及有效等位基因数(ne)
计算各同工酶酶带迁移率(Rf)及出现频率(Af),并利用popgene软件[7]计算出POD、EST和PPO同工酶多态性百分率(P)和有效等位基因数(ne)(表1)。
从表1可以看出,POD同工酶中的3条带(POD-03、POD-05和POD-10)为所有样本共有条带(ne为1.000 0),其余7条均为多态性条带,因此POD同工酶的多态性比率(P)为7/10=70%。出现频率Af最高的是POD-03、POD-05和POD-10,为100%;其次是POD-01和POD-02,Af为60%,POD-01对应样本编号为M1~M10、M15~M18和M27~M30,POD-02对应样本编号为M1~M10和M23~M30;POD-08出现频率Af为56.67%,对应样本编号为M1~M5和M19~M30;POD-06和POD-07,Af为33.33%,对应样本编号均为M1~M10;POD-04和POD-09,Af为16.67%,POD-04对应样本编号为M6~M10,POD-09对应样本编号为M1~M5。

图2 POD(A)、EST(B)、PPO(C)同工酶模式图Fig.2 POD(A), EST(B), PPO(C) isozyme pattern map
EST同工酶无共有条带(ne为1.000 0),所有条带(7条)均为多态性条带,因此EST同工酶的多态性比率(P)为7/7=100%。出现频率Af最高的是EST-05和EST-06,为83.33%,对应样本编号均为M6~M30;其次是EST-04,Af为66.67%,对应样本编号为M11~M30;EST-01,Af为60.00%,对应样本编号为M1~M10、M15~M18和M27~M30;EST-02和EST-07,Af为43.33%,EST-02对应样本编号为M6~M14和M19~M22,EST-07对应样本编号为M6~M10和M23~M30;EST-03,Af最低为30.00%,对应样本编号为M6~M14。
表1POD、EST、PPO同工酶酶带迁移率(Rf)、出现频率(Af)及有效等位基因数(ne)
Table1Enzyme band mobility (Rf), frequency of occurrence (Af) and effective allele number (ne) of POD, EST and PPO isozyme
酶带Enzyme bandRfAf/%nePOD-010.03460.001.9231POD-020.13860.001.9231POD-030.207100.001.0000POD-040.31016.671.3846POD-050.379100.001.0000POD-060.46633.331.8000POD-070.51733.331.8000POD-080.55256.671.9651POD-090.60316.671.3846POD-100.638100.001.0000EST-010.10260.001.9231EST-020.25443.331.9651EST-030.32230.001.7241EST-040.37366.671.8000EST-050.42483.331.3846EST-060.45883.331.3846EST-070.49243.331.9651PPO-010.03446.671.9231PPO-020.08616.671.3846PPO-030.20730.001.7241PPO-040.31033.331.8000PPO-050.37986.671.3006PPO-060.46646.671.9912PPO-070.51733.331.8000PPO-080.55216.671.3846PPO-090.60316.671.3846PPO-100.638100.001.0000PPO-110.69016.671.3846
PPO同工酶中的只有1条带(PPO-10)为所有样本共有条带(ne为1.000 0),其余10条均为多态性条带,因此PPO同工酶的多态性比率(P)为10/11=90.91%。出现频率Af最高的是PPO-10,为100.00%;其次是PPO-05,Af为86.67%,对应样本编号为M1~M18和M23~M30;PPO-01和PPO-06,Af为46.67%,PPO-01对应样本编号为M1~M10和M15~M18,PPO-06对应样本编号为M1~M14;PPO-04和PPO-07,Af为33.33%,对应样本编号均为M1~M10;PPO-03,Af为30.00%,对应样本编号为M6~M10和M27~M30;PPO-02、PPO-08、PPO-09和PPO-11,Af为16.67%,PPO-02、PPO-08和PPO-09对应样本编号均为M1~M5,PPO-11对应样本编号为M6~M10。
2.4 同工酶相似系数及聚类分析
使用NTsys2.1软件[7]计算各同工酶酶谱带的遗传相似系数(表2~4),用非加权组平均法进行聚类分析并绘制的相似系数树状聚类图如图3。
从图3-A的POD聚类分析图来看,在遗传相似系数0.650处,30个样本可分为二大组,M1~M10为第1组,而M11~M30为第2组,表明云南高山马铃薯和恩施高山马铃薯均属于同一类,而怀玉山高山马铃薯单独成一类。从图3-B的EST聚类分析图来看,在遗传相似系数0.398处,30个样本可分为2大组,M1~M5为第1组,而M6~M30为第2组,表明分别采用块茎和苗来做EST同工酶检测时,结果是不一样的,可能是由于块茎和苗的EST活力不一致造成的;但在遗传相似系数0.534处,M6~M30还可分为2个亚组,M6~M10为第1亚组,而M11~M30为第2亚组,表明云南高山马铃薯和恩施高山马铃薯均属于同一类,而怀玉山高山马铃薯单独成一类。从图3-C的PPO聚类分析图来看,在遗传相似系数0.486处,30个样本可分为2大组,M1~M10为第1组,而M11~M30为第2组,再次表明云南高山马铃薯和恩施高山马铃薯均属于同一类,而怀玉山高山马铃薯单独成一类。
表2POD同工酶相似系数
Table2Similarity coefficients of POD isozyme

NO12345678910111213141516171819202122232425262728293011.0021.001.0031.001.001.0041.001.001.001.0051.001.001.001.001.0060.700.700.700.700.701.0070.700.700.700.700.701.001.00880.700.700.700.700.701.001.001.0090.700.700.700.700.701.001.001.001.00100.700.700.700.700.701.001.001.001.001.00110.400.400.400.400.400.500.500.500.500.501.00120.400.400.400.400.400.500.500.500.500.501.001.00130.400.400.400.400.400.500.500.500.500.501.001.001.00140.400.400.400.400.400.500.500.500.500.501.001.001.001.00150.500.500.500.500.500.600.600.600.600.600.900.900.900.901.00160.500.500.500.500.500.600.600.600.600.600.900.900.900.901.001.00170.500.500.500.500.500.600.600.600.600.600.900.900.900.901.001.001.00180.500.500.500.500.500.600.600.600.600.600.900.900.900.901.001.001.001.00190.500.500.500.500.500.400.400.400.400.400.900.900.900.900.800.800.800.801.00200.500.500.500.500.500.400.400.400.400.400.900.900.900.900.800.800.800.801.001.00210.500.500.500.500.500.400.400.400.400.400.900.900.900.900.800.800.800.801.001.001.00220.500.500.500.500.500.400.400.400.400.400.900.900.900.900.800.800.800.801.001.001.001.00230.600.600.600.600.600.500.500.500.500.500.800.800.800.800.700.700.700.700.900.900.900.901.00240.600.600.600.600.600.500.500.500.500.500.800.800.800.800.700.700.700.700.900.900.900.901.001.00250.600.600.600.600.600.500.500.500.500.500.800.800.800.800.700.700.700.700.900.900.900.901.001.001.00260.600.600.600.600.600.500.500.500.500.500.800.800.800.800.700.700.700.700.700.900.900.900.901.001.001.001.00270.700.700.700.700.700.600.600.600.600.600.700.700.700.700.800.800.800.800.800.800.800.800.900.900.900.901.00280.700.700.700.700.700.600.600.600.600.600.700.700.700.700.800.800.800.800.800.800.800.800.900.900.900.901.001.00290.700.700.700.700.70.700.600.600.600.600.600.700.700.700.700.800.800.800.800.800.800.800.80.80.900.900.900.901.001.00300.700.700.700.700.700.600.600.600.600.600.700.700.700.700.800.800.800.800.800.800.800.800.800.900.900.900.901.001.001.00
表3EST同工酶相似系数
Table3Similarity coefficients of EST isozyme

NO12345678910111213141516171819202122232425262728293011.0021.001.0031.001.001.0041.001.001.001.0051.001.001.001.001.0060.290.290.290.290.291.0070.290.290.290.290.291.001.0080.290.290.290.290.291.001.001.0090.290.290.290.290.291.001.001.001.00100.290.290.290.290.291.001.001.001.001.00110.140.140.140.140.140.570.570.570.570.571.00120.140.140.140.140.140.570.570.570.570.571.001.00130.140.140.140.140.140.570.570.570.570.571.001.001.00140.140.140.140.140.140.570.570.570.570.571.001.001.001.00150.570.570.570.570.570.430.430.430.430.430.570.570.570.571.00160.570.570.570.570.570.430.430.430.430.430.570.570.570.571.001.00170.570.570.570.570.570.430.430.430.430.430.570.570.570.571.001.001.00180.570.570.570.570.570.430.430.430.430.430.570.570.570.571.001.001.001.00190.290.290.290.290.290.430.430.430.430.430.860.860.860.860.710.710.710.711.00200.290.290.290.290.290.430.430.430.430.430.860.860.860.860.710.710.710.711.001.00210.290.290.290.290.290.430.430.430.430.430.860.860.860.860.710.710.710.711.001.001.00220.290.290.290.290.290.430.430.430.430.430.860.860.860.860.710.710.710.711.001.001.001.00230.290.290.290.290.290.430.430.430.430.430.570.570.570.570.710.710.710.710.710.710.710.711.00240.290.290.290.290.290.430.430.430.430.430.570.570.570.570.710.710.710.710.710.710.710.711.001.00250.290.290.290.290.290.430.430.430.430.430.570.570.570.570.710.710.710.710.710.710.710.711.001.001.00260.290.290.290.290.290.430.430.430.430.430.570.570.570.570.710.710.710.710.710.710.710.711.001.001.001.00270.430.430.430.430.430.570.570.570.570.570.430.430.430.430.860.860.860.860.570.570.570.570.860.860.860.861.00280.430.430.430.430.430.570.570.570.570.570.430.430.430.430.860.860.860.860.570.570.570.570.860.860.860.861.001.00290.430.430.430.430.430.570.570.570.570.570.430.430.430.430.860.860.860.860.570.570.570.570.860.860.860.861.001.001.00300.430.430.430.430.430.570.570.570.570.570.430.430.430.430.860.860.860.860.570.570.570.570.860.860.860.861.001.001.001.00
表4PPO同工酶相似系数
Table4Similarity coefficients of PPO isozyme

NO12345678910111213141516171819202122232425262728293011.0021.001.0031.001.001.0041.001.001.001.0051.001.001.001.001.0060.700.700.700.700.701.0070.700.700.700.700.701.001.0080.700.700.700.700.701.001.001.0090.700.700.700.700.701.001.001.001.00100.700.700.700.700.701.001.001.001.001.00110.400.400.400.400.400.500.500.500.500.501.00120.400.400.400.400.400.500.500.500.500.501.001.00130.400.400.400.400.400.500.500.500.500.501.001.001.00140.400.400.400.400.400.500.500.500.500.501.001.001.001.00150.500.500.500.500.500.600.600.600.600.600.900.900.900.901.00160.500.500.500.500.500.600.600.600.600.600.900.900.900.901.001.00170.500.500.500.500.500.600.600.600.600.600.900.900.900.901.001.001.00180.500.500.500.500.500.600.600.600.600.600.900.900.900.901.001.001.001.00190.500.500.500.500.500.400.400.400.400.400.900.900.900.900.800.800.800.801.00200.500.500.500.500.500.400.400.400.400.400.900.900.900.900.800.800.800.801.001.00210.500.500.500.500.50.400.400.400.400.400.900.900.900.900.800.800.800.801.001.001.00220.500.500.500.500.500.400.400.400.400.400.900.900.900.900.800.800.800.801.001.001.001.00230.600.600.600.600.600.500.500.500.500.500.800.800.800.800.700.700.700.700.900.900.900.901.00240.600.600.600.600.600.500.500.500.500.500.800.800.800.800.700.700.700.700.900.900.900.901.001.00250.600.600.600.600.600.500.500.500.500.500.800.800.800.800.700.700.700.700.900.900.900.901.001.001.00260.600.600.600.600.600.500.500.500.500.500.800.800.800.800.700.700.700.700.900.900.900.901.001.001.001.00270.700.700.700.700.700.600.600.600.600.600.700.700.700.700.800.800.800.800.800.800.800.800.900.900.900.901.00280.700.700.700.700.700.600.600.600.600.600.700.700.700.700.800.800.800.800.800.800.800.800.900.900.900.901.001.00290.700.700.700.700.700.600.600.600.600.600.700.700.700.700.800.800.800.800.800.800.800.800.900.900.900.901.001.001.00300.700.700.700.700.700.600.600.600.600.600.700.700.700.700.800.800.800.800.800.800.800.800.900.900.900.901.001.001.001.00
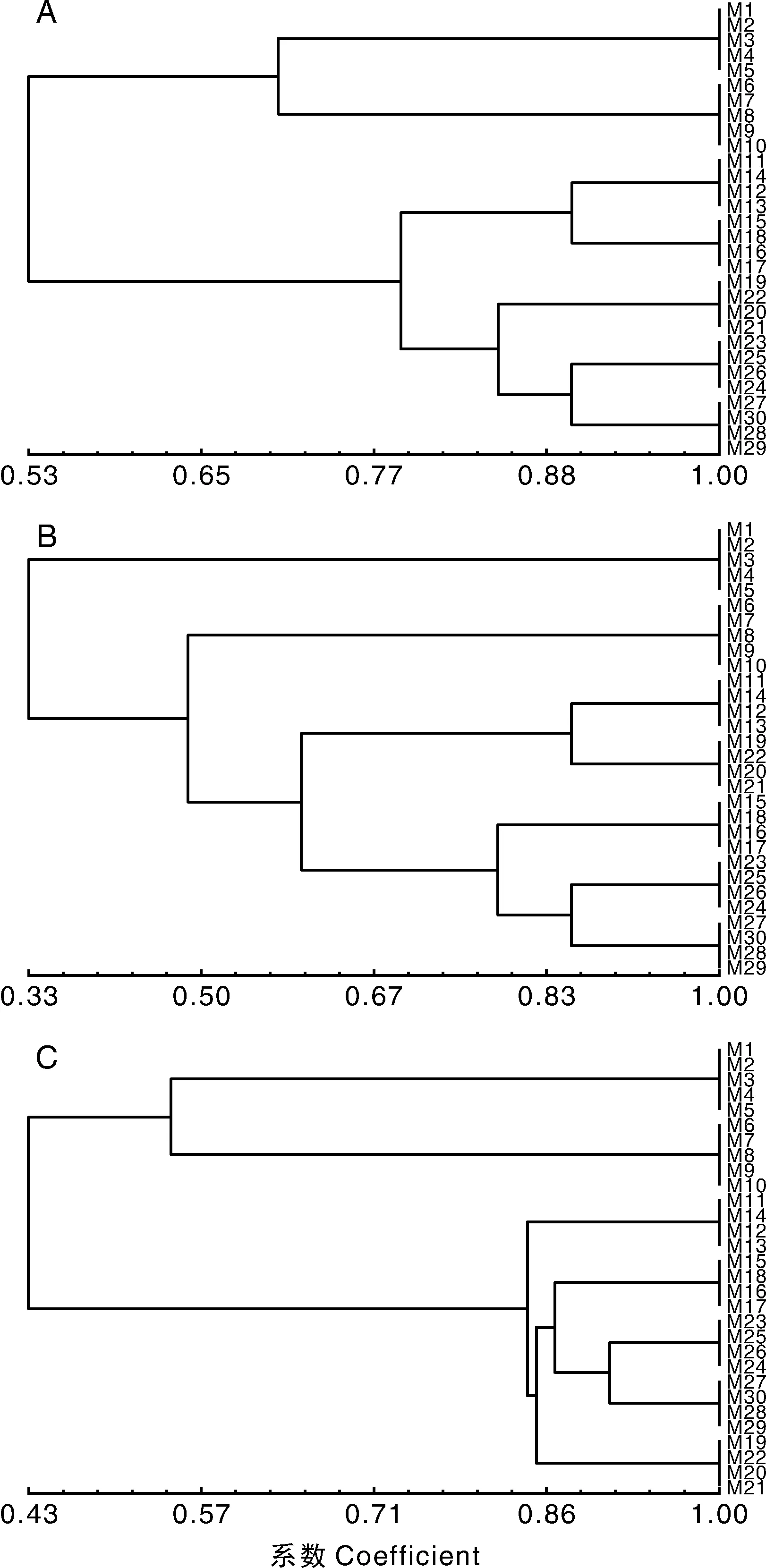
图3 POD(A)、EST(B)、PPO(C)同工酶聚类分析结果Fig.3 Cluster analysis results of POD(A), EST(B), PPO(C) isozyme
3 讨论
同工酶是指催化功能相同而结构及理化性质不同的一组特异性蛋白质,其蛋白多肽链结构中的氨基酸排列顺序是由DNA上结构基因所携带的遗传信息决定的[3]。同工酶分析是一种有效的生化标记技术,它与形态学及分子分类方法相比有自身的特点和优点,因而被广泛地应用于作物种质资源亲缘关系和遗传多样性分析[4]。近年来,随着分子生物学的发展,利用DNA分子标记技术研究生物多样性也越来越广泛,但相比而言,同工酶分析方法和设备操作简单,且具有试样需要量少、成本低、实验所需时间短、分析灵敏度高、共显性表达、能间接反映DNA水平的变化等优点,已被广泛应用于遗传、生物多样性和群体遗传学等研究[8]。常用的同工酶主要有过氧化物酶(POD)、过氧化氢酶(CAT)、超氧化物歧化酶(SOD)、酯酶(EST)、多酚氧化酶(PPO)、淀粉酶等。丁玲等[9-10]对菊花品种的遗传多样性进行了POD和EST同工酶检测,结果表明,菊花品种间POD和EST的遗传变异丰富,且菊属8个种27份材料聚类为两组,第Ⅰ组为野生菊、药用菊,第Ⅱ组为观赏菊。隋益虎等[4]基于叶片的POD、SOD及花蕾的EST同工酶酶谱的分析发现,32份辣椒材料的遗传分化程度较大。王述民等[11]对58份野生小豆和249份栽培小豆种质资源进行了EST、POD、苹果酸脱氢酶(MDH)和SOD的检测分析,共检测到6个基因位点,33个等位基因,可划分为5个组群,类群之间存在明显的遗传差异。刘贺贺等[5]对75份蕨麻进行了POD同工酶分析,结果与形态学标记、细胞水平、分子标记研究的结果一致,具有丰富的遗传多样性。胡树贵等[12]对44个百合品种叶样进行了POD、EST、SOD、PPO同工酶检测,结果表明,东方百合与野生百合的遗传相似度最小,亲缘关系最远,而日本百合与野生百合的遗传相似度最大,亲缘关系最近。陈万秋等[13]对猕猴桃10个品种进行EST同工酶和POD同工酶的遗传多样性检测,结果表明,猕猴桃种内遗传多样性水平较高。李景欣等[14]对内蒙古地区的16份野生冰草种质资源进行EST和POD同工酶分析,显示了较高的多态性,类群之间存在明显的遗传差异。沈镝等[15]用EST、POD、SOD、PPO、细胞色素氧化酶对云南48份芋材料进行同工酶分析,结果表明,云南省芋种质资源具有丰富的遗传多样性。在本实验中,6份高山马铃薯种质资源的POD、EST、PPO同工酶分析结果表明,云南高山马铃薯和恩施高山马铃薯均属于同一类,而怀玉山高山马铃薯单独成一类。关于马铃薯种质资源的遗传多样性分析,多采用DNA分子标记的方法,如SSR[16]、ISSR[17]、SRAP[17]、RAPD[18]、AFLP[19]等。对于马铃薯种质资源同工酶的检测仅限于转基因纯合四倍体马铃薯PPO检测[20]、马铃薯种间体细胞杂种植株POD检测[21]等,而关于高山马铃薯的研究也仅限于脱毒和繁育[22]。本实验通过POD、EST、PPO同工酶电泳分析了我国主要产地的高山马铃薯种质资源的遗传多样性,指出了云南高山马铃薯和恩施高山马铃薯的遗传分化较低,而怀玉山高山马铃薯为特有的种质。同时,本实验结果表明,对于同一马铃薯种质,取不同部位的材料(如块茎和叶片)来做同工酶检测会产生不一样的结果,这说明同工酶的检测需要保证材料选择的一致性。本实验结果可为我国高山马铃薯种质资源的保存和育种提供理论依据。

