宏条形码技术在食品物种鉴定中的应用及展望
邢冉冉,吴亚君,陈 颖
(中国检验检疫科学研究院,北京 100176)
近年来,国内外食品掺假造假事件时有曝光,由此产生的经济和社会影响引起世界各国的广泛关注。随着现代工业科技的迅速发展,掺假的手段和花样也在不断地翻新,从早期的缺斤短两、勾兑稀释等简单形式发展到利用现代科学技术手段进行的“弃真存伪”。同时,随着食品供应链触及的范围更宽泛,涉及到的因素更为复杂,食品的产地溯源也变得更加困难。食品生产中掺假造假的鉴定和食品标签制度的有效实施都必须有完善的食品物种鉴定和产地溯源方法作为检测的基础。因此,对食品中的物种来源进行有效鉴定是识别食品掺假造假问题的一项重要措施。
在过去的20 年中,大量基于DNA的分子检测方法,如聚合酶链式反应(polymerase chain reaction,PCR)技术、基因芯片技术和分子指纹图谱技术等得到迅速发展并在食品物种鉴定领域得到了广泛的应用[1]。但是随着检测需求的日益变化,现有方法在一定程度上存在如程序复杂、费时费力、鉴定结果精确性低等不足。2003年加拿大动物学家Paul Hebert教授首次提出了DNA条形码的概念,一经提出便迅速成为分子分类学以及分子鉴别技术的核心方法,在生物物种鉴定方面发挥了重要作用[2]。然而,无论是常规的基因检测技术,还是DNA条形码技术,通常一次只能检测出一种或者少数几种物种,而不能快速分析出数百万的基因序列。而多数情况下,待测样品通常是多种不同物种的混合物,且有些物种的含量占样品总量的比例极低(低于1%)[3]。特别是随着食品产业的发展,食品的品种越来越多,加工越来越精细,成分越来越复杂,物种的原始特征已经消失,因此同时对多物种进行定性定量检测的需求越来越大。基于高通量测序技术的宏条形码技术在该方面具有良好的应用前景。
本文对宏条形码技术进行了介绍,并对其在食品物种鉴定领域的应用、面临的挑战及发展前景进行了探讨。
1 宏条形码技术概述
1.1 DNA条形码技术
DNA条形码技术是利用基因组中一段标准的、相对较短的DNA片段作为物种标记,通过进化树分析来描述物种间的亲缘关系,进而对物种进行鉴定的一项技术[4]。在过去的十几年间,大量的科学研究机构和组织已经发现了多个具有物种学信息的基因片段。其中,线粒体细胞色素c氧化酶亚基I(cytochrome oxidase subunit I,COI)的一个特定片段DNA序列作为分辨动物界中近缘物种的标准片段[5];叶绿体核糖激酶1,5-二磷酸羧化酶基因(ribulose 1,5-bisphosphate carboxylase,rbcL)和蛋白成熟酶K基因(maturase K,matK)以及核基因片段(internal transcribed spacer,ITS)被用作植物DNA条形码[6];核糖体DNA则常被用作鉴定真菌和细菌等微生物的DNA条形码[7]。
随着生命条形码联盟(Consortium for the Barcode of Life,CBOL)的建立,DNA条形码技术被迅速用于生物多样性分析及保护、物种鉴定、生物的遗传进化、检验检疫、法医学、生物安全与公共健康、农业生产、道地药材产地溯源、食品真伪鉴别和产地溯源等多个方面[8-11]。与传统的分类手段相比,DNA条形码技术具有以下优点:1)不受个体发育阶段和基因片段的限制,甚至对已降解的样品也可以进行分析;2)加快了对已知物种的识别速度,同时便于发现新物种;3)加速了便携式手持设备的研发,可以尽快实现现场完成样品的处理、扩增、测序和鉴定;4)为生物的分类提供了条件[12-16]。然而,在显现优势的同时,基于Sanger测序法的DNA条形码技术的局限性也非常明显,其中一点就是无法同时完成对多个体多物种混合样品的高效快速分类及评估[17]。
1.2 基于高通量测序的宏条形码技术
随着对生命科学的深入研究与生物技术的进一步发展,人们对DNA测序技术的要求不断提高,传统的Sanger测序已经不能完全满足研究的需要。高通量测序技术的出现彻底改变了DNA分析的方式。以Illumina测序平台为例,其主要技术特点就是可以边合成边测序,DNA片段加上接头之后,可以随机附着于玻璃表面,通过桥式PCR扩增形成具有DNA分子克隆片段的DNA分子簇;基于可逆终止化学反应的原理,对数百万个片段同时进行大规模平行测序;测序反应结束后,成像系统能够捕捉荧光标记的核苷酸,其中一个簇的图像数据就是一个DNA序列;随后将图像数据转换为碱基序列信息,完成测序。与传统的Sanger测序技术只能对单一的DNA分子进行测序不同,高通量测序技术可以获得样本中每个DNA分子的序列,具有测序通量高、速度快、成本低等优点。近年来,随着测序技术的不断发展成熟、测序成本的不断降低和生物信息学分析软件的进一步完善,高通量测序技术的应用领域已经从最初的基因组科学研究逐渐延伸到生物技术、食品安全和医药卫生等各个领域。
宏条形码技术就是近年来随着高通量测序技术的发展而出现的一种新型物种鉴定技术。这种技术结合了DNA条形码技术和高通量测序技术的共同优点,可以获得来自于混合样本中所有目标DNA片段的序列,然后将这些序列与合适的数据库进行比对即可确定其代表的物种,从而可以分析混合样本中的物种组成[18]。2012年,Taberlet等[18]将宏条形码技术定义为“利用从环境样本中(例如土壤、水、粪便等)提取的总DNA和降解DNA来进行高通量多物种鉴定的技术”。需要指出的是,宏条形码与宏基因组是完全不同的两个术语,前者不涉及到基因组水平上的任何功能分析。
宏条形码技术最突出的优点就是可以更加快速、准确地对混合样本或环境样本中所包含的生物体进行鉴定,能够实现同时检测复杂样品中多个物种的目的。其优点主要包括:1)测序对象为短的PCR扩增子,有利于对降解和低分子质量的DNA样品进行分析;2)采用通用的PCR引物,因此可以对未知物种进行分析;3)经济,同时对大量样本分析降低了每个样本的成本;4)深度测序,增加了检出微量DNA的可能性[3]。这些优点对于食品类型鉴定,特别是对于未知食品、复杂食品或者深加工食品来说有着非常重要的作用,为分析食品中的物种成分提供了一个新的研究手段。
在对食品中的物种成分进行分析时,宏条形码技术的实验流程基本包括6 个步骤[19],具体见图1。宏条形码技术与DNA条形码技术在实验流程上大致相同,但是在具体操作中,两者又有很大不同,例如分子标记的选择、涉及到的测序技术和数据处理过程等。宏条形码技术所涉及到的这些步骤看似简单,但在实际操作过程中,遇到的问题及解决方案因研究目的而异,因此目前并没有统一的、标准的详细操作流程,各个步骤的具体操作还需要参考相关领域己有的研究来确定[20]。
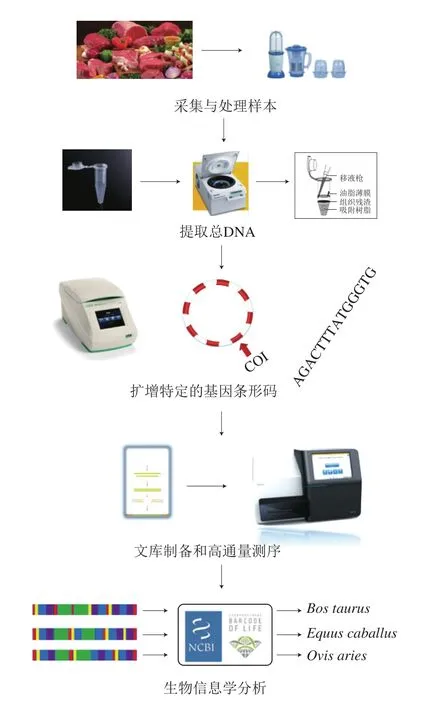
图1 宏条形码鉴定食品物种成分的流程Fig. 1 Major steps of metabarcoding for food species identification
2 宏条形码技术在食品物种鉴定中的应用
传统的食品物种来源主要依靠生物的表型及解剖特征等进行感官鉴定。但是对于加工处理后的食品,感官检验的难度及准确性将大大降低且感官检验对检验人员的经验要求较高。在分子生物学技术得到成熟应用之前,理化分析方法在食品种类和真伪鉴别中应用最广。随着科学的发展,基于核酸的分子生物学技术发展很快,与理化方法不同,分子生物学技术对物种的判别基础是遗传物质,通过遗传物质从而保证检测结果的确定性和重现性。如DNA芯片技术、指纹图谱技术、DNA条码技术等。但当检测对象是复杂食品、未知食品或者数量特别庞大的样品时,这些传统的分子检测方法和DNA条形码技术都开始显现出不足之处。宏条形码技术的出现为这一问题的解决提供了一种更加便捷经济的方案。
宏条形码技术在物种鉴定方面的应用早期主要集中在微生物群落多样性的研究[21-22],现已广泛应用在真菌[23]、无脊椎动物[24]、植物[25]的物种鉴定,食草和食肉动物的饮食成分分析[26-27]以及环境样品的生物多样性[18,28-29]等研究中。宏条形码技术已在食品物种鉴定方面有初步的研究,目前已经逐渐应用于动物源性食品、植物源性食品、复杂食品和深加工食品的物种鉴定等方面。
2.1 动物源性食品
不同肉类、禽类品种之间的价格差异很大,因此肉类产品的掺假主要是品种掺假。针对动物源性食品掺假现象,宏条形码技术的主要应用对象是混合肉制品,这也体现了宏条形码技术在混合样品物种鉴定方面的优势。在一项针对动物源食品DNA混合物物种成分鉴定的研究中,研究人员首先将猪、马、牛、羊、兔、鸡、火鸡、山鸡、鸭、鹅、鸽子、人类、老鼠共13 种生物的DNA按照不同比例进行混合,然后以12S_KH(215~222 bp)、16S_KH(112~121 bp)和16S_Ki(243~249 bp)作为分子标记,设计通用引物,分别进行PCR扩增后得到目的基因片段[30]。随后,在该实验中研究人员构建测序文库,并利用Ion Torrent(Life Technologies)测序平台对PCR扩增产物进行测序,最后利用不同的比对算法对测序数据进行分析。研究结果显示,以利用Sanger测序法获得的测序结果作为参照,利用此高通量测序方法测得的不同物种的序列错误率在0.000 3~0.020 0之间,并能很好地将样品中的不同物种成分进行鉴别。与此研究类似,Tillmar等[3]利用初级版的高通量测序仪GS Junior对包含大量哺乳动物物种的混合样品物种成分进行了准确的鉴定,并检测出了含量比例低至1%的物种成分。此项研究中所选用的分子标记是一段长度约为100 bp的线粒体16S rRNA基因片段,这类序列长度相对较短的条形码称为DNA微型条形码,主要应用于DNA发生降解的一些样品的鉴定。微型条形码的出现不仅在一定程度上弥补了高通量测序技术测序长度有限的缺陷,还能够有效地对DNA已大量降解的加工食品进行鉴定。基于微型条形码的这些特点,Galan等[31]以片段长度为136 bp的细胞色素b作为分子标记片段,设计出啮齿类动物的通用引物,然后利用高通量测序仪454 GS-FLX较为高效地鉴定出了混合样品中所包含的未知物种;此项研究的对象虽然是啮齿动物,但是对非啮齿类动物同样有效,也可应用于其他物种的鉴定,为宏条形码技术在加工食品鉴定中的广泛应用奠定了理论和技术基础。
DNA条形码技术已经广泛地应用于鱼类产品的分类和鉴定,绝大多数常见的鱼类都可以用DNA条形码技术进行鉴定[32]。高通量测序技术的出现则进一步推动了DNA条形码技术在水产业中的应用。最近,Galal-Khallaf等[33]利用基于454测序平台的宏条形码技术对埃及水产饲料样品中使用的鱼类是否属于濒危物种进行了研究;结果显示,草食性鱼和杂食性鱼的饲料组成存在着细微差异;此研究还发现,在所检测到的所有鱼类中有大约46%的鱼类存在被过度开发或者数量正在急剧减少的现象;在此项研究中,高通量测序技术为水产业中可追溯性系统的有效实施提供了工具。
2.2 植物源性食品
随着国际贸易的发展和经济的全球化,市场上的农产品和植物源性食品来源越来越广泛,种类也越来越多,对这类食品进行真伪鉴别和产地溯源就变得愈加困难。在欧盟,针对相关问题,已有相应的规定开始颁布并实施,例如食品中所含物种成分(包括过敏原)的正确标识,橄榄油、葡萄酒和面食的掺假鉴定以及可可和咖啡的合法贸易界定等[34]。相比传统的食品真伪鉴别方法,宏条形码技术作为一种基于高通量测序技术而发展起来的新的物种鉴定技术,在目前植物源性食品的鉴定工作中应用并不广泛,但是已经显现出很大的优势,例如在混合植物源食品、加工食品的物种鉴定方面都将具有更大的应用空间。
最近,Coutimho Moraes等[35]在基于DNA的植物和植源性膳食补充剂鉴别技术的综述中就强调了利用高通量测序技术对植源性膳食补充剂和中草药成分进行检测以及真伪鉴别的前沿性。综述指出,除宏条形码技术之外,基于高通量测序技术的靶向富集和叶绿体基因组测序同样具有鉴别植物物种的巨大潜力;但是,其中涉及到的复杂生物信息学知识和实验流程可能会限制此类技术的普遍使用。此外,对于食品的物种鉴定来说,对整个质体进行基因组测序还可能会存在一些其他问题,例如较难获得质体完整基因组或质体基因组出现断裂和降解等。因此,相比于其他基于高通量测序的食品物种鉴定技术,宏条形码技术更有可能成为一种可以得到广泛应用的标准化工具。
我国中医药理论有药食同源的观点。在中药分析领域,高通量测序技术已经开始得到应用。例如,Coghlan等[36]利用罗氏GS Junior测序平台对混合中药样品进行了分析,以trnL和16S rRNA作为分子标记,并利用宏条形码技术在一些中药中鉴定出了濒危物种和一些可能有毒或致过敏的成分。在另外的一项针对六味地黄丸物种成分鉴定的研究中,Cheng Xinwei等[37]利用了NCBI等数据库中所有已知的ITS2和trnL序列建立了小型数据库,并基于此数据库利用Parallel-Meta等方法搜索和鉴定六味地黄丸中的物种成分,利用Meta-Storms等方法对不同样本的物种来源进行比较,最后成功地对处方物种和非处方物种进行了鉴定,有效地鉴定出了中药的种类和产地。为了研究高通量测序技术在中药鉴定中的分析效果,Ivanova等[38]分别利用Sanger测序和高通量测序技术对来自不同药用植物以及不同生产商的共15 种中草药添加物进行了分类鉴定,研究结果显示,相比于Sanger测序,高通量测序技术对低含量物种的检出率更高。研究指出,宏条形码的方法可以为植物和真菌的DNA检测提供可靠的指导,并可用于原材料来源真实性保障以及生产过程和最终产品的污染控制。
2.3 复杂食品和深加工食品
对于复杂食品的真伪鉴别,从本质上可以追溯为鉴定其生物成分。当食品是多品种混合物时,其成分复杂且含量不一致。利用宏条形码技术对复杂食品的成分进行鉴定时,可以非特异性地将所有主要物种及杂质物种都检测出来,在生物混合体系研究方面具有更大的优势。
近年来消费者对蜂蜜的需求量逐年增加,然而由于利益驱动,蜂蜜掺杂使假的现象时有发生,已成为目前蜂产业生产中不容忽视的问题。蜂蜜中既包含植物源信息(蜜源植物),又包含动物源信息(蜂源),鉴于蜂蜜的多物种来源特点,Prosser等[39]基于Ion Torrent PGM(Life Technologies)测序平台,以ITS2、rbcLa和COI 3 个基因片段作为分子标记,利用宏条形码技术对7 种不同产地和加工方式的蜂蜜进行了真伪鉴别。其中,核基因ITS2(约350 bp)用以鉴定蜂蜜中的花粉来源;叶绿体基因片段rbcLa(162 bp)用以鉴定蜂蜜中痕量或者降解的植物DNA;COI(120 bp)用以鉴定其蜜蜂来源。此研究利用宏条形码技术对蜂蜜的植物来源和昆虫来源进行了较为准确地鉴定,为蜂蜜的真伪鉴别和产地溯源提供了一个新的解决方案。与此类似,还有研究利用宏条形码技术探寻动植物之间的联系,例如,Pornon等[40]利用宏条形码技术对植物-昆虫之间的联系进行了研究,通过将实验室检测和现场检测相结合,成功地对花粉混合物和昆虫携带的花粉中的物种来源进行了鉴别;此外,此项研究还显示出宏条形码技术在直接定量(以扩增序列数目代表数量)和半定量(相对定量)方面的潜力。在蜂蜜的鉴别方面,还有一些研究将宏条形码技术与其他检测技术进行了结合,例如Richardson等[41]利用Illumina MiSeq测序平台,以ITS2(461~469 bp)作为分子标记,利用宏条形码技术,结合传统的显微镜观察的方法对蜂蜜中花粉的来源进行了分析。该研究结果表明:如果研究目标是对花粉进行定性分析,宏条形码技术无论在灵敏度还是准确率上都更有优势;如果研究目标是对花粉进行定量分析,联合使用宏条形码技术和显微镜观察技术比单独采用其中任何一种技术的效果更好。这一研究也提示我们,在食品物种鉴定的研究过程中,任何一种研究方法都有一定的局限性,而多种检测方法联合使用,具有单种方式不可比拟的优势。
在深加工食品的鉴定中,宏条形码技术也已得到初步应用。Muñoz-Colmenero等[42]利用PGM测序平台对不同类型糖果中所包含的动物物种成分进行了分析,以16S核糖体基因作为分子标记,并将分析结果与常规的DNA条形码技术进行比较;研究结果显示:绝大多数利用PGM测序平台进行测序分析的结果与利用DNA条形码技术进行物种检测的结果一致;而基于PGM测序平台的高通量测序技术能够从糖果样品中检测到更多的动物物种,并且检测的灵敏度更高;但是,利用PGM测序平台获得的物种序列中含有更高的碱基对AT含量。除此之外,该研究还指出利用高通量测序技术在分析复杂度相对较低的食品方面具有更大的优势,但在对高度加工的食品进行物种鉴别和追溯的应用方面尚不成熟。
3 宏条形码技术面临的挑战
虽然基于高通量测序的宏条形码技术可以为生物学研究提供快速、简便与经济的物种鉴定方法,在操作层面的难度也并不大,但是这一技术却并非完美,仍有一些问题需要解决。首先,高通量测序技术虽然可以提供海量的数据,但却存在一定的错误率,测序质量有待提高。尤其对于宏条形码技术来说,因其是利用PCR扩增产物进行文库构建后对该文库进行测序,因此容易产生文库中碱基不平衡的现象(A、C、T、G 4 个碱基分布不均匀),导致测序仪在数据读取时会产生误差,使得测序数据质量降低、有效数据量减少。其次,不同的样品前处理条件和实验过程可能导致DNA的质量和浓度出现较大的差别[43],而DNA的完整性对宏条形码技术和其他用于物种鉴定的分子生物学实验效果都有较大的影响[37,44-45]。再次,虽然目前已有许多可用于宏条形码数据分析的生物信息学方法,但是这些方法的识别率很大程度上与条形码的选择以及参考数据库的组成直接相关[46-47]。此外,由物种间的可变引物-模板错配造成的PCR偏差可能会影响宏条形码技术在定量方面的应用,并且有可能导致某些物种无法检测[48-49]。最后,宏条形码技术的有效应用需要有较好分类和较高条形码覆盖率的条形码序列参考数据库做后盾。
3.1 条形码选择的局限性
高通量测序技术的测序读长普遍较短(长度为35~700 bp),而这可能是此技术在物种鉴定的应用方面存在的一个比较大的限制因素。虽然一些长度为200~300 bp的微条形码也可以用于物种鉴定,但是目前较常用的植物条形码(rbcL和matK)长度大概都在500~600 bp之间。罗氏454焦磷酸测序平台可以提供此长度范围的读长,这一平台也已经成功用于植物的宏条形码测定[50-51];但是,由于成本与应用范围过于狭小,454测序平台已停产。目前,各方面发展较好的是Illumina公司的MiSeq测序平台,其测序读长可以达到双侧长度2×300 bp,且其测序准确性相对较高(错误率0.003~0.004)[6,52];当然,还有其他一些长读长的测序平台,例如Pacific Biosciences和Oxford Nanopore Technologies。每个测序平台都有其优劣,因此,在应用宏条形码技术进行物种鉴定时,除了要考虑条形码的分辨率和引物的通用性等问题,还需要根据测序仪的测序长度来选择合适的条形码长度[53]。
对于DNA保存较为完整的样品,其DNA提取较为容易,能够扩增出较长的目的片段,因此可以采用较长的条形码,如长度为658 bp的COI基因片段[54];对于DNA高度降解的样品,其DNA提取较为困难,难以扩增出较长的基因片段,因此只能选择较短的条形码,如长度约为130 bp的COI基因片段[55]。在条形码的选择方面,宏条形码技术与传统的DNA条形码技术并不完全相同[18]。例如,在利用宏条形码和传统的DNA条形码技术对动物进行物种鉴定时,因为COI基因的高辨别度,所以通常都是选择COI基因片段作为条形码,但此基因的引物结合区域保守度并不高[56]。经验表明,当样品中所包含的物种覆盖分类范围特别广时,引物的变异性就会使得扩增结果变得不可靠[57]。在使用传统的DNA条形码技术时,可以通过优化实验方案从最初扩增失败的物种中获取数据;而当使用宏条形码技术对混合样品进行鉴定时,可能由于样品中其他类群的扩增子掩盖了某些未能扩增的特定类群,使得实验方案的优化较为困难。
在物种信息的确定方面,宏条形码技术与传统的DNA条形码技术相同,得到的未知靶标都必须要与参考数据库中已被鉴定的靶标进行比对才能获知物种信息;因此在利用宏条形码技术时,参考数据库的不完善成了一个很大的受限因素[58]。具体到宏条形码技术在食品中的应用,其局限性还包括:1)加工食品中的DNA通常是高度降解的,一些较长的基因长片段可能并不能完全准确地扩增出来;2)在一些复杂食品中,由于多个物种需要在同一个PCR管中扩增目的条带,这就要求所用的引物必须具有高度通用性,也就是说在各物种间扩增效率要一致。
简言之,在利用宏条形码技术对食品中的物种成分进行鉴定时,对于条形码的选择有一定的局限性,也因此变得非常关键。在实际的研究工作中,应该寻找通用性和特异性较强的基因片段作为标准DNA条形码。同时,为了减少由于条形码选择不当而带来的误差,多个条形码的联合使用有时非常有必要。大量的研究结果表明,使用多个条形码可以更全面地鉴定到样本中所包含的物种,更准确地区分不同物种[30,53,59];尤其当样本中包含的物种类型十分广泛时,由于每个条形码能够鉴定到的物种类群不同,联合使用多个条形码有助于更好地达到研究目的。此外,急需构建包含多种食品物种的条形码参考数据库,可为宏条形码技术的应用提供数据基础。
3.2 定量的局限性
定量问题是目前在应用宏条形码技术进行物种鉴定时存在的一个相对难以解决的问题,这是由于在此技术的应用过程中,存在PCR偏差、基因的多拷贝性以及实验流程的不同等问题,这会导致测得序列的数量与实际样品中的物种数量并没有很强的相关性;因此难以利用该技术对样品中的物种成分进行定量分析。目前普遍认为定量困难主要是由PCR过程中引物和模板错配以及纯粹的随机效应而造成的[20,60]。在利用宏条形码技术时,需要设计特异性探针,然后与基因组DNA进行杂交,经过PCR扩增后得到基因组目标区域的DNA片段;这个过程造成了该方法存在一个较大的缺陷,即PCR过程会产生偏差[20,60]。PCR偏差与引物-模板错配、寡聚核苷酸的浓度、退火温度和PCR循环数等因素有关[48];其中,引物-模板错配起最主要的作用[48],这与通用引物的选择有一定的关系;但是不管选择何种通用引物,都不能避免引物与模板的错配发生[61-62],最终都会导致一些物种的相对丰度增加,另外一些物种的相对丰度降低,甚至还会出现目标片段无法得到扩增的现象[48]。即使某种通用引物能够将所有的物种都扩增出来,但是由于不同物种间出现错配的情况不可能完全相同,也不能解决扩增效率不一致的问题。
事实上,关于利用高通量测序技术对PCR扩增产物进行测序的情况,很少有研究指出能够利用此技术对样品中的物种成分进行定量分析。虽然有报道提到测序的相对丰度与样品中的物种含量存在一定的相关性[63],但也仅是变化趋势大致相同而已,并不完全相关,而且测定序列的相对丰度与样品中的物种含量在一些细节上也有出入。在宏条形码技术的应用研究中,很多研究都会对定量问题进行讨论,但是至今还未得到一个完美的解决方案;大部分文献都认为利用宏条形码技术进行物种鉴定时,序列的相对丰度与样品中物种含量的相对丰度不存在相关性[39,48,64]。因此,目前看来,测定序列的相对丰度并不能作为对样品中物种含量进行定量分析的依据。
目前,针对宏条形码技术实验过程中出现的偏差因素进行改善的措施主要集中在改变单个物种产生的偏差,或者是改善实验步骤方面[65-66]。最近也有一些研究针对偏差的修正问题进行了探讨,认为不同物种间模板DNA的拷贝数或者DNA的浓度不同可能造成一些物种过量扩增,而另外一些物种扩增量较低的现象[67-68]。通过修正拷贝数和优化实验方法可以在一定程度上提高宏条形码技术对于物种定量的能力;此外,还可以通过设置对照组来修正单个样品在实验过程中产生的偏差[69-70]。但是,除此之外还存在其他的技术因素阻碍了研究人员利用测定序列数的比例来判断样品中各物种的量或者比例。为了控制实验过程中由偏差带来的影响,Thomas等[71]通过将目标物种和对照物种按照50∶50的比例进行混合,得到可以修正多种来源偏差的修正因子。这种通过计算修正因子来降低实验偏差影响的方法可以在一定程度上评估和修正宏条形码研究中出现的偏差;但是,此方法仅适用于在目标物种已知,且目标物种种类有限的情况下对混合物中的物种成分进行定量。
为了解决由于PCR扩增过程中的偏差引起的定量困难问题,一些研究人员开始尝试绕过PCR步骤,直接对提取的DNA进行测序分析。这种实验方法与目前的宏基因组测序方法类似[72-73],但是其实验目标并不是对全基因组进行拼接或者寻找功能基因,而是通过具有代表性的DNA片段来进行物种鉴定。具体做法是,对感兴趣的基因组区域设计特异性探针,与基因组DNA进行杂交,将目标基因组区域的DNA片段进行富集后,再利用高通量测序技术进行测序。这种测序方法称为目标捕获测序,但是这种方法还是无法彻底解决定量的问题;因为目前尚无法确定此类实验中涉及到的包含物种信息的序列读长,如线粒体、叶绿体和核糖体DNA的比例等信息[18]。例如,不同物种间核糖体的拷贝数各不相同[74],即使是同一物种,不同组织部位不同细胞中的线粒体基因组的数量也有不同[75],这些因素都会影响到定量结果的准确性。
鉴于以上方法都无法完全解决利用高通量测序技术对食品中的物种成分进行定量分析的问题,一些研究也在尝试利用全基因组测序的方法来解决这一难题。2014年,Ripp等[76]利用Illumina公司的 HiSeq 2000测序平台对包含哺乳动物(猪、牛、马、羊)和禽类(鸡、火鸡)在内的肉制品的肉肠进行了全基因组深度测序,随后对测定的序列进行生物信息学分析;其所建立的方法一方面可以从复杂的物种中准确地鉴别出特定物种,另一方面也可以实现对复杂食品中的主要成分和未知成分进行定量分析。但是,这项研究仅仅实现了对已知物种成分的定量分析,所建立的参考数据库也仅仅包含少数几种物种。而且到目前为止,全基因组测序的成本仍然很高,且参考基因组的数量有限。因此,对复杂食品中的所有未知物种成分进行全基因组测序,不管是从成本还是数据分析方面来说,都有很大的挑战。
4 结 语
目前,在食品物种鉴定方面,高通量测序技术虽然已经得到了应用,但在国内外尚处于起步阶段。与此形成鲜明对比的是该技术在人类疾病诊断和预防中的研究及应用实例已经不计其数,并且在国内外均有经审批上市的诊断产品用于无创产前、肿瘤分型、遗传病筛查等方面的日常临床实践。一方面,由于高通量测序技术对于食品物种鉴定领域来说相对较新颖,且目前的测序成本相对来说较高,所以只有在基础设施相当完善的实验室才能应用此项技术;另一方面,测序之前的准备工作也缺少统一的标准,例如样品处理、DNA提取等。最重要的一点,这一系列的生物信息学分析流程掌握起来相对困难,对测序结果的解读也有一定的技术要求,尤其是当检测的基序比对不到具体物种时;例如,当检测的样品为深度加工食品和复杂食品时,测序结果的完整解读就存在很大的困难。在数据分析阶段,虽然目前已有一些专门的软件可以对测序结果进行分析,但是仍然需要更加准确有效的分析手段;同时为了完成一系列的生物信息学分析流程,高技能的生物信息学专业人员也是必不可少的[77-78]。只有当高通量测序技术达到操作自动化和标准化之后,这种方法才能真正广泛地应用于食品安全领域的相关工作中。
虽然基于高通量测序的宏条形码技术在食品物种鉴定的实际应用中还需要不断改进,但其仍然具有快速、经济、准确等特点。随着测序读长的增加、理论与技术的不断完善,宏条形码技术的应用将使复杂食品的物种鉴定研究变得更加快速简便。尤其是在过去的10 年中,技术的不断发展已经大大降低了测序的成本,并且显著增加了测序的通量;全(半)自动的生物信息学分析软件也已开发出来;所有这些技术进步都预示着高通量测序技术的成本在未来会不断降低至可接受水平。
此外,随着测序技术的发展,单分子测序或第三代测序已经开始兴起并得到应用。单分子测序技术的突出优点为:1)无需PCR扩增,直接对单个分子进行测序;2)具有更快的速度和更低的成本;3)测序读长更长。这些特点对于宏条形码技术的应用将会起到推动作用。相信在不久的将来,宏条形码技术会更加简单、便宜,并能得到更广泛的应用,这类检测方法在食品安全领域也将占据更大的空间;在未来的食品安全监管中,这一分析技术也将成为不可或缺的重要组成部分。

