克雷伯肺炎杆菌2,3-丁二醇合成途径功能基因研究
周吉东,郝晓蔚,王 敏,史吉平,郝 健*
1.天津科技大学生物工程学院,工业发酵微生物教育部重点实验室,天津市工业微生物重点实验室,天津 300457
2.中国科学院上海高等研究院,上海 201210
3.河北科技大学,河北 石家庄 050018
克雷伯肺炎杆菌(Klebsiella pneumoniae)是一种重要的工业微生物,可用于生产1,3-丙二醇[1]、2,3-丁二醇[2]、2-酮基葡萄糖酸[3]、乙偶姻等[4]。2,3-丁二醇作为一种平台化合物,被广泛应用于塑料、溶剂、合成橡胶等领域,并且是一种重要的潜在生物能源[5]。自然界中有许多微生物可以合成2,3-丁二醇,其中生产效率较高的有克雷伯肺炎杆菌(Klebsiella pneumoniae)、产酸克雷伯氏杆菌(Klebsi ella oxytoca)[6]、粘质沙雷氏菌(Serratia marcescens)[7]和多粘芽孢杆菌(Bacillus polymyxa)[8]。
2,3-丁二醇生物合成途径从丙酮酸开始,两分子丙酮酸在α-乙酰乳酸合成酶的催化下形成乙酰乳酸,乙酰乳酸在脱羧酶催化下形成乙偶姻,乙偶姻进一步还原形成2,3-丁二醇。在克雷伯肺炎杆菌中2,3-丁二醇代谢途径相关基因形成bud操纵子[9]。budB编码α-乙酰乳酸合成酶,budA编码乙酰乳酸脱羧酶,budC编码丁二醇脱氢酶。乙酰乳酸除了用于合成2,3-丁二醇外,还是合成缬氨酸的前体,在缬氨酸合成途径中同时存在ilvB和ilvI两个乙酰乳酸合成酶编码基因。但是目前这三个乙酰乳酸合成酶基因在克雷伯氏肺炎杆菌合成2,3-丁二醇过程中的贡献并不清楚。
课题组先期关于2,3-丁二醇异构体合成机理的研究中发现:budC编码的丁二醇合成酶是菌株中主要催化乙偶姻到2,3-丁二醇的酶,budC突变株2,3-丁二醇的产量明显降低,并大量积累乙偶姻,但是菌体仍然合成少量2,3-丁二醇。这是因为除了budC编码的丁二醇脱氢酶外,dhaD编码的甘油脱氢酶也具有丁二醇脱氢酶活性,并且特异性的将乙偶姻的酮基转化成R构型的羟基[10]。另外gldA编码的甘油脱氢酶和acyI编码的短链酰基脱氢酶也被报道具有丁二醇脱氢酶活性[11,12]。但是这些基因编码的酶在菌株合成2,3-丁二醇中的贡献程度,以及除了这三个丁二醇脱氢酶外,菌株中是否还有其他丁二醇脱氢酶,并不清楚。
针对以上问题,本文构建了budB、ilvB、ilvI单突变株,和kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl四基因突变株。通过考察突变株合成2,3-丁二醇的变化情况,确定budB、ilvB、ilvI编码的三个乙酰乳酸合成酶在乙酰乳酸合成中的贡献,同时确定dhaD,gldA和acyl对菌株合成2,3-丁二醇的贡献。
1 材料与方法
1.1 材料
1.1.1 菌株、质粒及引物 本实验所用到的菌株及质粒见表1,所用引物见表2。
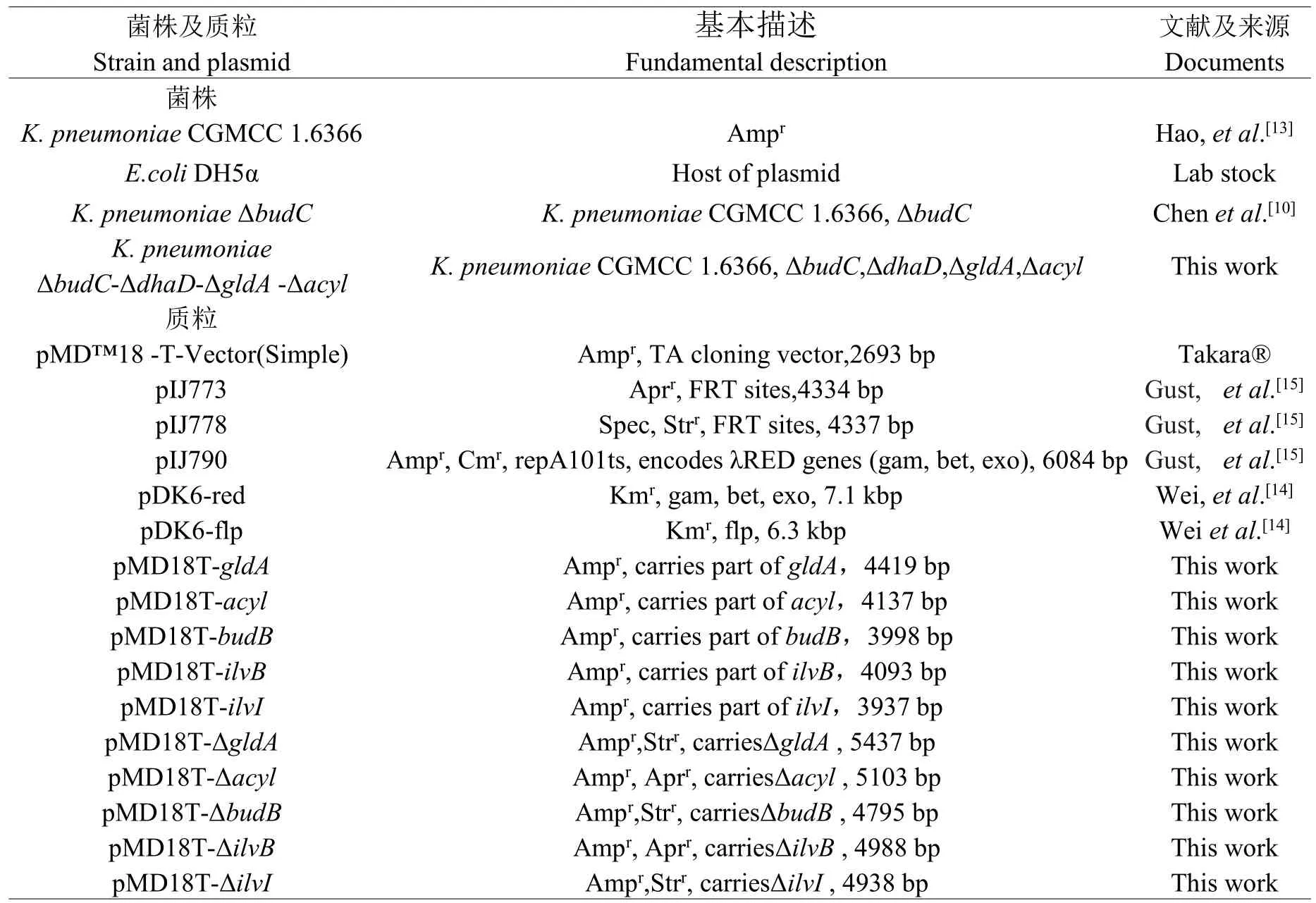
表1 实验用菌株及质粒Table 1 Experiment strains and plasmids
1.1.2 培养基及制备方法 种子培养基(g/L):氯化钠10,胰蛋白粉10,酵母粉5。种子培养基制备方法:称量好相应种子培养基试剂后用水定容至1 L,搅拌混匀后分别分装50 mL种子培养基于250 mL三角摇瓶内灭菌待用。发酵培养基(g/L):葡萄糖100,玉米浆4,(NH4)2SO45,KCL 0.4,NaAc 3,MgSO40.1,FeSO40.02,MnSO40.01,酵母粉 5。

表2 PCR实验所需引物Table 2 Primer for PCR experiment
1.2 方法
1.2.1 重组菌株的构建 克雷伯肺炎杆菌基因突变株构建利用Red重组酶辅助的同源重组系统,具体操作原料和方法过程见Wei D.et al.[14]。
以敲除gldA基因为例:首先是要构建带有目的基因长同源臂的同源重组质粒,以gldA-s1和gldA-a1为引物扩增克雷伯肺炎杆菌基因组,得到gldA片段,与PMD18T-vector(simple)连接,转化大肠杆菌DH5α感受态中,涂于氨苄霉素抗性平板,待长出菌落,利用菌落PCR筛选阳性克隆,得到菌株DH5α-T-gldA。提取质粒pMD18T-gldA,化转入带有表达Red重组酶质粒pIJ790的大肠杆菌感受态DH5α-PIJ790中,涂布于氯霉素和氨苄霉素双抗平板,待长出菌落,利用菌落PCR筛选阳性克隆,筛选获得的重组大肠杆菌DH5α-PIJ790-T-gldA。以质粒PIJ778为模板,利用引物gldA-FRT-s1和gldA-FRT-a1扩增获得链霉素抗性盒,该DNA两侧连接有39 bp的gldA同源臂。利用该抗性盒转化DH5α-PIJ790-T-gldA感受态细胞。涂布于链霉素抗性平板,菌落PCR验证筛选阳性单菌落,得到菌株 DH5α-T-ΔgldA。菌株中pMD18T-gldA质粒的 gldA基因已经被重组失活,重命名为pMD18T-ΔgldA。提取质粒pMD18T-ΔgldA为模板,以引物gldA-s1和gldA-s1扩增得到含有长gld同源臂的抗性盒。将该片段电转化入带有pDK6-red质粒的克雷伯肺炎杆菌感受态细胞,利用链霉素抗性平板筛选。待长出菌落利用菌落PCR验证,即为gldA基因缺失克雷伯氏肺炎杆菌突变株。基因acyl、budB、ilvB、ilvI的突变株构建方法同上。
1.2.2 重组菌株抗性的消除 在突变株构建成功后需要将其抗性进行消除,以方便构建多基因敲除的突变株。首先需要消除pDK6-red质粒,在无抗LB试管培养基中,加入10 μL/1 mL LB的10%(w/v)SDS,之后接种含pDK6-red质粒的突变菌株,每隔12 h传代1次,3~4次传代后,将菌液稀释107涂布于无抗LB平板,利用无抗LB平板和卡那霉素抗性平板进行印章法筛选,当无抗板上可以生长而卡那霉素抗性平板上不能生长的,即为pDK6-red质粒已消除的重组菌株。接着将pDK6-flp质粒电转入已消除pDK6-red质粒的重组菌中,每隔12 h传代的同时加入终浓度为1 mmol/L的IPTG进行诱导,将多次传代后的菌液稀释107后涂布于无抗LB平板培养,利用无抗LB平板和抗性平板进行印章法筛选,当无抗板上可以生长而抗性平板上不能生长的,即为抗性基因已消除的重组菌株。最后同样方法消除pDK6-flp质粒,菌株可以进行下一轮基因重组。
1.2.3 发酵条件 种子菌株培养于装有50 mL LB培养基的250 mL三角摇瓶内,于摇床培养箱中37℃,200 r/min培养9~10 h。将50 mL已培养好的种子液接种于5 L发酵罐中(BIOSTAT-A plus Sartorius),发酵罐装液量为3 L。发酵温度37℃,采用流加NaOH的方式自动控制pH稳定在6.0,通风量2 L/min,转速300 r/min。发酵结果均为3次平行实验数据的平均值。
1.2.4 分析方法 使用紫外分光光度计(DU730 Beckman)检测OD600 nm下菌体浓度数值,发酵液中葡萄糖、琥珀酸、乳酸、乙酸、乙偶姻、meso-2,3-丁二醇、2R,3R-丁二醇、乙醇等产物利用高效液相色谱检测。采用岛津20AVP色谱仪,装备RID-10A示差检测器,采用有机酸分析色谱柱Aminex HPX-87H(Bio-Rad)色谱柱,柱温65℃,流动相为0.005 mol/L的H2SO4,流速为0.8 mL/min。取1 mL发酵液于1.5 mL离心管中,以12000 r/min在离心机中离心10 min,取上清液用超纯水稀释20倍,经水相膜过滤后进样。
2 结果与分析
2.1 α-乙酰乳酸合成酶功能基因研究
budB、ilvB以及ilvI这三个基因都编码α-乙酰乳酸合成酶,分别构建三个基因单独突变株,筛选到的突变株菌落PCR验证结果如图1,2所示。

图1 budB和ilvB突变株单菌落PCR结果Fig.1 PCR result of bud and ilvB mutant single colony
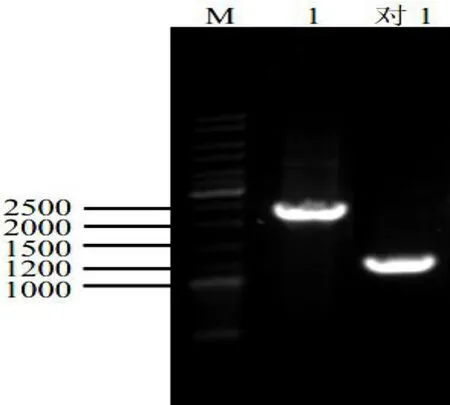
图2 ilvI突变株单菌落PCR结果Fig.2 PCR result of ilvI mutant single colony
将构建成功的kp-ΔbudB,kp-ΔilvB,kp-ΔilvI三株突变株以野生型克雷伯氏肺炎杆菌K.pneumoniaeCGMCC 1.6366(kp)为对照进行批次发酵实验,并检测发酵过程中主要代谢产物和菌体浓度,发酵结果如图3所示。

图3 kp和 kp-ΔbudB,kp-ΔilvB,kp-ΔilvI发酵生产 2,3-丁二醇过程Fig.3 2,3-butanediol production by kp,kp-ΔbudB,kp-ΔilvB and kp-ΔilvI
图3 A显示,kp-ΔilvB和kp-ΔilvI两株突变株与对照菌株的菌体生长情况形似,在前10 h内菌体生长迅速,而kp-ΔbudB菌体浓度不高,只有上述菌株菌体浓度的一半左右。在15 h左右菌体浓度达到最高值,图3 B中,kp-ΔilvB与kp-ΔilvI两株突变株和对照菌株kp葡萄糖消耗速率相似,都在19 h左右耗尽底物,而kp-ΔbudB突变株葡萄糖消耗速率十分缓慢,在19 h时还有37.8 g/L葡萄糖。在图3 C,D,E中,kp-ΔilvB与kp-ΔilvI两株突变株与对照菌株在meso-2,3-丁二醇,2R,3R-丁二醇和乙偶姻的产量以及生产速率上都相似,kp-ΔilvB和kp-ΔilvI与对照菌株的meso-2,3-丁二醇产量分别为27.5 g/L,26.9 g/L,27.1 g/L;2R,3R-丁二醇产量分别为2.7 g/L,2 g/L,2.6 g/L;乙偶姻产量分别为0.38 g/L,0.36 g/L,0.36 g/L,但在kp-ΔbudB突变株中,这三种产物都没有积累。在图3 F,H中kp-ΔilvB和kp-ΔilvI两株突变株表现出了十分相似的产物生产速率与产量,琥珀酸,乙醇的产量分别为2.5 g/L,2.7 g/L;5.5 g/L,5.7 g/L,略高于对照菌株kp的2.3 g/L,3.9 g/L。kp-ΔbudB突变株中这两种产物积累较少,分别为1.2 g/L和1.3 g/L。而在图3 G中,kp-ΔbudB突变株乳酸的产量相较其它菌株十分高,达到了26.3 g/L,kp-ΔilvB与kp-ΔilvI两株突变株和对照菌株kp的乳酸的生产速率相似,产量较低。
从发酵结果可以看出kp-ΔbudB突变株2,3-丁二醇合成完全消失,而kp-ΔilvB与kp-ΔilvI两个突变株对于2,3-丁二醇积累合成并无明显影响。表明budB编码的乙酰乳酸合成酶是菌体中唯一有功能催化乙酰乳酸合成的酶。
2.2 丁二醇脱氢酶功能基因研究
已报道的能够催化乙偶姻转化成2,3-丁二醇的酶包括:丁二醇脱氢酶(budC编码)、甘油脱氢酶(dhaD编码)、甘油脱氢酶(gldA编码),和短链酰基脱氢酶(acyI编码),其中budC编码的丁二醇脱氢酶对2,3-丁二醇贡献最大,但是其他酶对菌株合成2,3-丁二醇的贡献并不清楚。在kp-ΔbudC的基础上,本文构建了kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl四基因缺失突变株,筛选到的突变株菌落PCR验证结果如图4所示。
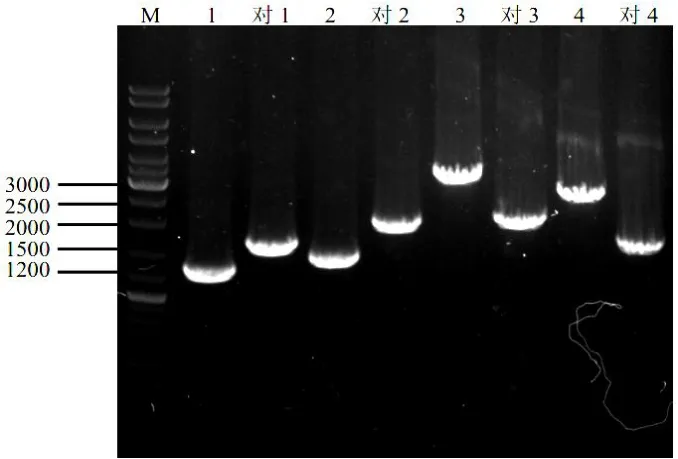
图4 kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl单菌落 PCR 结果Fig.4 PCR results of single colony kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl
将构建成功的kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl四敲菌株以kp-ΔbudC为对照进行批次发酵实验,并检测发酵过程中主要代谢产物和菌体浓度,发酵结果如图5所示。
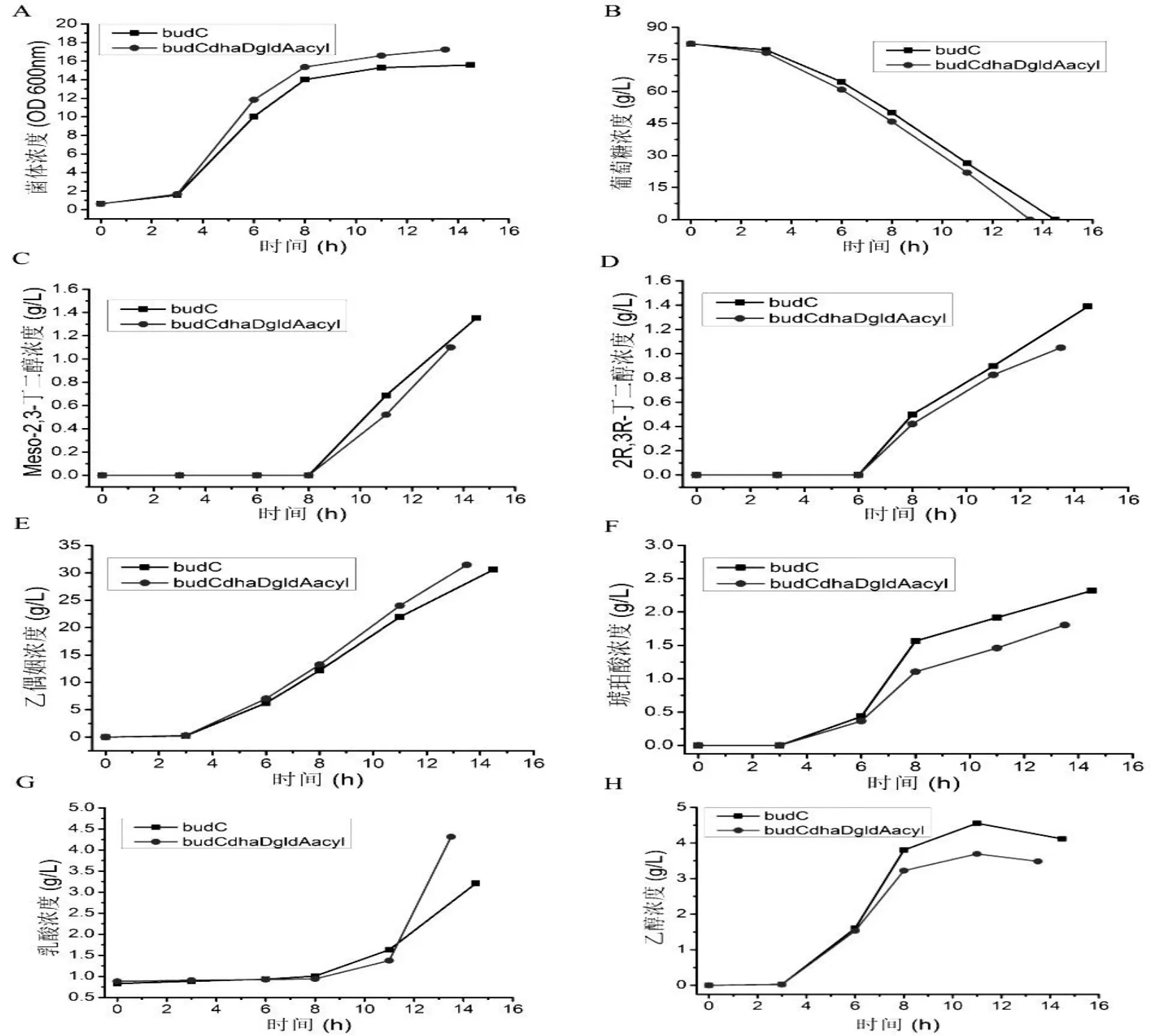
图5 kp-ΔbudC 和 kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl发酵生产乙偶姻过程Fig.5 Acetoin fermentation process by kp-ΔbudC and kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl
根据图5 A结果显示,kp-ΔbudC与kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl两株突变株菌体生长情况类似,在 4~8 h内两突变株菌体生长迅速,但kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl生长速率快于kp-ΔbudC,并且在发酵结束时kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl的菌体浓度高于kp-ΔbudC。从图5 B 可以看出,kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl的 发 酵 周 期 快 于kp-ΔbudC, 在 耗 尽 葡 萄 糖 底 物 时 ,kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl用了 13.5 h,kp-ΔbudC则用了 14.5 h。从图5 C,D 中可以看出 meso-2,3-丁二醇和2R,3R-丁二醇分别在8 h和6 h后开始合成积累,发酵终止时,对照kp-ΔbudC的meso-2,3-丁二醇与2R,3R-丁二醇产量分别为1.35 g/L和1.39 g/L,略高于kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl的1.1 g/L和1.05 g/L。图5 E中可以看出对照kp-ΔbudC和kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl的乙偶姻生产速率和产量相似,发酵结束时kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl乙偶姻产量为31.5 g/L,对照kp-ΔbudC乙偶姻产量为30.5 g/L。在图5 F,H中两株菌的琥珀酸、乙醇生产趋势相同,而对照kp-ΔbudC的琥珀酸,乙醇产量为2.3 g/L,4.1 g/L都高于kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl的1.8 g/L,3.5 g/L。而在图3 G中,kp-ΔbudC-ΔdhaD-ΔgldA-Δacyl突变株在发酵终点时乳酸的产量高于对照kp-ΔbudC。
从结果可以看出在kp-ΔbudC基础上敲除了甘油脱氢酶基因dhaD、gldA以及短链酰基脱氢酶acyl后,虽然两种构型的2,3-丁二醇产量相较于kp-ΔbudC对照略微有所下降,但还是会有2,3-丁二醇的生成。表明dhaD、gldA和acyl编码的酶蛋白对于菌株合成2,3-丁二醇的贡献微弱。
3 讨论
2,3-丁二醇合成途径是K.pneumoniae中非常重要的一条代谢途径,菌株利用糖类为碳源时,2,3-丁二醇是菌株主要的代谢产物。K.pneumoniae中2,3-丁二醇合成途径最早由Stormer于1975年提出[16],代谢途径中酶蛋白的编码基因由Blomqvist等于1993年在土生克雷伯氏菌(Klebsiella terrigena)中进行了鉴定[9]。乙酰乳酸不单单是乙偶姻的前体物,还是合成缬氨酸的前体物。缬氨酸合成途径中的乙酰乳酸合成酶是否对丁二醇合成有贡献,并无研究报道。本文研究结果表明,在2,3-丁二醇发酵条件下,缬氨酸合成途径的乙酰乳酸合成酶对2,3-丁二醇合成无贡献。ilvB和ilvI是缬氨酸合成途径上乙酰乳酸合成酶基因,而本文中用于2,3-丁二醇生产的培养基是一种完全培养基,在此条件下,细胞表达缬氨酸合成的相关基因可能并不表达,或表达微弱。
在2,3-丁二醇合成途径中budC编码的丁二醇脱氢酶催化乙偶姻与2,3-丁二醇之间的反应。Ui等人对于乙偶姻转化成2,3-丁二醇的过程有很多研究,他们提出细胞中有三种丁二醇脱氢酶,催化不同2,3-丁二醇的合成[17]。我们对这一反应进行研究后,得到dhaD编码的甘油脱氢酶催化R-乙偶姻合成2R,3R丁二醇的结论[10]。同时又有人提出了gldA编码的甘油脱氢酶和arcyI编码的的短链酰基脱氢酶也催化乙偶姻到2,3-丁二醇的反应[11,12]。本文研究表明,在利用葡萄糖为碳源情况下dhaD、gldA和acyl对于菌株合成2,3-丁二醇的贡献微弱。同时菌株中还存在其他具有丁二醇脱氢酶活性的酶蛋白。根据目前的研究报道,细胞合成的2,3-丁二醇仅通过乙偶姻还原生成。四突变株中依然存在能催化乙偶姻转化成2,3-丁二醇的酶类,这是由于细胞中存在多种醇脱氢酶,而醇脱氢酶的底物转移性并不很严格,包括dhaD和gldA编码的甘油脱氢酶和arcyI编码的的短链酰基脱氢酶等多种醇类脱氢酶都具有2,3-丁二醇脱氢酶活性。虽然在细胞合成2,3-丁二醇过程中,这些醇脱氢酶的活性比budC编码的2,3-丁二醇脱氢酶活性低,但是这些脱氢酶的存在使得budC突变株依然能够合成少量2,3-丁二醇。acyl的表达调控目前未见报道。dhaD和gldA都是甘油脱氢酶,这两个基因的表达受到甘油诱导,dhaD负责厌氧下甘油代谢,gldA负责好氧条件下甘油代谢[18]。在以甘油为碳源时,菌株2R.3R-丁二醇合成量提高,既是dhaD表达量升高的结果[10]。而在以葡萄糖为碳源时,dhaD和gldA表达水平受限,所以对2,3-丁二醇合成贡献很小。
4 结论
本研究利用Red重组系统在K.pneumoniae中对2,3-丁二醇合成途径中催化乙酰乳酸合成和2,3-丁二醇合成的相关基因进行了突变。通过突变株生理特性变化对以葡萄糖等为碳源时细胞中2,3-丁二醇合成途径的功能基因进行了研究。结果表明,在生产2,3-丁二醇时,budB编码的乙酰乳酸合成酶是细胞中唯一的功能性乙酰乳酸合成酶;催化乙偶姻转化成2,3-丁二醇反应的主要功能酶酶为budC编码的丁二醇脱氢酶,其他醇脱氢酶对于该反应的贡献微弱。本研究结果对于利用克雷伯氏肺炎杆菌生产2,3-丁二醇的代谢工程改造具有指导作用。
[1]Biebl H,Menzel K,Zeng AP,et al.Microbial production of 1,3-propanediol[J].Applied Microbiology and Biotechnology,1999,52(3):289-297
[2]Ma C,Wang A,Qin J,et al.Enhanced 2,3-butanediol production byKlebsiella pneumoniaeSDM[J].Applied Microbiology and Biotechnology,2009,82(1):49-57
[3]Wei D,Xu J,Sun J,et al.2-Ketogluconic acid production byKlebsiella pneumoniaeCGMCC 1.6366[J].Journal of Industrial Microbiology&Biotechnology,2013,40(6):561-570
[4]Wang D,Zhou J,Chen C,et al.R-acetoin accumulation and dissimilation inKlebsiella pneumoniae[J].Journal of Industrial Microbiology&Biotechnology,2015,42(8):1105-1115
[6]Celińska E,Grajek W.Biotechnological production of 2,3-butanediol-current state and prospects[J].Biotechnology Advances,2009,27(6):715-725
[7]Park JM,Song H,Lee HJ,et al.Genome-scale reconstruction and in silico analysis ofklebsiella oxytocafor 2,3-butanediol production[J].Microbial Cell Factories,2013,12(1):1-11
[8]Rao B,Zhang L-Y,Sun J,et al.Characterization and regulation of the 2,3-butanediol pathway inSerratia marcescens[J].Appl Microbiol Biotechnol,2012,93(5):2147-2159
[9]Gao J,Xu H,Li Q J,et al.Optimization of medium for one-step fermentation of inulin extract from jerusalem artichoke tubers usingpaenibacilluspolymyxazj-9toproducer,r-2,3-butanediol[J].BioresourceTechnology,2010,101(18):7076-7082
[10]Blomqvist K,Nikkola M,Lehtovaara P,et al.Characterization of the genes of the 2,3-butanediol operons fromKlebsiella terrigenaandEnterobacter aerogenes[J].Journal of Bacteriology,1993,175(5):1392-1404.
[11]Chen C,Wei D,Shi J,et al.Mechanism of 2,3-butanediol stereoisomer formation inKlebsiella pneumoniae[J].Applied Microbiology and Biotechnology,2014,98(10):4603-4613
[12]Wang Y,Tao F,Xu P.Glycerol dehydrogenase plays a dual role in glycerol metabolism and 2,3-butanediol formation inKlebsiella pneumoniae[J].Journal of Biological Chemistry,2014,289(9):6080-6090
[13]Park JM,Hong WK,Lee SM,et al.Identification and characterization of a short-chain acyl dehydrogenase fromKlebsiella pneumoniaeand its application for high-level production of L-2,3-butanediol[J].Journal of Industrial Microbiology&Biotechnology,2014,41(9):1425-1433
[14]Hao J,Lin R,Zheng Z,et al.Isolation and characterization of microorganisms able to produce 1,3-propanediol under aerobic conditions[J].World Journal of Microbiology and Biotechnology,2008,24(9):1731-1740
[15]Wei D,Wang M,Shi J,et al.Red recombinase assisted gene replacement inKlebsiella pneumoniae[J].Journal of Industrial Microbiology&Biotechnology,2012,39(8):1219-1226
[16]Gust B,Challis GL,Fowler K,et al.PCR-targeted Streptomyces gene replacement identifies a protein domain needed for biosynthesis of the sesquiterpene soil odor geosmin[J].Proceedings of the National Academy of Sciences,2003,100(4):1541-1546
[17]Stormer F.2,3-Butanediol biosynthetic system inAerobacter aerogenes[J].Methods in Enzymology,1975,41:518-533
[18]Ui S,Matsuda N,Masuda H,et al.Mechanism for the formation of 2,3-butanediol stereoisomers inKlebsiella pneumoniae[J].Journal Fermentation Technology,1986,64(6):481-486
[19]Wei D,Wang M,Jiang B,et al.Role of dihydroxyacetone kinases I and II in thedharegulon ofKlebsiella pneumoniae[J].Journal of Biotechnology,2014,177(2):13-19

