表油菜素内酯影响水稻幼苗响应低温胁迫的蛋白质组学分析
王道平 徐 江 牟永莹, 闫文秀, 赵梦洁 马 博 李 群 张丽娜, 潘映红,,*
表油菜素内酯影响水稻幼苗响应低温胁迫的蛋白质组学分析
王道平1,2徐 江2牟永莹2,3闫文秀2,3赵梦洁3马 博3李 群1,*张丽娜2,3潘映红2,3,*
1新疆大学生命科学与技术学院, 新疆乌鲁木齐 830046;2中国农业科学院作物科学研究所, 北京 100081;3农作物基因资源与基因改良国家重大科学工程, 北京 100081
表油菜素内酯(2,4-Epibrassinolide, EBR)是一种被广泛研究和应用的油菜素内酯类(brassinosteroids, BRs)植物生长调节剂, 它能有效增强植物对低温的耐受性, 但EBR在蛋白质组水平上对水稻幼苗响应低温胁迫的影响尚不清楚。本研究用0.1 mg L–1EBR和蒸馏水分别浸泡萌发的日本晴种子, 然后提取4°C胁迫培养和26°C正常培养幼苗的总蛋白, 进行质谱非标(label-free)定量分析和平行反应监测(parallel reaction monitoring, PRM)验证。最终共鉴定出5778个蛋白质, 其中, 在有定量信息的4834个蛋白中, 401个上调和220个下调蛋白与EBR影响水稻幼苗响应低温胁迫有关。功能分析和代谢通路富集分析发现, 上调蛋白主要与RNA结合或水解酶活性等分子功能相关, 并富集在碳代谢、叶酸合成和氨基酸生物合成等途径中; 下调蛋白主要与催化活性和氧化还原酶活性相关, 主要涉及卟啉和叶绿素代谢等代谢途径。PRM验证结果和文献证据显示, 分布在碳代谢和苯丙素代谢通路中的NADP-苹果酸酶、过氧化物酶、3-磷酸甘油酸脱氢酶、烯醇化酶、甘油醛-3-磷酸脱氢酶和丙酮酸激酶参与了EBR对低温胁迫水稻幼苗的调控, 提示BRs可通过多种途径影响水稻幼苗对低温胁迫的响应。
表油菜素内酯; 低温胁迫; 蛋白质组学; 水稻
作为植物内源信号的乙烯、生长素、细胞分裂素、脱落酸、赤霉素、油菜素内酯(brassinosteroids, BRs)等激素在植物生长发育和逆境响应中发挥着重要的调节作用[1-2]。其中BRs是一类在体内分布广泛但不长距离运输的甾体激素, 这类激素全面参与植物的细胞扩增、分化、生长发育、衰老以及光形态建成等生物过程[3-5], 对植物响应高温、低温、干旱和盐胁迫均有调控作用[5-6]。有研究用BRs类植物生长调节剂表油菜素内酯(2,4-Epibrassinolide, EBR)处理拟南芥和欧洲油菜的幼苗, 增强了植物对干旱和低温胁迫的耐受性[7]; BRs生物合成基因在种子中过表达也可提高拟南芥幼苗的耐冷性[8]。在苦瓜中发现BRs可以通过增加植物鲜重、叶绿素含量、可溶性蛋白质含量以及增强超氧化物歧化酶(SOD)活性等来降低低温胁迫对苦瓜生长的影响[9]。对玉米的研究发现, EBR处理幼苗可增加株高, 提高叶绿素和可溶性蛋白质含量, 并显著增强植株的低温耐受性[10]。番茄叶面喷施EBR后, 可缓解低温对光合作用和氮代谢的影响, 提高低温弱光胁迫下番茄的鲜重和干重[11]。此外, BRs还可以通过增强抗氧化系统和维持光系统II来缓解辣椒中由低温诱导产生的氧化胁迫[12]。这些研究均表明BRs可以增强植物的低温抗性, 但有关调控机制的研究尚不多见。从现有的BRs影响植物低温胁迫蛋白质组学研究结果来看, 绿豆中的上调蛋白质主要参与甲硫氨酸的同化、ATP合成、细胞壁构建和应激反应[13]; 辣椒的差异蛋白主要参与催化及活性调节、蛋白质结构性分子活性、能量转运过程和蛋白质结合等生物过程及光合固碳途径、氧化磷酸化途径、谷胱甘肽代谢途径、苯丙素生物合成途径、苯丙氨酸代谢途径和核糖体途径等[12]。上述结果提示, BRs可能通过调节多种蛋白质的丰度来影响植物对低温胁迫的响应。
水稻是一种被广泛研究的重要作物, 与水稻相关的蛋白质组研究主要集中在响应盐胁迫、干旱胁迫等方面, 有关低温胁迫的研究相对较少。对1周龄水稻幼苗进行低温处理后发现, 膜结构受损, 代谢变化剧烈, 水稻防御系统在低温胁迫下被激活[14]。2周龄幼苗低温胁迫6 h后, 一些蛋白质包括应激抗性蛋白质和负调控蛋白质等出现上调[15]; 低温胁迫12 h后, 根部组织的烯醇化酶、甘油醛-3-磷酸脱氢酶、核苷二磷酸激酶、抗坏血酸过氧化物酶等的蛋白丰度可增加2倍以上[16]; 低温胁迫24 h后叶片的差异蛋白主要包括蛋白质生物合成因子、分子伴侣、细胞壁组分生物合成酶、抗氧化/解毒酶以及与能量代谢通路相关的蛋白质, 这些蛋白主要定位于叶绿体中[17]; 幼苗经48 h低温胁迫, 能量代谢和应激相关蛋白质明显上调, 而防御相关蛋白质下调[18]。进一步对水稻幼苗12~14°C处理48 h、72 h和96 h的研究发现, 差异蛋白质主要是物质转运、光合作用、前体代谢产物和能量代谢相关类蛋白, 组蛋白和维生素B生物合成相关蛋白的丰度明显受到冷胁迫的影响[19]。这些蛋白质组学研究结果说明水稻幼苗受不同程度低温胁迫处理后, 光合作用、能量代谢和应激相关蛋白会出现明显的变化, 但是目前仍然不清楚BRs如何在蛋白质水平上影响水稻幼苗对低温胁迫的响应。
本研究采用表油菜素内酯(EBR)处理萌发的水稻种子, 利用蛋白质组学技术筛选和分析可能与EBR影响水稻幼苗响应低温胁迫有关的蛋白, 旨在为进一步研究BRs影响植物响应低温胁迫机制提供新的线索。
1 材料与方法
1.1 实验材料
日本晴种子在16 h光照(26°C)/8 h黑暗(22°C)条件下萌发培养7 d后, 用0.1 mg L–1EBR浸泡12 h, 对照为蒸馏水浸泡, 浸泡后的萌发种子分别在16 h光照/8 h黑暗的4°C和26°C培养箱中培养24 h后取芽, EBR处理4°C培养样品, 单独4°C培养样品, EBR处理26°C培养样品和单独26°C培养样品分别标记为4B、4、26B和26,-80°C保存备用。
1.2 叶绿素测定方法
取每个样品新鲜芽0.2 g,-80°C冷冻1 h, 加入10 mL80%丙酮溶液, 50°C水浴3 h至叶片发白。以80%丙酮为空白, 分别测定叶绿素提取液在663 nm和645 nm波长的光密度, 按下列公式计算叶绿素和含量(D645和D663表示在645 nm和663 nm波长下的光密度, V为定容体积10 mL, W为称样量0.2 g)。
叶绿素(mg g–1) = (12.72D663-2.69D645) V/(W1000)
叶绿素(mg g–1) = (22.88D645-4.68D663) V/(W1000)
1.3 蛋白提取及酶切
称取0.2 g样品用细胞破碎仪20 Hz s–1振荡破碎, 加入0.4 mL提取缓冲液(8mol L–1Urea, 2 mmol L–1EDTA, 20 mmol L–1CaCl2, 200 mmol L–1NaCl, 100 mmol L–1Tris-HCl, pH 8.1), 13 000´离心30 min; 转移上清液到10 K超滤管内超滤离心(13 000´, 30 min, 下同), 弃滤出液, 加入200 µL现配的积二硫苏糖醇(dithiothreitol, DTT)溶液(50 mmol L–1DTT, 8 mol L–1Urea, 100 mmol L–1Tris-HCl, pH 8.1), 30°C孵育60 min; 超滤离心除去滤出液, 分3次加入200 µL尿素缓冲液(8mol L–1Urea, 100mmol L–1Tris-HCl, pH 8.1)洗涤超滤, 加入100 µL现配碘乙酰胺(iodoacetamide, IAA)溶液(50 mmol L–1IAA, 8 mol L–1Urea, 100 mmol L–1Tris-HCl, pH 8.1)暗置30 min; 超滤离心除去滤出液, 用100 µL尿素缓冲液(8mol L–1Urea, H2O)洗涤超滤3次, 加入200 µL NH4HCO3溶液(50 mmol L–1)洗涤超滤2次; 加入50 µL蛋白酶解液(含0.5 µg Trypsin, 50 mmol L–1NH4HCO3), 同时更换超滤管外管, 37°C酶切10 h, 超滤离心得到质谱分析样品。
1.4 质谱分析
液相色谱分离在Easy nLC 1000纳升级液相色谱仪(Thermo Fisher公司产品)上完成。流动相A为0.1%甲酸水(Formic Acid, FA), 双蒸水; 流动相B: 0.1% FA, CAN。设置流动相的梯度为流速0.4 mL min–1, 3%~6% 3 min, 6%~22% 78 min, 22%~100% 1 min, 100% 8 min。质谱分析在依靠静电场轨道离子阱(Orbitrap)检测离子质量的Q Exactive Plus (QE Plus, Thermo Fisher公司产品)质谱仪上完成, 采集模式为数据依赖型(data dependent acquisition, DDA)。设一级质谱的分辨率为70 000, 扫描范围为300~1800 m/z, 自动增益控制值为3e6, 注入时间为50 ms。二级质谱的分辨率为17 500, 自动增益控制值1e5, 注入时间45 ms, 碰撞能量27 NCE。二级采集的标准以前20个最强离子为母离子, 再进行Orbitrap检测。每个样品进行3次质谱技术重复。
1.5 蛋白质鉴定及数据库搜索
利用Proteome Discoverer 2.0定性分析软件(Thermo Fisher公司产品), 对质谱的原始数据RAW文件进行定性分析。检索引擎设定为SEQUEST HT, 检索数据库为植物基因组中水稻数据库(https:// phytozome.jgi.doe.gov/, 版本为Osativa_323_v7.0. protein)。设定如下定性搜库参数, 即肽段置信度为高, 最多蛋白漏切位点数2个, 肽段的长度范围6~144个氨基酸, 母离子质量偏差为±10 μmol L–1, 碎片离子质量偏差0.02 Da, 固定修饰选择半胱氨酸(Cys)碘乙酰胺化(carbamidomethy/+57.021 Da), 可变修饰选择甲硫氨酸(Met)氧化(oxidation/+15.995 Da)和N-乙酰化(Acetyl/+42.011 Da), 肽段搜库假阳性率FDR为1%。将每个样品3次重复的数据一起检索, 得到每个样品的定性结果, 分别以Excel表格的形式输出。
采用Maxquant软件(http://www.coxdocs.org/, 版本为1.3.0.5) 定量分析, 检索数据库同上。用全部样品(各自重复3次)的质谱数据一起进行非标定量(label-free)分析, 设定如下参数, 即胰蛋白酶为默认的蛋白酶, 2个漏切位点, 固定修饰选择半胱氨酸碘乙酰胺化, 可变修饰选择甲硫氨酸氧化和N-乙酰化, 先驱离子质量偏差为±20 μmol L–1, 碎片离子质量偏差0.02 Da, 能够检测到的肽段长度的最小值是7个氨基酸, 肽段搜库假阳性率FDR为1%。4个样品的相对定量结果以Excel表格的形式输出, 供后续比较分析。
1.6 数据相关性分析
使用SPSS 22.0数据处理软件(https://www.ibm. com/analytics)对各样品定量数据进行皮尔逊(Pearson)相关性分析。通过绝对值的大小表示样品间线性相关强弱的程度, 绝对值越大表示相关性越高, 如果样品间相关系数的绝对值为0则表示无关, 在0.9以上为高度相关, 1表示变量完全正相关。
1.7 生物信息学分析
用于生物信息分析的定量蛋白质假阳性率q-value值<0.01, 4个样品的总信号强度Intensity>0, 蛋白质信号强度的相对比值大于或等于2倍即视为差异蛋白质。从植物基因组水稻数据库中分别批量提取上调和下调差异蛋白质的FASTA格式序列导入Omicsbean软件(http://www.omicsbean.cn/)进行本体(Gene Ontology, GO)和代谢通路(Kyoto Encyclopedia of Genes and Genomes, KEGG)富集分析。
1.8 PRM靶向定量
4个样品混合样的上样量为1mL, DDA分析和定性搜库参数设置同上。将混合样中待验证蛋白的定性结果导入Skyline软件(https://skyline.ms/), 选择待验证目标蛋白肽段并编辑PRM检测方法, 使用QE-Plus分别采集4个样品中待验证蛋白的PRM定量数据, 离子隔离窗口(isolation window)为1.6 m z–1, 一级全扫描1次结合30次PRM扫描, 一级全扫描的分辨率为70 000, PRM扫描分辨率设为17 500, 用Skyline完成待验证蛋白的定量分析。
2 结果与分析
2.1 水稻低温胁迫表型观察及叶绿素含量测定
EBR处理26°C常温培养的样品与单独26°C培养样品在表型上无差异, 低温培养的两组样品中, 单独4°C培养样品明显偏黄, 芽也相对细弱(图1-A)。常温培养样品的叶绿素和叶绿素的含量均没有显著差异, 但明显高于低温培养的样品; EBR处理4°C培养样品与单独4°C培养样品相比, 叶绿素含量差异显著, 叶绿素含量差异不显著(图1-B); 全株称重3次, 每次10粒发芽种子, 结果显示4个样品间的全株鲜重无显著差异(图1-C)。
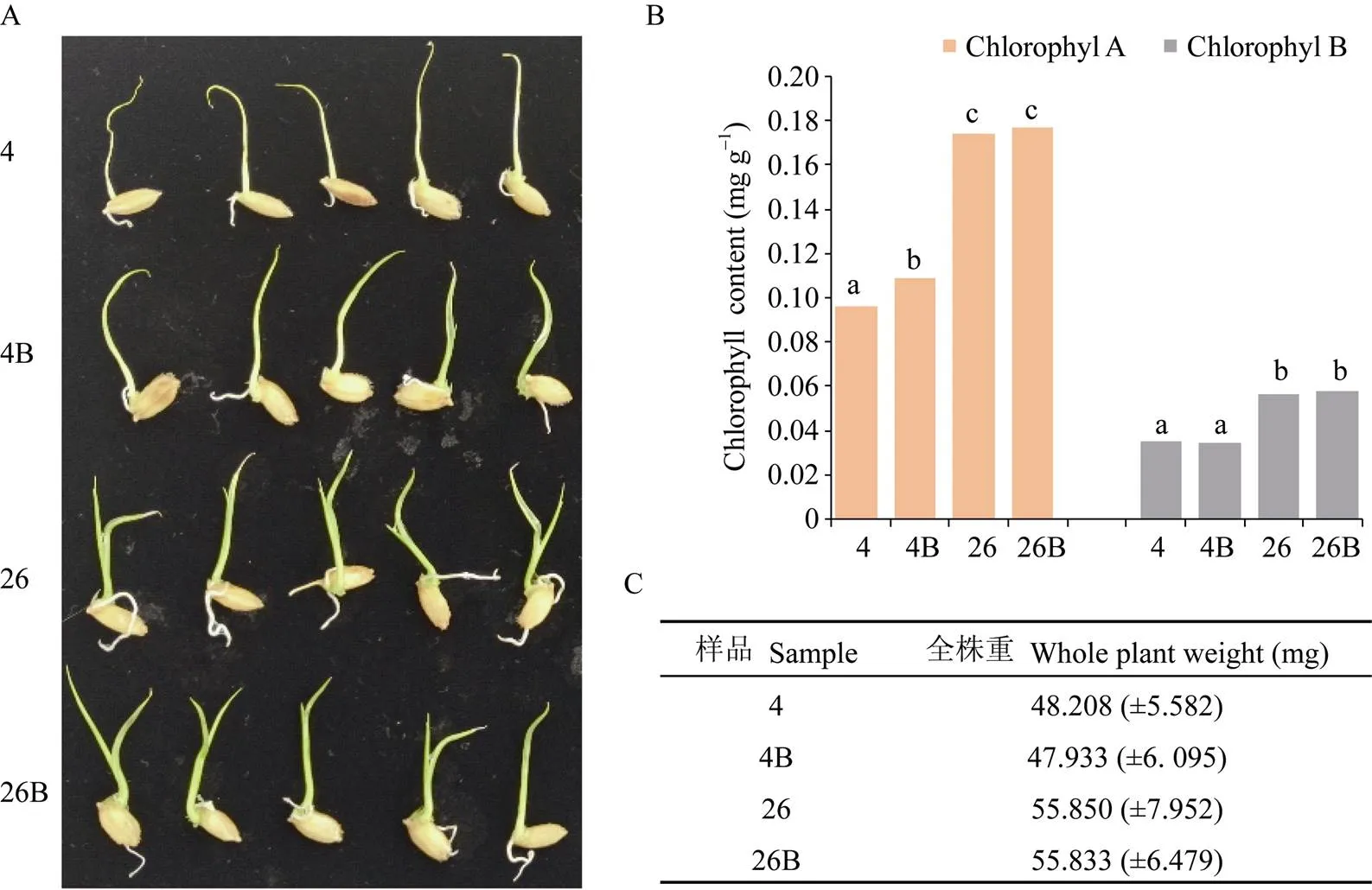
图1 水稻低温胁迫表型及叶绿素含量
A: 4组样品表型图; B: 4组样品叶绿素和叶绿素含量比较; C: 4组样品全株重比较。随机选取10株幼苗称重, 重复3次, 算其平均值作为该组样品的全株鲜重。4B: EBR处理4°C培养样品, 4: 单独4°C培养样品, 26B: EBR处理26°C培养样品, 26: 单独26°C培养样品。B和C中标以不同小字母的同组数值在0.05水平差异显著。
A: four groups of sample phenotype; B: comparison of chlorophylland chlorophyllcontents in four groups of samples; C: comparison of whole plant weight in four groups of samples, randomly selected 10 seedlings and weighed which was repeated three times and calculate the average value as the whole plant fresh weight of a group. 4B: samples treated with EBR then cultured at 4°C, 4: samples cultured at 4°C, 26B: samples treated with EBR then cultured at 26°C, 26: samples cultured at 26°C. Values within a group followed by a different small letter are significantly different at the 0.05 probability level in B and C.
2.2 质谱分析
从4个样品定性鉴定到5778个蛋白质, 平均鉴定到每个样品4331个蛋白质, 低温胁迫样品与正常培养样品(4 vs 26, 4B vs 26B)相比鉴定出的蛋白质数量较少, 但同温度培养的EBR处理样品蛋白鉴定数有所增加。2个样品间共有的蛋白质数在3508到3631之间, 其中正常培养的2个样品的共有蛋白质数量最多, 占2个样品总蛋白数的72.4%; 低温胁迫处理的2个样品间共有蛋白质数量最少, 仅占2个样品总蛋白数的69.3%; 4个样品共有蛋白数为3112, 占4个样品总蛋白数的53.9% (图2-A)。对质谱定量数据的Pearson相关分析表明, 样品4B与其他几组样品的相似度普遍较高, 样品4B和26的定量结果相似度最高, 样品4B和26B以及不加EBR处理的两组样品的相似度居中, 样品4和26B的相似度低于其他的组合(图2-B)。4组样品定量鉴定结果还显示, EBR处理后正常培养及低温胁迫的样品中检测到有定量信息的蛋白质均增加, 且蛋白质信号强度多为107~108(图2-C)。
A:定性鉴定蛋白数及样品间共有蛋白数; B: 样品间的皮尔逊相关性分析图; C: 定量鉴定蛋白质信号强度分布表。
A: qualitatively identified proteins and common proteins between samples; B: Pearson correlation analysis between samples; C: signal intensity distribution of quantitative identified proteins.
2.3 定量蛋白差异特性分析
利用DDA质谱数据, 采用非标定量方法共鉴定到4834个有可靠定量信息的蛋白质, 其中样品4和26 (4 vs 26), 样品4B和26B (4B vs 26B), 样品4B和4 (4B vs 4), 以及样品26B和26 (26B vs 26)两两相比丰度差异大于2倍的上、下调蛋白数见图3-A。进一步分析后发现, 在“4B vs 26B”和“4B vs 4”上调蛋白的并集(4BH∪B4H)中, 即EBR处理4°C培养样品分别与EBR处理26°C培养及无EBR处理4°C培养样品相比上调的1429个蛋白中, 与“26B vs 26”和“4 vs 26”下调蛋白有交集的有401个, 记为与EBR影响水稻幼苗响应低温胁迫有关的上调蛋白(图3-B); “4B vs 26B”和“4B vs 4”下调蛋白的并集(4BL∪B4L)有1293个, 其中与“26B vs 26”和“4 vs 26”上调蛋白共有的有220个, 记为与EBR影响水稻幼苗响应低温胁迫有关的下调蛋白(图3-C)。
2.4 GO分析
在与EBR影响水稻幼苗响应低温胁迫有关差异蛋白中, 上调蛋白质的分子功能主要涉及RNA结合、小分子结合、转移酶活性、氧化还原酶活性和辅酶结合等, 多数蛋白位于细胞质、叶绿体和有膜细胞器等, 参与了小分子代谢、有机氮化合物代谢、羧酸代谢、含氧酸代谢、有机酸代谢、碳水化合物生物合成、生物合成及基因表达等生物学过程(图4-A); 下调蛋白质其分子功能主要涉及辅因子结合、催化活性、辅酶结合、水解酶活性等, 多数位于细胞质、内质网和内膜系统, 生物过程富集分析显示这些蛋白主要参与了氧化还原、分解、含氧酸代谢、有机氮化合物代谢和小分子代谢等过程(图4-B)。
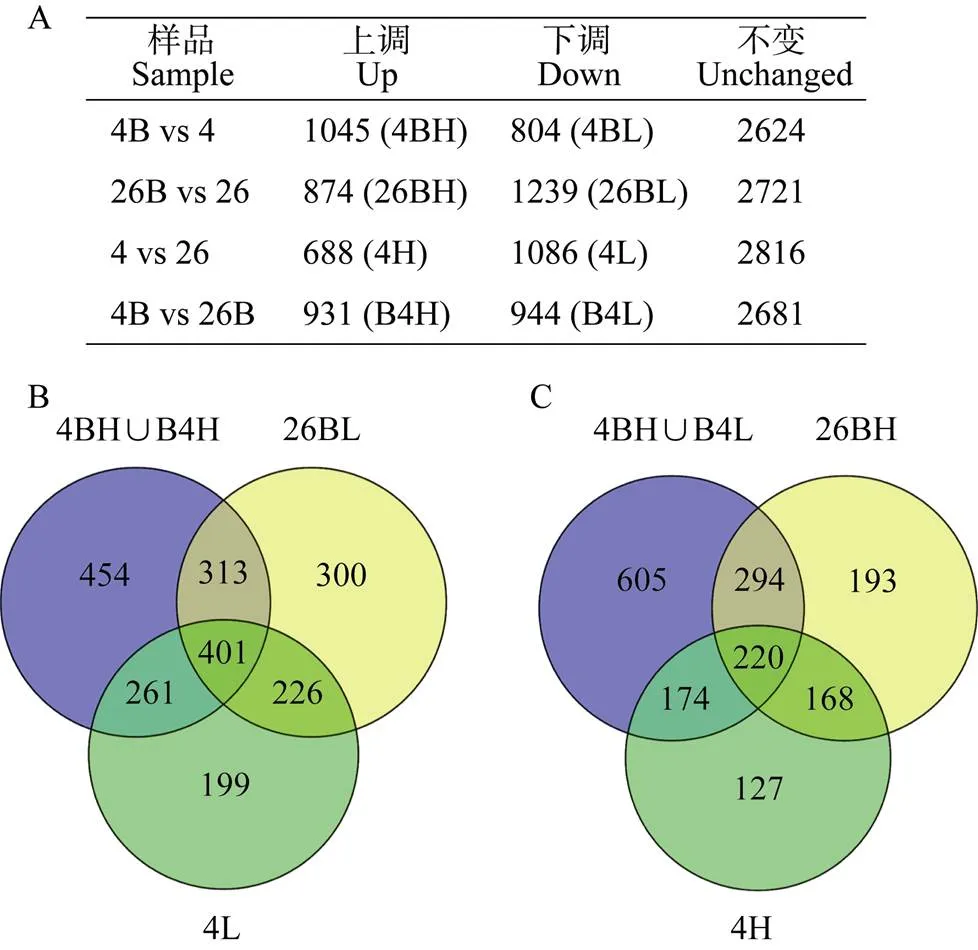
图3 定量蛋白的差异性比较
A: 样品间的差异蛋白数; B:与EBR影响水稻幼苗响应低温胁迫有关上调蛋白维恩图; C: 与EBR影响水稻幼苗响应低温胁迫有关下调蛋白维恩图。H表示上调, L表示下调。
A: proteins with intensity difference between every two samples; B: Venn diagram shows the 401 up-regulated proteins related to the effect of EBR on rice seedings response to cold stress; C: Venn diagram shows the 220 down-regulated proteins related to the effect of EBR on rice seeding response to cold stress. H means up regulated and L means down regulated.
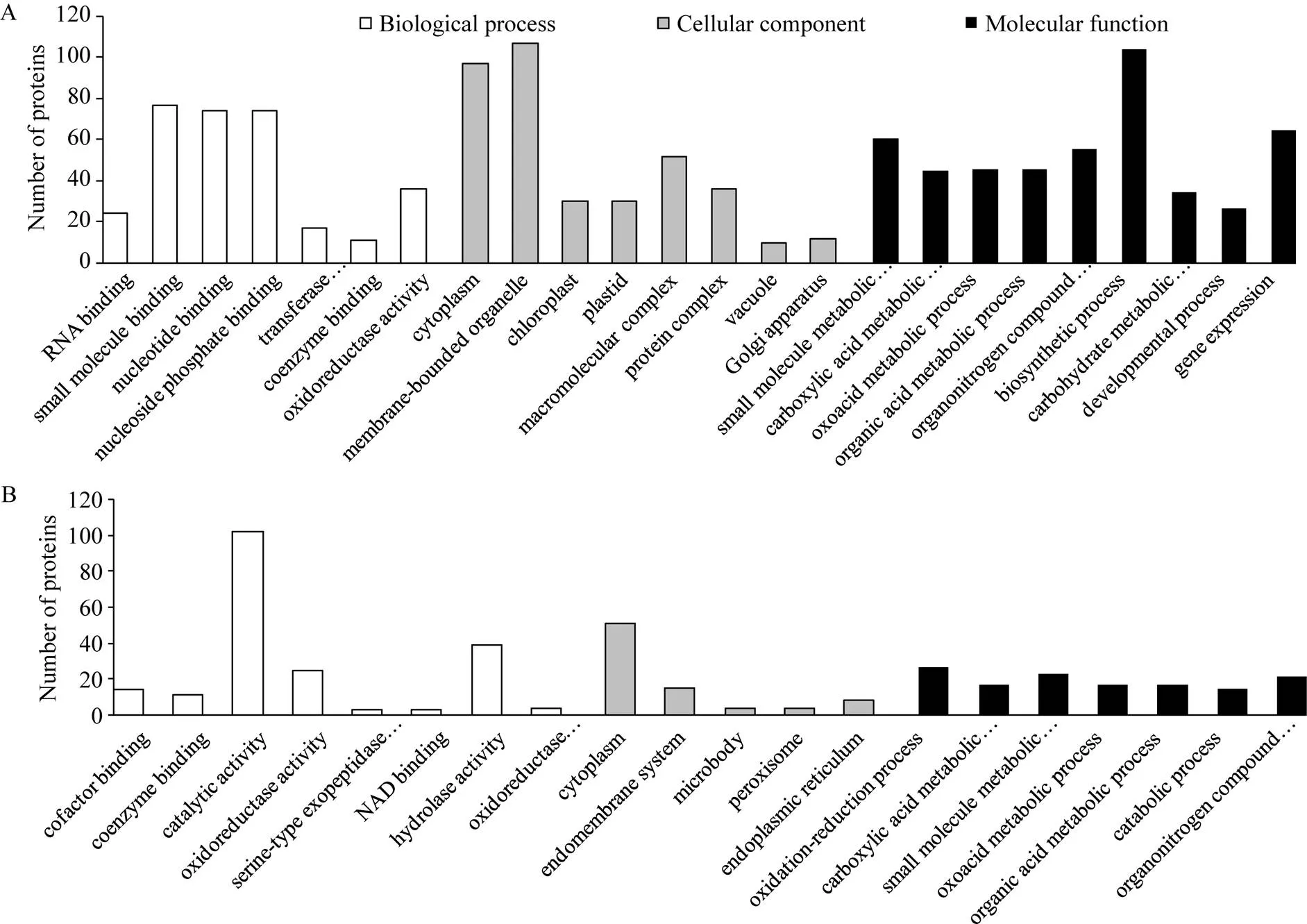
图4 影响水稻幼苗响应低温胁迫相关蛋白的本体分析
A: 401个上调蛋白质的本体分析; B: 220个下调蛋白质的本体分析。
A: gene ontology analysis of 401 up-regulated proteins; B: gene ontology analysis of 220 down-regulated proteins.
2.5 代谢途径分析
对差异蛋白进行代谢通路富集分析, 发现在<0.05的范围内, 上调蛋白与叶酸生物合成、嘧啶代谢、氨基酸的生物合成、剪接体、碳代谢等代谢途径相关(图5-A)。下调蛋白与卟啉和叶绿素代谢、核黄素代谢、柠檬烯和蒎烯降解、其他代谢途径等相关(图5-B)。
在KEGG富集分析中,值低于0.16且富集蛋白数多于4的重要代谢通路及其富集到的蛋白质种类见表1。
如表1所示, 富集在碳代谢途径中的NADP依赖性苹果酸酶、甘油醛-3-磷酸脱氢酶、磷酸果糖激酶和丙酮酸激酶(LOC_Os11g10980.1), 苯丙素的生物合成途径中的过氧化物酶(LOC_Os01g73200.1)等仅在样品4B中被检测到。碳代谢途径中的烯醇化酶、卟啉和叶绿素代谢途径中的联乙烯还原酶(divinyl reductase, DVR)在样品4B中的蛋白丰度是样品4和26B中的2倍以上。
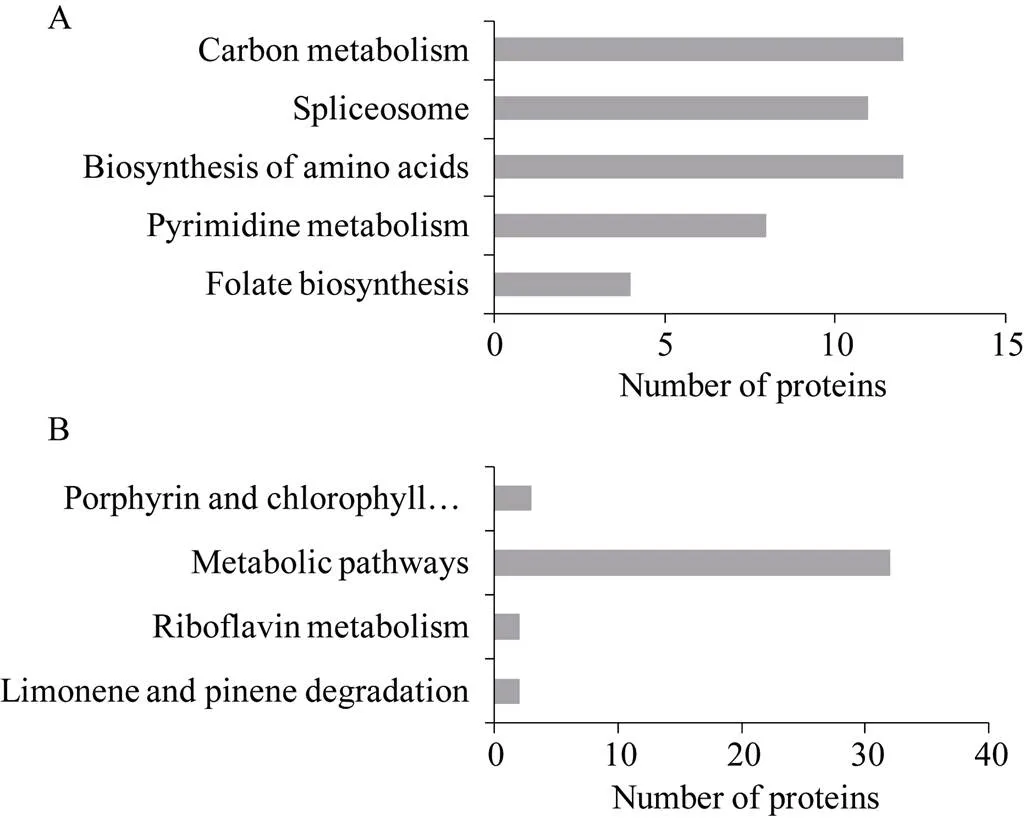
图5 代谢通路分析
A: 401个上调差异蛋白质代谢通路分析; B: 220个下调差异蛋白质的代谢通路分析。
A: KEGG pathways analysis of 401 up-regulated proteins; B: KEGG pathways analysis of 220 down-regulated proteins.
表1 与EBR影响水稻幼苗响应低温胁迫相关的重要通路及富集的蛋白质
Table 1 Important pathways and enriched proteins related to the effect of EBR on rice seedings response to cold stress
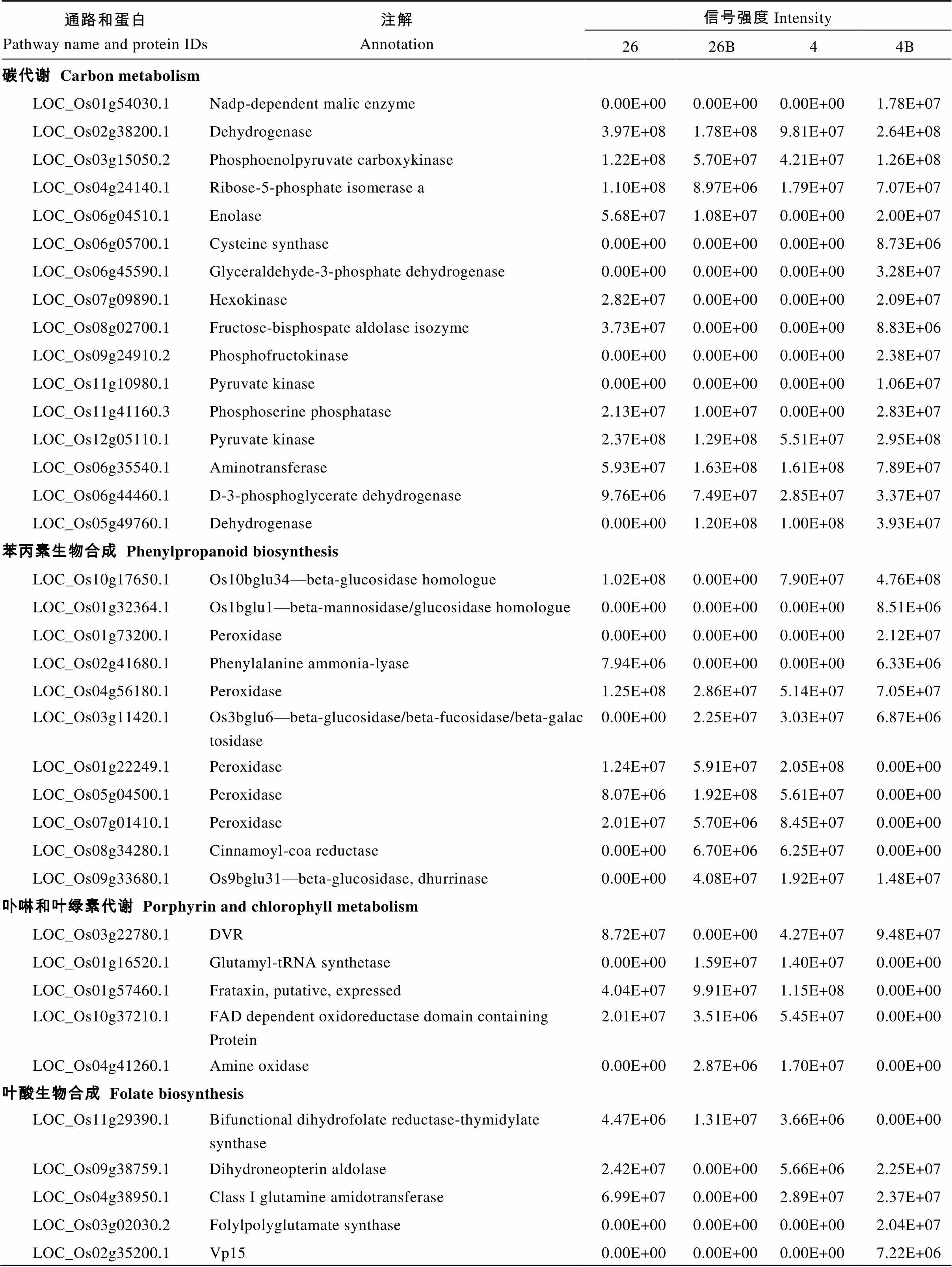
通路和蛋白Pathway name and protein IDs注解Annotation信号强度Intensity 2626B44B 碳代谢 Carbon metabolism LOC_Os01g54030.1Nadp-dependent malic enzyme0.00E+000.00E+000.00E+001.78E+07 LOC_Os02g38200.1Dehydrogenase3.97E+081.78E+089.81E+072.64E+08 LOC_Os03g15050.2Phosphoenolpyruvate carboxykinase1.22E+085.70E+074.21E+071.26E+08 LOC_Os04g24140.1Ribose-5-phosphate isomerase a1.10E+088.97E+061.79E+077.07E+07 LOC_Os06g04510.1Enolase5.68E+071.08E+070.00E+002.00E+07 LOC_Os06g05700.1Cysteine synthase0.00E+000.00E+000.00E+008.73E+06 LOC_Os06g45590.1Glyceraldehyde-3-phosphate dehydrogenase0.00E+000.00E+000.00E+003.28E+07 LOC_Os07g09890.1Hexokinase2.82E+070.00E+000.00E+002.09E+07 LOC_Os08g02700.1Fructose-bisphospate aldolase isozyme3.73E+070.00E+000.00E+008.83E+06 LOC_Os09g24910.2Phosphofructokinase0.00E+000.00E+000.00E+002.38E+07 LOC_Os11g10980.1Pyruvate kinase0.00E+000.00E+000.00E+001.06E+07 LOC_Os11g41160.3Phosphoserine phosphatase2.13E+071.00E+070.00E+002.83E+07 LOC_Os12g05110.1Pyruvate kinase2.37E+081.29E+085.51E+072.95E+08 LOC_Os06g35540.1Aminotransferase5.93E+071.63E+081.61E+087.89E+07 LOC_Os06g44460.1D-3-phosphoglycerate dehydrogenase9.76E+067.49E+072.85E+073.37E+07 LOC_Os05g49760.1Dehydrogenase0.00E+001.20E+081.00E+083.93E+07 苯丙素生物合成 Phenylpropanoid biosynthesis LOC_Os10g17650.1Os10bglu34—beta-glucosidase homologue1.02E+080.00E+007.90E+074.76E+08 LOC_Os01g32364.1Os1bglu1—beta-mannosidase/glucosidase homologue0.00E+000.00E+000.00E+008.51E+06 LOC_Os01g73200.1Peroxidase0.00E+000.00E+000.00E+002.12E+07 LOC_Os02g41680.1Phenylalanine ammonia-lyase7.94E+060.00E+000.00E+006.33E+06 LOC_Os04g56180.1Peroxidase1.25E+082.86E+075.14E+077.05E+07 LOC_Os03g11420.1Os3bglu6—beta-glucosidase/beta-fucosidase/beta-galactosidase0.00E+002.25E+073.03E+076.87E+06 LOC_Os01g22249.1Peroxidase1.24E+075.91E+072.05E+080.00E+00 LOC_Os05g04500.1Peroxidase8.07E+061.92E+085.61E+070.00E+00 LOC_Os07g01410.1Peroxidase2.01E+075.70E+068.45E+070.00E+00 LOC_Os08g34280.1Cinnamoyl-coa reductase0.00E+006.70E+066.25E+070.00E+00 LOC_Os09g33680.1Os9bglu31—beta-glucosidase, dhurrinase0.00E+004.08E+071.92E+071.48E+07 卟啉和叶绿素代谢 Porphyrin and chlorophyll metabolism LOC_Os03g22780.1DVR8.72E+070.00E+004.27E+079.48E+07 LOC_Os01g16520.1Glutamyl-tRNA synthetase0.00E+001.59E+071.40E+070.00E+00 LOC_Os01g57460.1Frataxin, putative, expressed4.04E+079.91E+071.15E+080.00E+00 LOC_Os10g37210.1FAD dependent oxidoreductase domain containing Protein2.01E+073.51E+065.45E+070.00E+00 LOC_Os04g41260.1Amine oxidase0.00E+002.87E+061.70E+070.00E+00 叶酸生物合成 Folate biosynthesis LOC_Os11g29390.1Bifunctional dihydrofolate reductase-thymidylate synthase4.47E+061.31E+073.66E+060.00E+00 LOC_Os09g38759.1Dihydroneopterin aldolase2.42E+070.00E+005.66E+062.25E+07 LOC_Os04g38950.1Class I glutamine amidotransferase6.99E+070.00E+002.89E+072.37E+07 LOC_Os03g02030.2Folylpolyglutamate synthase0.00E+000.00E+000.00E+002.04E+07 LOC_Os02g35200.1Vp150.00E+000.00E+000.00E+007.22E+06
(续表1)
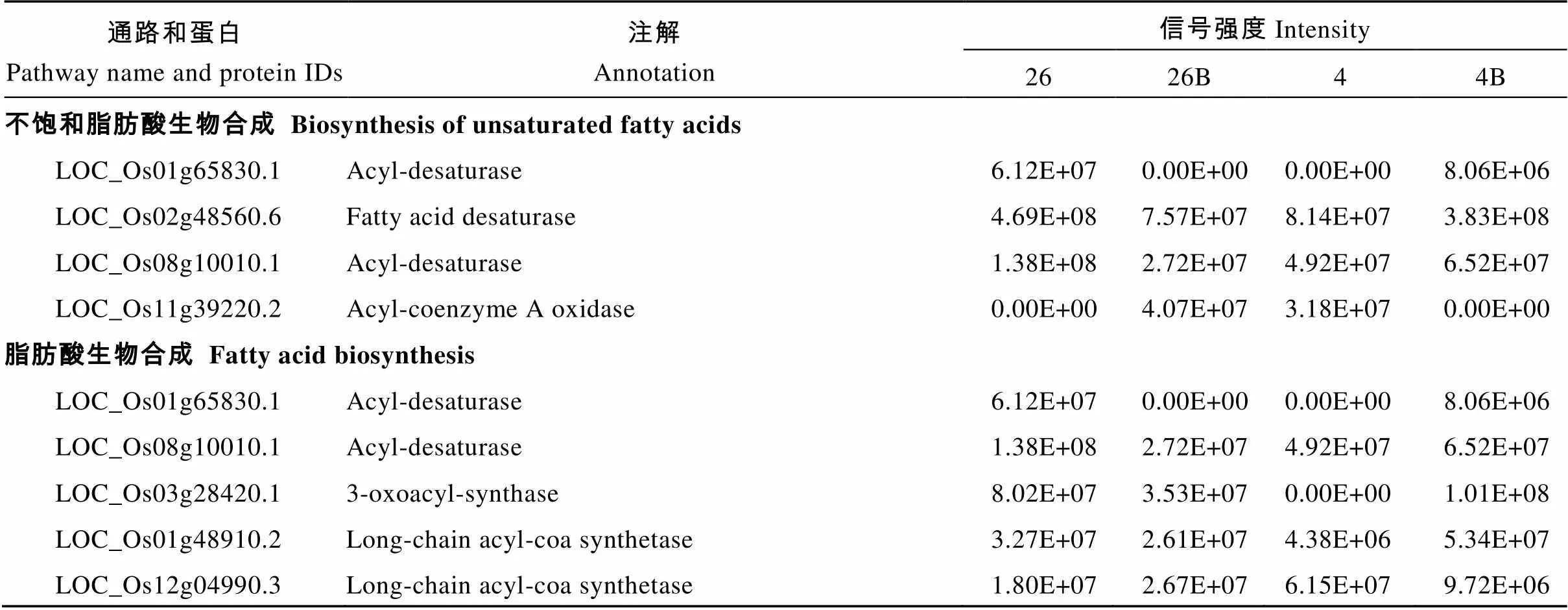
通路和蛋白Pathway name and protein IDs注解Annotation信号强度Intensity 2626B44B 不饱和脂肪酸生物合成 Biosynthesis of unsaturated fatty acids LOC_Os01g65830.1Acyl-desaturase6.12E+070.00E+000.00E+008.06E+06 LOC_Os02g48560.6Fatty acid desaturase4.69E+087.57E+078.14E+073.83E+08 LOC_Os08g10010.1Acyl-desaturase1.38E+082.72E+074.92E+076.52E+07 LOC_Os11g39220.2Acyl-coenzyme A oxidase0.00E+004.07E+073.18E+070.00E+00 脂肪酸生物合成 Fatty acid biosynthesis LOC_Os01g65830.1Acyl-desaturase6.12E+070.00E+000.00E+008.06E+06 LOC_Os08g10010.1Acyl-desaturase1.38E+082.72E+074.92E+076.52E+07 LOC_Os03g28420.13-oxoacyl-synthase8.02E+073.53E+070.00E+001.01E+08 LOC_Os01g48910.2Long-chain acyl-coa synthetase3.27E+072.61E+074.38E+065.34E+07 LOC_Os12g04990.3Long-chain acyl-coa synthetase1.80E+072.67E+076.15E+079.72E+06
2.6 PRM验证
PRM技术可对样品中的目标肽段进行准确的特异性分析, 因此可用于验证非标记定量分析得到的差异蛋白。在对所有差异蛋白的生物学功能分析的基础上, 选取NADP-苹果酸酶(LOC_Os01g540 30.1)、过氧化物酶(LOC_Os01g73200.1)、3-磷酸甘油酸脱氢酶(LOC_Os06g44460.1)、烯醇化酶(LOC_ Os06g04510.1)、甘油醛-3-磷酸脱氢酶(LOC_Os06g 45590.1)和丙酮酸激酶(LOC_Os11g10980.1)共6个蛋白进行PRM分析(图6)。表明EBR影响水稻幼苗响应低温胁迫相关的1个下调和5个上调蛋白在4个样品中的变化趋势与DDA数据(表1)基本一致。
3 讨论
以往的研究证实, 低温胁迫可影响光合作用、细胞膜、活性氧累积等相关生物学过程[20], 而激素在植物对低温胁迫的响应中发挥着重要作用[21]。本研究利用蛋白质组学技术分析了EBR影响水稻幼苗响应低温胁迫的相关蛋白和代谢通路, 为进一步的相关机制研究提供了新的线索。
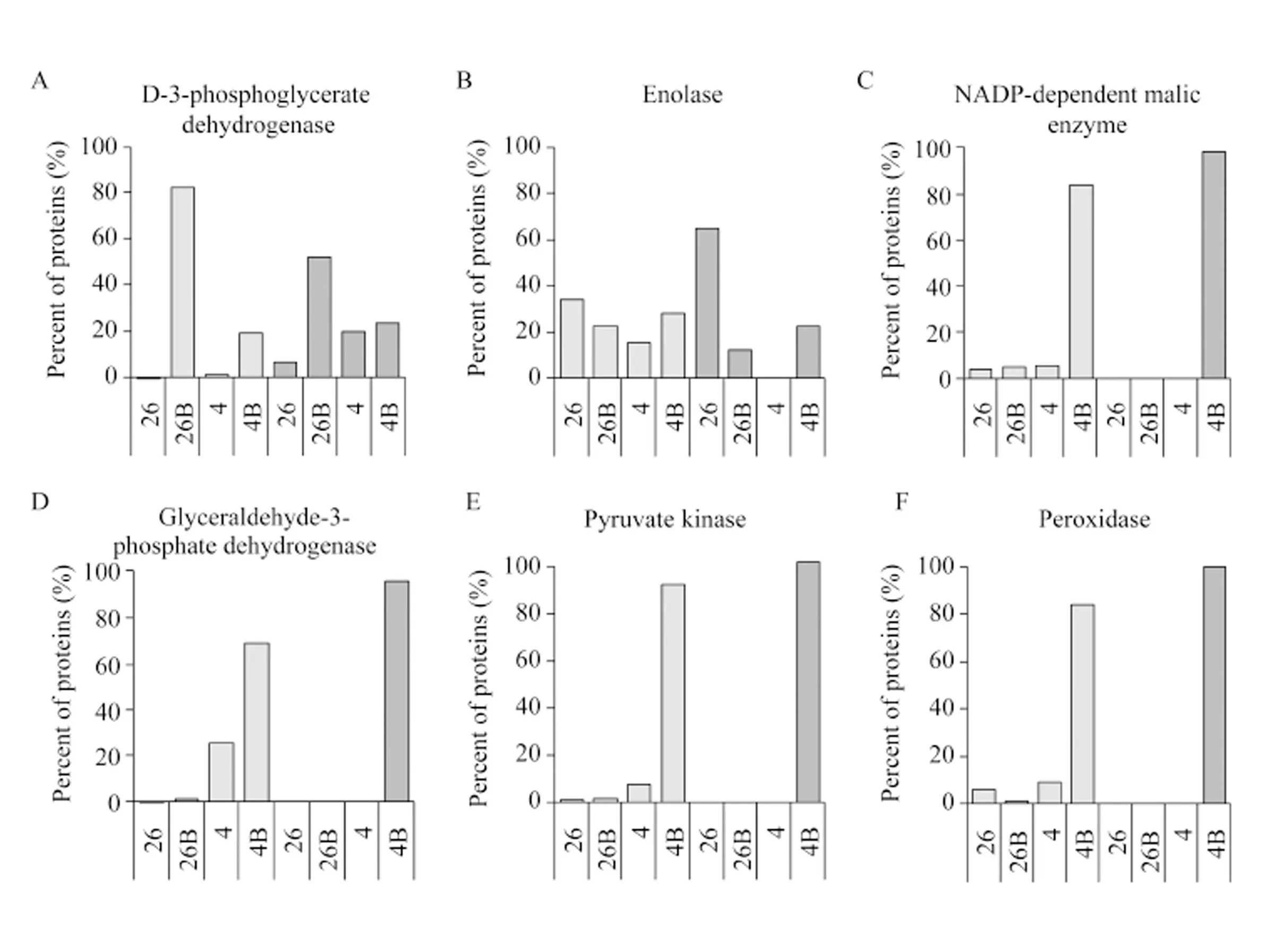
图6 部分差异丰度蛋白PRM 验证

