基于ICD-10诊断编码的慢性病并发症聚类算法
王晓霞,蒋伏松,王宇,熊贇
1. 复旦大学计算机科学技术学院,上海 201203;
2. 上海市数据科学重点实验室,上海 201203;
3. 西北师范大学计算机科学与工程学院,甘肃 兰州 730070;
4. 上海市第六人民医院,上海 200233;
5. 上海市普陀区疾病预防控制中心,上海 200333
1 引言
在长时间的发展过程中,很多慢性病都会引发并发症,比如慢性阻塞性肺病、抑郁症等,这些并发症是导致慢性病患者残疾、死亡的重要原因之一。了解慢性病与其并发症的关系以及研究相关并发症的发展,无论是对控制和预防慢性病并发症的发生、提高患者的自我监管能力,还是对慢性病的临床防治以及药物研发都有重要意义。
在医疗大数据环境下,对电子病历(electronic medical record)中的海量数据进行利用与分析能够促进医疗服务水平的提高以及对疾病的理解[1]。在电子病历中,对患者的诊断结果一般使用国际疾病分类(international classification of diseases,ICD)编码进行分类标注,近年来使用的是全世界通用的第10次修订本《疾病和有关健康问题的国际统计分类》,即ICD-10诊断编码。例如,对于糖尿病及其并发症[2],虽然在ICD-10诊断编码中根据疾病已知的病因、病理、临床表现和解剖位置等特性进行了一些详细的分类编码,但对于一些有可能由糖尿病引发的并发症并没有加以体现(比如抑郁症)。为了更好地利用电子病历中的数据分析各种目标疾病及其并发症的关系,将大量疾病诊断合理归类到若干种并发症类型是一项重要的任务。由于数据量巨大,对这些诊断结果按照研究目标的逻辑进行分类的工作,很难全部由具有医学知识的医生们来完成。本文设计了一种基于半监督聚类的慢性病并发症归类方法,聚类的依据是ICD-10诊断编码系统自身的编码特点:对于那些依据ICD-10诊断编码的分类规则划分为类似的疾病,其代码也会相邻。
2 相关工作
在机器学习领域,根据学习任务的不同分为两种学习方法:一种是无监督学习,在无人工标记的数据集中发现数据自身蕴含的结构信息;另一种是有监督学习,在有人工标记的训练数据集中寻找数据与给定标记之间的映射关系,对测试数据集中的未标记数据进行分类或预测[3]。在很多具体的应用中,获取大量有标记的数据是很困难、很昂贵且很费时的,也就是说有标记的数据数量是相对有限的,对于有监督学习,学习结果令人不满意的重要原因之一是有限的标记数据很难为机器学习算法提供足够的分布信息,而对于不使用标记数据的无监督学习方法,在处理海量数据时很难保证算法的精度[4]。在此背景下,半监督学习方法引起了人们的关注,它的学习目标是综合利用少量有标记数据和大量无标记数据来更好地完成学习任务。半监督聚类分析基本思想是通过引入标记数据(或约束关系)来指导聚类过程,帮助算法更好地划分样本数据,提高聚类算法的性能[5]。
电子病历存储了大量的健康信息,其绝大部分数据的获取、收集、存储与管理是面向医疗机构信息管理系统的,针对不同的研究任务,可能会遇到以下一些问题:标记与未标记数据同时存在、有标记数据常常是少量的、需要医学先验知识来指导数据的处理等。为了应对这些问题,越来越多的半监督学习方法被用来处理与分析电子病历。Huyen D T T等人[6]在对电子病历中的医疗行为与住院费用信息进行分析的过程中,认为在聚类过程中融合先验知识非常有必要,其通过设计的半监督模糊协同聚类方法,发现了一些能在降低治疗费用的同时提高服务质量的方案,能辅助医院更好地对医疗行为进行管理与决策。为了使更多的研究者能够共享临床数据,电子病历的去身份化是一项重要任务,参考文献[7]提出的混合半监督学习方法,能够准确地识别以及划分受保护的健康信息。Khan F M等人[8]构造了一种直推式的半监督神经网络回归学习框架,利用在预后生存分析中存在的大量无标记数据,显著提升了前列腺癌和乳腺癌的预后分析性能。在这些工作中可以看到,在对医疗数据的分析与研究工作中,采用能够融合医学先验知识的半监督算法能够获得更好的结果。
3 慢性病并发症半监督聚类算法
3.1 问题描述
设目标慢性病的N个疾病诊断编码集合为C,C={C1,C2,…,CN},Ci表示ICD-10诊断编码,已知目标疾病有K类并发症,学习任务是得到一个数据集C的K-划分使得C中所有的ICD-10诊断编码能够依据先验医学知识被尽可能地划分到正确的目标疾病并发症中。目标疾病并发症的先验医学知识是以种子数据的形式给出的,设种子数据集为,对于目标疾病的K类并发症,即C中的每一个划分Ck都至少包含一个种子数据Ci∈Sk,这样种子数据集S也是一个K-划分
例如,设某目标慢性病的疾病诊断编码集合:C={A05.9,B17.1,H25.1,H28.0,H43.3,I12.0,I74.3,N02.0,N17.0,Z49.2},其中,有10个不同的诊断编码,即N=10,已知该慢性病有3种并发症,分别是眼睛病变、肾病变和其他,为方便描述,分别用第1、2、3类表示,即K=3。医生给的种子数据集为S={A05.9,也就是已知S中所有疾病诊断编码的并发症类别,在此例中,A05.9属于第3类,H28.0属于第1类,N17.0属于第2类,可表示为S={A05.9:3,H28.0:1,N17.0:2},本文算法的任务是在所给种子数据的指导下,将集合中所有的诊断编码尽可能正确地归到相应的并发症分组中。
3.2 半监督聚类算法Constrained K-means
半监督聚类算法ConstrainedK-means[9]是一种基于约束的算法,在对目标慢性病并发症的聚类问题中,首先利用种子数据约束集(有并发症类别标签的ICD-10疾病诊断编码集合)初始化K个簇中心,K是给定的目标慢性病并发症种类数量,然后利用期望最大化(expectation maximization,EM)算法将在ICD-10诊断编码空间上距离相近的疾病聚在一起。由于直到聚类结束种子数据的标签都不允许被更改,因此种子数据自始至终都指导着整个聚类过程,使得与种子数据在ICD-10空间上相近的编码被聚到同一类,从而达到将数据集中的ICD-10编码按照医学先验知识聚为K类并发症的目的。为了使聚类效果更精确,半监督聚类算法ConstrainedK-means对种子数据集的要求是聚类集合中的每一类并发症都至少包含一个种子数据。
3.3 基于ICD-10诊断编码的疾病距离
世界卫生组织提供了4位ICD-10国际标准诊断编码,虽然我国在医疗系统中使用的是在此基础上扩展到6位的代码,但为了增加算法的适应性,也为了更好地利用ICD-10诊断编码自身的分类信息,本文提出的算法只使用ICD-10诊断编码的前4位进行聚类。ICD-10诊断编码的前4位是字母与数字的混合编码,第一位是英文字母,其后是阿拉伯数字,编码的前3位与后面的数字用点号隔开,是具有实际意义的类目码,第4位数字编码是前3位码的亚分类。例如:在编码“E11.3”中,前3位“E11”表示“2型糖尿病”,与第4位“3”合起来表示“2型糖尿病伴眼并发症”,而编码“E11.4”表示“2型糖尿病伴神经并发症”。根据ICD-10的编码规律,越相似的疾病使用越相近的编码空间,比如E10~E14代表的是各种类型的糖尿病,而编码首字母为“E”(即编码区间E00~E90)代表内分泌、营养和代谢疾病。
按照ICD-10诊断编码本身的编码规则,当两个诊断编码的首字母不同时,很难使用编码自身的信息度量其表示的疾病之间的距离,比如,从编码的角度看,E00.0(先天性碘缺乏综合征,神经病型)距离F00.0(阿尔茨海默(Alzheimer)病,早老型)比E00.0距离G00.0(嗜血杆菌脑膜炎)更近,然而从它们所代表疾病的角度来看,编码E00.0与F00.0之间的距离很难说就一定比 E00.0与G00.0之间的距离小。因此本文采用将疾病诊断编码按首字母不同进行分组的策略进行聚类,每组中编码的首字母是相同的,只需考虑首字母相同时疾病诊断编码之间的距离。疾病诊断编码空间中的第2位、第3位之间的远近关系在一定程度上反映了疾病之间的差异程度,首先将编码的第2位作为十位数、第3位作为个位数,合起来转换为一个两位的十进制整数,然后使用曼哈顿距离(Manhattan distance)来度量两个ICD-10诊断编码之间的距离。
设C1与C2是首字母相同的两个不同ICD-10疾病诊断编码,基于ICD-10诊断编码的疾病距离DICD(C1,C2)定义如下:

其中,int_C(C)函数的作用是将ICD-10诊断编码C中的第2位、第3位取出并转换为整数,该距离的取值范围是0~99。
3.4 慢性病并发症聚类算法
本文提出的采用分组策略的慢性病并发症聚类(complications semisupervised clustering by grouping,CSSC-G)算法是基于半监督聚类算法(ConstrainedK-means)算法框架的,完整的算法如算法1所示。CSSC-G算法的基本思想是,首先将所有ICD-10诊断编码以及种子数据按照将首字母是否相同进行分组,然后对每一组数据按照ConstrainedK-means算法进行聚类,以种子数据形式给出的先验知识指导每一组聚类过程的完成,且在整个聚类过程中不改变种子数据的类别,最后再将所有分组的聚类结果进行整合。设Ω为ICD-10诊断编码集C的首字母集,α∈Ω是其中的某个首字母,用表示首字母为α的ICD-10诊断编码分组,Kα≤K表示此分组中并发症的类别数量,其类别分别为l1,l2,…,lKα,Sα∈S表示在分组Cα中的种子数据,Sα中的种子数据是一个Kα-划分,为了使ConstrainedK-means算法能够有效进行,要求Cα中的每一个划分,都至少包含一个种子数据x∈Sα。组Cα的聚类任务是通过该组的种子数据,指导聚类过程得到一个该组的Kα-划分为相应划分的中心,使得Cα中所有的ICD-10诊断编码能够利用以种子数据形式给出的先验医学知识,尽可能正确地将目标疾病的相同并发症聚在一起。
算法1: 分组的半监督并发症聚类算法。
输入:ICD-10编码集以及科室就诊频率;种子数据集。
输出:ICD-10编码集的一个K-划分。
算法描述:
初始化Ω为数据集C的首字母集

4 实验结果
4.1 糖尿病并发症
对于糖尿病并发症的分类,从不同的角度以及不同的应用出发,有很多不同的划分。在参考文献[10]中依据身体系统的不同,将糖尿病慢性并发症基于ICD-9编码划分为心血管疾病、肾病、视网膜病、周围血管疾病、中风、神经病变、新陈代谢病七大类,用来计算糖尿病并发症严重指数(diabetes complications severity index,DCSI)。在对此工作的扩展研究中[11],将中风修改为脑血管疾病。在参考文献[2]中,首先将糖尿病并发症分为急性并发症、慢性并发症以及其他三大类,然后对每一大类再进行细分,具体的分类结果如图1所示。
在我国早期的糖尿病报告卡管理中,对于新增并发症记录的选项有酮酸症中毒、高渗性高血糖状态、糖尿病乳酸性酸中毒、糖尿病肾脏病变、视网膜病变和失明、糖尿病神经病变、糖尿病心脑血管病、下肢血管病变、糖尿病足,这也是对糖尿病并发症的一种划分。
国际糖尿病联合会将糖尿病并发症首先分为两大类:急性并发症与慢性并发症。其中急性并发症被细分为低血糖、酮酸症中毒、高渗性高血糖状态、高血糖糖尿病昏迷、癫痫或失去意识、感染;慢性并发症包括微血管并发症和大血管并发症。微血管并发症包括肾病、视网膜病变以及神经病变,大血管并发症包括冠心病(会导致心绞痛、心肌梗死)、外周动脉疾病(会导致中风、糖尿病脑病和糖尿病足)。另外,糖尿病还会使癌症、肺结核、生理残疾和认知障碍、抑郁症等疾病的发病率上升[12]。
根据以上医疗领域的知识,本文将糖尿病并发症的类别设为13种,分别是:急性并发症、感染性疾病、心血管类疾病、肾病、眼病、周围血管疾病、脑血管疾病、神经病变、代谢类疾病、癌症、肌肉骨骼类疾病、免疫系统疾病、精神疾病。考虑到算法的需要,对糖尿病自身的编码单独设置一类,另外再设置一类不属于以上13种并发症的其他并发症,则算法中的并发症总类别数为15,即K=15。
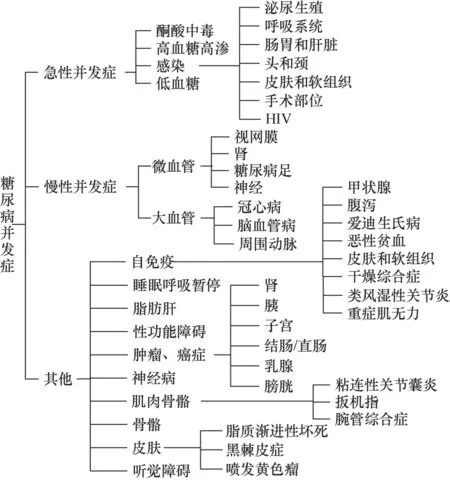
图1 医学领域中关于糖尿病并发症的一种分类方法
4.2 数据描述
本文的数据来源是整合了上海市7家医院以及10家社区医院临床诊疗数据的区域卫生平台,按照 ICD-10诊断编码从中筛选了至少有一次诊断在 E10~E14范围内的患者长达4年(2012—2015年)的电子病历,在28 910条有科室信息的记录中共整理出758种ICD-10诊断(只取前4位)。出现次数最频繁的前15个科室与ICD-10诊断编码的具体频数分别如图2(a)与图2(b)所示。
4.3 聚类结果评价
本文对CSSC-G算法与没有分组的聚类算法进行了比较,并分别展示了不同数量的种子数据对算法聚类效果的影响,从实验结果来看,采用分组策略的聚类算法明显优于没有进行分组的聚类方法。不分组的慢性病并发症聚类(complications semi-supervised clustering,CSSC)算法也是基于ConstrainedK-means算法框架的,但是没有根据首字母的不同对数据集进行分组。
CSSC算法将所有的疾病诊断编码放在一起进行聚类,但首字母的不同也意味着数据点之间的距离增大。为了体现首字母对数据点之间距离的影响,CSSC算法采用一个分段函数来度量包含首字母的不同ICD-10诊断编码间的距离。当疾病诊断编码的首字母相同时,使用同CSSC-G算法相同的距离计算方法,当编码首字母不同时,给定一个常数作为距离,因为首字母不同意味着诊断编码所代表的疾病之间的距离要比首字母相同的时候大,所以该常数的取值应该大于首字母相同情况下编码距离的最大值。具体如式(2)所示。

距离|int_C(C1)-int_C(C2)|的取值范围是0~99,因此当编码首字母不同时,CSSC算法取常数值100作为其距离。
两种算法采用的聚类策略不同,对种子数据的要求也不同。在CSSC-G算法中每一个分组是分别进行聚类的,因此要求每一个分组中的每一个簇至少包含一个种子数据,即任意一个ICD-10诊断编码分组中的每一个簇至少包含一个种子数据。而CSSC算法只需要所有数据中的每一个簇至少包含一个种子数据,即对于K类并发症,每一类至少包含一个种子数据。图3展示了CSSC算法与CSSC-G算法在以上两种情况下,不同种子数量占比对聚类效果的影响。
从图3(a)中可以看到,即使种子数据只满足CSSC算法的要求,CSSC-G算法对聚类效果也有明显的提升,且随着种子数据数量占比的增加,两种算法的聚类效果都有不同程度的提升。CSSC-G算法的聚类性能在有些情况下会有明显的下降,比如在种子数量占比为0.26时,造成这种情况的主要原因是给定的种子数据不满足CSSC-G算法的要求,尽管如此,CSSC-G算法的聚类效果总是优于CSSC算法的。
图3(b)说明,在种子数据满足CSSC-G算法要求的情况下,CSSC算法的性能会随着种子数据数量占比的增加而稳步提升,而CSSC-G算法在种子数据量较少的情况下(数量占比为0.25)就达到了较好的聚类效果(正确率在80%以上)。随着种子数据量的增加,聚类效果提升很快,当种子数据量占比是0.31时就达到了90%以上,之后再增加种子数据对CSSC-G算法的效果提升不是很大(尽管还是在提升)。造成这种结果的原因是在CSSC-G算法中,当种子数据量达到一定比例以后(在本实验中是0.31),每一组中种子数据的分布已经能够较好地表达并发症的分类特点,在增加的种子数据中有很多是不用再增加其他先验知识就可以得到正确分类的数据,而且新增加的种子数据对每一组中其他数据聚类结果的影响也不大,因此再增加种子数据对此算法的提升作用不是很大。而对于CSSC算法,由于没有采用分组策略,需要更多的种子数据来更好地表达并发症在整体数据集中的分类特点,因此随着种子数据的增加,CSSC算法的效果一直在逐步提升。但从总体来看CSSC-G算法的聚类效果在各种种子数据占比的情况下一直优于CSSC算法。
综上所述,无论是在所给种子数据满足CSSC-G算法要求的情况下,还是在只满足CSSC算法要求的情况下,本文提出的CSSC-G算法在聚类效果上都有很大的优势,并且CSSC-G算法能在比CSSC算法拥有更少种子数据的情况下,获得更好的聚类效果。
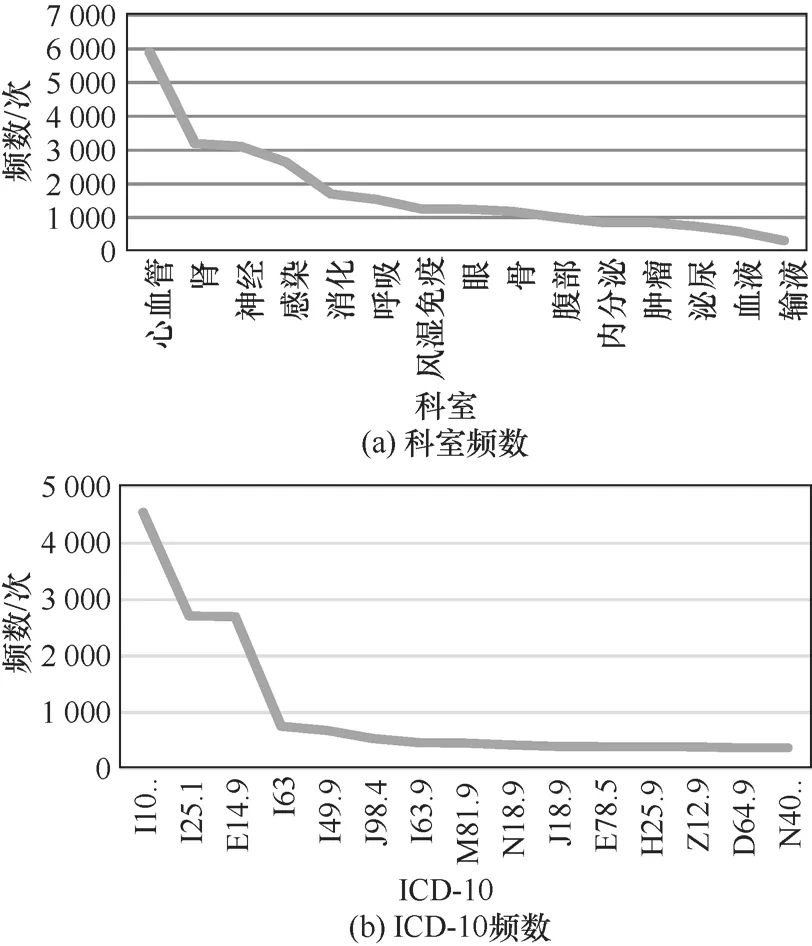
图2 科室和ICD-10编码的频数

图3 种子数据满足不同算法要求情况下不同种子数占比对聚类效果的影响
5 结束语
本文提出的基于ICD-10诊断编码的慢性病并发症半监督聚类算法,利用以种子数据形式给出的目标慢性病并发症先验分类知识,辅助临床医师完成对电子病历中出现的所有与目标疾病患者有关的相关并发症归类工作,在数据的分析与处理方面减轻了临床医师们的负担。本文所提算法只应用到了糖尿病及其并发症分析中,若提供其他目标慢性病数据集以及相关种子数据集,也能用于其他慢性病的并发症分析。实验结果表明,对于基于ICD-10诊断编码的慢性病并发症聚类问题,采用分组策略的算法要明显优于不采用分组策略的算法,并且采用分组策略的算法利用更少的种子数据就能够较好地反映并发症的分布特点,从而获得更好的聚类效果。
[1]WANG X, SONTAG D, WANG F.Unsupervised learning of disease progression models[C]// The 20th ACM SIGKDD International Conference on Knowledge Discovery and Data Mining,August 24-27, 2014, New York, USA.New York: ACM Press, 2014: 85-94.
[2]JOHNSON L W, WEINSTOCK R S. Medical complications of diabetes mellitus[M].Hoboken: John Wiley & Sons, Inc, 2014.
[3]CHAPELLE O, SCHÖLKOPF B, ZIEN A.Semi-supervised learning[M]. Cambridge:MIT Press, 2006.
[4]李昆仑, 曹铮, 曹丽苹, 等. 半监督聚类的若干新进展[J]. 模式识别与人工智能, 2009,22(5): 735-742.LI K L, CAO Z, CAO L P, et al. Some developments on semi-supervised clustering[J]. Pattern Recognition And Artificial Intelligence, 2009, 22(5): 735-742.
[5]ZHU X J, GOLDBERG A B. Introduction to semi-supervised learning[J]. Synthesis Lectures on Artificial Intelligence and Machine Learning, 2009, 3(1): 130.
[6]HUYEN D T T, LE H S, TUAN T M, et al.Semi-supervised fuzzy co-clustering for hospital-cost analysis from electronic medical records[C]// The 8th IEEE International Conference on Knowledge and Systems Engineering, October 6-8,2016, Hanoi, Vietnam. Piscataway: IEEE Press, 2016: 25-30.
[7]PHUONG N D, CHAU V T N, BAO H T. A hybrid semi-supervised learning approach to identifying protected health information in electronic medical records[C]// The 2016 ACM International Conference on Ubiquitous Information Management and Communication, January 4-6, 2016,Danang, Vietnam. New York: ACM Press,2016: 80.
[8]KHAN F M, KULIKOWSKI C A.Survival analysis via transduction for semi-supervised neural networks in medical prognosis[C]// The 2015 IEEE International Conference on Bioinformatics and Biomedicine, November 9-12, 2015,Washington, DC, USA. Piscataway: IEEE Press, 2015: 433-437.
[9]BASU S, BANERJEE A, MOONEY R J. Semi-supervised clustering by seeding[C]// The 19th International Conference on Machine Learning, July 8-12, 2002, Sydney, Australia. New York: ACM Press, 2002: 27-34.
[10]YOUNG B A, LIN E, KORFF M V, et al.Diabetes complications severity index and risk of mortality, hospitalization, and healthcare utilization[J]. American Journal of Managed Care, 2008, 14(1): 15-23.
[11]GLASHEEN W P, RENDA A, DONG Y T.Diabetes complications severity index(DCSI)-update and ICD-10 translation[J].Diabetes Complications, 2017, 31(6):1007-1013.
[12]International Diabetes Federation[R]. IDF Diabetes Atlas 8th Edition, 2017.

