蛋白质组学技术在肉类鉴别及肉质分析中的应用进展
胡争艳, 王军淋, 吴平谷, 王天娇, 王立媛, 汤 鋆
浙江省疾病预防控制中心, 杭州 310051
随着社会经济的发展和人民生活水平的提高,人们对肉食的需求量越来越大,同时也对肉类的品质提出了更高的要求。此外,多种市售深加工肉制品因其食用方便受到了广大消费者的欢迎,但是这也对深加工肉制品(特别是熟肉制品)中肉类的鉴别及其品质把控提出了挑战。
目前,肉制品中肉类的鉴别方法主要包括[1~3]:以DNA检测为基础的多重PCR法、限制性片段长度多态性PCR法、随机扩增多态性PCR法、荧光定量PCR法等;以蛋白质检测为基础的电泳分析法、免疫分析法、色谱分析法等;还有红外光谱法、核磁法、电子鼻技术等。虽然这些检测技术的发展已经明显提高了肉类鉴别的精确度和灵敏度,但是也都存在着较大的局限性。其中,PCR法因其具有较好的检测灵敏度和特异性,目前应用较为广泛,但由于PCR法容易出现交叉污染,因此对于样本制备的要求较高,而且容易受到肉制品加工过程中出现的DNA降解、复杂基质干扰等因素的影响而出现假阳性或假阴性的鉴定结果[4]。而蛋白质电泳法不适用于深加工及混合不同肉类的肉制品的分析。免疫分析法对于亲缘关系较近的物种易出现交叉反应,产生假阳性结果,且单克隆抗体虽然能大幅提高检测特异性,但其制备成本昂贵、步骤繁杂,因此难以推广应用。另外,在肉制品的加工过程中蛋白质的结构会发生不可逆的改变,导致其溶解性降低,难以采用色谱分析法进行检测。虽然,红外光谱法、核磁法具有检测快速无损耗、无需样品前处理的优点,但其精确度较低且不适用于复杂混合样品的检测。而电子鼻技术目前仍处于发展阶段,其在肉制品检测中的灵敏度、识别率等尚未达到令人满意的程度。因此,蛋白质组学技术的快速发展为探索肉制品中肉类的鉴别提供了新的研究思路。
肉的品质是指与肉的外观、适口性和营养价值等有关的物理、化学特性的综合体现,包括肉的颜色、pH、保水性指标(滴水损失)等,是决定消费者感官享受的重要因素[5]。一般来说,肉质的形成受到多种因素的影响,其中包括遗传(品种、基因型等)、营养因素(氨基酸水平、饲料添加剂等)、饲养方式、屠宰和储存条件等[6],然而,影响其品质形成的潜在分子机制尚未研究清楚。而蛋白质组学技术的应用为探索肉类品质形成的影响因素提供了新的研究视角。虽然,目前蛋白质组学在肉类鉴别和肉质研究中的应用尚处于早期阶段,但已经展现出了较好的前景。本文将对蛋白质组学的定义、主要研究策略及其在肉类鉴别和肉质研究中的应用进行介绍。
1 蛋白质组学及其主要研究策略
作为后基因组时代的核心,蛋白质组学(proteomics)是指通过生物及化学方法对细胞、组织等生物体内的蛋白质进行全面分析的一门学科[7]。传统的蛋白质样品分析通常是基于二维凝胶电泳(2D SDS-PAGE)色谱系统,它具有蛋白分离效率高的特点,但也存在操作步骤繁琐、重现性差、不适合分离疏水性蛋白及分子量极大或极小的蛋白等问题。此外,分离后蛋白质的鉴定需要通过Edman降解法进行氨基酸测序,极大程度上限制了蛋白质样品分析的通量[8~10]。20世纪90年代以来,生物质谱技术的不断发展,实现了蛋白质样品的大规模分析,使蛋白质组学研究取得了突破性进展。基于生物质谱的蛋白质分析法现已成为蛋白质组学研究的核心内容[11,12]。
随着蛋白质组学研究的不断深入,仅提供蛋白质鉴定信息的分析策略已不能满足当前研究工作的需要,而准确地定位生物体不同状态下发生变化的蛋白质则是目前研究的难点,这对蛋白质定量分析技术即差异蛋白质组学的发展提出了要求。传统的蛋白质定量分析主要是通过2D SDS-PAGE显色反应来实现的,其能同时定量上千种蛋白质的表达差异,是蛋白质组学技术早期应用于肉质分析中的主要研究方法。
近年来,随着质谱技术的发展,基于质谱分析的定量蛋白质组学技术也得到了快速发展,被广泛应用于生命活动的研究中。基于质谱技术的蛋白质定量策略主要有无标记定量和同位素标记相对定量两种方法(表1)。无标记定量技术是基于多次质谱分析的结果,因此,样品分析时要求稳定的液相色谱分离系统及良好的质谱采集数据重现性,以保证定量结果的准确性和可靠性。但同时由于其不需要对样品进行同位素标记等步骤,不会增加样品的复杂程度,操作简单,现多应用于肉质分析领域。而同位素标记定量技术根据同位素标记引入的方式又分为体内标记和体外标记。其中,体内标记方法省去了标记反应和分离纯化的步骤,减少了样品的损失,提高了蛋白质定量的准确性,但是,重同位素标记蛋白质的标记周期较长、成本较高。体外标记方法主要包括通过将标记试剂与蛋白质或肽段中的氨基酸反应引入稳定同位素的化学标记方法以及在蛋白质的酶解过程中引入稳定同位素的酶解标记方法(酶促标记)。其中,二甲基标记技术因具有标记效率高、反应迅速、标记试剂价格低等优点,已经成为应用最为广泛的化学标记方法。而酶解标记方法主要是指蛋白质在酶解的过程中使用H218O,在蛋白酶的作用下使肽段C端增加2个18O原子,从而实现蛋白质的定量分析。目前,基于同位素标记的定量蛋白质组学技术也已开始逐渐应用于肉质分析的研究中。
然而,上述蛋白质组学的定量方法均是通过高分辨质谱检测而实现的生物样本中信号强度较强(较高丰度)的蛋白质的相对定量,具有高通量的优点,但也表现出明显的局限性,即上述方法中蛋白质的定量结果具有非靶向性,而且无法实现样品中信号强度较弱(较低丰度)的蛋白质的定量,约束了检测的灵敏度。因此,为了增加蛋白质定量分析的特异性和准确性,提高对于目标蛋白质(尤其是低丰度目标蛋白质)定量分析的灵敏度,以蛋白质的特异性肽段为分析对象的靶向蛋白质组学定量方法逐步得到了发展[25]。靶向蛋白质组学定量方法主要包括2种技术:选择/多反应监测(selected/multiple reaction monitoring,SRM/MRM)[26,27]和平行反应监测(parallel reaction monitoring,PRM)[28,29]。SRM/MRM技术是利用三重四级杆质谱对目标蛋白质中特定肽段的母离子和子离子依次进行分选,从而实现对目标蛋白的定量分析,具有灵敏度高、准确性好的特点。而PRM技术则是在SRM/MRM的基础上,同时利用了四级杆的高选择性以及静电场轨道阱(Orbitrap)的高分辨率、高精度的特性来实现对目标蛋白的定量分析,具有更加优异的抗干扰能力和检测灵敏度。目前,靶向蛋白质组学定量方法在肉类鉴定方面得到了广泛的应用。
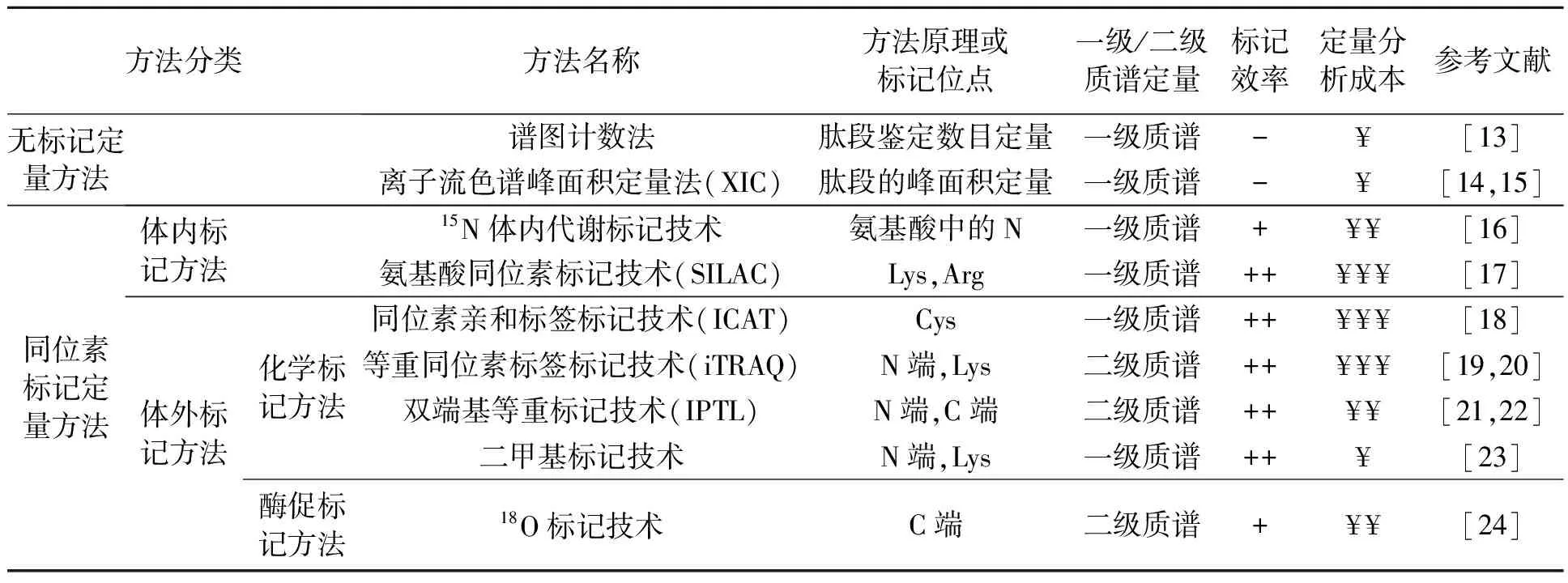
表1 定量蛋白质组学主要分析方法Table 1 The main analysis methods of quantitative proteomics.
注:+和++分别代表标记效率较高、非常高;¥、¥¥和¥¥¥分别代表定量分析成本较便宜、较高、非常高。
通过数据分析对蛋白质进行鉴定是蛋白质组学技术的核心步骤之一。主要利用生物信息学技术,采用特定的算法,根据蛋白质或蛋白质酶解得到的多肽分子量与蛋白质数据库进行比对,从而鉴定出样品中的蛋白质种类。目前,用于蛋白质组学数据鉴定的数据库主要是NCBI(National Center for Biotechnology Information database)数据库和UniProt KB(Universal Protein Knowledgebase)数据库;用于蛋白质组学数据鉴定的软件较多,较为常用的有Mascot、Protein Pilot、MaxQuant和X!Tandem等。
2 蛋白质组学技术在肉类鉴别及肉质分析中的应用
近年来,基于生物质谱技术的蛋白质组学方法得到了较快的发展,已被广泛用于深加工肉制品中肉类的鉴别、肉质特性(颜色、嫩度等)分析的相关研究中。而蛋白质组学的研究结果表明,肉质特性与其所含的蛋白质分子密切相关。
2.1 肉类品种鉴别
蛋白质组学技术应用于肉类品种的鉴别时,其主要研究策略为:首先提取待测肉制品所使用的原料肉中的蛋白质,经蛋白酶解、除杂净化后,利用高分辨质谱技术进行多肽鉴定(自下而上的蛋白质组学分析方法,bottom-up strategy),并将鉴定结果与蛋白质数据库(如UniProt等)进行比对,筛选出具有良好稳定性、物种特异性的1条或多条多肽标志物,然后建立上述多肽标志物的质谱MRM定性、定量分析方法,从而实现对肉制品中肉类的鉴别和掺假鉴定。该方法与应用较为广泛的PCR法相比,不仅具有良好的检测灵敏度和特异性,还具有更为突出的优点:多肽的稳定性强于DNA,待检样品不易受到肉制品加工过程的影响;可通过多条肽段标志物进行定性、定量分析,不仅能够保证检测结果的准确性,且能避免出现假阳性现象;样品处理相对简单,且MRM分析速度快、检测通量高,可以对肉制品中多物种进行同时溯源分析和鉴定。例如,Wulff等[30]通过自下而上的蛋白质组学分析方法,建立了22种不同种类鱼肉组织的肽段质谱图谱数据库,从而通过将未知鱼肉组织的肽段质谱分析图谱与已建立的质谱图谱数据库进行比对来实现鉴定鱼肉种类的目的,而且该方法适用于经过深加工的鱼肉种类的鉴别。另外,Montowska等[31]利用无标记相对定量蛋白质组学的方法建立了禽类深加工肉制品中是否掺假的判断方法。该研究通过定量蛋白质组学技术得到了鸡肉、鸭肉和鹅肉3种禽肉中具有热稳定性和物种特异性的20个肽段标志物,以用于3种禽肉加工产品中肉类的鉴定,该方法可以识别出3种禽肉混合物中掺杂低至1%的猪肉以及市售禽肉香肠中掺杂低至0.8%的牛肉蛋白,具有非常高的灵敏度。
2.2 新鲜肉与冻融肉的鉴别
肉类的储存和加工方式会影响其肉质的变化。冷冻一直被视作是肉类保存的最佳方法,然而,肉在冷冻的同时会伴随着品质的下降。这是由于冷冻时冰晶的形成会导致肉的组织结构破坏、机械张力破坏、蛋白变性等一系列生化和物理指标的变化,因而,一般来说冷冻肉的价格会低于新鲜肉,然而,一些不法商家为了追求利益最大化会将冷冻肉融化来冒充新鲜肉进行销售,因此,开发有效的区分新鲜肉和冻融肉的方法成为了研究热点。Kim等[32]首次利用2D SDS-PAGE结合生物质谱的方法对新鲜猪胸最长肌(1.0±0.5℃储存60 h)和经过冻融过程的猪胸最长肌(-20℃冷冻48 h后,1.0±0.5℃解冻12 h)的肉渗出液进行了差异蛋白质组学分析。结果显示,2种猪胸最长肌渗出液2D SDS-PAGE分别检测到450个蛋白点,其中24个蛋白点的丰度表现出显著性差异,经质谱分析进一步确证后,共发现22个可用于区分新鲜猪胸最长肌和经过冻融过程的猪胸最长肌的潜在蛋白标志物(包括钙网蛋白、脯氨酰羟化酶β亚基前体蛋白、血红蛋白α链、血红蛋白β链和丙酮酸激酶亚型3等)。这一研究表明差异蛋白质组学技术的应用成功实现了新鲜肉和冻融肉的判别,同时也为蛋白质组学技术用于研究其他储存方式和加工方式对肉质的影响奠定了理论基础。
2.3 肉的嫩度
肉的嫩度、色泽和风味等均是关系到消费者满意度的重要属性(肉的适口性),其中,嫩度是最重要的组成因素,同时也是不同肉类间差异最大的属性之一。肉的嫩度通常被认为是生物体内溶酶体酶、组织蛋白酶、钙激活中性蛋白酶和蛋白酶体等多种内源性蛋白酶经过一系列的生化反应导致的肌原纤维和结缔组织弱化的结果[33]。而随着双向电泳(2-DE)、生物质谱和生物信息学的不断发展,蛋白质组学技术已成为阐释肉的嫩度与α-肌动蛋白(α-actin)和肌球蛋白轻链-1(myosin light chain 1,MLC1)等部分结构蛋白、热休克蛋白27(heat shock protein,HSP27)等热休克蛋白、脂质抗氧化酶(peroxiredoxin-6,Prdx-6)等嫩度相关蛋白标志物之间关联的不可或缺的研究工具[34,35]。例如,Morzel等[36]通过对屠宰后的法国布朗德·安奎坦牛的新鲜胸最长肌和老化14 d的胸最长肌进行2-DE分析,发现HSP27可能是与其嫩度相关的重要蛋白标志物。同样地,Carvalho等[37]利用2-DE结合生物质谱检测的方法对剪切力大小不同(即嫩度不同)的2组牛肉进行分析,发现2组牛肉之间的蛋白质种类和丰度均存在明显差异。质谱鉴定结果发现2组不同嫩度的牛肉中的差异蛋白质主要包括α-actin、MLC1、MLC3、MLC2F和原肌球蛋白等结构蛋白,HSPB1和HSP70等与细胞组织相关的热休克蛋白以及β-乳球蛋白(β-lactoglobulin,β-LG)、酰基辅酶A结合结构域蛋白6(ACBD6)等与代谢过程相关的蛋白,这一结果证明不同嫩度的牛肉中结构蛋白含量存在显著性差异。此外,Jia等[38]利用2-DE结合质谱鉴定的方法对不同嫩度的牛背长肌肌肉进行了蛋白质组学分析,发现过氧化物氧还酶6(peroxiredoxin 6,Prdx6)可能是影响其嫩度的潜在蛋白质标志物。而Bjarnadóttir等[39]则通过iTRAQ同位素标记定量蛋白质组学方法对不同嫩度的牛肉进行了分析,发现了3个均未被报道过的与嫩度差异相关的蛋白质:三羧酸循环相关蛋白2(oxoglutarate dehydrogenase complex component E2)、细胞凋亡相关蛋白(galectin-1)以及控制细胞内钙离子释放的蛋白(annexin A6)。另外,肉类的包装方式和储存时间也会影响其嫩度。Moczkowska等[40]研究发现,真空贴体包装条件下的牛背最长肌的剪切力在其储存14 d和28 d后显著低于气调保鲜包装储存条件下的牛背最长肌,同时嫩度也更佳,这是由于气调保鲜包装方式中氧气的存在会促进牛背最长肌中肌球蛋白的聚合,从而使肌肉老化。尽管上述研究结果尚未完全阐明蛋白质标志物与肉的嫩度之间的潜在分子机制,但为肉产业中肉质嫩度预测和动物肉质嫩度改良方法的研究提供了理论基础。
2.4 肉色
从销售的角度来讲,肉色是影响消费者购买欲望的关键因素之一,消费者通常认为樱桃红色是新鲜、可靠的健康肉的标志,而肉色的变化会直接影响其销售情况和价格,因此,从蛋白质水平上开展对于不同品种或不同部位肌肉肉色差异的研究具有非常重要的意义。以往关于肉色的研究多关注于肌红蛋白与少数小分子之间的相互作用,认为肉色变化与脂质氧化引起的肌红蛋白氧化密切相关。近年来,蛋白质组学和质谱技术开始成为研究肉色差异的潜在生化机制的高通量分析工具。Li等[41,42]发现绵羊肉中肌浆蛋白质的磷酸化水平可能与其颜色的形成密切相关。实验结果表明,绵羊肉颜色的稳定性与肌红蛋白的磷酸化水平呈负相关,而蛋白质的磷酸化水平则可能通过调节肌红蛋白的糖基化和氧化还原稳定性来实现,进而影响其颜色的形成。Joseph等[43]利用无标记定量蛋白质组学的方法对具有不同肌肉颜色稳定性的牛背长肌肌肉(颜色稳定)和腰大肌肌肉(颜色易变)的肌浆蛋白质组分别进行了分析,结果发现,与肌肉红色及颜色稳定性正相关的抗氧化蛋白、分子伴侣等蛋白质均在背长肌肌肉中过表达,而在腰大肌肌肉中过表达的蛋白质均与肌肉红色呈负相关,这一结果也证实了要针对不同组织部位开发不同的牛肉处理方法以改善牛肉颜色的必要性。Yu等[44]利用无标记定量蛋白质组学的方法阐明了荷斯坦牛肉在宰后4℃储存过程中腰最长肌和腰大肌的颜色稳定性。结果表明在储存4 d和9 d后,腰最长肌比腰大肌表现出更好的红色,而腰大肌中高铁肌红蛋白的增加比例要明显高于腰最长肌。另外,腰最长肌和腰大肌的比较蛋白质组学研究结果表明,大量具有抗氧化、保护和修复作用的有益于颜色稳定的蛋白质在腰最长肌中过表达;而大量主要参与氧化还原过程、三羧酸循环和线粒体电子传递过程的蛋白质则在腰大肌中过表达,从而导致其具有较差的颜色稳定性。这一研究从蛋白质组学的角度阐明了牛肉组织呈现颜色特异性的分子机制。
综上所述,目前鉴定到的与肉色稳定性相关的蛋白质标志物主要与动物宰后肉的氧化代谢过程相关,其存在能够延迟肉类pH的下降并且减缓蛋白质的变性,因此,上述研究为开发保持肉色的方法(抗氧化方法或包装方式等)提供了新思路。
2.5 持水性
持水性是肉质的一个重要特性,通常用滴水损失来评价,与动物宰后的新陈代谢变化有着密切联系。Lawson等[45]研究表明宰后肉中整合素(integrin)蛋白的降解会导致其滴水通道的形成,即integrin降解越少,滴水损失率越小,肌肉持水力越强,因此,肉中蛋白质的组成与变化对于肉的持水性具有重要影响。而利用差异蛋白质组学技术研究不同持水能力的肉中蛋白质的组成与变化对于揭示与肉持水性相关的分子机制是十分必要的。研究发现,不同肉类的持水能力与其表达的多种代谢酶和热休克蛋白等的含量密切相关。例如,Phongpa-Ngan等[46]对具有不同持水能力的鸡肉进行了差异蛋白质组学分析,发现高持水能力的鸡肉与低持水能力的鸡肉相比,含有5个高表达、4个低表达的蛋白点,经基质辅助激光解吸/电离飞行时间质谱(MALDI-TOF-MS)鉴定后发现,这些差异蛋白质主要包括丙酮酸激酶、磷酸丙糖异构酶等代谢酶及热休克蛋白等。同样地,Di等[47]通过对不同持水能力的猪肉离心滴出液进行蛋白质组学分析,质谱鉴定结果表明,44个蛋白质的丰度会随着猪肉宰后时间的增加而发生变化,但这些蛋白质中只有HSP70的丰度在低滴水损失样品的滴出液中显著高于其他样品,由此推断,在不同滴水损失的样品中,细胞内HSP70的含量存在明显差异,即HSP70可能作为猪肉样品中持水性预测的潜在标志物。Zuo等[48]通过利用2D SDS-PAGE结合基质辅助激光解吸离子飞行时间串联(MALDI TOF/TOF)质谱分析方法,分别对屠宰后0 d、1 d、7 d内的不同持水性的牦牛背长肌进行了差异蛋白质组学分析。结果表明,55个蛋白质的丰度随着宰后时间的增加发生了显著变化,这些蛋白质主要包括4类:代谢酶、细胞结构蛋白、压力相关蛋白和转运蛋白,其中,肌球蛋白轻链、热休克蛋白27和磷酸丙糖异构酶3个蛋白质在高、低滴水损失率样品中表现出显著性差异,可以作为肉持水能力预测的潜在标志物。另外,生物信息学分析结果表明,这些鉴定到的蛋白质主要参与了碳代谢、氨基酸的糖基化和生物合成等生物学过程,这一结果对于揭示与肉类持水性相关的分子机制具有重要意义。
2.6 宰后代谢机制
宰后新陈代谢一直被认为是影响肉质变化的重要因素。研究表明,宰后肉类会发生一系列的生化反应(pH下降、滴水损失等),这些生化反应发生的过程可能会极大地影响肉类的品质,然而,目前宰后代谢过程中肉质变化的相关分子机制仍不清晰。现阶段,蛋白质组学技术的应用开始从分子水平上揭示宰后蛋白质磷酸化修饰水平的变化与肉质变化之间存在着的密切联系。例如,Huang等[49]通过利用SDS-PAGE结合MALDI-TOF质谱检测的方法,研究发现随着屠宰后猪背长肌放置时间的延长,肌纤维蛋白的磷酸化水平发生了明显变化,而肌纤维蛋白磷酸化水平的变化则可能是与屠宰后发生猪肉肌肉僵直和肉质变化关联的直接原因。近期,Huang等[50]又利用高通量的二甲基标记定量蛋白质组学技术对屠宰24 h内的猪背长肌肌肉的磷酸化蛋白质组进行了分析,定量结果发现93个磷酸化蛋白质的184个磷酸化位点发生了显著变化,而这些发生变化的蛋白质的生物学功能主要是与葡萄糖的代谢以及肌肉的收缩过程相关。由此说明,由宰后猪肉的新陈代谢引起的肉质变化与蛋白质的磷酸化水平具有紧密的关联。因此,蛋白质组学技术的应用对于动物宰后新陈代谢分子机制的研究及其与宰后肉质变化之间的关联性研究具有重要价值。
2.7 肉质的营养调控
一般来说,肉类的品质受到遗传、饲养条件(包括营养条件和饲养方式等)、屠宰和储存条件等多种因素的影响。在动物的饲养过程中,可通过吸收饲料中的可消化营养物质来改变机体的能量和蛋白质的代谢水平,并最终影响动物的肉质,因此,可以通过调整动物饲料中营养物质的组成(如增加脂肪、氨基酸、维生素、矿物质的含量等)和供给水平(短期禁饲等)来达到改善肉质的目的[51~55]。目前,蛋白质组学技术已逐渐应用于阐述膳食营养物质的添加对于肉质调控的研究中。例如,Ma等[54]研究发现在猪饲料中添加1%L-精氨酸,能够在不影响其生长速度的前提下,使猪肉的肌内脂肪含量增加约32%,同时降低其宰后48 h的滴水损失。这是由于向猪饲料中添加1%L-精氨酸会显著影响猪肉中与能量代谢、肌纤维类型和结构形成等相关的蛋白质的丰度,从而使猪肉的肌内脂肪含量增加,并达到改善肉质的目的。另外,della Malva等[55]通过分析添加了亚麻籽和藜麦的饲料喂养的意大利美利奴羊的差异蛋白质组信息,评价了饲料中亚麻籽和藜麦的添加对于羊肉嫩度的影响,研究结果显示,添加了亚麻籽的饲料喂养的羊肉与普通无脂肪添加的饲料喂养的羊肉相比,其肌间线蛋白和肌钙蛋白复合物等发生了显著降解,即羊肉嫩度增加,这一结果与不同喂养方式下羊肉剪切力的测定结果一致(添加了亚麻籽的饲料喂养的羊肉剪切力最小);而喂养添加了藜麦的饲料对于羊肉的嫩度的改善作用略低于添加了亚麻籽的饲料的喂养结果。因此,蛋白质组学技术的应用从分子水平上阐明了动物饲料中营养物质的添加对于肉质改善的作用机理。
3 展望
目前,基于2-DE和生物质谱的蛋白质组学技术已经成为肉类鉴别和肉质分析中高通量、结果可靠的强有力的分析工具。蛋白质组学技术的发展与应用使肉的新鲜度鉴别及深加工肉制品中肉的种类鉴定成为了可能,同时在揭示肉质特性形成的潜在分子机制方面已经取得了一系列的进展,在一定程度上,从分子水平上阐明了蛋白质及生物分子之间的相互作用是影响肉质相关生化、物理化学特性的根本原因。然而,由于生物体是其基因、蛋白质等相互作用的有机体,蛋白质组学技术并不能完全解释各种生物学系统的现象,因此,综合利用系统生物学中多组学技术(蛋白质组学、基因组学、翻译后修饰组学、代谢组学等)将成为未来肉质研究的重要方向。与此同时,基于SRM/MRM和PRM的靶向蛋白质组学定量技术也将作为非靶向蛋白质组学方法的补充,在肉质分析相关的蛋白标志物的高通量验证中发挥越来越重要的作用。
参 考 文 献
[1] Ballin N Z. Authentication of meat and meat products[J]. Meat Sci., 2010, 86(3): 577-587.
[2] Danezis G P, Tsagkaris A S, Camin F,etal.. Food authentication: Techniques, trends & emerging approaches[J]. Trend. Anal. Chem., 2016, 85: 123-132.
[3] Abbas O, Zadravec M, Baeten V,etal.. Analytical methods used for the authentication of food of animal origin[J]. Food Chem., 2018, 246: 6-17.
[4] Di Pinto A, Bottaro M, Bonerba E,etal.. Occurrence of mislabeling in meat products using DNA-based assay[J]. J. Food Sci. Technol., 2015, 52(4): 2479-2484.
[5] 杨金宝, 何若钢, 秦小娥, 等. 营养与猪肉品质[J]. 猪业科学, 2006, 23(12):68-69.
[6] 杨建成,徐日峰,胡建民. 影响猪肉品质因素的研究进展[J]. 湖北农业科学, 2013, 52(20): 4849-4852.
[7] Pandey A, Mann M. Proteomics to study genes and genomes[J]. Nature, 2000, 405(6788): 837-846.
[8] Tonge R, Shaw J, Middleton B,etal.. Validation and development of fluorescence two-dimensional differential gel electrophoresis proteomics technology[J]. Proteomics, 2001, 1(3): 377-396.
[9] Rabilloud T. Two-dimensional gel electrophoresis in proteomics: Old, old fashioned, but it still climbs up the mountains[J]. Proteomics, 2002, 2(1): 3-10.
[10] Cox J, Mann M. Is proteomics the new genomics?[J]. Cell, 2007, 130(3): 395-398.
[11] Aebersold R, Goodlett D R. Mass spectrometry in proteomics[J]. Chem. Rev., 2001, 101(2): 269-296.
[12] Mann M, Hendrickson R C, Pandey A. Analysis of proteins and proteomes by mass spectrometry[J]. Ann. Rev. Biochem., 2001, 70(1): 437-473.
[13] Arike L, Peil L. Spectral counting label-free proteomics[A]. In: Martins-de-Souza D. Shotgun Proteomics[M]. New York: Humana Press, 2014, 213-222.
[14] Han C L, Chen J S, Chan E C,etal.. An informatics-assisted label-free approach for personalized tissue membrane proteomics: Case study on colorectal cancer[J]. Mol. Cell. Proteomics, 2011, 10(4): M110.003087.
[15] Cox J, Hein M Y, Luber C A,etal.. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ[J]. Mol. Cell. Proteom., 2014, 13(9): 2513-2526.
[16] Krijgsveld J, Ketting R F, Mahmoudi T,etal.. Metabolic labeling ofC.elegansandD.melanogasterfor quantitative proteomics[J]. Nat. Biotechnol., 2003, 21(8): 927-931.
[17] Mann M. Functional and quantitative proteomics using silac [J]. Nat. Rev. Mol. Cell Biol., 2006, 7: 952-958.
[18] Gygi S P, Rist B, Gerber S A,etal.. Quantitative analysis of complex protein mixtures using isotope-coded affinity tags[J]. Nat. Biotechnol., 1999, 17(10): 994-999.
[19] Zieske L R. A perspective on the use of iTRAQTMreagent technology for protein complex and profiling studies[J]. J. Exp. Bot., 2006, 57(7): 1501-1508.
[20] Wiese S, Reidegeld K A, Meyer H E,etal.. Protein labeling by iTRAQ: A new tool for quantitative mass spectrometry in proteome research[J]. Proteomics, 2007, 7(3): 340-350.
[21] Koehler C J, Arntzen M Ø, de Souza G A,etal.. An approach for triplex-isobaric peptide termini labeling (triplex-IPTL)[J]. Anal. Chem., 2013, 85(4): 2478-2485.
[22] Koehler C J, Strozynski M, Kozielski F,etal.. Isobaric peptide termini labeling for MS/MS-based quantitative proteomics[J]. J. Proteome Res., 2009, 8(9): 4333-4341.
[23] Boersema P J, Raijmakers R, Lemeer S,etal.. Multiplex peptide stable isotope dimethyl labeling for quantitative proteomics[J]. Nat. Protoc., 2009, 4(4): 484-494.
[24] Yao X, Freas A, Ramirez J,etal.. Proteolytic18O labeling for comparative proteomics: Model studies with two serotypes of adenovirus[J]. Anal. Chem., 2001, 73(13): 2836-2842.
[25] Vidova V, Spacil Z. A review on mass spectrometry-based quantitative proteomics: Targeted and data independent acquisition[J]. Anal. Chim. Acta, 2017, 964: 7-23.
[26] Picotti P, Aebersold R. Selected reaction monitoring-based proteomics: Workflows, potential, pitfalls and future directions[J]. Nat. Methods, 2012, 9(6): 555-566.
[27] Kitteringham N R, Jenkins R E, Lane C S,etal.. Multiple reaction monitoring for quantitative biomarker analysis in proteomics and metabolomics[J]. J. Chromatog. B, 2009, 877(13): 1229-1239.
[28] Gallien S, Kim S Y, Domon B. Large-scale targeted proteomics using internal standard triggered-parallel reaction monitoring (IS-PRM)[J]. Mol. Cell. Proteom., 2015, 14(6): 1630-1644.
[29] Peterson A C, Russell J D, Bailey D J,etal.. Parallel reaction monitoring for high resolution and high mass accuracy quantitative, targeted proteomics[J]. Mol. Cell. Proteom., 2012, 11(11): 1475-1488.
[30] Wulff T, Nielsen M E, Deelder A M,etal.. Authentication of fish products by large-scale comparison of tandem mass spectra[J]. J. Proteome Res., 2013, 12(11): 5253-5259.
[31] Montowska M, Fornal E. Label-free quantification of meat proteins for evaluation of species composition of processed meat products[J]. Food Chem., 2017, 237: 1092-1100.
[32] Kim G D, Jeong T C, Yang H S,etal.. Proteomic analysis of meat exudates to discriminate fresh and freeze-thawed porcine longissimus thoracis muscle[J]. LWT-Food Sci. Technol., 2015, 62(2): 1235-1238.
[33] Dransfield E. Optimization of tenderization, aging and tenderness [J]. Meat Sci., 1994, 36(1-2): 105-121.
[34] della Malva A, Marino R, Santillo A,etal.. Proteomic approach to investigate the impact of different dietary supplementation on lamb meat tenderness[J]. Meat Sci., 2017, 131: 74-81.
[35] Rosa A F, Moncau C T, Poleti M D,etal.. Proteome changes of beef in Nellore cattle with different genotypes for tenderness[J]. Meat Sci., 2018, 138: 1-9.
[36] Morzel M, Terlouw C, Chambon C,etal.. Muscle proteome and meat eating qualities of Longissimus thoracis of “Blonde d’Aquitaine” young bulls: A central role of HSP27 isoforms[J]. Meat Sci., 2008, 78(3): 297-304.
[37] Carvalho M E, Gasparin G, Poleti M D,etal.. Heat shock and structural proteins associated with meat tenderness in Nellore beef cattle, aBosindicusbreed[J]. Meat Sci., 2014, 96(3): 1318-1324.
[38] Jia X, Veiseth-Kent E, Grove H,etal.. Peroxiredoxin-6——A potential protein marker for meat tenderness in bovine longissimus thoracis muscle[J]. J. Anim. Sci., 2009, 87(7): 2391-2399.
[39] Bjarnadoóttir S G, Hollung K, Høy M,etal.. Changes in protein abundance between tender and tough meat from bovineLongissimusthoracismuscle assessed by isobaric tag for relative and absolute quantitation (iTRAQ) and 2-dimensional gel electrophoresis analysis[J]. J. Anim. Sci., 2012, 90(6): 2035-2043.
[40] Moczkowska M, Pótorak A, Montowska M,etal.. The effect of the packaging system and storage time on myofibrillar protein degradation and oxidation process in relation to beef tenderness [J]. Meat Sci., 2017, 130: 7-15.
[41] Li M, Li X, Xin J,etal.. Effects of protein phosphorylation on color stability of ground meat[J]. Food Chem., 2017, 219: 304-310.
[42] Li M, Li Z, Li X,etal.. Comparative profiling of sarcoplasmic phosphoproteins in ovine muscle with different color stability[J]. Food Chem., 2018, 240: 104-111.
[43] Joseph P, Suman S P, Rentfrow G,etal.. Proteomics of muscle-specific beef color stability[J]. J. Agric. Food Chem., 2012, 60(12): 3196-3203.
[44] Yu Q, Wu W, Tian X,etal.. Comparative proteomics to reveal muscle-specific beef color stability of Holstein cattle during post-mortem storage[J]. Food Chem., 2017, 229: 769-778.
[45] Lawson M A. The role of integrin degradation in post-mortem drip loss in pork[J]. Meat Sci., 2004, 68(4): 559-566.
[46] Phongpa-Ngan P, Grider A, Mulligan J H,etal.. Proteomic analysis and differential expression in protein extracted from chicken with a varying growth rate and water-holding capacity[J]. J. Agric. food Chem., 2011, 59(24): 13181-13187.
[47] Di Luca A, Mullen A M, Elia G,etal.. Centrifugal drip is an accessible source for protein indicators of pork ageing and water-holding capacity[J]. Meat Sci., 2011, 88(2): 261-270.
[48] Zuo H, Han L, Yu Q,etal.. Proteome changes on water-holding capacity of yak longissimus lumborum during postmortem aging[J]. Meat Sci., 2016, 121: 409-419.
[49] Huang H, Larsen M R, Lametsch R. Changes in phosphorylation of myofibrillar proteins during postmortem development of porcine muscle[J]. Food Chem., 2012, 134(4): 1999-2006.
[50] Huang H, Larsen M R, Palmisano G,etal.. Quantitative phosphoproteomic analysis of porcine muscle within 24 h postmortem[J]. J. Proteom., 2014, 106: 125-139.
[51] Calvo L, Toldrá F, Rodríguez A I,etal.. Effect of dietary selenium source (organic vs. mineral) and muscle pH on meat quality characteristics of pigs[J]. Food Sci. Nutr., 2017, 5(1): 94-102.
[53] Andersen H J, Oksbjerg N, Young J F,etal.. Feeding and meat quality——A future approach[J]. Meat Sci., 2005, 70(3): 543-554.
[54] Ma X, Zheng C, Hu Y,etal.. Dietary L-arginine supplementation affects the skeletal longissimus muscle proteome in finishing pigs[J]. PLoS ONE, 2015, 10(1): e0117294.
[55] della Malva A, Marino R, Santillo A,etal.. Proteomic approach to investigate the impact of different dietary supplementation on lamb meat tenderness[J]. Meat Sci., 2017, 131: 74-81.

