宏基因组来源果胶酸裂解酶在毕赤酵母中的表达及应用
董俊帅, 秦 星, 张志伟*, 张宇宏*, 张 伟
1.山西农业大学林学院, 山西 太谷 030801;2.中国农业科学院生物技术研究所, 北京100081
果胶(pectin)广泛存在于植物细胞初生壁及中胶层,是一种复杂的高分子聚合物,其主链由α-1,4-糖苷键连接的聚半乳糖醛酸组成[1,2]。在多种农产品加工过程中,果胶的存在会带来一些不利影响。如在果汁加工中,果胶的存在导致果浆粘稠,果汁压榨率低而且浑浊;在动物的肠胃中,果胶的存在会增加食糜粘性,影响消化吸收。在苎麻纺织中,果胶将韧皮组织中的单纤维粘结成片状,导致纤维粘结、不易染色、吸水性差。在传统精炼脱胶工艺中,需要把麻纤维在强碱和高温条件下煮炼数小时以脱除果胶,不仅使棉织物纤维机械强度降低,还存在高能耗、高污染的问题。近年来,果胶酶尤其是果胶裂解酶在棉麻脱胶领域得到广泛关注。在新型的酶法精炼工艺中,生产过程在较低温度下进行,降低了生产能耗,减少了污水排放,经酶处理所得的纺织品吸水率高、易染色、手感更好。因而酶精炼工艺有望成为未来纺织工业生产技术的重要发展方向[3,4]。
果胶酶(pectinase)是一类催化降解果胶分子的酶的总称,通常可分为聚半乳糖醛酸酶(PG,EC 3.2.1.15)、果胶酯酶(PE,EC 3.1.1.11)和果胶解聚酶[5]。果胶酸裂解酶(Pel, EC 4.2.2.2)是果胶解聚酶的一种。果胶酸裂解酶来源广泛,如:来源于微生物(Pseudoalteromonassp.[6]、Bacillussp.[7~9]、Aspergillussp.[10,11])、植物[12]、动物[13~15]等。除前述用于棉麻脱胶外,果胶酸裂解酶还可应用于食品加工、造纸、环保、油料提取和茶叶发酵等领域[5]。但目前商品化的果胶酸裂解酶仍然不能满足各种应用环境对酶制剂的特殊性质要求,例如在苎麻脱胶中,为了降低能耗和减少环境污染,需要酶在较低的催化温度和偏碱pH环境下保持高催化效率,而且要求酶在最适操作条件下具有较好的稳定性。然而目前大多数果胶酸裂解酶仍然难以完全满足苎麻脱胶对酶性质的要求。因此,有待于进一步挖掘和开发性质优良的新型果胶酸裂解酶。
宏基因组(metagenome)是指特定生态环境中可培养和不可培养的所有生物遗传物质的总和。据报道,99%的微生物应用现有实验条件和技术还不能进行培养,仅1%为可培养微生物。传统分离纯培养的方法极大的限制了人们对微生物资源的开发和认识,大量潜在的新的微生物和基因资源仍未被发现。从宏基因组中挖掘新基因成为研究热点,传统方法是构建宏基因组文库,基于序列驱动筛选或者功能驱动筛选策略从中发现新基因。但构建文库及筛选过程需要耗费大量人工和时间。近年来,随着高通量测序技术的发展,越来越多的宏基因组DNA信息被解析,使得不依赖于文库构建的新基因挖掘成为可能。
本研究从富含果胶的土壤中,基于高通量宏基因组测序技术挖掘到1个果胶酸裂解酶基因,并在毕赤酵母中成功实现了异源表达,并对其性质进行了研究,以期为微生物基因资源利用及苎麻脱胶相关研究提供参考。
1 材料与方法
1.1 实验材料
1.1.1宏基因组的制备及测序 取果园土壤,加入大量果胶并自然发酵两个月,取土壤样品委托百迈克生物科技有限公司进行宏基因组测序。
1.1.2菌株、酶和试剂 Trans1-T1大肠杆菌感受态细胞和FastpfuDNA 聚合酶为北京全式金生物技术有限公司产品;表达载体pPIC9和酵母菌株PichiapastorisGS115为Invitrogen公司产品;质粒小提试剂盒、琼脂糖凝胶DNA回收试剂盒为北京天根生化技术有限公司产品;限制性内切酶(EcoRⅠ、NotⅠ、BglⅡ)为Thermo公司产品;聚半乳糖醛酸(PGA)、酯化度约34%的果胶(DE34)、酯化度约70%的果胶(DE70)和酯化度约85%的果胶(DE85)为Sigma公司产品。酵母提取物(yeast extract)和蛋白胨(tryptone)为Oxford公司产品;氨苄青霉素和牛血清白蛋白(BSA)标准品为经科宏达产品;内切β-N-乙酰氨基葡萄糖苷酶(Endo-H)为New England Biolabs公司产品;其他试剂均为国产分析纯。
1.1.3实验仪器 PCR仪(BioRad公司);基因电击导入仪(BioRad公司);发酵罐(Bioengineering AG公司);蛋白纯化系统和镍亲和层析柱(GE Healthcare公司);切向流超滤系统(Sartorius公司);Stop-flow停留反应分析仪(Applied Photophysics公司)。
1.1.4培养基配方 YPD培养基:20 g/L蛋白胨,10 g/L酵母提取物,20 g/L葡萄糖;MD 固体培养基:20 g/L葡萄糖,0.000 4 g/L生物素,13.4 g/L 酵母氨基氮源(YNB),20 g/L琼脂糖;BMGY培养基:20 g/L蛋白胨,10 g/L酵母提取物,11.8 g/L KH2PO4,3 g/L K2HPO4·3H2O,13.4 g/L YNB,0.000 4 g/L生物素,20 g/L甘油;BMMY培养基:20 g/L蛋白胨,10 g/L酵母提取物,11.8 g/L KH2PO4,3 g/LK2HPO4·3H2O,13.4 g/L YNB,0.000 4 g/L生物素,1%(V/V)甲醇;LB培养基:10 g/L蛋白胨,5 g/L酵母提取物,10 g/L NaCl。固体培养基含 20 g/L琼脂。
1.2 实验方法
1.2.1果胶酸裂解酶基因序列分析 从宏基因组测序数据中,将注释为果胶酸裂解酶基因的序列提取出来,使用在线比对工具Nucleotide Blast(http://www.ncbi.nlm.nih.gov/BLAST/)进行序列比对。收集文献报道已经验证功能的果胶酸裂解酶、GenBank中登录的果胶酸裂解酶以及本研究从宏基因组中搜索得到的果胶酸裂解酶氨基酸序列,用MEGA 5软件Neighbor-Joining方法构建系统发育进化树。使用Vector NTI 10.3软件预测蛋白理论分子量和等电点。使用在线工具SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/)预测蛋白信号肽;用NetNGlyc 1.0 Server(http://www.cbs.dtu.dk/services/NetNGlyc/)和NetOGlyc 4.0 Server(http://www.cbs.dtu.dk/services/NetOGlyc/)分别预测蛋白潜在的N-糖基化位点和O-糖基化位点。
1.2.2果胶酸裂解酶基因扩增引物设计 根据果胶酸裂解酶pela基因测序序列,设计不含信号肽的扩增引物P1(5′-CCGGAATTCAATACAAATACTGACAATTCGTACAAAGAGAATGCTA-CTCCCGACCGTTC-3′,下划线为EcoRⅠ位点)和P2(5′-ATAAGAATGCGGCCGCTTAGTGGTGGTGG-TGGTGGTGTTTTTTAGAATCCGCAGGATAC-3′,下划线为NotⅠ位点,斜体为His-tag序列)。引物由生工生物工程(上海)股份有限公司合成。
1.2.3果胶酸裂解酶基因的扩增及pPIC9-pela构建 参考Sean[16]报道的土壤宏基因组提取方法,将提取的宏基因组经DNA回收试剂盒回收大片段DNA。以回收的宏基因组DNA为模板,以P1和P2为引物,使用FastpfuDNA聚合酶扩增果胶酸裂解酶基因。扩增产物经EcoRⅠ和NotⅠ酶切,连接表达载体pPIC9,连接产物转化Trans1-T1大肠杆菌感受态细胞,涂布于含有氨苄青霉素的LB培养基平板上,37℃过夜培养,挑取单菌落进行PCR验证。验证正确的菌落经扩大培养后抽提得到pPIC9-pela重组质粒。
1.2.4重组质粒pPIC9-pela转化及诱导表达 重组质粒pPIC9-pela经BglⅡ线性化后电击转化P.pastorisGS115感受态细胞并涂布于MD培养基平板,28℃培养48~72 h至转化子长出。挑取MD平板上阳性转化子于含有500 μL BMGY培养基的48孔细胞培养板中,28℃振荡培养48 h,4 000 g离心10 min弃去上清,然后加入500 μL BMMY诱导培养基重悬菌体,28℃振荡培养48 h。每隔24 h补加一次甲醇,补加量为体系终浓度的1%。培养结束后,4 000 g离心10 min分离菌体,测定培养液上清中的果胶酸裂解酶活力。
1.2.5高活力转化子复筛及高细胞密度发酵
挑取酶活力较高的转化子接种于10 mL BMGY培养基中,28℃ 200 r/min培养48 h,4 000 g离心10 min,弃去上清后加入5 mL BMMY诱导培养基,28℃ 200 r/min继续培养48 h。每隔24 h补加1次甲醇,补加量为终浓度的1%。培养结束后,4 000 g离心10 min分离菌体,测定培养液上清中的果胶酸裂解酶活力。将筛选出的酶活力最高的转化子在3 L发酵罐进行高细胞密度发酵。发酵工艺参照Invitrogen毕赤酵母操作手册。
1.2.6重组果胶酸裂解酶的纯化 将所得发酵液在6 000 g,4℃离心10 min以除去细胞和其他不溶物。上清液用0.1 μm微滤膜去除残留的菌体,滤液经截留分子量为10 kDa的超滤膜进行超滤浓缩,并去除色素和高浓度的盐分。所得超滤浓缩液经镍离子亲和层析柱进行纯化。纯化的重组蛋白PELA经聚丙烯酰胺凝胶电泳(SDS-PAGE)检测纯化结果。纯化的重组蛋白经内切β-N-乙酰氨基葡萄糖苷酶(Endo-H)处理去除糖基化。
1.2.7果胶酸裂解酶活力测定 果胶酸裂解酶活力测定方法参考Zhou等[17]的方法:380 μL的0.2%(w/V)聚半乳糖醛酸底物预热2 min,加入20 μL酶液(适当稀释),45℃反应10 min后立即加入600 μL 0.03 mol/L的H3PO4终止反应,于波长235 nm处测定光吸收值。空白对照380 μL的底物预热2 min,先加入600 μL 0.03 mol/L的H3PO4反应10 min后再加入20 μL酶液。酶活力(X)计算公式:
式中,5 500表示双键在235 nm波长处的消光系数为5 500/mol·cm[18,19]。
1个酶活力单位定义为在1 min内生成1 μmol的双键所需蛋白量。
1.2.8重组PELA酶学性质测定 以牛血清白蛋白为标准品,根据Bradford方法测定纯化重组PELA的蛋白浓度,并计算出酶的比活力。酶的比活力定义为每毫克蛋白所具有酶活力的单位数。
在40℃,不同pH(4.0~11.0)条件下测定重组酶最适pH。测定所用的缓冲液为:HAc-NaAc缓冲液pH 4.0~6.0;Na2HPO4-NaH2PO4缓冲液pH 6.0~8.0;Tris-HCl缓冲液pH 8.0~9.0;Gly-NaOH缓冲液pH 9.0~12.0。在最适pH条件下,测定不同温度(20~70℃)下的重组酶活力以确定重组酶蛋白的最适温度。
将纯化的酶蛋白置于不同pH(4.0~12.0)的缓冲液中,室温处理1 h后,测定重组酶的剩余活力。在最适pH下,将重组酶在不同的温度(20~65℃)下保温1 h,取出立即置于冰上,测定重组酶的剩余活力。
以较低浓度(0.25 mg/mL)聚半乳糖醛酸为底物,使用Stop-flow停留反应分析仪测定重组酶的一级反应时间。在一级反应时间内测定不同底物浓度下(0.25~2.5 mg/mL)重组酶的反应速率。用GraphPad Prism 5软件非线性拟合工具计算重组酶的Km和Vmax。
在最适温度和最适pH下,酶活力测定体系中分别加入不同的金属离子(Ni2+,Co2+,Cd2+,Mg2+,Zn2+,Mn2+,Ca2+,Fe3+,Cu2+,Li+,K+)或化学试剂(EDTA,SDS,CTAB),使其终浓度分别为1 mmol/L和2 mmol/L,以研究其对重组酶活性的影响。以未加上述金属离子和化学试剂的样品测得的酶活力记为100%,计算不同的金属离子或化学试剂对酶活性的影响程度。
1.2.9重组果胶酸裂解酶在苎麻脱胶中的应用 用不同的处理方法处理苎麻纤维并计算质量损失率。①化学法:准确称取苎麻1 g,在20 mL 0.5% NaOH中,100℃处理1 h。处理结束后,用去离子水冲洗干净,100℃烘干至恒重(w)。②酶处理法:准确称取苎麻1 g,在20 mL含有30 U/mL重组酶PELA的Gly-NaOH的缓冲液(pH 9.0)中,40℃处理4 h,每间隔20 min震荡1次。处理结束后,用去离子水冲洗干净后,100℃烘干至恒重(w)。以不含重组PELA的Gly-NaOH缓冲液pH 9.0作为对照。③酶-化学联合法:将经过酶处理的苎麻浸入20 mL 0.5% NaOH,100℃处理1 h,100℃烘干至恒重(w)。
质量损失率(%)=(1-w)/1×100%
2 结果与分析
2.1 果胶酸裂解酶pela基因序列分析
从宏基因组测序数据中,挖掘得到1个果胶酸裂解酶基因pela。该基因全长1 494 bp,编码497个氨基酸和1个终止密码子,预测前20个氨基酸为信号肽序列。Blast比对结果显示该酶DNA序列与Bacteroidesthetaiotaomicron来源果胶酸裂解酶基因(WP_011109109.1)的相似性达到99%,但该基因仍未进行功能鉴定。使用已知的果胶酸裂解酶氨基酸序列构建系统发育进化树(图1),发现这些基因可以分为3个簇(Cluster)。大部分已经验证功能的果胶酸裂解酶属于Cluster Ⅱ和Cluster Ⅲ,而PELA所属的Cluster Ⅰ中,仅有来源于Pseudoalteromonashaloplanktisstrain ANT/505的果胶酸裂解酶被克隆并进行了性质研究[6],其余绝大部分仅报道了基因序列,而未进行功能验证,说明Cluster Ⅰ中蛋白可能是一类新的未被开发利用的果胶酸裂解酶。
PELA蛋白的理论分子量为53.3 kDa,等电点(pI)为6.19,预测存在2个N-糖基化位点(124 NYTF,409 NYTY)和8个O-糖基化位点(33 T,37 S,52 T,53 T,405 T,437 S,440 T和442 T)(图2)。
2.2 重组果胶酸裂解酶PELA的表达和纯化
从100个阳性转化子中,筛选到1个高活力转化子。该重组菌株在3 L发酵罐水平表达果胶酸裂解酶PELA,甲醇诱导10 h后酶活力达到最高为10.8 U/mL,粗酶液经微滤和超滤浓缩后,经镍离子亲和层析纯化得到电泳纯蛋白,SDS-PAGE结果显示单一条带,其分子量大小约为60.0 kDa(图3泳道2,箭头所示),大于预测理论分子量53.3 kDa。经糖基化酶(Endo-H)处理后,可见其分子量仅略微变小(图3泳道3,箭头所示),可能原因是蛋白表达过程中PELA还经过了O-糖基化、脂酰化或者磷酸化等其他翻译后修饰。
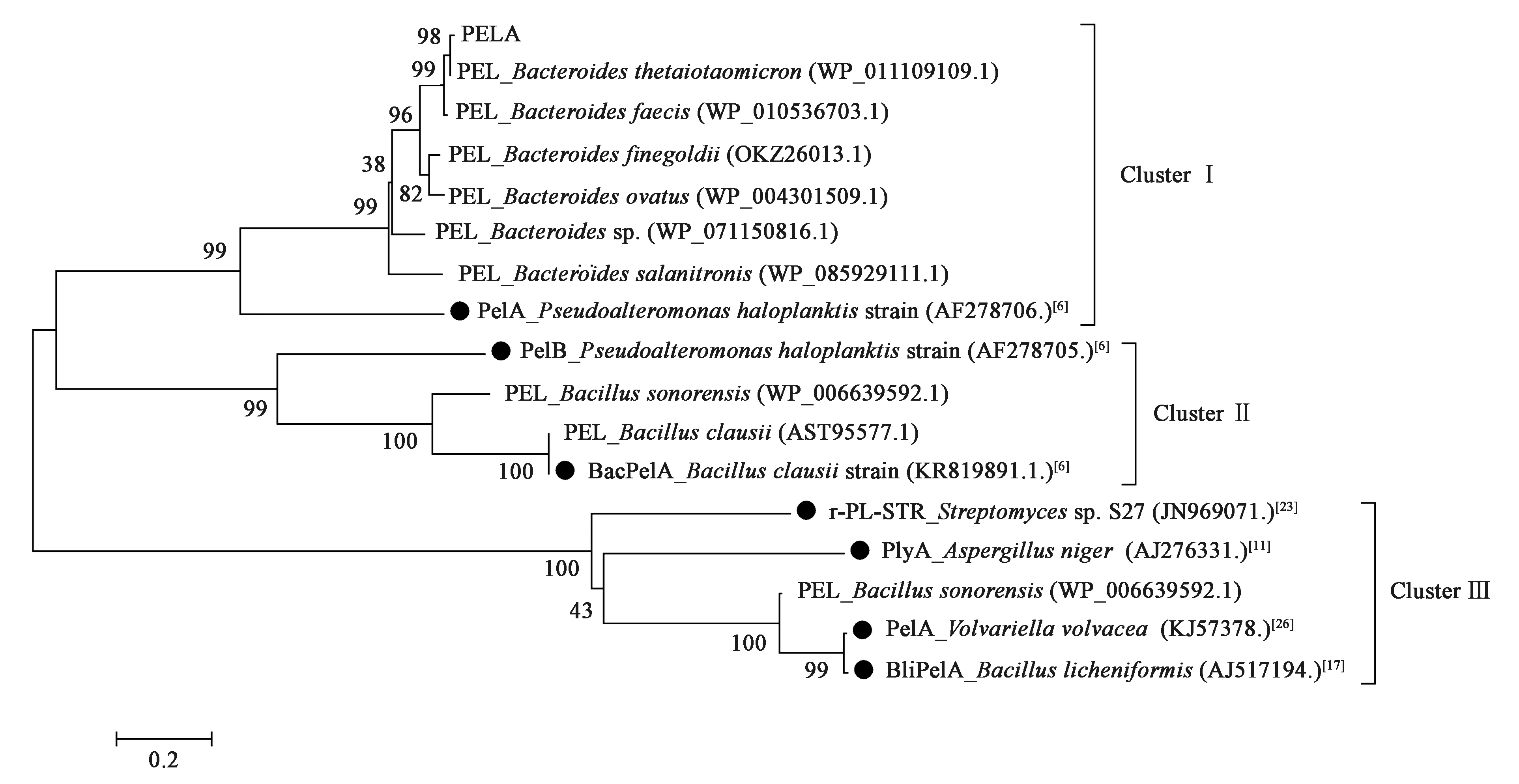
图1 果胶酸裂解酶进化树Fig.1 Phylogenetic trees of the pectate lyases.注:图中进化树分支所示数字为自举检验置信值(1 000个复制序列)。●表示已进行功能验证的果胶酸裂解酶。

图2 pela的DNA序列和推导的氨基酸序列Fig.2 The DNA sequence and deduced amino acid sequence of the pela.注:加下划线处为信号肽序列;星号为终止密码子;预测N-糖基化位点以方框突出显示;预测O-糖基化位点以灰色底纹突出显示。
2.3 重组果胶酸裂解酶PELA酶学性质测定
重组PELA的最适温度为45℃,高于50℃时酶活力急剧下降(图4A)。重组PELA在40℃时表现出良好的稳定性,在低于40℃条件下处理1 h,酶活力不变(图4C)。重组PELA的最适pH为9.0(图4B)。在pH 9.0~9.5范围内表现出较高活性,在低于pH 8.5或高于pH 9.5时酶活力急剧下降,在pH 8.5和pH 10.0时仅表现出40%的相对活力(图4B)。重组酶在碱性范围内稳定性较好,在pH 7.5~11.0条件下处理1 h,剩余酶活力在60%以上,当pH高于11.0时,酶变得极不稳定,在pH 12.0时,酶活力完全丧失(图4D)。
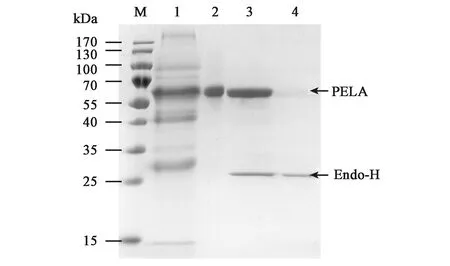
图3 重组果胶酸裂解酶PELA的SDS-PAGE分析Fig.3 SDS-PAGE analysis of the recombinant pectate lyase PELA.M:蛋白分子量Marker; 1:发酵粗酶液; 2:纯化重组果胶酸裂解酶PELA; 3:Endo-H处理的重组果胶酸裂解酶PELA; 4:Endo-H。
在最适条件下测得重组果胶酸裂解酶PELA的比活力为244.12 U/mg。在设定条件下,Stop-flow分析PELA酶催化一级反应时间为20 s,在一级反应时间内反应速率维持在1.96 μmol/s·mg左右(图5A)。在一级反应时间内,不同底物浓度下,PELA的Km值是0.26 mg/mL,最大反应速度Vmax值为488.40 μmol/min·mg(图5B)。
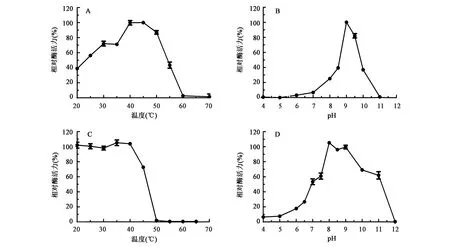
图4 温度和pH对重组PELA活力的影响Fig.4 Effect of temperature and pH on the recombinant PELA.A:温度对果胶酸裂解酶活力的影响;B:pH对果胶酸裂解酶活力的影响;C:果胶酸裂解酶温度稳定性;D:果胶酸裂解酶pH稳定性。
化学试剂EDTA及金属离子Ni2+、Co2+、Cu2+、Cd2+、Zn2+、Mn2+和Cu2+对PELA酶活力具有不同程度的抑制作用。其中Cd2+、Zn2+、Mn2+和Cu2+表现为对酶活性的强烈抑制(表1)。1 mmol/L SDS对酶活性没有明显的影响,当浓度达到2 mmol/L时,表现出明显的抑制作用,但是仍有70%以上活力。Mg2+、Li+、K+和Ca2+均能促进酶的活力。2 mmol/L的K+或1 mmol/L的Ca2+对酶活力具有明显的促进作用(表1),但2 mmol/L的Ca2+却表现出对酶活性的抑制。
2.4 重组果胶酸裂解酶PELA在苎麻脱胶中的应用
用PELA在40℃作用于苎麻4 h,苎麻纤维质量损失率达到9.2%,与单独使用0.5% NaOH效果相当,而对照质量损失率仅1.3%;当PELA和0.5% NaOH共同作用时,质量损失率达到了16.6%(图6A,彩图见图版一)。在苎麻形态变化上,用PELA处理苎麻4 h后,与对照相比,苎麻纤维分散度和白度得到明显地提高;当PELA和0.5% NaOH共同作用时,苎麻纤维的分散度和白度均优于单独使用PELA或者0.5% NaOH(图6B)。
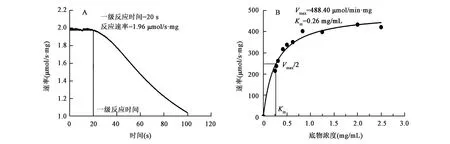
图5 PELA酶动力学测定Fig.5 Kinetic parameters of the recombinant PELA.A:PELA一级反应时间测定;B:PELA催化动力学参数测定。
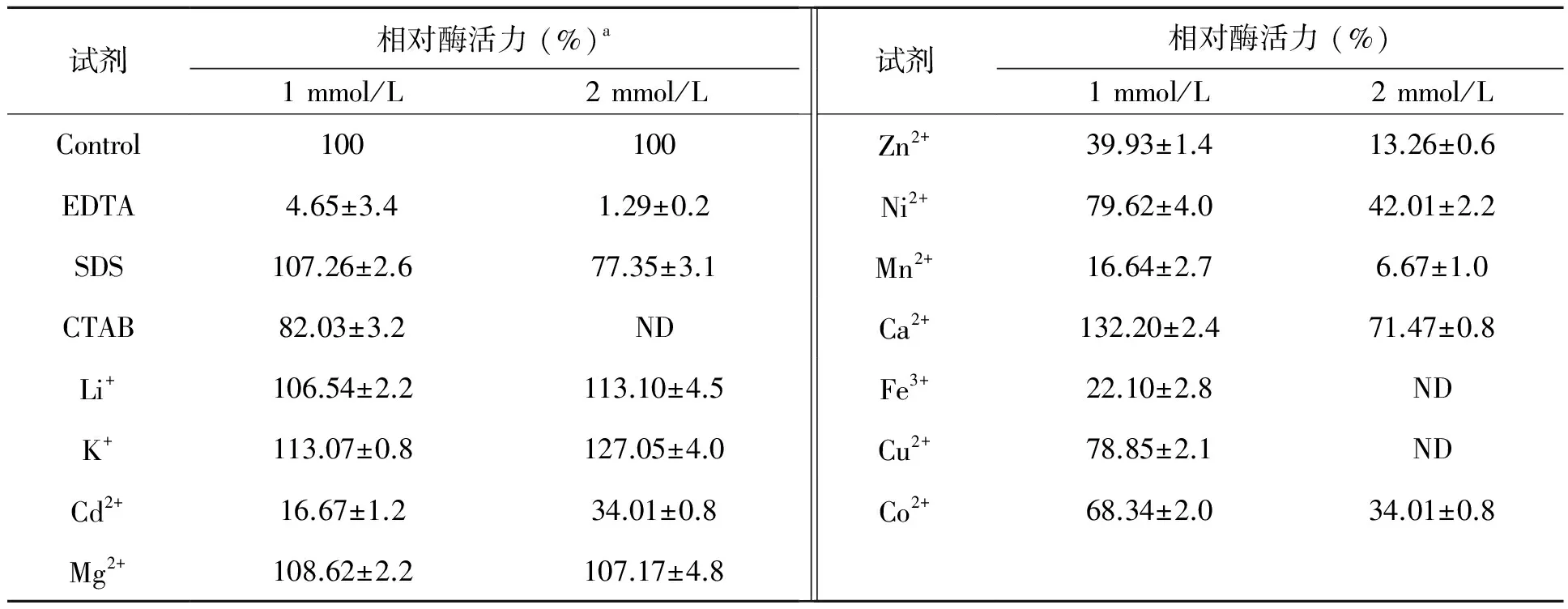
表1 不同化学试剂和金属离子对重组酶活性的影响Table 1 The effects of various chemical reagents and metal ions on the recombinant PELA activity.
注:a所得的值为3次所测酶活力的平均值。ND:无数据。当部分离子或化合物浓度为2 mmol/L时,在波长235 nm处不能测出其相应吸光值。
3 讨论
高通量宏基因组测序技术一次可以对上百万条DNA分子序列进行测定,具有成本低、数据量大的特点。海量的新基因从宏基因组测序数据库得到挖掘,如Roehe等[20]的研究中通过宏基因组测序揭示的与甲烷排放相关的2 700多个基因中,只有0.6%在NCBI的蛋白质数据库中能够匹配。Venter等[21]对马尾海藻的宏基因组测序发现了超过120万个未知新基因。本研究获得的pela基因经进化树分析,其与已经验证功能的果胶酸裂解酶属于不同的Cluster,可能是一类新的未被开发利用的果胶酸裂解酶,同时也说明宏基因组测序能够高效挖掘到新基因。另外,本研究通过宏基因组测序挖掘到多个果胶酸裂解酶基因并在P.pastoris中进行了异源表达,但仅有pela异源表达产物显示出了果胶酸裂解酶活性。P.pastoris并不是所有蛋白的最佳表达宿主,对于挖掘到的大量的果胶酸裂解酶基因,后期将尝试在不同的宿主中进行异源表达。
pela在P.pastoris中的表达量为10.8 U/mL,低于大多数已报道的果胶酸裂解酶,如Aspergillusnidulans和Paenibacillussp. 0602来源的果胶酸裂解酶基因在E.coli中的表达量分别为360.0 U/mL[10]和2 467.4 U/mL[22]。为了降低酶制剂的生产成本,提高PELA的表达量,我们将进一步在表达系统选择、发酵条件优化以及基因序列的优化上开展后续研究。但值得指出的是,PELA的Vmax为488.40 μmol/min·mg,Km为0.26 mg/mL,几乎低于所有已报道的果胶酸裂解酶的Km值,如来源于Bacilluslicheniformis、Aspergillusniger和Streptomycessp. S27的Km分别为0.38 mg/mL[17]、1.16 mg/mL[11]和7.90 mg/mL[23]。PELA的最大反应速度Vmax也高于大多数果胶酸裂解酶的数值[11,22,24,25],说明PELA具有很高的催化效率。另外,本研究中的PELA具有较高比活力(244.12 U/mg),高于大多数已报道果胶酸裂解酶,如来源于Streptomycessp. S27(23.00 U/mg[23])和Bacilluslicheniformis14A(45.40 U/mg[26])的果胶酸裂解酶。
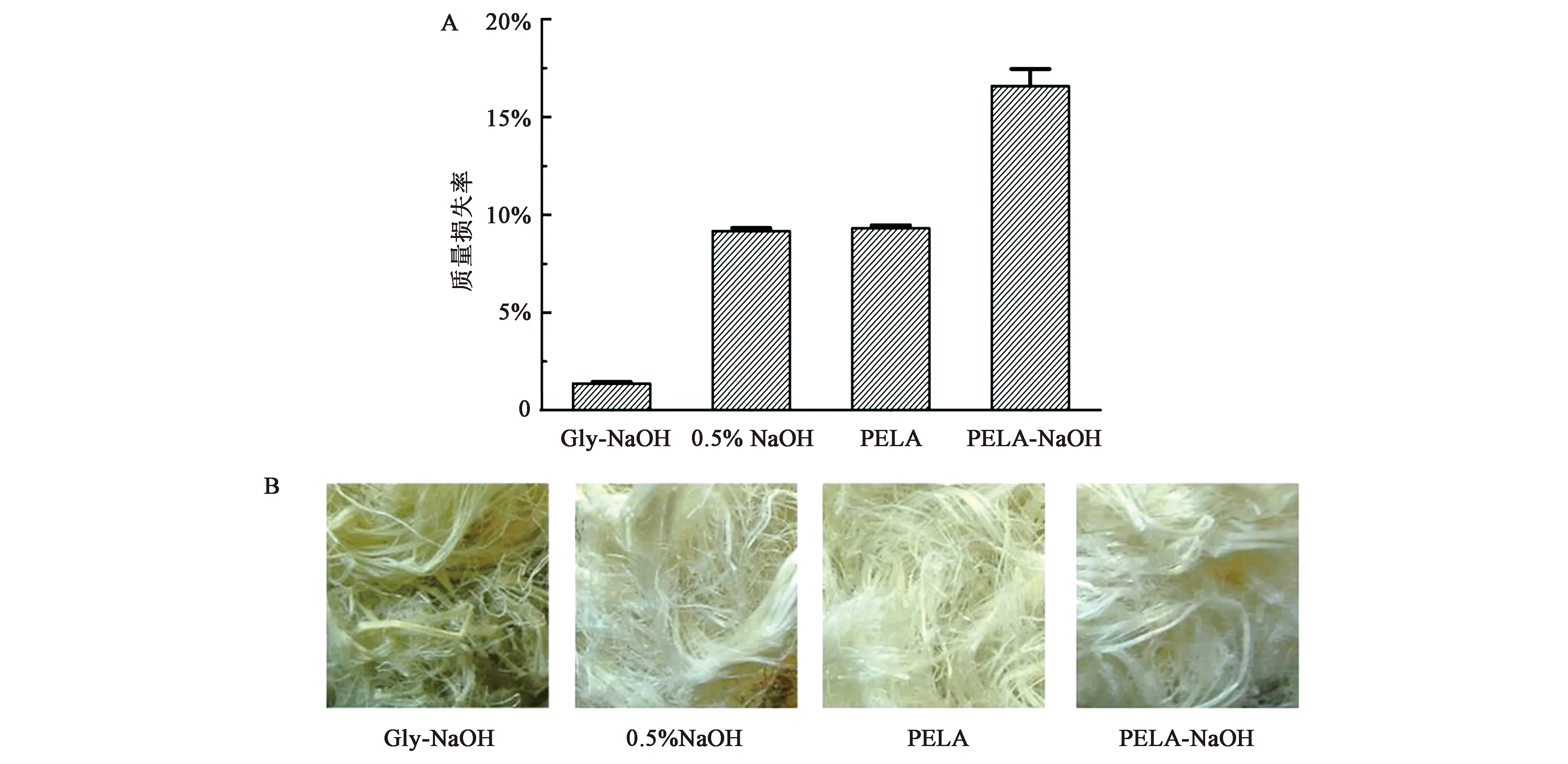
图6 不同处理后苎麻纤维质量损失率(A)及形态变化(B)Fig.6 Percentage of ramie fiber weight loss (A) and morphological change (B) after different treatments.(彩图见图版一)
在纺织工业中应用果胶酶往往要兼顾催化效率和稳定性。通过对纯化的PELA进行酶学性质研究,结果表明PELA在最适反应条件下的稳定性很好,能够兼顾催化效率与稳定性。PELA最适温度为45℃,在40℃具有良好的稳定性,处理1 h酶活力仍有100%的活性,优于已报道的来源于Paenibacillussp.的PelN,PelN在最适温度下处理1 h,剩余酶活力不足25%[22]。来源于Bacillussubtilis的PL,其在最适温度下处理20 min,剩余酶活力不足40%[27]。PELA的最适pH为9.0,在最适pH条件下处理1 h,剩余90%以上的酶活力。
目前报道的大多数适用于纺织行业的果胶酸裂解酶多为高温酶,酶的最适温度在50~70℃范围内的较多,如BacillustequilensisSV11来源的果胶酸裂解酶的最适温度为60℃[28],BacilluspumilusBK2来源的PL在70℃具有最高酶活性[29]。这些酶在实际工业应用中可能仍然存在高耗能的问题,更重要的是对纤维质量造成影响。PELA的最适温度为45℃,并且PELA对纤维素没有催化活性,不会降低纤维质量。因此PELA更适合应用于纺织脱胶过程中。这些结果表明PELA在苎麻脱胶中具有良好的应用潜力。
参 考 文 献
[1] Zhang J, Henriksson G, Johansson G. Polygalacturonase is the key component in enzymatic retting of flax [J]. J. Biotechnol., 2000, 81(1):85-89.
[2] Kashyap D, Vohra P, Chopra S,etal.. Applications of pectinases in the commercial sector: A review [J]. Bioresour. Technol., 2001, 77(3):215-227.
[3] 刘惠娟, 陈 晟, 华兆哲, 等. 纺织清洁生产用碱性果胶酶的研究进展 [A]. 见: 第七届功能性纺织品及纳米技术应用研讨会[C].浙江杭州,2007,4.
[4] 董云舟, 赵 政, 堵国成, 等. 碱性果胶酶及其在棉纺织预处理中的应用 [J]. 工业微生物, 2004(02):30-34.
[5] Sharma N, Rathore M, Sharma M. Microbial pectinase: sources, characterization and applications [J]. Rev. Environ. Sci. Biotechnol., 2013, 12(1):45-60.
[6] Van T L, Tuyen H, Helmke E,etal.. Cloning of two pectate lyase genes from the marine antarctic bacteriumPseudoalteromonashaloplanktisstrain ANT/505 and characterization of the enzymes [J]. Extremophiles, 2001, 5(1):35-44.
[7] Kobayashi T, Hatada Y, Suzumatsu A,etal.. Highly alkaline pectate lyase Pel-4A from alkaliphilicBacillussp. strain P-4-N: Its catalytic properties and deduced amino acid sequence [J]. Extremophiles, 2000, 4:377-383.
[8] Liu Y, Chen G, Wang J,etal.. Efficient expression of an alkaline pectate lyase gene fromBacillussubtilisand the characterization of the recombinant protein [J]. Biotechnol. Lett., 2011, 34(1):109-115.
[9] Zhou C, Xue Y, Ma Y. Cloning, evaluation, and high-level expression of a thermo-alkaline pectate lyase from alkaliphilicBacillusclausiiwith potential in ramie degumming [J]. Appl. Microbiol. Biot., 2017, 101(9):3663-3676.
[10] Zhao Q, Yuan S, Zhang Y,etal.. Expression, purification and characterization of pectate lyase A fromAspergillusnidulansinEscherichiacoli[J]. World J. Microb. Biotech., 2006, 23(8):1057-1064.
[11] Jacques A E, Benen H C M K, Lucie P,etal.. Characterization ofAspergillusnigerpectate lyase A [J]. Biochemistry-us, 2000, 39(50):15563-15569.
[12] Payasi A, Misra P C, Sanwal G G. Purification and characterization of pectate lyase from banana (Musaacuminata) fruits [J]. Phytochemistry, 2006, 67(9):861-869.
[13] 彭 焕, 彭德良, 黄文坤, 等. 大豆孢囊线虫果胶酸裂解酶基因Hg-pel-5的克隆与分析 [J]. 中国农业科学, 2012(05):854-866.
[14] 李 新, 顾晓川, 龙海波, 等. 禾谷孢囊线虫果胶酸裂解酶新基因Ha-pel-1的鉴定与表达特征分析 [J]. 中国农业科学, 2017(19):3723-3732.
[15] 龙海波, 孙燕芳, 曾凡云, 等. 象耳豆根结线虫果胶酸裂解酶新基因Me-pel2的鉴定及发育表达分析 [J]. 热带作物学报, 2016(01):92-98.
[16] Brady S F. Construction of soil environmental DNA cosmid libraries and screening for clones that produce biologically active small molecules [J]. Nat. Protoc., 2007, 2(5): 1297-1305.
[17] Zhou C, Xue Y, Ma Y. Characterization and overproduction of a thermo-alkaline pectate lyase from alkaliphilicBacilluslicheniformiswith potential in ramie degumming [J]. Proc. Biochem., 2017, 54:49-58.
[18] Yadav S, Dubey A K, Anand G,etal.. Purification and biochemical characterization of an alkaline pectin lyase fromFusariumdecemcellulareMTCC 2079 suitable forCrotolariajunceafiber retting [J]. J. Basic. Microb., 2014, 54:S161-S169.
[19] Yadav S, Yadav P K, Yadav D,etal.. Purification and characterization of an alkaline pectin lyase fromAspergillusflavus[J]. Proc. Biochem., 2008, 43(5):547-552.
[20] Roehe R, Dewhurst R J, Duthie C A,etal.. Bovine host genetic variation influences rumen microbial methane production with best selection criterion for low methane emitting and efficiently feed converting hosts based on metagenomic gene abundance [J]. PLoS Genet., 2016, 12(2):1-20.
[21] Venter J C, Remington K, Heidelberg J F,etal.. Environmental genome shotgun sequencing of theSargassosea[J]. Science, 2004, 304(5667):66-74.
[22] Li X, Wang H, Zhou C,etal.. Cloning, expression and characterization of a pectate lyase fromPaenibacillussp. 0602 in recombinantEscherichiacoli[J]. BMC Biotechnol., 2014, 14(1):1-12.
[23] Yuan P, Meng K, Shi P,etal.. An alkaline-active and alkali-stable pectate lyase fromStreptomycessp. S27 with potential in textile industry [J]. J. Ind. Microbiol. Biotechnol., 2012, 39(6):909-915.
[24] Su H, Qiu W, Kong Q,etal.. Thermostable pectate lyase fromCaldicellulosiruptorkronotskyensisprovides an efficient addition for plant biomass deconstruction [J]. J. Mol. Catal B-enzym., 2015, 121:104-112.
[25] Shi A, Hu H, Zheng F,etal.. Biochemical characteristics of an alkaline pectate lyase PelA fromVolvariellavolvacea: roles of the highly conservedN-glycosylation site in its secretion and activity [J]. Appl. Microbiol. Biotechnol., 2015, 99(8):3447-3458.
[26] Berensmeier S, Singh S A, Meens J,etal.. Cloning of the pelA gene fromBacilluslicheniformis14A and biochemical characterization of recombinant, thermostable, high-alkaline pectate lyase [J]. Appl. Microbiol. Biotechnol., 2004, 64(4):560-567.
[27] Zhuge B, Du G C, Shen W,etal.. Expression of aBacillussubtilispectate lyase gene inPichiapastoris[J]. Biochem. Eng. J., 2008, 40(1):92-98.
[28] Chiliveri S R, Linga V R. A novel thermostable, alkaline pectate lyase fromBacillustequilensisSV11 with potential in textile industry [J]. Carbohydr. Polym., 2014, 111:264-272.
[29] Klug-Santner B G, Schnitzhofer W, Vršanská M,etal.. Purification and characterization of a new bioscouring pectate lyase fromBacilluspumilusBK2 [J]. J. Biotechnol., 2006, 121(3):390-401.

