美国近20年主要食源性致病菌的分布及耐药性分析
——对我国细菌耐药性监控工作的启示
潘航,李肖梁,方维焕,乐敏
(浙江大学动物科学学院,浙江省动物预防医学重点实验室,杭州310058)
进入21世纪以来,食源性疾病已成为影响公众健康的重要因素,其中病原微生物引起的食源性疾病比例达50%以上[1],世界卫生组织估测这一比例为70%[2]。抗生素在临床医疗及动物养殖领域广泛和大量使用,导致耐药性问题日益严重,成为全球最为重要的公共卫生问题之一[1-3]。从产超广谱β-内酰胺酶的肠杆菌和耐甲氧西林金黄色葡萄球菌的流行,到耐碳青霉烯类肠杆菌的涌现,再到mcr基因携带菌株的出现,不断压缩着人类可使用的抗菌药物的范围。我国食源性病原污染及其引发的疾病和耐药率近年来呈不断上升态势,已成为现代医学的重要难题。
据估计,2010年全球在牛、鸡和猪中抗生素的平均用量分别是45、148和172 mg/kg,到2030年,全球的抗菌药物消费总量将在此基础上增加67%[4]。我国作为发展中国家,由于人口因素和营养结构上对肉类食品的客观需求,因此近20年来抗菌药的使用量(尤其是在兽医领域)呈上升趋势,且总量巨大。据报道,2010年我国食品动物中抗菌药物使用比例已达全球总额的23%,位列世界第一,远超第二位的美国(13%)[4]。2013年我国抗菌药物使用约16.2万t,其中52%用于动物[5]。2017年上海疾病预防与控制中心监测的动物源数据表明,鸡源和猪源分离株的耐药率可达20%~96%,其中复方新诺明和四环素的耐药率分别达84%和93%。在食品动物养殖过程中抗菌药物滥用导致食源性细菌耐药率不断上升;同时,由于食源性病原菌能够通过食物链传播而感染人体,所以抗菌药物滥用也增加了耐药基因的散播风险。为此,农业部近年来逐步限制饲料中部分抗生素作为促生长剂的使用,并出台了一系列政策,如全国遏制动物源细菌耐药行动计划(2017—2020年)。但由于监测体系不完善,监测数据仍不完整,所以目前采取的限用措施对降低耐药细菌,特别是降低人体感染耐药细菌的实际效果还无法评价。
面对如此庞大的抗生素使用量和不断上升的耐药率,全国范围内系统而完善的耐药性细菌监测系统不可或缺。目前,我国并没有对食品产业链中食源性病原菌及其耐药情况进行系统、全面的官方监测,相关研究报道也很少。美国肠道细菌耐抗生素监测系统(National Antimicrobial Resistance Monitoring System for Enteric Bacteria,NARMS)从20世纪90年代中期开始,系统地监测了美国本土动物性食品(肉类为主)、食品动物及人类来源分离株的耐药情况[6]。为了较系统地评价抗生素耐药性监测平台的成果及优势,本研究对美国NARMS在20余年间的监测数据成果进行分析,并与我国现有权威数据进行比较,力求为我国开展从农场到餐桌的食源性病原及其耐药性监测与控制提供理论参考和有益借鉴。
1 材料与方法
统计菌株来源于1996—2016年间美国NARMS系统所监测统计的190 365株分离株[7],包括5种常见食源性微生物菌属的至少8个菌种:沙门菌属(包括肠道沙门菌Salmonella enterica),弯曲杆菌属(主要包括空肠弯曲菌Campylobacter jejuni,结肠弯曲菌Campylobacter coli),埃希菌属(包括大肠埃希菌Escherichia coli),志贺菌属(Shigella),肠球菌属(主要包括粪肠球菌Enterococcus faecalis,屎肠球菌Enterococcus faecium,海氏肠球菌Enterococcus hirae)。来自美国10大区域的14个州(图1)的屠宰场、食品店及医院,其样品来源主要包括畜禽(宰后胴体、盲肠内容物)、屠宰厂肉样与零售肉类、病人。参照美国临床和实验室标准化协会(Clinical and Laboratory Standards Institute,CLSI)推荐的药敏试验方法测定44种常用抗生素对细菌的最小抑菌浓度(minimum inhibitory concentration,MIC),并以此为基础分析耐药情况。
图1 美国NARMS所监测的10大区域中的14个州示意图Fig.1 Map of 14 U.S.states in 10 geographical regions monitored by NARMS
2016年我国人源分离株耐药数据来源于中国细菌耐药监测网(China Antimicrobial Resistance Surveillance Network,CHINET)[8]。参考我国主要食源性致病菌及NARMS监测菌株的菌属,整理出共55 554个分离株数据,包括大肠埃希菌、金黄色葡萄球菌、肠球菌属细菌(屎肠球菌和粪肠球菌)、沙门菌、志贺菌。样品来自我国30家医院的住院患者(86.6%)和门诊(急诊)患者(13.4%),样品类型主要包括呼吸道标本、尿液、血液、伤口脓液、粪便等,参照2016年新版美国CLSI推荐的方法测定32种常用抗生素的MIC值。
2000—2015年间美国及中国抗生素用量数据来源于艾美仕市场研究公司(IMSHealth)[9]。
2 结果与分析
2.1 美国NARMS监测数据分析
1996—2016年间NARMS监测菌株来源与种属分布情况如图2所示。

图2 1996—2016年间NARMS监测菌株来源与种属分布Fig.2 Sources and genus distributions of the bacterial strains monitored by NARMSfrom 1996 to 2016
2.1.1 宿主构成比及代表性
由图2A可知:在近19万个菌株中,约29%来源于病人,绝大部分来自于动物源(71%);在动物源菌株中,13%来源于牛(健康和带病动物牛,以及市售牛肉,下同),41%来源于鸡,8%来源于猪,9%来源于火鸡。而分离自肉类的菌株数占动物源菌株数的37%(图2B)。监测区域内各类宿主比例约为人∶牛∶鸡∶猪∶火鸡=10∶1.2∶252∶3.7∶7.7,而监测获得菌株的宿主来源比例为10∶4.5∶14.1∶2.8∶3.1,基本能代表监测区域内的情况。
2.1.2 各菌属比例
由图2C可知,监测的食源性病原菌涉及5个属,按比例从大到小排列依次为沙门菌属(40%)、肠球菌属(20%)、埃希菌属(20%)、弯曲杆菌属(17%)和志贺菌属(3%)。
2.1.3 各来源样品中的主要菌属
动物样本中检出率最高的病原是沙门菌(45%);畜禽肉样品中检出率最高的是肠球菌属(42%),其中粪链球菌占64%;病人样本中检出率最高的也是沙门菌(66%)。
2.1.4 用药量走势
从图3A可以看出:广谱青霉素用量最大,但总体呈下降趋势,2000—2015年间减少近25%;头孢菌素、四环素和大环内酯类使用量为每1 000人约2 000~5 000标准单位,这些药物的使用被有计划地控制,用量逐年稳步减少;甲氧苄啶和氟喹诺酮类的用量没有明显变化,维持在每1 000人约2 000标准单位的水平;其他药物用量水平极低或处于禁用状态。
2.1.5 NARMS分离株耐药情况
将目前44种抗生素分为影响细胞壁或胞外被膜、影响DNA合成和影响蛋白质合成3大类(图3B)。其中:影响蛋白合成类的耐药情况最为严重,尤其是林可酰胺(clindamycin,CLI)和链阳菌素(奎奴普汀-达福普汀)(quinupristin-dalfopristin,QDA),耐药率接近90%;在氨基糖苷类和四环素类中,除阿米卡星(amikacin,AMI)和安普霉素(apramycin,APR)外,其余的耐药率在15%~50%之间;在大环内酯类中红霉素(erythromycin,ERY)和泰乐菌素(tylosin,TYL)的耐药率也高于40%。
在影响细胞壁或胞外被膜大类的抗生素中,耐药情况也较为严重,尤其是头孢菌素类、单环β-内酰胺类和广谱青霉素类,耐药率可达40%~90%[达托霉素(daptomycin,DAP)、盘尼西林(penicillin,PEN)、头孢西丁(cefoxitin,FOX)、头孢曲松(ceftriaxone,AXO)、头孢噻呋(ceftifur,TIO)、头孢吡肟(cefepime,FEP)除外];杆菌肽(bacitracin,BAC)的耐药率高达92%。
在影响DNA合成大类中,除复方新诺明(COT)的耐药率在10%~20%的可接受范围内,其余叶酸拮抗物类抗生素和硝基呋喃(nitrofurantoin,NIT)的耐药率都超过20%的可接受上限,因此不推荐继续使用;而氟喹诺酮类药物中并未发现高程度的耐药。
联系用药趋势图(图3A)及耐药情况(图3B)可以发现,常用的广谱青霉素类、头孢菌素、四环素、大环内酯类、叶酸拮抗物类抗生素(主要指含甲氧苄啶的复方新诺明)及氟喹诺酮类药物都呈现不同程度的耐药,而用量较少的如碳青霉烯类、恶唑烷酮类等几乎没有出现耐药。但也有例外,如氨基糖苷类(除AMI和APR外)和单环β-内酰胺类药物(除DAP外)的用量并不高,但耐药率也较高。综合用药趋势可以发现,四环素类、大环内酯类及头孢菌素类的用量被控制而逐年下降,说明系统的耐药率监测可指导用药政策调整,以控制耐药性的发生和发展。
2.1.6 沙门菌分离株耐药动态
以检出率最高的沙门菌为对象,分析其在21年间耐药谱的动态变化。结果(图4)发现,沙门菌中较高水平耐药(耐药率≥20%)的抗生素有头孢他啶(CAZ)、头孢噻肟(CTX)、头孢噻肟-克拉维酸(CTC)、头孢他啶-克拉维酸(CCV)、头孢喹肟(CEQ)、氨曲南(ATM)、哌拉西林-他唑巴坦(PTZ)和四环素(TET),应对这些药物的使用加以限制。耐药率在安全范围内(<10%)的理想抗生素主要有亚胺培南(IMI)、头孢西丁(FOX)、头孢曲松(AXO)、头孢噻呋(TIO)、复方新诺明(COT)、环丙沙星(CIP)、阿米卡星(AMI)、庆大霉素(GEN)、氯霉素(CHL)和阿奇霉素(AZM),推荐使用。而头孢吡肟(FEP)和萘啶酸(NAL)因近年来出现较高耐药水平应当谨慎使用,尤其是萘啶酸,耐药率呈逐年升高趋势。
2.2 我国抗生素用量及耐药性
2.2.1 抗生素用量趋势
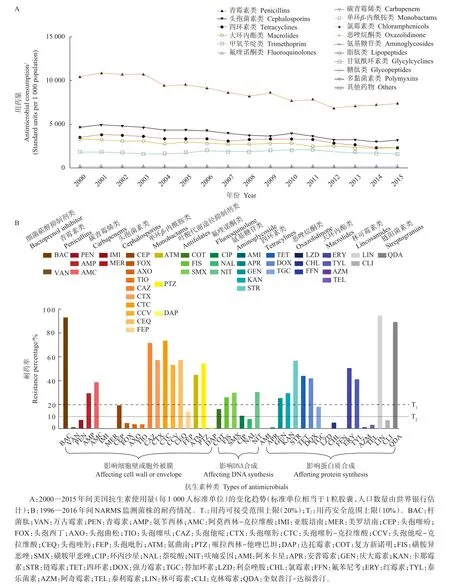
图3 美国主要抗生素使用量及耐药性Fig.3 Consumption and resistanceof major antimicrobialsin the United States
从2000—2015年间的使用趋势(图5A)看,我国抗生素用量总体呈上升趋势,尤其是头孢菌素类、广谱青霉素类、氟喹诺酮类及大环内酯类的用量明显偏高。虽然没有确切的监测数据显示抗生素在动物中的使用比例,但这些药物是畜禽养殖业中使用最多的几类抗生素。此外,从2012年起上述这些用量偏高的抗生素使用量不再增长,提示政策性控制可能显示成效。
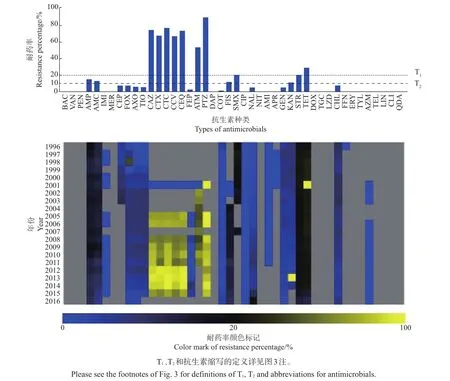
图4 1996—2016年NARMS监测的沙门菌耐药性的变化趋势Fig.4 Dynamicsof Salmonella resistanceto major antimicrobialsmonitored by NARMSfrom 1996 to 2016
2.2.2 2016年人源分离株菌属比例
参考我国主要食源性致病菌及NARMS监测菌株的菌属,2016年人源分离株在5个属中按所占比例从大到小排列依次为埃希菌属(53.6%,皆为大肠埃希菌)、葡萄球菌属(23.8%,皆为金黄色葡萄球菌)、肠球菌属(21.4%,其中粪肠球菌占45.5%,屎肠球菌占54.5%)、沙门菌属(1.1%)和志贺菌属(0.1%)。
2.2.3 2016年人源分离株耐药情况
图5B显示人源临床分离株的耐药情况以及与抗生素使用量的可能关系。在影响细菌细胞壁合成大类中,用量最大的头孢菌素类和广谱青霉素类的耐药情况严重(分别为12.7%~60.1%和39.7%~94.5%),而用药量较少的阿莫西林-克拉维酸(AMC)的耐药率也达到11.6%。在影响DNA合成大类中,用量较大的氟喹诺酮类的耐药率较高,如环丙沙星(CIP)为49.4%,左氧氟沙星(LVX)为43.3%,而较少使用的复方新诺明(COT)的耐药率也高达42.9%。在影响蛋白质合成大类药物中,大环内酯类耐药率较高,如红霉素(ERY)为72.1%;用量较少的氨基糖苷类中的庆大霉素(GEN)、利福平(RIF)和恶唑烷酮类中的氯霉素(CHL)耐药率分别为38.4%、40.1%和14.6%。
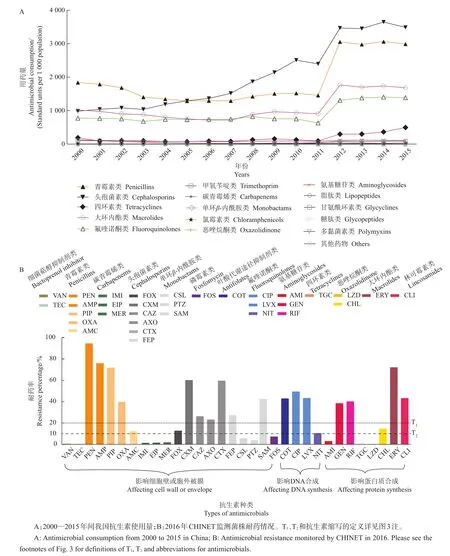
图5 我国抗生素用量及人源分离菌耐药性Fig.5 Consumption of antimicrobials and antimicrobial resistance of human isolates in China
在未知使用量的药物中,林可霉素类的克林霉素(CLI)耐药率达43.2%。此外,较高用量的四环素类药物中仅监测了替加环素(TGC)(耐药率0.4%),缺少四环素、土霉素等其他药物的耐药性数据,无法推测用药量和耐药率之间的关系。由于缺少畜产品从农场到餐桌阶段分离菌株的耐药数据,因此无法分析人源耐药菌是否与动物源耐药菌存在关联。
2.3 中美两国2015年抗生素使用量比较
对比中美两国抗生素使用量(图6)可以发现,我国人均使用量远小于美国(2015年用量比约为3∶5),但考虑到人口因素,在总使用量上中国应接近美国的2倍。
从主要用药种类上看,两国都大量使用了广谱青霉素及头孢菌素类药物,但美国更侧重于使用广谱青霉素类,而我国侧重于使用头孢菌素类;氟喹诺酮类、大环内酯类、氨基糖苷类药物也被两国较多使用;另外,美国使用了一定量的四环素类药物和甲氧苄啶,以及少量窄谱青霉素类和糖肽类药物,而我国仅使用少量四环素类药物。
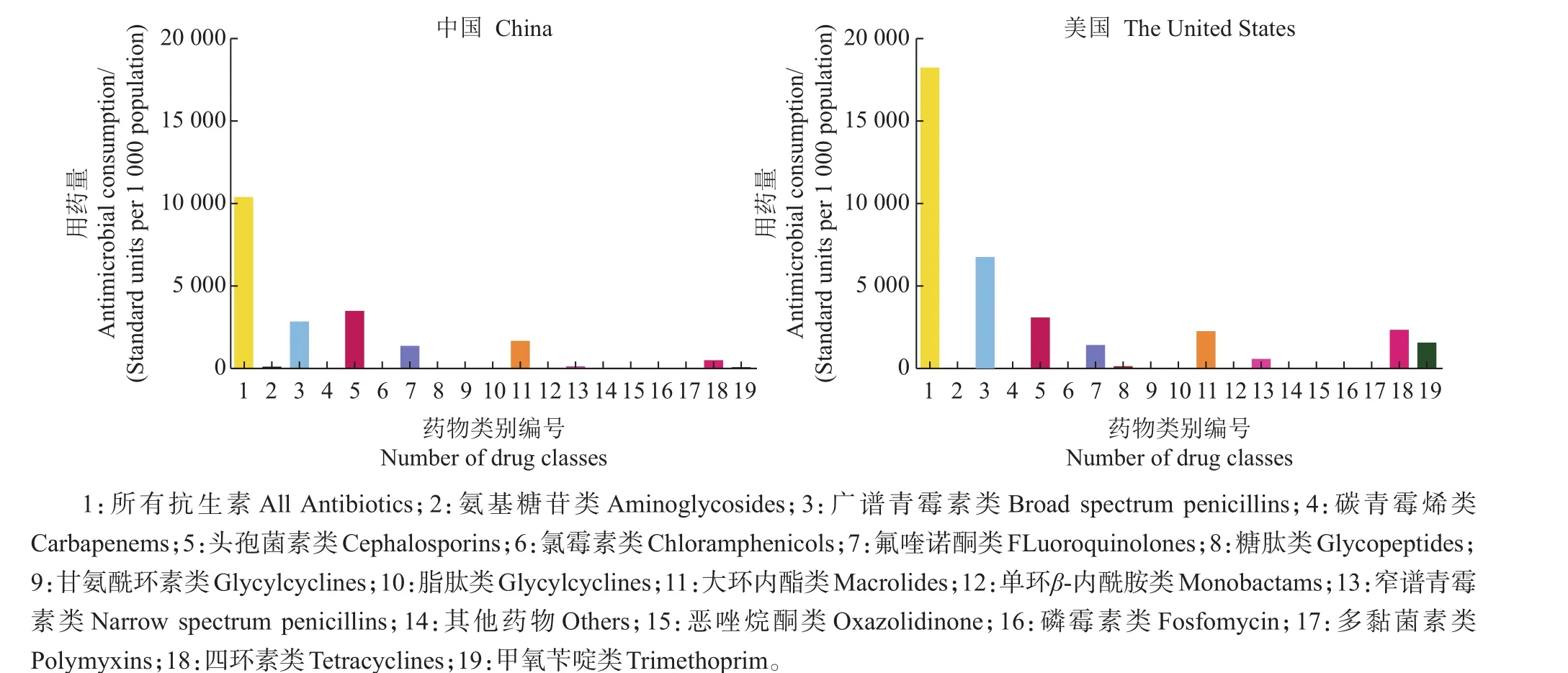
图6 2015年中美两国抗生素使用量比较Fig.6 Comparison of antimicrobial consumption between Chinaand the United Statesin 2015
3 讨论
食物全产业链一般包含环境、饲料、养殖、屠宰、加工、销售和消费7大环节。食品动物养殖业广泛使用甚至滥用抗生素是导致细菌耐药性问题日趋严重的重要原因[4],由养殖环节(尤其是畜禽养殖场和水产养殖系统)蔓延的耐药菌及耐药基因转移对整个产业链、环境生态和人群有着巨大影响。此外,在屠宰及加工过程中理化因子如含氯类化合物、醇类消毒剂、紫外照射也能够诱导病原菌的耐药性形成[10-11]。有研究显示,亚抑菌浓度的酸类物质或冷藏处理可以显著提高单核细胞增生李斯特菌敏感株对氨基糖苷、四环素、青霉素等的耐药性[12]。另外,细菌耐药性可通过食物链、人畜直接接触或环境散播等途径实现耐药基因的水平转移。对人体而言,耐药菌株及其耐药基因可能通过空气、水和食物链等多种途径侵入肠道菌群而导致耐药基因增多[13-14]。在这些途径中,食源性病原菌既是耐药基因的接受者,也是耐药基因的天然携带者或散播者,其中,肠道或环境中的微生物菌群成为多重耐药菌产生、形成的重要“温床”。
美国肠道细菌耐抗生素监测系统(NARMS)是美国疾病预防控制中心(Centers for Disease Control and Prevention,CDC)、食品药品监督管理局(Food and Drug Administration,FDA)和美国农业部(United States Department of Agriculture,USDA)合作承担的监测项目,主要监控动物性食品(肉类为主)、养殖动物及人类来源菌株的耐药情况,通过系统地比较耐药细菌的种类、耐药状况、不同物种间分布等信息,为有关政府部门制定合理使用抗生素的决策提供重要依据[6]。本研究发现,沙门菌和肠球菌属细菌在美国的人、食品动物以及食品动物产品中的检出率较高,空肠弯曲菌引发的食源性疾病和耐药性近年来增长最快,这些细菌应作为主要的监测和控制对象。另外,部分抗生素使用过多可能是导致食源性病原菌高度耐药的主要因素。因此,持续动态的耐药监测数据能在有效指导合理用药中发挥关键作用。而在我国,仅从医学领域的数据也能发现抗生素使用与耐药性之间的联系。但由于对食源性病原在食品动物、食品动物产品以及在食物链中的传播缺乏系统研究,也没有相应的监测数据积累,加之中美两国在不同种抗生素的使用频率和使用量上各异,因此,两国间的总体耐药性数据可比性不大。初步分析两国数据(图3和图5)可以发现:我国青霉素(PEN)和氨苄西林(AMP)耐药率都超过70%,但这2种药物在美国都不超过30%;我国环丙沙星(CIP)耐药率接近50%,而美国只有约10%;另外,我国哌拉西林-他唑巴坦(PTZ)耐药率不到5%,但美国高达50%以上。结合用药量来看,非主要使用的抗生素但出现较高水平耐药的有氨基糖苷类(中美皆有)和氯霉素(仅中国),因此应进一步探究其耐药率及耐药性产生机制。应指出的是,四环素类是我国主要使用的抗生素之一,且主要为兽用,应深入分析动物源菌株对四环素类的耐药性,以指导兽医临床合理使用,并进一步评估兽用四环素类抗生素使用对相应医源性耐药菌株产生的影响。
目前,我国对食品全产业链中耐药菌/耐药基因的监测包括2个区块:第一区块是农业部牵头的全国动物源细菌耐药性监测网络(覆盖22个省及自治区、4个直辖市);第二区块是卫生系统中的临床细菌耐药性监测网络,主要有全国细菌耐药性监测网、中国细菌耐药监测网和浙江大学医学院附属第一医院牵头的血源性感染细菌耐药监测网。总体来看,监测格局零散化、片段化,缺乏系统性和连贯性,并且很多环节(比如养殖场、屠宰厂、农贸市场、医院)的设施以及环境数据等方面存在缺口,特别是养殖环境和医院住院部,前者存在大量的抗生素及其他化学药物残留,可持续性提供选择压力富集耐药菌;而医院住院部往往是耐药菌长期滞留、交叉感染频繁发生的场所,对这2个重要场所的耐药菌进行分离和监测及相关研究应引起足够重视。
由于我国过去一段时期对养殖业使用抗生素没有进行十分严格的控制,因而动物源和人源细菌在耐药谱上的差异是否与美国的情况相似尚不得而知,耐药性传播机制也需要进一步研究。建议统筹中国疾病预防控制中心、国家食品安全风险评估中心和中国动物疫病预防控制中心的相关工作,定期协同监测国内临床分离株、动物及其产品中的菌型及其耐药情况,从而探明动物、动物源性食品、临床病人以及相关环境中耐药菌株的分布情况及同型菌株的潜在传播途径。根据美国NARMS的运作经验,若满足如下条件,将大大提高监测的成效:1)在动物源食品产业链各环节中能获得明确清晰的数据;2)有职能明确的管理机构负责监测数据的整合、分析及共享;3)各监测机构间定期交流;4)有清晰界定的监测对象(如大肠埃希菌、金黄色葡萄球菌、沙门菌、弯曲杆菌、副溶血弧菌、肠球菌等)[15];5)建立存放监测数据的专用数据库;6)每年定期的数据整合、分析及信息公开。总之,系统的基础监测工作对及时调整用药相关政策和法规有重要的指导意义,有望从源头上控制细菌耐药性的产生,并在食物链不同阶段或关键控制点上遏制耐药病原菌、耐药细菌、耐药基因的传播。兽医与人医携手紧密合作,为保护国民健康、公共卫生和食品安全作出重要贡献。
参考文献(References):
[1] 吴清平.食品微生物安全快速检测与高效控制技术研究.第147场中国工程科技论坛:轻工科技发展论坛.江苏无锡,2012-09-01.https://airitilibrary.com/Publication/alDetailedmesh?DocID=7753217.
WU Q P.Research on fast detection and efficient control technology towards food microorganism.The 147th Chinese Engineering Science and Technology Symposium:Development of light industrial technology.Wuxi,Jiangsu,2012-09-01.https://airitilibrary.com/Publication/alDetailedmesh?DocID=7753217.(in Chinese)
[2] World Health Organization.Antimicrobial resistance:Global report on surveillance2014.Geneva,Switzerland,2014.
[3]杜鹃,谢峻,郑颖城,等.《抗生素耐药:全球监测报告2014》解读与反思.华南国防医学杂志,2014(8):814-817.
DU J,XIE J,ZHENG Y C,et al.Interspersion and learning of antimicrobial resistance:Global report on surveillance 2014 from WHO.Military Medical Journal of South China,2014(8):814-817.(in Chinese with English abstract)
[4]VAN BOECKEL T P,BROWER C,GILBERT M,et al.Global trends in antimicrobial use in food animals.Proceedings of the National Academy of Science of the USA,2015,112(18):5649-5654.
[5]ZHANG Q Q,YING G G,PAN C G,et al.Comprehensive evaluation of antibiotics emission and fate in the river basins of China:Source analysis,multimedia modeling,and linkage to bacterial resistance.Environmental Science&Technology,2015,49(11):6772-6782.
[6]KARP B E,TATE H,PLUMBLEE J R,et al.National antimicrobial resistance monitoring system:Two decades of advancing public health through integrated surveillance of antimicrobial resistance.Foodborne Pathogen and Disease,2017,14(10):545-557.
[7] U.S.Food and Drug Administration.NARMSNow:Integrated Data.1996—2016.https://www.fda.gov/AnimalVeterinary/SafetyHealth/AntimicrobialResistance/NationalAntimicrobial ResistanceMonitoringSystem/ucm570685.htm.
[8] 胡付品,郭燕,朱德妹,等.2016年中国CHINET细菌耐药性监测.中国感染与化疗杂志,2017,17(5):481-491.
HU F P,GUO Y,ZHU D M,et al.CHINET surveillance of bacterial resistance across China:Report of the results in 2016.Chinese Journal of Infection and Chemotherapy,2017,17(5):481-491.(in Chinesewith English abstract)
[9] IMS Health and Quintiles.Data of antibiotic use.ResistanceMap,2000—2015.https://resistancemap.cddep.org/AntibioticUse.php.
[10]ALONSO-HEMANDO A,CAPITA R,PRIETO M,et al.Comparison of antibiotic resistance patterns in Listeria monocytogenes and Salmonella enterica strains pre-exposed and exposed to poultry decontaminants.Food Control,2009,20(12):1108-1111.
[11]MOLINA-GONZÁLEZD,ALONSO-CALLEJA C,ALONSOHEMANDO A,et al.Effect of sub-lethal concentrations of biocides on the susceptiblity to antibiotics of multi-drug resistant Salmonella enterica strains.Food Control,2014,40:329-334.
[12]AL-NABULSI A A,OSALI T M,SHAKER R R,et al.Effects of osmotic pressure,acid,or cold stresses on antibiotic susceptibility of Listeria monocytogenes.Food Microbiology,2015,46:154-160.
[13]PAN H,PAUDYAL N,LIX L,et al.Multiple food-animalborne route in transmission of antibiotic-resistant Salmonella newport to humans.Frontiersin Microbiology,2018,9:23.
[14]GIBSON M K,CROFTST S,DANTASG.Antibiotics and the developing infant gut microbiota and resistome.Current Opinion Microbiology,2015,27:51-56.
[15]PAUDYAL N,PAN H,LIAO X Y,et al.A meta-analysis of major foodborne pathogens in Chinese food commodities between 2006 and 2016.Foodborne Pathogens and Disease,2018.doi:10.1089/fpd.2017.2417.

