鲍曼不动杆菌基因分型技术的研究进展
汤全珍,陆坚
深圳市第三人民医院感染科,深圳 518040
鲍曼不动杆菌(Acinetobacterbaumannii)为氧化酶阴性的无动力革兰阴性杆菌,镜下呈粗短球杆状或球形,是引起医院内感染的重要病原菌之一。近年来研究发现,导致患者终末期感染的致病菌以鲍曼不动杆菌最为多见,其也是导致重症监护室(intensive care unit,ICU)住院患者重症感染的最主要病原菌之一[1]。鲍曼不动杆菌在医院甚至全球范围内的克隆传播、暴发流行屡见报道,可引起呼吸机相关性肺炎、泌尿系统感染、血流感染、复杂性皮肤软组织感染、腹膜炎、中枢神经系统感染等,病死率高[2-4]。因此,掌握和使用高效、重复性好、分辨率高的细菌流行株克隆分型方法对抗感染治疗和医院内感染控制尤为重要。目前细菌分型方法很多,其中基于基因分型的方法最准确。本文就近年来鲍曼不动杆菌基因分型技术的进展进行综述。
1 鲍曼不动杆菌的基因分型技术
1.1 脉冲场凝胶电泳(pulsed-field gel electrophoresis,PFGE)
PFGE的原理如图1所示:将完整分离后的细菌基因组DNA嵌入低熔点琼脂糖,基因组被特定内切酶切成大小不等的片段,在特定电泳系统中这些片段在电场方向交替变换及合适的脉冲时间等条件下获得良好的分离。酶切的基因组片段电泳后呈现出条带,进而依据条带的数量、大小及位置,判定其同源性。PFGE配合新型软件BioNumerics可呈现出更直观的条带和聚类分析树状图,已广泛用于细菌同源性分析。尽管新的分型方法不断出现,PFGE仍是微生物基因分型的“金标准”[4-5],具有高分辨力、操作相对简便、费用适中等优点,广泛用于鲍曼不动杆菌暴发流行的监测,并可发现未知的克隆株[6-7]。菌株传播时会有突变、重组等遗传变异事件发生,若突变位点正好发生于酶切割位点,则导致分型结果有差异,因此 PFGE不适用于长时间、大规模的研究,且操作耗时(2~4 d)。其最主要的局限性是不同实验室之间缺乏可重复性,这是因为凝胶电泳、DNA制备等操作高度敏感,且目前全球范围内没有统一的参考标准。为此, Seifert等制定了统一标准流程,并在多个实验室对鲍曼不动杆菌进行分型,从而为建立网络数据库提供了资源[8]。Cetin等采用PFGE对一家土耳其大学附属医院66株鲍曼不动杆菌的流行病学特征和基因型进行分析,发现了36种PFGE类型,并明确A型和K型为流行克隆株[9]。目前,PulseNet网站已公布了霍乱弧菌、副溶血性弧菌、沙门菌、大肠埃希菌、志贺菌、空肠弯曲菌、单核细胞增生性李斯特菌、鼠疫耶尔森菌、肉毒杆菌PFGE的标准操作方案(http://www.pulsenetintertnational.org)[10]。

图1PFGE原理图
Fig.1SchematicdiagramofPFGE
1.2 随机扩增多态性DNA(randomly amplified polymorphic DNA,RAPD)分析
RAPD分析以分离自基因组中的DNA为模板,以一系列碱基对随机排列的特定核苷酸单链为引物(一般为10个碱基对),进行聚合酶链反应(polymerase chain reaction,PCR)扩增。这些引物在基因组DNA序列上存在特定结合位点,当这些区域发生DNA片段缺失、插入、重组或碱基突变,这些特定结合位点的分布可能发生变化,导致扩增产物因大小和数目不同而显示出多态性。用琼脂糖凝胶电泳检测PCR产物,根据分离后条带的差异即可分析菌株间的克隆同源关系。因此,RAPD分析相对简易、快速、分型能力好,并能为鲍曼不动杆菌流行病学的调查提供可靠信息,广泛用于医院内菌株的同源性分析[11-12]。Zanganeh等利用RAPD分析研究鲍曼不动杆菌基因的多态性,发现烧伤组患者中检出的鲍曼不动杆菌基因异质性高于非烧伤组[13]。然而,RAPD分析因各实验室之间尚未有统一设备及标准试剂,且操作时易受退火温度、技术人员及其他环节的影响[14],导致各时间段及区域显示的条带不能进行统一比较,从而缺乏可重复性。最近,Deschaght等采用RAPD熔解曲线分析法(melting curve analysis of RAPD-generated DNA fragments,McRAPD),对鲍曼不动杆菌进行基因分型,结果表明McRAPD耗时更少,更经济、环保[15]。
1.3 基因外重复回文序列PCR(repetitive extragenic palindromic-PCR,REP-PCR)
REP-PCR的原理如图2所示:短小重复序列广泛分布于细菌基因组,且具有高度保守的特性,包括REP。PCR分析可显示不同种属、同种属非克隆株的细菌重复序列的拷贝数和分布差异,以此判断其亲缘关系。REP-PCR进行基因分型迅速可靠,简易灵活,可作为大量菌株分型调查的首选,是鲍曼不动杆菌分子流行病学研究和克隆传播监测的有力工具[16-18]。但由于各实验室之间缺乏可重复性,且数据无法进行交换,REP-PCR难以广泛应用。近年来出现的半自动化DiversiLab系统,可使DNA提取、REP-PCR扩增操作标准化,克服了其早期重复性差的缺陷;PCR产物经高分辨率微流体芯片分离并染色后,采用统一的数据处理软件,对检测到的不同强度荧光进行分析,可获得直观图谱,代替了肉眼读取;其还能动态检测数据,在不同区域建立的数据库可上传至中心数据库,从而便于数据共享、各实验室之间比较及与其他分型方法的比较。Higgins等采用REP-PCR研究了492株亚胺培南耐药鲍曼不动杆菌的分子流行病学特点,共鉴定出8株全球范围内传播的克隆株, 显示欧洲克隆Ⅱ型株为最广泛的流行株[19]。
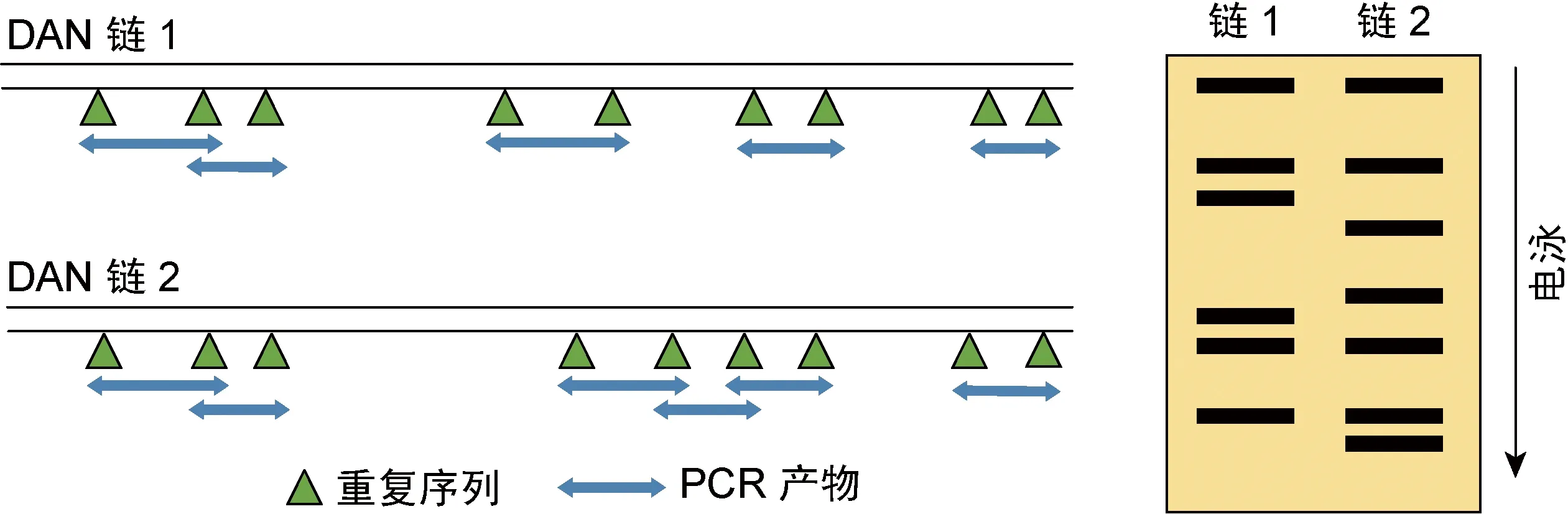
图2REP-PCR原理图
Fig.2SchematicdiagramofREP-PCR
1.4 多位点可变数目串联重复序列分析(multiple-locus variable-number tandem-repeat analysis,MLVA)
细菌基因组中广泛存在可变数目串联重复序列(variable-number tandem-repeat,VNTR),同类菌株串联重复单位的碱基数和类型基本固定,而重复次数不同。MLVA结合PCR技术可检出VNTR,对其进行扩增并测定重复拷贝数,从而根据VNTR位点拷贝数的差异对菌株进行分型。MLVA对菌株间同源性较高的细菌分型能力更强,且简单、快速,便于自动化,对实验室设备和技术人员要求不高,不仅可调查鲍曼不动杆菌的菌群结构,还可用于流行病学分析。其缺点是不适用于快速突变菌株的研究[20-22]。MLVABank专业数据库已建立(http://minisatellites.u-psud.fr/MLVAnet/),囊括了鲍曼不动杆菌相关信息,表明MLVA在鲍曼不动杆菌菌株亲缘关系中的应用有更广阔的前景,有助于鲍曼不动杆菌数据库的建立和完善[23],从而快速进行大范围调查,提高溯源分析的效率和准确率。
1.5 扩增片段长度多态性(amplified fragment length polymorphism,AFLP)分析
AFLP分析是一种分子标记技术,其基本原理如图3所示:基因组DNA被限制性内切酶切割后产生大小不等的限制性片段和不同的酶切位点,利用特定且与酶切位点配对的双链接头连接DNA酶切片段后,作为模板,与含有多个选择性碱基的引物进行扩增,扩增产物通过凝胶电泳等分离后进行检测,最后对DNA片段条带图进行判读分析,从而获得菌株间的亲缘关系。作为将限制性片段长度多态性(restriction fragment length polymorphism,RFLP)分析与PCR结合的分子分型技术,AFLP分析具有高效、可靠、分辨率高等优点,建立的数据库可进行资料共享与交流,便于长期观察菌株耐药性变化和监测医院内感染,已用于鲍曼不动杆菌克隆株的鉴定和流行株的检测,尤其在种的鉴定方面优势更加明显[24];其同样适用于种群结构的研究,且研究所需时间和区域范围不受限制[5]。AFLP分析的缺点是费用昂贵,对DNA模板质量要求高。Bahador等利用AFLP分析确定91株分布在伊朗3个地区的鲍曼不动杆菌的亲缘关系,检出24种类型(包括A~E型),其中贡巴德以S型、M型分布最广[25]。Metan 等利用AFLP分析进行大规模研究时,发现其对鲍曼不动杆菌菌株的鉴定结果与多位点序列分型(multilocus sequence typing,MLST)基本一致[26]。
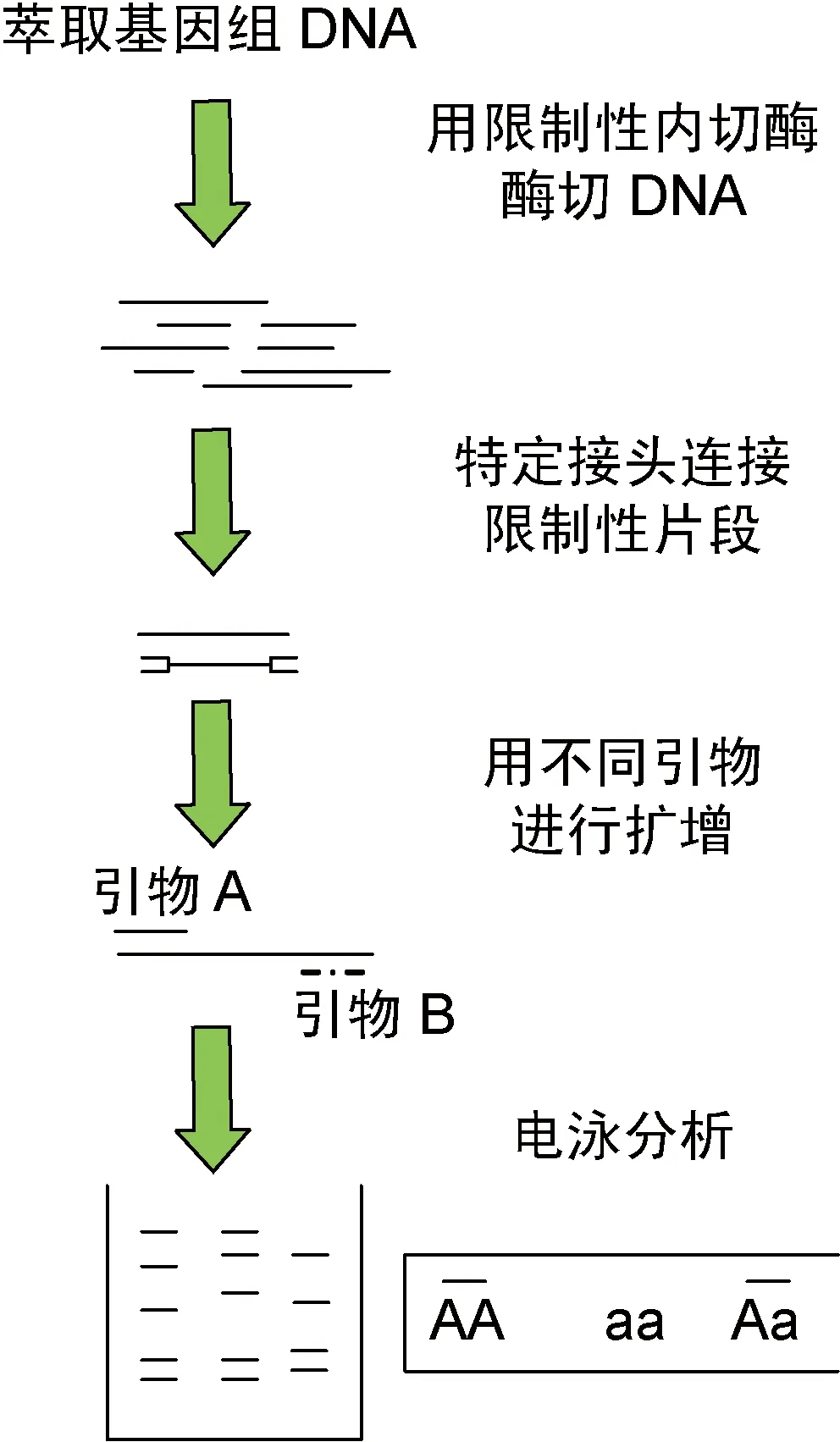
图3AFLP原理图
Fig.3SchematicdiagramofAFLP
1.6 MLST
MLST原理如图4所示:扩增细菌基因组管家基因片段,测定扩增序列,根据序列的差异进行分型,进而揭示菌株间的克隆同源相关性[14]。管家基因具有保守性、动态性、变异性等特点。如选择管家基因数目过少,则变异度太窄,分型效能降低;数目过多,则操作繁琐且费用较高。研究分析显示,选择7个管家基因既可达到有效分型目的,又不耗费过多资源[27]。MLST不适用于管家基因变异程度低的菌种或在短期内暴发的流行病学研究,而全球范围内的流行病学研究可能需10年或更长并在各区域进行评估,因此MLST逐渐成为种群结构调查和全球细菌流行病学研究的“金标准”[28]。针对鲍曼不动杆菌所选择管家基因的不同,MLST可分为 Bartual’s MLST (gltA、gyrB、gdhB、recA、cpn60、gpi、rpoD)和 Pasteur’s MLST(cpn60、fusA、gltA、pyrG、recA、rplB、rpoB),并已建立共享数据库 (http://pubmlst.org/abaumannii)[29]。目前MLST已用于鲍曼不动杆菌分子流行病学研究,发现了数个克隆复合体和多重耐药序列型。Hu等采用MLST对中国台湾地区耐碳青酶烯类鲍曼不动杆菌进行分型,发现ST787为最主要的序列型[30]。同时,MLST也可用于支原体、李斯特菌、链霉菌等的研究[31-33]。
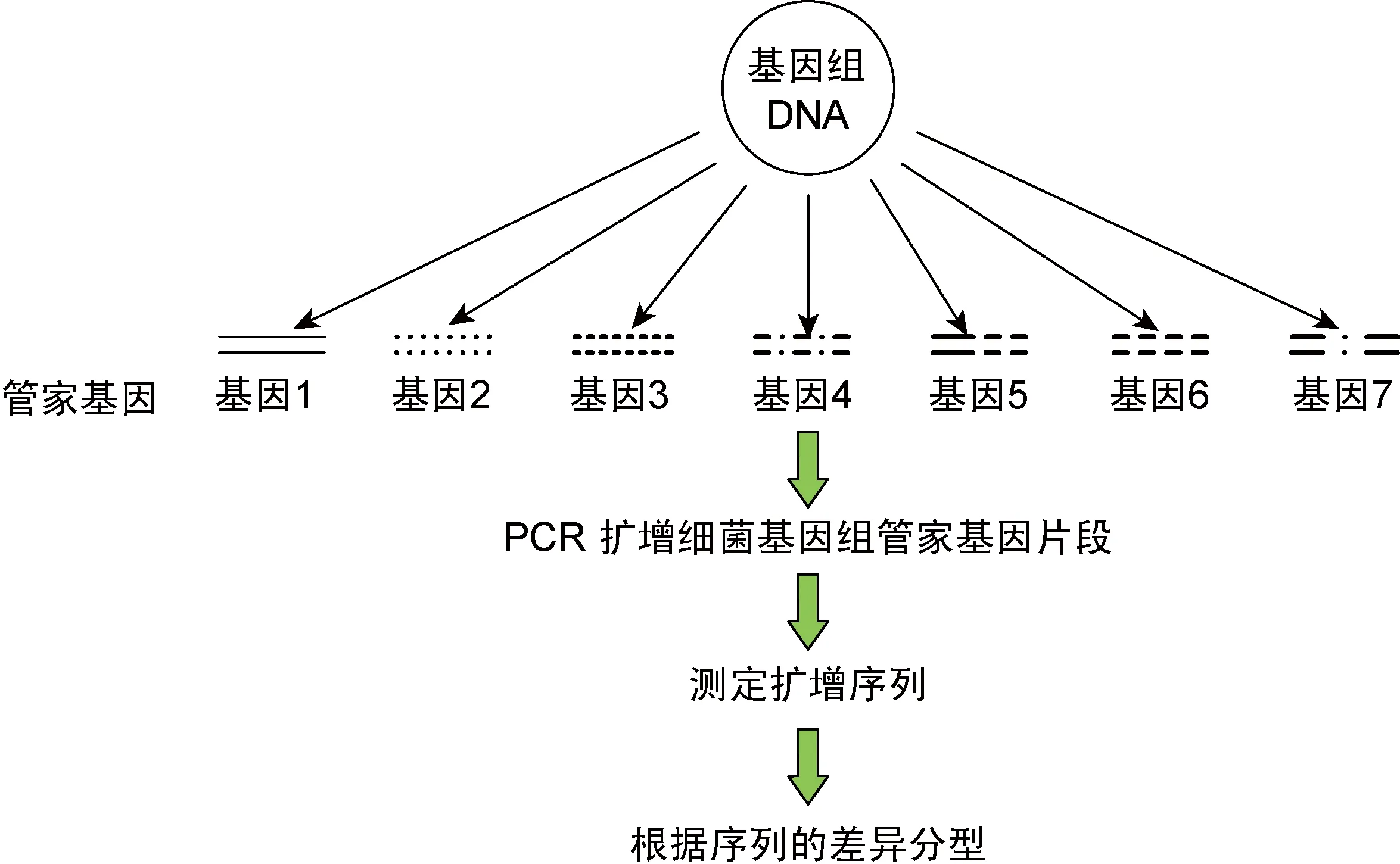
图4MLST原理图
Fig.4SchematicdiagramofMLST
1.7 全基因组测序(whole-genome sequencing,WGS)
WGS是指对整个基因组的核苷酸序列进行测序,通过分析这些核苷酸携带的全部遗传信息而达到分子分型的目的。目前,微生物WGS主要有两种:单核苷酸多态性分型WGS(wgSNP)和多位点序列分型WGS(wgMLST)。WGS可全面分析病原菌遗传信息,帮助研究病原菌基因多态性、遗传进化规律、致病机制、耐药基因传播途径及规律,从而精准确认暴发流行的源头,及时切断传播途径,为防控病原菌暴发流行提供基础数据;其还可对整个生物系统和种群结构实现从小范围到大范围乃至全球的研究。其中,wgSNP的突出优点是可鉴别体现种群多态性的关键突变。目前,WGS已用于鲍曼不动杆菌研究,可发现基因组结构变异、毒力基因、耐药基因等,从而从多方面研究多重耐药鲍曼不动杆菌与敏感株的区别。Di Nocera等发现了鲍曼不动杆菌菌株部分基因丢失被非同源菌株DNA序列替代的现象,为流行病学研究提供了新思路[34]。Lewis等应用WGS对6株多重耐药鲍曼不动杆菌菌株进行检测,揭示了英国一家医院该菌暴发流行期间菌株如何在人群之间进行传播[35]。Wright等调查了鲍曼不动杆菌在医院环境中的种群动态[36]。由此可见,WGS 将是未来细菌基因分型的“金标准”。
2 鲍曼不动杆菌基因分型方法的比较
对以上几种鲍曼不动杆菌基因分型技术的原理、优点、局限性及应用范围进行比较,结果详见表1。
3 结语
本文仅对近年来常用鲍曼不动杆菌基因分型技术予以简要叙述,实际研发应用的还包括核糖体分型、质粒图谱等。上述方法各有特色,在选择分型方法时应结合研究目的、规模、收集标本的时间、实验条件、经费预算等综合考虑。为更全面地研究流行病学和分析菌株间的同源性,可选择两种及以上的方法进行结果对比。随着生物信息学技术的提升及测序成本的降低,WGS将在微生物分型、遗传规律、菌株演变和暴发溯源等研究中突显其应用价值和发展潜力。
表1基因分型方法的比较
Tab.1Comparisonofgenotypingmethods
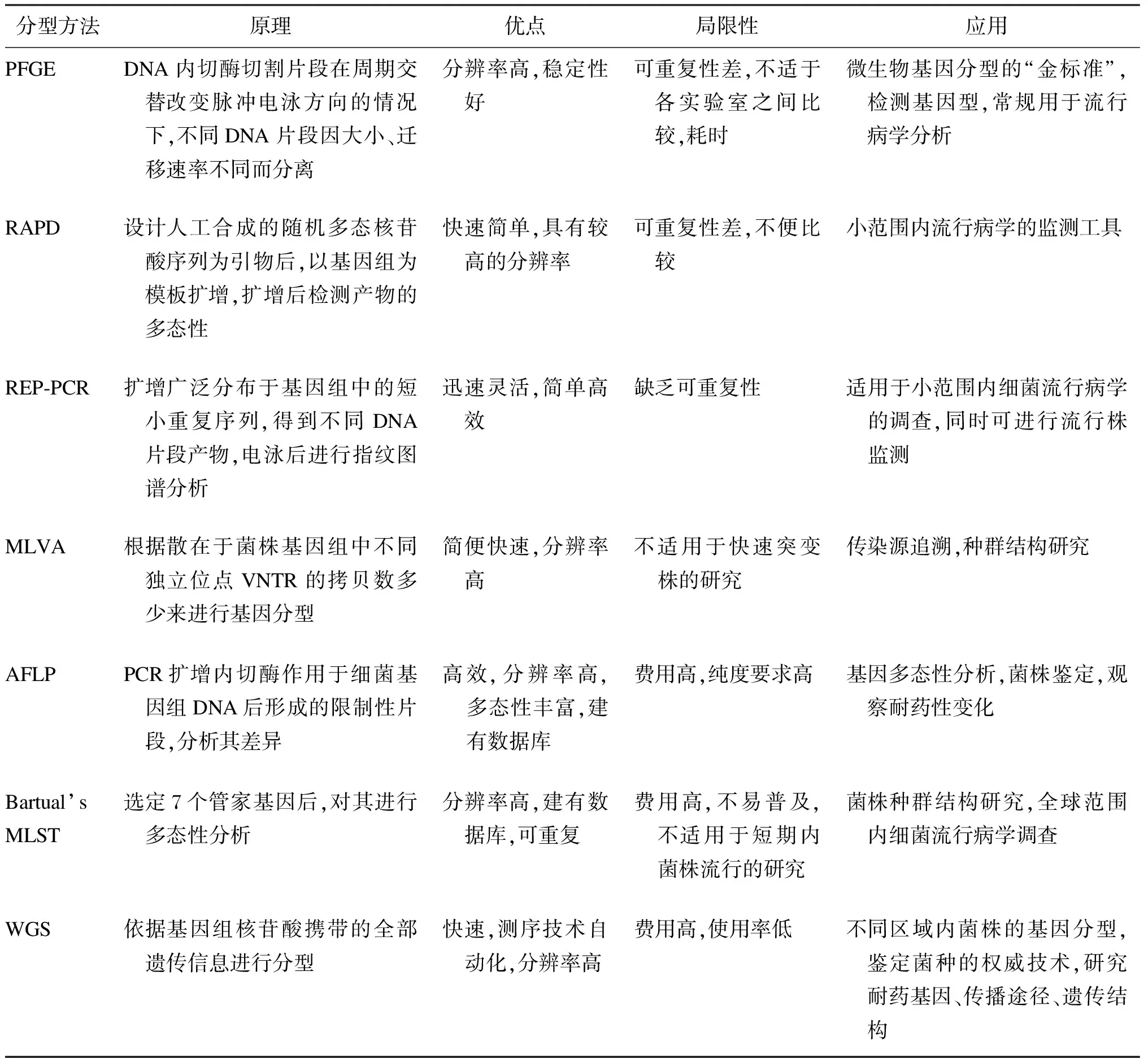
分型方法原理优点局限性应用PFGEDNA内切酶切割片段在周期交替改变脉冲电泳方向的情况下,不同DNA片段因大小、迁移速率不同而分离分辨率高,稳定性好可重复性差,不适于各实验室之间比较,耗时微生物基因分型的“金标准”,检测基因型,常规用于流行病学分析RAPD设计人工合成的随机多态核苷酸序列为引物后,以基因组为模板扩增,扩增后检测产物的多态性快速简单,具有较高的分辨率可重复性差,不便比较小范围内流行病学的监测工具REP⁃PCR扩增广泛分布于基因组中的短小重复序列,得到不同DNA片段产物,电泳后进行指纹图谱分析迅速灵活,简单高效缺乏可重复性适用于小范围内细菌流行病学的调查,同时可进行流行株监测MLVA根据散在于菌株基因组中不同独立位点VNTR的拷贝数多少来进行基因分型简便快速,分辨率高不适用于快速突变株的研究传染源追溯,种群结构研究AFLPPCR扩增内切酶作用于细菌基因组DNA后形成的限制性片段,分析其差异高效,分辨率高,多态性丰富,建有数据库费用高,纯度要求高基因多态性分析,菌株鉴定,观察耐药性变化Bartual’sMLST选定7个管家基因后,对其进行多态性分析分辨率高,建有数据库,可重复费用高,不易普及,不适用于短期内菌株流行的研究菌株种群结构研究,全球范围内细菌流行病学调查WGS依据基因组核苷酸携带的全部遗传信息进行分型快速,测序技术自动化,分辨率高费用高,使用率低不同区域内菌株的基因分型,鉴定菌种的权威技术,研究耐药基因、传播途径、遗传结构
[1] Lee CR,Lee JH,Park M,Park KS,Bae IK,Kim YB,Cha CJ,Jeong BC,Lee SH.Biology of Acinetobacter baumannii: pathogenesis,antibiotic resistance mechanisms,and prospective treatment options [J].Front Cell Infect Microbiol,2017,7: 55.
[2] Kröger C,Kary SC,Schauer K,Cameron AD.Genetic regulation of virulence and antibiotic resistance in Acinetobacter baumannii [J].Genes (Basel),2016,8(1): 12.doi: 10.3390/genes8010012.
[3] Ahmed SS,Alp E.Genotyping methods for monitoring the epidemic evolution of A.baumannii strains [J].J Infect Dev Ctries,2015,9(4): 347-354.
[4] Glickman MS,Cox JS,Jacobs WR Jr.A novel mycolic acid cyclopropane synthetase is required for cording,persistence,and virulence of Mycobacterium tuberculosis [J].Mol Cell,2000,5(4): 717-727.
[5] Rafei R,Kempf M,Eveillard M,Dabboussi F,Hamze M,Joly-Guillou ML.Current molecular methods in epidemiological typing of Acinetobacter baumannii [J].Future Microbiol,2014,9(10): 1179-1194.
[6] Mohajeri P,Farahani A,Mehrabzadeh RS.Molecular characterization of multidrug resistant strains of Acinetobacter baumannii isolated from intensive care units in West of Iran [J].J Clin Diagn Res,2017,11(2): DC20-DC22.
[7] Turton JF,Kaufmann ME,Warner M,Coelho J,Dijkshoorn L,van der Reijden T,Pitt TL.A prevalent,multiresistant clone of Acinetobacter baumannii in Southeast England [J].J Hosp Infect,2004,58(3): 170-179.
[8] Seifert H,Dolzani L,Bressan R,van der Reijden T,van Strijen B,Stefanik D,Heersma H,Dijkshoorn L.Standardization and interlaboratory reproducibility assessment of pulsed-field gel electrophoresis-generated fingerprints of Acinetobacter baumannii [J].J Clin Microbiol,2005,43(9): 4328-4335.
[9] Cetin ES,Durmaz R,Tetik T,Otlu B,Kaya S,Çaliskan A.Epidemiologic characterization of nosocomial Acinetobacter baumannii infections in a Turkish university hospital by pulsed-field gel electrophoresis [J].Am J Infect Control,2009,37(1): 56-64.
[10] 崔晶花,杜小莉,崔志刚,刁保卫,娄静,周海健,阚飙,李伟.PulseNet China网络实验室能力考核结果评价与分析 [J].疾病监测,2011,26(10): 766-769.
[11] Chang HL,Tang CH,Hsu YM,Wan L,Chang YF,Lin CT,Tseng YR,Lin YJ,Sheu JJ,Lin CW,Chang YC,Ho MW,Lin CD,Ho CM,Lai CH.Nosocomial outbreak of infection with multidrug-resistant Acinetobacter baumannii in a medical center in Taiwan [J].Infect Control Hosp Epidemiol,2009,30(1): 34-38.
[12] Szejbach A,Mikucka A,Bogiel T,Gospodarek E.Usefulness of phenotypic and genotypic methods for metallo-beta-lactamases detection in carbapenem-resistant Acinetobacter baumannii strains [J].Med Sci Monit Basic Res,2013,19: 32-36.
[13] Zanganeh Z,Eftekhar F.Correlation of oxacillinase gene carriage with the genetic fingerprints of imipenem-resistant clinical isolates of Acinetobacter baumannii [J].Jundishapur J Microbiol,2015,8(9): e26545.
[14] Bonofiglio L,Gardella N,Mollerach M.Application of molecular typing methods to the study of medically relevant Gram-positive cocci [M/OL].InTech,2012.https://cdn.intechopen.com/pdfs-wm/34915.pdf.
[15] Deschaght P,Van Simaey L,Decat E,Van Mechelen E,Brisse S,Vaneechoutte M.Rapid genotyping of Achromobacter xylosoxidans,Acinetobacter baumannii,Klebsiella pneumoniae,Pseudomonas aeruginosa and Stenotrophomonas maltophilia isolates using melting curve analysis of RAPD-generated DNA fragments (McRAPD) [J].Res Microbiol,2011,162(4): 386-392.
[16] Chen CH,Kuo HY,Hsu PJ,Chang CM,Chen JY,Lu HH,Chen HY,Liou ML.Clonal spread of carbapenem-resistant Acinetobacter baumannii across a community hospital and its affiliated long-term care facilities: A cross sectional study [J].J Microbiol Immunol Infect,2017.doi: 10.1016/j.jmii.2017.08.001.
[17] Kateete DP,Nakanjako R,Okee M,Joloba ML,Najjuka CF.Genotypic diversity among multidrug resistant Pseudomonas aeruginosa and Acinetobacter species at Mulago Hospital in Kampala,Uganda [J].BMC Res Notes,2017,10(1): 284.doi: 10.1186/s13104-017-2612-y.
[18] Bianco A,Quirino A,Giordano M,Marano V,Rizzo C,Liberto MC,Focà A,Pavia M.Control of carbapenem-resistant Acinetobacter baumannii,outbreak in an intensive care unit of a teaching hospital in Southern Italy [J].BMC Infect Dis,2016,16(1): 747.doi: 10.1186/s12879-016-2036-7.
[19] Higgins PG,Dammhayn C,Hackel M,Seifert H.Global spread of carbapenem-resistant Acinetobacter baumannii [J].J Antimicrob Chemother,2010,65(2): 233-238.
[20] Azimi L,Talebi M,Khodaei F,Najafi M,Lari AR.Comparison of multiple-locus variable-number tandem-repeat analysis with pulsed-field gel electrophoresis typing of carbapenemases producing Acinetobacter baumannii isolated from burn patients [J].Burns,2016,42(2): 441-445.
[21] Hu Y,Li B,Jin D,Cui Z,Tao X,Zhang B,Zhang J.Comparison of multiple-locus variable-number tandem-repeat analysis with pulsed-field gel electrophoresis typing of Acinetobacter baumannii in China [J].J Clin Microbiol,2013,51(4): 1263-1268.
[22] Hauck Y,Soler C,Jault P,Mérens A,Gérome P,Nab CM,Trueba F,Bargues L,Thien HV,Vergnaud G,Pourcel C.Diversity of Acinetobacter baumannii in four French military hospitals,as assessed by multiple locus variable number of tandem repeats analysis [J].PLoS One,2012,7(9): e44597.
[23] Pourcel C,Minandri F,Hauck Y,D’Arezzo S,Imperi F,Vergnaud G,Visca P.Identification of variable-number tandem-repeat (VNTR) sequences in Acinetobacter baumannii and interlaboratory validation of an optimized multiple-locus VNTR analysis typing scheme[J].J Clin Microbiol,2011,49(2): 539-548.
[24] Carretto E,Barbarini D,Dijkshoorn L,van der Reijden TJ,Brisse S,Passet V,Farina C;APSI Acinetobacter Study Group.Widespread carbapenem resistant Acinetobacter baumannii clones in Italian hospitals revealed by a multicenter study [J].Infect Genet Evol,2011,11(6): 1319-1326.
[25] Bahador A,Taheri M,Pourakbari B,Hashemizadeh Z,Rostami H,Mansoori N,Raoofian R.Emergence of rifampicin,tigecycline,and colistin-resistant Acinetobacter baumannii in Iran;spreading of MDR strains of novel International Clone variants [J].Microb Drug Resist,2013,19(5): 397.
[26] Metan G,Sariguzel F,Sumerkan B,van der Reijden T,Dijkshoorn L.Clonal diversity and high prevalence of OXA-58 among Acinetobacter baumannii,isolates from blood cultures in a tertiary care centre in Turkey [J].Infect Genet Evol,2013,14: 92-97.doi: 10.1016/j.meegid.2012.11.003.
[27] Hamouda A,Evans BA,Towner KJ,Amyes SG.Characterization of epidemiologically unrelated Acinetobacter baumannii isolates from four continents by use of multilocus sequence typing,pulsed-field gel electrophoresis,and sequence-based typing of bla(OXA-51-like) genes [J].J Clin Microbiol,2010,48(7): 2476-2483.
[28] Spratt BG.Exploring the concept of clonality in bacteria [J].Methods Mol Biol,2004,266: 323-352.
[29] Hamidian M,Nigro SJ,Hall RM.Problems with the Oxford MLST scheme for Acinetobacter baumannii: do ST92 and ST109 exist?[J].J Clin Microbiol,2017,55(7): 2287-2289.doi: 10.1128/JCM.00533-17.
[30] Hu YF,Hou CJ,Kuo CF,Wang NY,Wu AY,Leung CH,Liu C,Yeh HI.Emergence of carbapenem-resistant Acinetobacter baumannii ST787 in clinical isolates from blood in a tertiary teaching hospital in Northern Taiwan [J].J Microbiol Immunol Infect,2017,50(5): 640-645.
[31] El-Gazzar M,Ghanem M,McDonald K,Ferguson-Noel N,Raviv Z,Slemons RD.Development of multilocus sequence typing (MLST) for Mycoplasma synoviae [J].Avian Dis,2017,61(1): 25-32.
[32] Takahashi H,Iwakawa A,Ohshima C,Kyoui D,Kumano S,Kuda T,Kimura B.A rapid typing method for Listeria monocytogenes based on high-throughput multilocus sequence typing (Hi-MLST) [J].Int J Food Microbiol,2017,243: 84-89.
[33] Labeda DP,Dunlap CA,Rong X,Huang Y,Doroghazi JR,Ju KS,Metcalf WW.Phylogenetic relationships in the family Streptomycetaceae using multi-locus sequence analysis [J].Antonie Van Leeuwenhoek,2017,110(4): 563-583.
[34] Di Nocera PP,Rocco F,Giannouli M,Triassi M,Zarrilli R.Genome organization of epidemic Acinetobacter baumannii strains [J].BMC Microbiol,2011,11: 224.doi: 10.1186/1471-2180-11-224.
[35] Lewis T,Loman NJ,Bingle L,Jumaa P,Weinstock GM,Mortiboy D,Pallen MJ.High-throughput whole-genome sequencing to dissect the epidemiology of Acinetobacter baumannii isolates from a hospital outbreak [J].J Hosp Infect,2010,75(1): 37-41.
[36] Wright MS,Haft DH,Harkins DM,Perez F,Hujer KM,Bajaksouzian S,Benard MF,Jacobs MR,Bonomo RA,Adams MD.New insights into dissemination and variation of the health care-associated pathogen Acinetobacter baumannii from genomic analysis [J].MBio,2014,5(1): e00963-13.doi: 10.1128/mBio.00963-13.

