基于PLFA的高山栎和高山松林松茸菌塘土壤微生物群落特征研究
杨淑娇,杨永平,石玲玲,毕迎凤,李建文,杨锦超,杨雪飞,*
1 中国科学院昆明植物研究所中国西南野生生物种质资源库,昆明 650201 2 中国科学院大学,北京 100049 3 中国科学院昆明植物研究所资源植物与生物技术重点实验室,昆明 650201 4 西南林业大学, 昆明 655024
外生菌根(Ectomycorrhiza,ECM)是由土壤真菌与植物根系形成的互惠共生体[1-2],是森林生态系统的重要组成部分,在维持生态系统稳定和多样性等方面发挥着重要作用[3]。松口蘑(Tricholomamatsutake),又名松茸,是一种北半球广布的典型ECM真菌,主要与松科和壳斗科多种植物共生形成外生菌根,包括日本的赤松(Pinusdensiflora)、偃松(Pinuspumila)、南日本铁杉(Tsugasiebordii)[4- 10],芬兰的欧洲赤松(Pinussylvestris)、欧洲云杉(Piceaabies)[11],以及中国的高山松(Pinusdensata)、云南松(Pinusyunnanensis)、高山栎(Quercussemecarpifolia)和元江锥(Castanopsisorthacantha)等[12, 13]。最新研究还发现,松茸能被成功接种到其他科属的根系,在试验条件下产生共生关系甚至形成菌塘结构,例如,蔷薇科的Prunusspeciosa[14]、桦木科的Betulaplatyphyllavar. japonica[15]以及楝科的Cedrelaherrerae[16]。这充分说明松茸宿主植物的多样性和广谱性。
松茸子实体是名贵的野生食用菌,主要出口日本,在当地市场上价格高达1000美元/kg[17]。以日本学者为主的科学家对松茸的人工栽培进行了近百年的探索[10-11, 18-19],取得一定成果。目前能实现菌根合成并生成一定的菌塘结构,但尚未完全实现人工栽培出菇[20-21]。究其原因,一方面是我们对松茸与其宿主植物之间的相互关系和作用机制的了解不全面;另一方面,对松茸菌塘及其内的微生物群落特征了解较少。
菌塘(Shiro)是由松茸的地下菌丝、宿主根系及周围的土壤结合形成的白色海绵状结构[4],是松茸生长的重要载体和完成主要生活史的具体场所[22]。关于松茸菌塘内的微生物,前人已做了一些探讨[23- 26],研究结果的广度和深度随着技术的发展不断提高。日本学者Ohara等人早在1967年便采用分离培养的方式发现菌塘中可培养的细菌和放线菌减少[23]。随着技术的发展,Lian[24]、Kataoka[25]、Vaario[26]、马大龙[27]等人分别通过形态学鉴定、ITS(Internal transcribed spacer,ITS)多态性分析、PCR-变性梯度凝胶电泳(Denaturing gradient gel electrophoresis,DGGE)技术、磷脂脂肪酸(Phospholipid fatty acid,PLFA)生物标记法等手段对松茸菌塘土壤微生物群落特征进行了深入研究。近年来,由于新一代测序技术的发展,宏基因组学方法也运用到松茸菌塘的研究中来[28-29]。尽管如此,前人对松茸菌塘微生物的研究只针对松科某一种宿主森林类型,如Ogawa[4, 7- 9],Lian[24],Ohara[23],Kataoka[25]和Kim[28-29]等人的研究,尚未见壳斗科宿主森林菌塘内微生物群落的研究报道。除此之外,还未见不同宿主森林类型内松茸菌塘微生物群落组成和结构的对比分析,缺乏对规律性的认知。
本研究以松茸典型产区云南省迪庆州香格里拉县分布的高山栎林和高山松林为研究对象,采用磷脂脂肪酸(PLFA)生物标记法[30]对比研究上述两种松茸宿主森林类型内的菌塘和非菌塘的土壤微生物群落特征,分析与其相关的土壤化学性质。目的是回答松茸菌塘微生物群落特征在不同林型间是否存在异同的科学问题,为科学管理具有高附加值的松茸资源,保障其产量和质量,以及探索人工栽培条件奠定研究基础。
1 研究地区与研究方法
1.1 研究点概况
选择松茸产量较高且具有高山松和高山栎分布的云南省迪庆州香格里拉县建塘镇吉迪村为研究地点。该村所在的香格里拉县平均海拔3300m,干湿季分明且昼夜温差较大。年平均气温为7.3℃,年平均最低气温为1.9℃,年平均最高气温为14.3℃,年平均降水量为38.5mm,年平均相对湿度为61%(中国气象数据网http: //data.cma.cn/site/index.html香格里拉气象站数据)。其中高山栎林研究点(99°37′52.59″E,28°05′38.48″N)平均海拔为3747m,森林建群种为高山栎(Quercussemecarpifolia),约占45.19%,平均高度为7.71m;高山松林研究点(99°38′28.73″ E,28°04′11.09″N)平均海拔为3452m,森林建群种为高山松(Pinusdensata),约占64.06%,平均高度为6.20m。两种森林类型下均有杜鹃灌丛分布。
1.2 样品采集
土样采集工作于2014年8月上旬完成,该时间段为松茸出菇的活跃期。为使取样具有代表性,在两种林型各设置了5个20m×20m的重复样方。为避免假重复,样方间的距离均大于25m,且每个样方中至少有3个距离大于2m的松茸菌塘。去除土壤腐殖质层后,在每个样方中,用土钻采集3个菌塘的矿质层0—10cm处的土壤。作为对照,再根据五点取样法采集5个非菌塘的矿质层0—10cm处的土壤。采集的土样先过4mm筛,然后用冰袋保鲜及时运回实验室。所有土样一部分风干用于土壤化学性质测定,另一部分保存在-20℃冰箱用于土壤微生物分析。
本实验共10个样方,80份土壤样品。后期处理中,按样方将重复样品均匀合并,每个样方有2份土壤样品:一份菌塘矿质层土样和一份非菌塘矿质层土样。最终共有20份土壤样品用于后面的土壤微生物群落结构分析和土壤化学性质测定。
1.3 土壤化学性质测定
用于化学性质测定的土壤首先过2mm的筛,一部分用碳氮分析仪(Vario MAX CN,Germany)测定土壤全碳和全氮含量,用ICP-AES分析仪测定土壤全磷含量。另一部分经研磨再过0.25mm筛后用精密酸度计(PHS- 3C,Shanghai)测定土壤pH值。测定过程交由中国科学院西双版纳热带植物园生物地球化学实验室完成。
1.4 土壤微生物群落PLFAs测定
土壤微生物群落组成的测定,采用PLFA生物标记法。PLFA的提取过程参考Bossio和Scow[31]的方法,测定过程由中国科学院华南植物园退化生态系统植被恢复与管理重点实验室完成。
1.5 数据处理
对PLFA原始数据进行整理,包括每个样品中PLFA的组成、百分含量和生物量等信息。其中,单个PLFA的百分含量表示该特征PLFA占总提取的PLFAs量的百分数[30]。单个PLFA的生物量由内标19:0的反应值转换而来,单位为nmol/g,表示每克干土中该PLFA的含量,公式如下:
用R软件包(vegan)进行数据分析。测定微生物多样性的指标,包括Shannon-Wiener多样性指数(H)、Pielou均匀度指数(Evenness,J)和PLFAs丰度(Richness,S)。具体计算公式如下:
(1)Shannon-Wiener多样性指数(H)[30,32]:
式中,S表示样品中 PLFA生物标记的数目,即丰度,Pi表示第i个种占总数的比例。
(2)Pielou均匀度指数(J)[30, 32]:
用百分含量、生物量和多样性指数表征土壤微生物群落组成。根据各个变量的数据分布属性,分别采用二项分布检验(Binomial test,针对百分含量),双因素方差分析(Two-way ANOVA,针对生物量)和泊松分布检验(Poisson test,针对总PLFAs多样性、均匀度和丰度)进行菌塘、林型及其交互作用对土壤微生物组成影响的显著性检验。用非参数多元方差分析法(Permutational analysis of variance,PERMANOVA)分析群落结构差异,及其与菌塘和林型的相互关系,并通过典范对应分析(Canonical correspondence analysis,CCA,CANOCO 4.5软件)偶联与之相关的土壤化学因子。
2 结果与分析
2.1 菌塘的土壤微生物群落组成
不同的磷脂脂肪酸表征不同的微生物类群[33],表1显示了所有样品中出现的PLFAs及其代表的微生物类群。所有样品中共出现了76种PLFAs,每个样品中平均有(40±5)种。
菌塘中共计出现63种PLFAs,其中主要的10种PLFAs分别为指代真菌的18:2ω6,9c(8.49%)、18:1ω9c(7.51%)和18:3 w6c (6,9,12)(2.44%),共占18.44%;指代革兰氏阴性菌的19:0 cyclo w8c,占3.78%;指代革兰氏阳性菌的15:0 iso,占3.51%;未被鉴定的非特异性脂肪酸16:0(16.96%)、17:1 anteiso B(13.77%)、18:1 w6c(6.85%)、16:1 w6c(4.75%)和18:0(3.16%),共占45.49%。

表1 用于微生物生物标记的磷脂脂肪酸
GNB: 革兰氏阴性菌 Gram-negative bacteria;GPB: 革兰氏阳性菌 Gram-positive bacteria;AMF: 丛枝菌根真菌 Arbuscular mycorrhizal fungi;F/B: 真菌/细菌比 Ratio of fungal to bacterial PLFAs;GNB/GPB: 革兰氏阴性菌/阳性菌比 Ratio of Gram-negative bacterial to Gram-positive bacterial PLFAs
2.2 菌塘和林型对土壤微生物群落组成的影响
总结土壤微生物群落组成的各个指标在菌塘和林型间的差异(表2),统计检验结果发现,土壤微生物的百分含量、生物量和多样性均在不同程度上受菌塘的影响,植被类型对土壤微生物群落的影响仅体现在个别类群的生物量方面。
菌塘内真菌百分含量显著增加(P< 0.001),革兰氏阴性菌(GNB,P< 0.05)和革兰氏阳性菌(GPB,P< 0.01)的百分含量显著降低。例如,真菌百分含量在高山栎林内由12.03±0.25增加为19.98±3.38,在高山松林内由13.92±0.39增加到16.89±1.02。菌塘内放线菌和丛枝菌根真菌(AMF)的百分含量减少,但不显著(P> 0.05)。菌塘内真菌/细菌生物量比(F/B)显著高于非菌塘(P< 0.01)。总的来说,菌塘并不影响土壤微生物总PLFAs多样性(Shannon多样性指数)和丰度,及各微生物类群的PLFAs丰度,但菌塘内土壤微生物总PLFAs均匀度(Pielou均匀度指数)显著低于非菌塘(P< 0.05)。
除了真菌和AMF的生物量,林型对大部分表征土壤微生物群落组成指标的影响并不显著,也不与菌塘产生交互作用。林型对真菌和AMF生物量的影响表现为:真菌和AMF生物量在高山栎林内均显著高于高山松林(P< 0.05)。
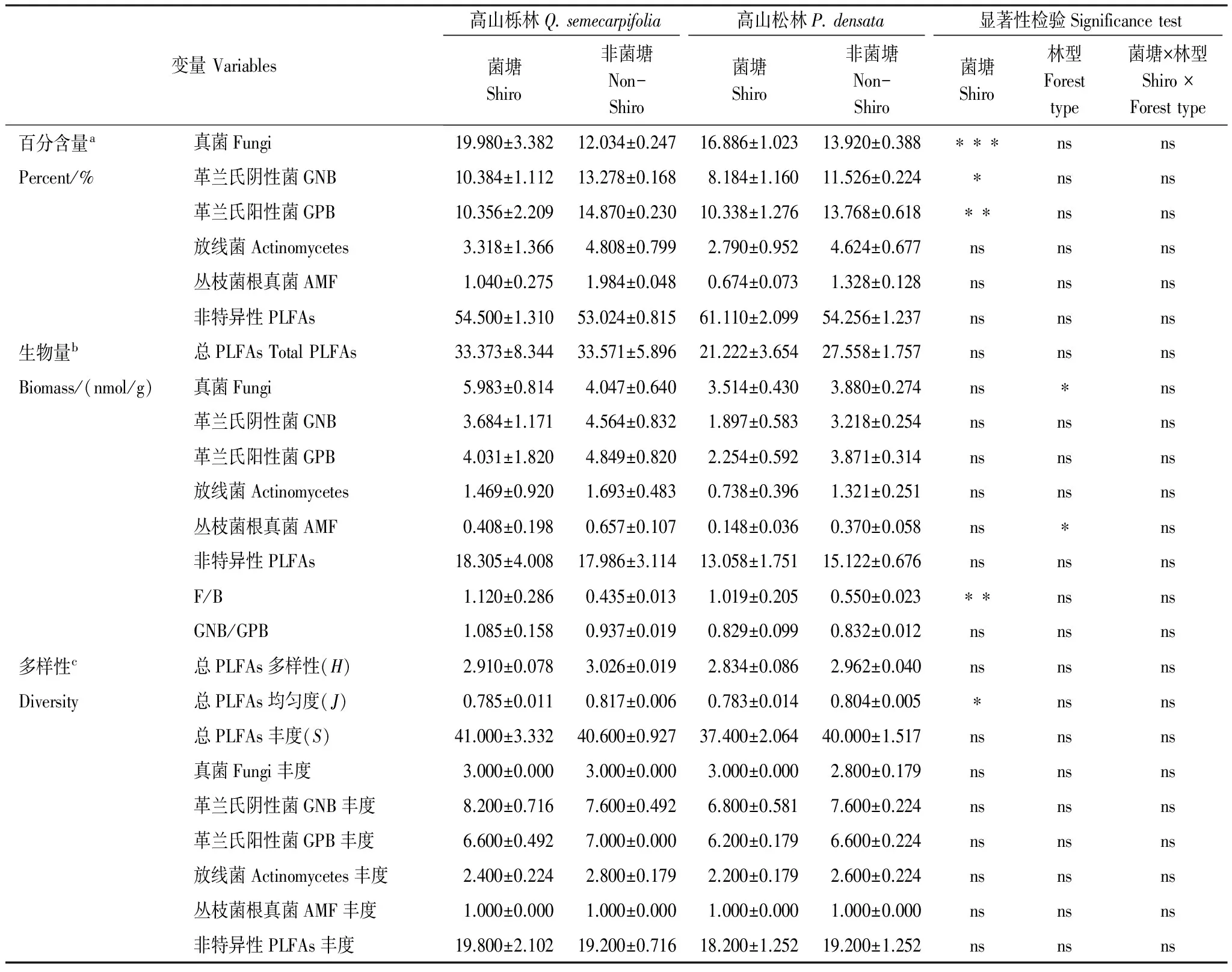
表2 菌塘、林型及其交互作用对土壤微生物百分含量、生物量和多样性的影响
ns:P> 0.05, *P< 0.05,**P< 0.01,***P< 0.001;a: 采用二项分布检验;b: 采用双因素方差分析;c: 采用泊松分布检验
2.3 菌塘和林型对土壤微生物群落结构的影响
通过非参数多元方差分析发现(表3),菌塘、林型均显著影响土壤微生物群落结构,其中菌塘(R2=0.263,P< 0.01)的影响大于林型(R2=0.173,P< 0.05)的影响,但两者的交互作用对微生物群落结构的影响不显著(P> 0.05)。
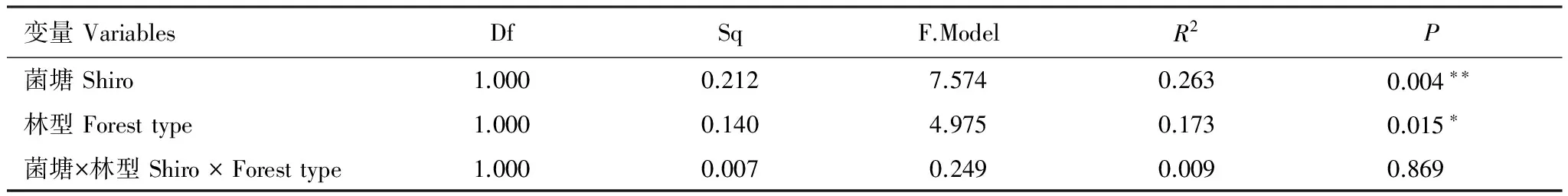
表3 菌塘、林型及其交互作用对土壤微生物群落结构的非参数多元方差(PERMANOVA)分析
*P< 0.05,**P< 0.01;df:自由度Degree of freedom;Sq:均方Mean Square;F.Model:F检验的结果The result of F-test
2.4 微生物群落结构与菌塘、林型和土壤化学性质的关系
对样品中出现频率最高的40种磷脂脂肪酸进行典范对应分析(Canonical correspondence analysis,CCA),发现除菌塘和森林类型外,各个土壤化学指标也在不同程度上与土壤微生物的群落结构相关(图1)。然而进一步分析表明,这些土壤化学指标主要与林型相关联(表4),表现为高山栎林的全碳(P< 0.001)、全氮(P< 0.05)、全磷(P< 0.001)和碳氮比(P< 0.001)显著高于高山松林,而pH值显著低于高山松林(P< 0.05)。对于菌塘和非菌塘,土壤化学性质间并无显著差异(表4)。
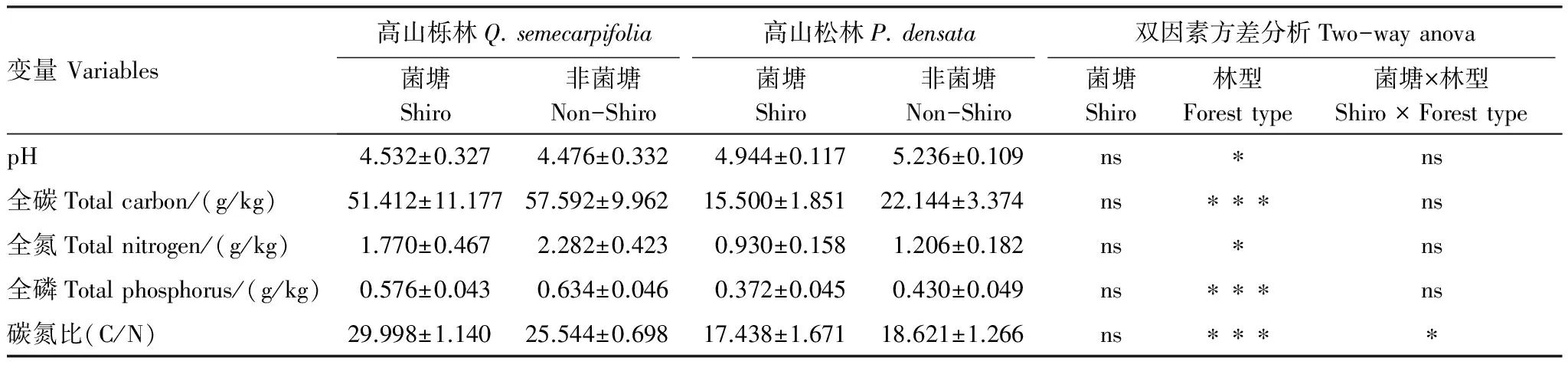
表4 菌塘、林型及其交互作用对土壤化学性质的影响
ns:P> 0.05, *P< 0.05,**P< 0.01,***P< 0.001
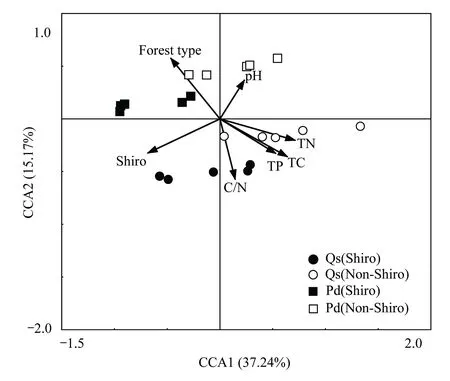
图1 土壤微生物PLFAs与土壤化学性质的典范对应分析 Fig.1 Canonical correspondence analysis of soil microbial PLFAs and soil chemical factorsQs: 高山栎林 Q. semecarpifolia; Pd: 高山松林 P. densata; Shiro: 菌塘; Non-Shiro: 非菌塘;TC: 全碳 Total carbon; TN: 全氮 Total nitrogen; TP: 全磷 Total phosphorus; C/N: 碳氮比 Ratio of total carbon to total nitrogen
3 讨论
掌握菌塘内微生物群落结构及其与宿主植物的相互关系,对科学管理具有高附加值的松茸资源,保障其产量和质量、并最终实现人工栽培具有重要意义。在自然界松茸与松科和壳斗科的多个物种产生共生关系。过去针对赤松(Pinusdensiflora)林[23- 25, 28-29]、偃松(Pinuspumila)林[7]、云杉(Piceaglehnii)林[8]、铁杉(Tsugasieboldii)林[4]等宿主森林的研究为我们认知松茸菌塘内的微生物群落组成奠定了基础。但这些研究结果通常相对独立、缺乏横向对比,限制了我们对松茸菌塘微生物群落特征的规律性认知。本研究不仅首次研究了壳斗科高山栎林内的菌塘微生物群落特征,还将其与高山松林进行对比分析,浅析其共性和规律。
研究主要发现,尽管高山栎林的真菌和AMF的生物量显著高于高山松林(表2),且两种林型在微生物群落结构上存在显著差异(表3、图1),但高山栎林和高山松林间菌塘微生物群落特征的差异并不显著。此外,非参数多元方差分析也表明菌塘对土壤微生物群落结构的影响大于林型(表3),且两者间不产生交互作用。综上所述,研究认为本研究涉及的两种林型间松茸菌塘微生物群落的特征基本相似,具有共性。然而该共性是否普遍存在并具有规律?有待更加广泛的取样和深入的研究来解答。
松茸菌塘具有特定的微生物群落特征。与非菌塘相比,菌塘内的真菌含量增加,可能主要由松茸本身为优势类群所决定。无论采用哪种方法,前人已知的研究结果均表明菌塘内细菌减少[23, 25]。本研究再次印证了该观点,发现GPB和GNB在菌塘中的百分含量显著下降。分析其原因,目前存在两种可能性解释。首先,根据最新研究,松茸子实体在实验室离体条件下会分泌由吡喃糖氧化酶(pyranose oxidase)产生的过氧化氢,可抑制致病菌立枯丝核菌(Rhizoctoniasolani)的生长[42]。本研究中土壤取样时间恰好是出菇期,如果在野外条件下松茸子实体也能分泌过氧化氢,可能是解释GPB和GNB减少的原因。其次,Park等人[43]从松茸担子果、赤松根尖和菌塘土壤中均分离到一个木霉属新种Trichodermasongyi,该属物种通常为植物病原体的生物控制媒介,而Vaario等[26]的文章也提出,木霉属真菌与松茸总是存在共生关系,因此另一种可能即是松茸在木霉属物种的协助下抑制菌塘内细菌生长。
本研究还发现,从PLFAs多样性看,菌塘与非菌塘并不存在显著差异,也就是说菌塘也具有丰富的类群多样性(表2),这与Vaario等[26]的观点相似。另外,菌塘与非菌塘的总PLFAs生物量也基本等量。有趣的是,针对北方温带森林外生菌根菌Piloderma菌丝垫和非菌丝垫(与本文菌塘和非菌塘概念相似)的定量PCR研究也有类似发现[44]。这可能说明在外生菌根菌主导的土壤菌丝际,微生物总生物量与周边土壤保持一致。然而,菌塘的PLFAs均匀度显著下降,说明尽管总生物量和多样性维持稳定,但菌塘内各组分间的相对含量较非菌塘发生了变化,这种变化的结果使得各组分间的相对含量差异增大。最为典型的代表便是真菌与细菌的相对变化上,菌塘内真菌比例增加,细菌比例下降,导致菌塘内F/B显著增加(表2)。
分析发现尽管两种林型间的pH、全碳、全氮、全磷和碳氮比存在显著差异(表4),但菌塘和非菌塘的土壤化学特征基本相同,说明松茸主导菌塘后并未改变土壤的化学性质。该结果与Vaario等[26]在的芬兰的研究结果相同。最后,值得注意的是,仍有超过一半的PLFAs未能被鉴定(表2),在一定程度上限制了我们对菌塘微生物群落的全面理解,未来可结合其他研究手段,特别是结合下一代测序技术深入解析松茸菌塘的微生物群落特征。
4 结论
综上所述,松茸菌塘内的土壤微生物群落组成和结构特征与非菌塘存在差异,菌塘的真菌含量和F/B增加,细菌含量减少,表明松茸改变了菌塘的土壤微生物群落组成和结构,导致菌塘具有特定的微生物群落特征。此外,菌塘和非菌塘微生物群落特征的差异在高山栎和高山松林内表现相似,说明松茸对菌塘微生物群落结构的影响不随宿主森林类型而改变。
参考文献(References):
[1] Pennisi E. The secret life of fungi. Science, 2004, 304(5677): 1620- 1622.
[2] 高程, 郭良栋. 外生菌根真菌多样性的分布格局与维持机制研究进展. 生物多样性, 2013, 21(4): 488- 498.
[3] Van Der Heijden M G, Bardgett R D, Van Straalen N M. The unseen majority: soil microbes as drivers of plant diversity and productivity in terrestrial ecosystems. Ecology Letters, 2008, 11(3): 296- 310.
[4] Ogawa M. Microbial ecology of ‘Shiro’ inTricholomamatsutake(S. Ito et Imai) Sing. and its allied species. V.TricholomamatsutakeinTsugasieboldiiforests. Transactions of the Mycological Society of Japan, 1977, 18(1): 34- 46.
[5] Hamada M. General introduction toTricholomamatsutake(in Japanese)//The Matsutake Research Association, ed. Matsutake (TricholomamatsutakeSinger)-its Fundamental Studies and Economic Production of the Fruit-Body. Kyoto: The Matsutake Research Association, 1964: 6- 6.
[6] Endo N, Dokmai P, Suwannasai N, Phosri C, Horimai Y, Hirai N, Fukuda M, Yamada A. Ectomycorrhization ofTricholomamatsutakewithAbiesveitchiiandTsugadiversifoliain the subalpine forests of Japan. Mycoscience, 2015, 56(4): 402- 412.
[7] Ogawa M. Microbial ecology of ‘Shiro’ inTricholomamatsutake(S. Ito et Imai) Sing. and its allied species. II:TricholomamatsutakeinPinuspumilavar. yezoalpina forest. Transactions of the Mycological Society of Japan, 1976, 17: 176- 187.
[8] Ogawa M. Microbial ecology of ‘Shiro’ inTricholomamatsutake(S. Ito et Imai) Sing. and its allied species. III:TricholomamatsutakeinPiceaglehniiandPiceaglehniiAbiessachalinensisforests. Transactions of the Mycological Society of Japan, 1976, 17: 188- 198.
[9] Ogawa M. Microbial ecology of ‘Shiro’ inTricholomamatsutake(S. Ito et Imai) Sing. and its allied species. IV:TricholomamatsutakeinTsugadiversifoliaforests. Transactions of the Mycological Society of Japan, 1977, 18: 20- 33.
[10] Ogawa M. The Biology of Matsutake. Tokyo: Tsukiji-Shokan, 1978.
[11] Vaario L M, Pennanen T, Sarjala T, Savonen E M, Heinonsalo J. Ectomycorrhization ofTricholomamatsutakeand two major conifers in Finland—an assessment of in vitro mycorrhiza formation. Mycorrhiza, 2010, 20(7): 511- 518.
[12] Yamanaka K, Aimi T, Wan J, Cao H, Chen M J. Species of host trees associated withTricholomamatsutakeand close allies in Asia. Mushroom Science & Biotechnology, 2011, 19: 79- 87.
[13] 苏开美. 云南楚雄、中甸地区松茸生态环境调查研究. 中国食用菌, 2002, 21(3): 19- 20.
[14] Murata H, Yamada A, Yokota S, Maruyama T, Endo N, Yamamoto K, Ohira T, Neda H. Root endophyte symbiosis in vitro between the ectomycorrhizal basidiomyceteTricholomamatsutakeand the arbuscular mycorrhizal plantPrunusspeciosa. Mycorrhiza, 2014, 24(4): 315- 321.
[15] Murata H, Yamada A, Maruyama T, Neda H. Ectomycorrhizas in vitro betweenTricholomamatsutake, a basidiomycete that associates with Pinaceae, andBetulaplatyphyllavar. japonica, an early-successional birch species, in cool-temperate forests. Mycorrhiza, 2015, 25(3): 237- 241.
[16] Murata H, Yamada A, Maruyama T, Endo N, Yamamoto K, Hayakawa N, Neda H. In vitro shiro formation between the ectomycorrhizal basidiomyceteTricholomamatsutakeandCedrelaherreraein the Mahogany family (Meliaceae). Mycoscience, 2014, 55(4): 275- 279.
[17] 陈荣民, 张春凤, 张虎. 松茸生态及人工增产技术综述. 中国林副特产, 2003, (4): 21- 22.
[18] Tominaga Y.Tricholomamatsutake//Chang S T, Hayes W A, Eds. The Biology and Cultivation of Edible Mushrooms. New York: Academic Press, 1978: 683- 697.
[19] Yamada A, Kanekawa S, Ohmasa M. Ectomycorrhiza formation ofTricholomamatsutakeonPinusdensiflora. Mycoscience, 1999, 40(2): 193- 198.
[20] Yamada A, Maeda K, Kobayashi H, Murata H. Ectomycorrhizal symbiosis in vitro betweenTricholomamatsutakeandPinusdensifloraseedlings that resembles naturally occurring ‘shiro’. Mycorrhiza, 2006, 16(2): 111- 116.
[21] Vaario L M, Guerin-Laguette A, Gill W M, Lapeyrie F, Suzuki K. Only two weeks are required forTricholomamatsutaketo differentiate ectomycorrhizal Hartig net structures in roots ofPinusdensifloraseedlings cultivated on artificial substrate. Journal of Forest Research, 2000, 5(4): 293- 297.
[22] 李小林, 金鑫, 李强, 熊川, 郑林用. 生态环境因子对四川松茸菌塘土壤微生物的影响. 应用与环境生物学报, 2015, 21(1): 164- 169.
[23] Ohara H, Hamada M. Disappearance of bacteria from the zone of active mycorrhizas inTricholomamatsutake(S. Ito et Imai) Singer. Nature, 1967, 213(5075): 528- 529.
[24] Lian C L, Narimatsu M, Nara K, Hogetsu T.Tricholomamatsutakein a naturalPinusdensifloraforest: correspondence between above- and below-ground genets, association with multiple host trees and alteration of existing ectomycorrhizal communities. New Phytologist, 2006, 171(4): 825- 836.
[25] Kataoka R, Siddiqui Z A, Kikuchi J, Ando M, Sriwati R, Nozaki A, Futai K. Detecting nonculturable bacteria in the active mycorrhizal zone of the pine mushroomTricholomamatsutake. The Journal of Microbiology, 2012, 50(2): 199- 206.
[26] Vaario L M, Fritze H, Spetz P, Heinonsalo J, Hanajík P, Pennanen T.Tricholomamatsutakedominates diverse microbial communities in different forest soils. Applied and Environmental Microbiology, 2011, 77(24): 8523- 8531.
[27] 马大龙, 臧淑英, 李莹. 基于磷脂脂肪酸生物标记法的松茸生长土壤微生物测定. 贵州农业科学, 2015, 43(3): 106- 109.
[28] Kim M, Yoon H, Kim Y E, Kim Y J, Kong W S, Kim J G. Comparative analysis of bacterial diversity and communities inhabiting the fairy ring ofTricholomamatsutakeby barcoded pyrosequencing. Journal of Applied Microbiology, 2014, 117(3): 699- 710.
[29] Kim M, Yoon H, You Y H, Kim Y E, Woo J R, Seo Y, Lee G M, Kim Y J, Kong W S, Kim J G. Metagenomic analysis of fungal Communities Inhabiting the Fairy Ring Zone ofTricholomamatsutake. Journal of Microbiology and Biotechnology, 2013, 23(10): 1347- 1356.
[30] 张秋芳, 刘波, 林营志, 史怀, 杨述省, 周先冶. 土壤微生物群落磷脂脂肪酸PLFA生物标记多样性. 生态学报, 2009, 29(8): 4127- 4137.
[31] Bossio D A, Scow K M. Impacts of carbon and flooding on soil microbial communities: Phospholipid fatty acid profiles and substrate utilization patterns. Microbial Ecology, 1998, 35(3): 265- 278.
[32] 戈峰. 现代生态学. 北京: 科学出版社, 2002: 252- 254.
[33] Wu J P, Liu Z F, Wang X L, Sun Y X, Zhou L X, Lin Y B, Fu S L. Effects of understory removal and tree girdling on soil microbial community composition and litter decomposition in two Eucalyptus plantations in South China. Functional Ecology, 2011, 25(4): 921- 931.
[34] Bossio D A, Fleck J A, Scow K M, Fujii R. Alteration of soil microbial communities and water quality in restored wetlands. Soil Biology and Biochemistry, 2006, 38(6): 1223- 1233.
[35] Vestal J R, White D C. Lipid analysis in microbial ecology-quantitative approaches to the study of microbial communities. Bioscience, 1989, 39: 535- 541.
[36] Zelles L. Phospholipid fatty acid profiles in selected members of soil microbial communities. Chemosphere, 1997, 35(1/2): 275- 294.
[37] Frostegård Å, Tunlid A, Bååth E. Phospholipid fatty acid composition, biomass, and activity of microbial communities from two soil types experimentally exposed to different heavy metals. Applied and Environmental Microbiology, 1993, 59(11): 3605- 3617.
[38] Olsson P A. Signature fatty acids provide tools for determination of the distribution and interactions of mycorrhizal fungi in soil. FEMS Microbiology Ecology, 1999, 29(4): 303- 310.
[39] Fierer N, Schimel J P, Holden P A. Variations in microbial community composition through two soil depth profiles. Soil Biology and Biochemistry, 2003, 35(1): 167- 176.
[40] Parker J H, Smith G A, Fredrickson H L, Vestal J R, White D C. Sensitive assay, based on hydroxy fatty acids from lipopolysaccharide lipid A, for Gram-negative bacteria in sediments. Applied and Environmental Microbiology, 1982, 44(5): 1170- 1177.
[41] Wilkinson S. Gram-negative bacteria//Ratledge C, Wilkinson S G, eds. Microbial Lipids. London: Academic Press, 1988, 1: 299- 488.
[42] Takakura Y.Tricholomamatsutakefruit bodies secrete hydrogen peroxide as a potent inhibitor of fungal growth. Canadian Journal of Microbiology, 2015, 61(6): 447- 450.
[43] Park M S, Oh S Y, Cho H J, Fong J J, Cheon W J, Lim Y W.Trichodermasongyisp. nov., a new species associated with the pine mushroom (Tricholomamatsutake). Antonie van Leeuwenhoek, 2014, 106(4): 593- 603.
[44] Kluber L A, Smith J E, Myrold D D. Distinctive fungal and bacterial communities are associated with mats formed by ectomycorrhizal fungi. Soil Biology and Biochemistry, 2011, 43(5): 1042- 1050.

