长链非编码RNA作为肝细胞癌诊断新型血清标志物的meta分析
王秀月,赵 川,陈 彻,王 晶,胡立冬
甘肃中医药大学临床医学院,甘肃 兰州 730000
肝细胞癌(hepatocellular carcinoma,HCC)是常见的恶性肿瘤之一,其发病率居全球第5位,死亡率居第3位。HCC是一种高侵袭性和高致命性肿瘤,容易复发和转移,且预后较差[1]。HCC的发生、发展过程非常复杂,涉及很多基因和通路。因此,治疗HCC是全球性问题。根治性手术通常被用于HCC患者的治疗,但是对于失去手术机会的患者这是一个难题[2]。近年来,随着对基因的不断认识和对长链非编码RNA(long noncoding RNA,lncRNA)的研究,越来越多的证据表明,HCC患者中lncRNA的表达水平发生了重大变化[3]。
LncRNA是一类长度超过200个核苷酸的非编码RNA,在调控基因表达方面具有重要的生物学功能[4]。近年来有研究表明,lncRNA在肿瘤的发生、发展过程中起促进肿瘤或肿瘤抑制因子的作用,参与细胞凋亡、肿瘤浸润和转移过程[5]。正常组织和肿瘤组织中lncRNA的差异性表达可被用作肿瘤预防、治疗和预后的指标。有文献已经通过meta分析的方法报告了lncRNA与肿瘤预后的关系[6]。
目前,仅有极少数的研究通过meta分析的方法评价肿瘤诊断中lncRNA的整体价值[6]。肝癌患者血清中能够检测到lncRNA,这为肝癌诊断提供了新的途径。该研究在全面文献检索的基础上,运用meta分析的方法评价肝癌患者中lncRNA的整体诊断价值。
1 资料和方法
1.1 文献检索
使用计算机检索PubMed、EMbase、Cochrane Library、CBM、CNKI及万方数据库相关的中英文文献,同时通过其他资源(谷歌学术和百度学术)补充获得相关文献,检索时限均从建库至2017年2月。检索词包括lncRNA、肝癌、血清、诊断灵敏度、特异度、受试者工作特征(receiver operating characteristic,ROC)曲线和曲线下面积(area under curve,AUC)。
1.2 纳入和排除标准
所有研究均由2名研究员通过题目和摘要独立进行初步筛选,然后通过阅读全文筛选满足纳入和排除标准的研究。通过讨论或咨询第3名研究员来解决有分歧的地方。此外,如果有必要可联系原作者来获取缺失的数据。文献纳入标准如下:HCC是基于组织学检查,与之匹配的对照组检查结果阴性且无其他肿瘤史,所有的血样均在病理检查和治疗前收集,研究人员单独评估血样中的lncRNA,研究应包含灵敏度、特异度或从数据中能够获得这些数值。排除标准如下:重复发表的研究,系统评价、会议摘要、案例报告和评论,不合格的患者、对照及其血样,数据不充分,样本量小于20。
1.3 数据提取和质量评价
从每项研究中提取的数据包括第一作者、发表时间、国家、对照来源、年龄、样本量、检测方法、lncRNA表达模式和诊断结果。任何分歧通过讨论或咨询第3名研究员来解决。2名研究员采用Cochrane协作网推荐的诊断准确性研究质量评价工具(Quality Assessment of Diagnostic Accuracy Studies 2,QUADAS-2)独立评价每个研究的偏倚风险[7]。QUADAS-2主要由病例的选择、待评价试验、金标准、病例流程和进展情况组成。根据每部分纳入的相关标志性问题的回答为“是”、“否”或“不确定”,可对应将偏倚风险等级判断为“低”、“高”或“不确定”。
1.4 统计学处理
该研究采用Stata 12和Meta-Disc 1.4对纳入文献进行统计学分析。选取灵敏度、特异度、阳性似然率、阴性似然率和ROC效应指标进行该诊断试验的meta分析。采用Q统计量和I2检验评估异质性,如果P<0.05(Q统计)或I2>50%,则采用随机效应模型计算合并效应量,否则采用固定效应模型。对于发表偏倚,采用Deeks检验来评估纳入的研究,P<0.05为差异有统计学意义。
2 结 果
2.1 文献检索和研究的基本特征
共检索到385篇相关的研究,通过认真阅读题目和摘要,228篇研究由于是系统评价、病例报告或基础研究等被进一步排除。全文阅读剩下的39篇研究,26篇研究由于数据不完整、对照标准不明确或信件被排除。因此,在剩余的13篇研究中很多lncRNA被纳入该meta分析[8-20],如UCA1、MALAT1、HULC、Linc00152、uc003wbd、AF085935、RP11-160H22.5、XLOC_014172、LOC149086、uc001ncr、AX800134、WRAP53、SPRY4-IT1、GAS5、H19、HEIH和JPX。筛选流程图见图1。纳入研究的基本信息见表1。所有符合纳入标准的研究均发表在2013—2017年,其中HCC患者1 296例,健康对照者1 304人。病理检查被视为诊断HCC的金标准。纳入研究的基本特征包括第一作者、发表年份、国家、患者和对照者的数量、年龄、分析类型、内参、临界值、lncRNA名称、灵敏度、特异度及ROC曲线的AUC。
2.2 质量评估
使用QUADAS-2评估纳入研究的质量,发现纳入的所有研究的质量较高(图2)。然而,在这些纳入的研究中存在一个明显的偏倚,即在“index text”中存在明显的缺陷,占46%(6/13)。
2.3 异质性分析和阈值效应
为评估纳入研究的lncRNA是否存在异质性,用Spearman检验计算真阳性率的对数值与假阳性率的对数值之间的相关系数和P值来排除阈值效应。Spearman相关系数为0.191(P=0.478),表明不存在阈值效应。诊断比值比的Cochran-Q值为64.21,I2值为81.3%,P<0.1,表明不存在阈值效应。上述结果表明,研究间存在异质性,可能与人群、年龄等有关;因此,在该meta分析中应用DerSimonian-Laird法。

图1 文献筛选流程及结果Fig. 1 Flow diagram of study selection process
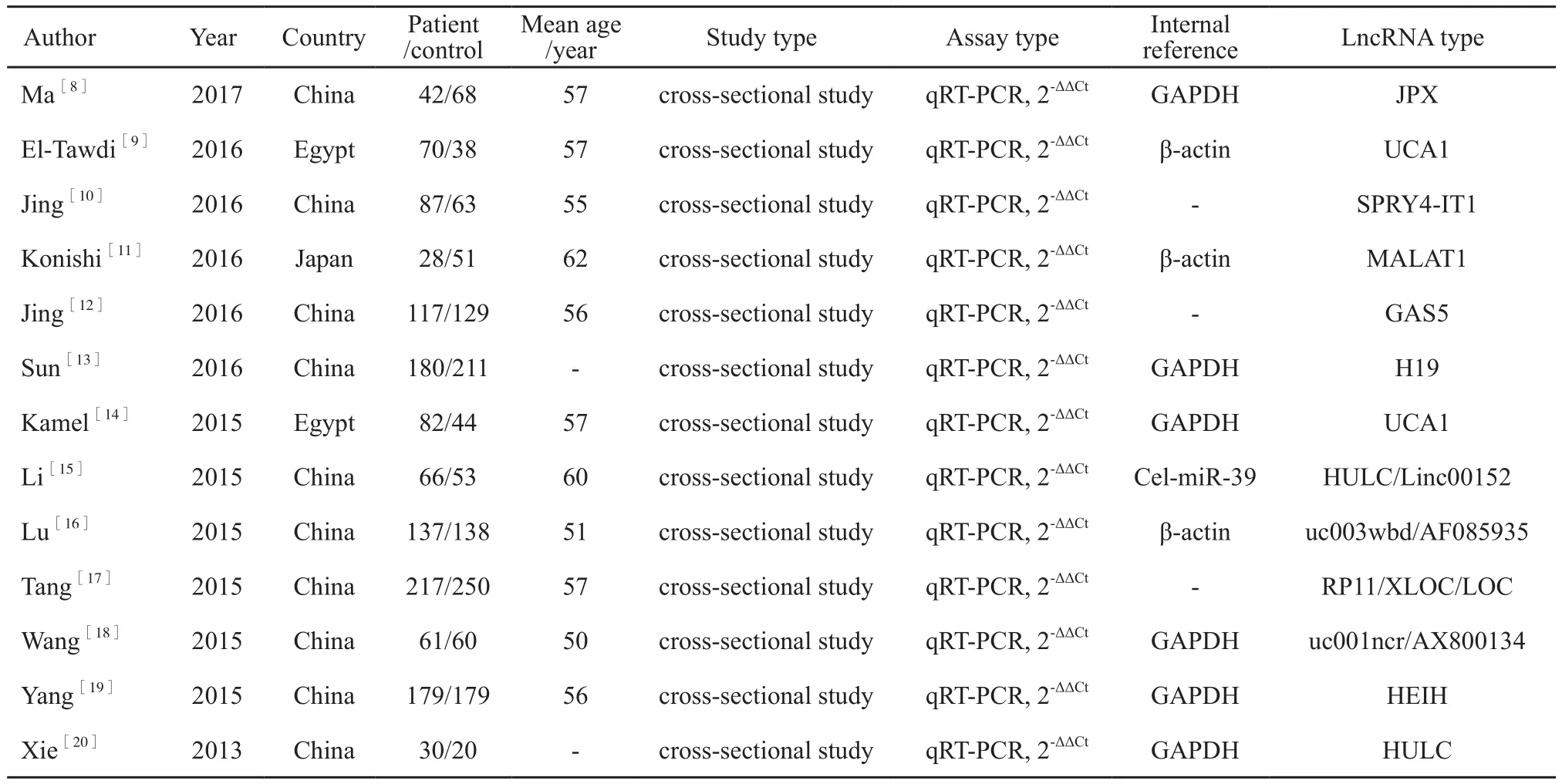
表1 纳入研究的基本特征Tab. 1 General characteristics of included studies
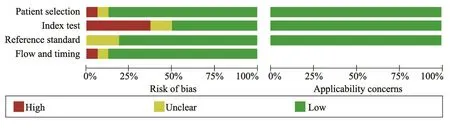
图2 采用QUADAS-2对纳入研究进行质量评价Fig.2 Quality assessment of the included studies by QUADAS-2
2.4 诊断价值评价
随机效应模型用于评价lncRNA在诊断HCC中的整体效果(图3)。整体lncRNA诊断HCC的灵敏度为83%(95%CI:81%~85%),特异性为82%(95%CI:80%~84%),阳性似然比为5.32(95%CI:3.37~8.42),阴性似然比是0.21(95%CI:0.15~50.28),诊断比值比为30.38(95%CI:16.76~55.05)。ROC曲线的AUC为0.91(95%CI:0.88~0.93),具体见图4。上述结果表明,lncRNA对HCC具有较好的诊断价值。然而,因为由非阈值引起的异质性能够在诊断指标的森林图中清晰地观察到,我们试图利用meta回归(如国家、年龄、内参和临界值)来探索研究的特点。因为在纳入的研究中,lncRNA均取自HCC患者和健康人群的血液,标本类型和对照不是异质性的来源。如果协变量有缺失值,在meta回归中用0代替。根据国家、年龄和内参做meta回归显示,P>0.05,表明未找到异质性的来源(表2)。研究的总体分布总结见图5,Fagan列线图描述了lncRNA检测确定或排除肿瘤患者的可能性。对于任何检测前有20%患HCC可能性的人来说,如果在肿瘤检测中lncRNA是阳性,则患HCC的检测后概率将达到56%;然而,lncRNA检测结果阴性则意味着检测后概率将降至4%。因此,lncRNA检测在初始筛查HCC中发挥着重要作用。
2.5 发表偏倚
发表偏倚被认为是影响诊断准确性的又一因素。Deeks检验用于评估纳入文献是否存在发表偏倚。Deeks检验的P=0.53,表明lncRNA在研究中没有发表偏倚(图6)。
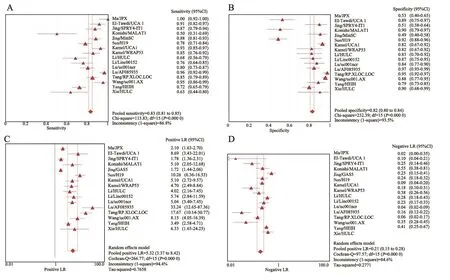
图3 森林图展示lncRNA诊断HCC的合并诊断指标Fig. 3 Results of pooled diagnostic performance for lncRNAA: Sensitivity; B: Specificity; C: Positive LR; D: Negative LR. LR: Likelihood ratio
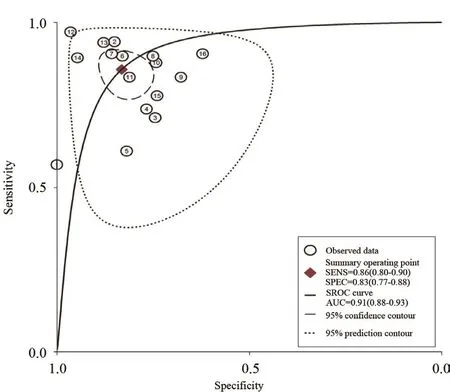
图4 LncRNA诊断HCC的ROC曲线Fig. 4 The ROC curve for lncRNA expression in the diagnosis of HCC
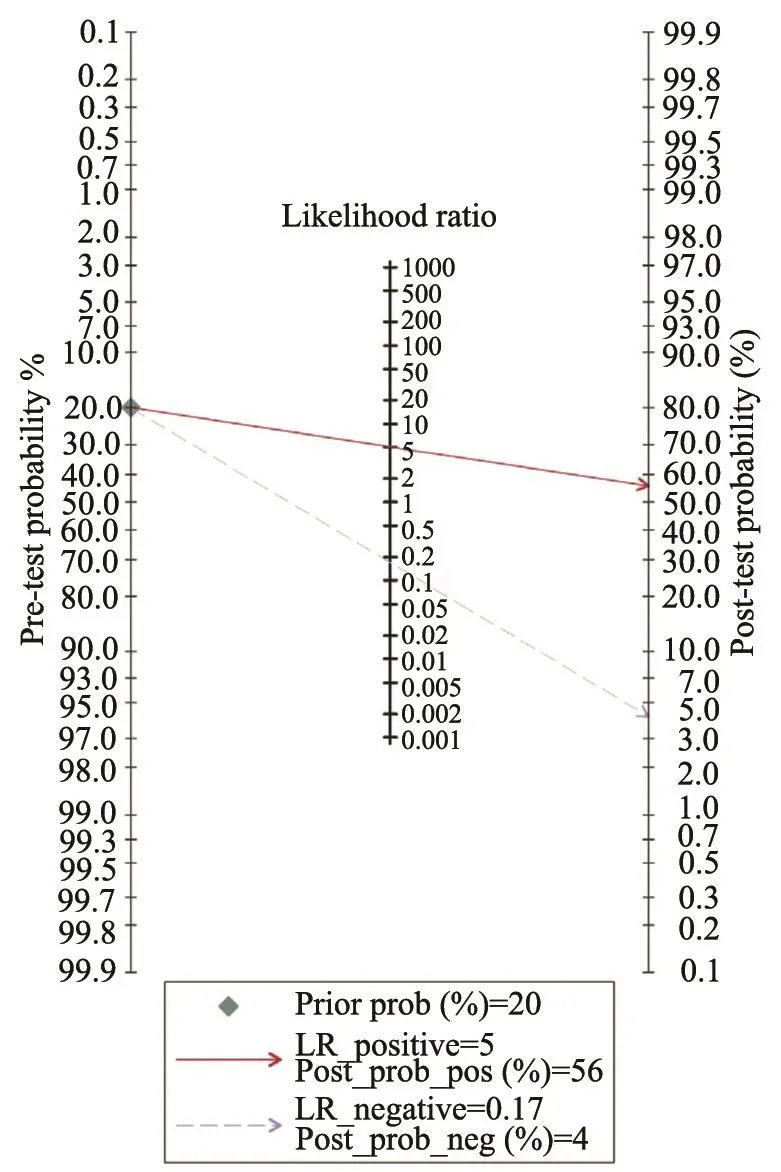
图5 lncRNA诊断HCC的Fagan列线图Fig. 5 Fagan’s nomogram describes the ability of lncRNA assay to confirm or exclude HCC patients
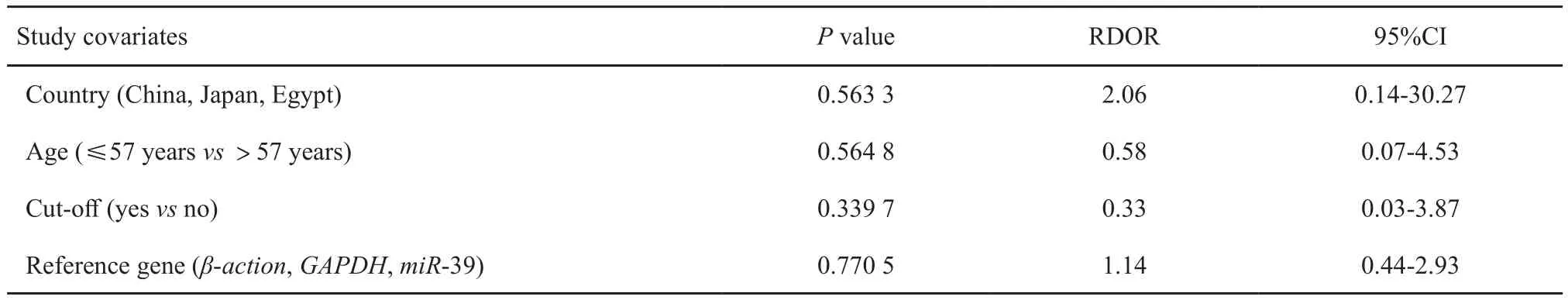
表2 Meta回归检测异质性的潜在来源Tab. 2 Meta-regression for the potential source of heterogeneity

图6 lncRNA诊断HCC来自Deeks检验的发表偏倚Fig. 6 Publication bias from Deek’s test for lncRNA expression in the diagnosis of HCC
3 讨 论
随着基因测序的完成,发现仅有1%~2%的基因具有编码蛋白的功能,大多数非编码序列被转录成非编码RNA,80%~90%具有非蛋白编码基因的转录活性,即lncRNA[21]。尽管有大量的lncRNA,但有关lncRNA的研究远少于microRNA。与lncRNA相比,microRNA更早被发现。目前,lncRNA研究还处于起步阶段。最近的研究表明,lncRNA能够调节转录和表观遗传学水平的重要细胞信号通路[22]。很多lncRNA具有调节细胞稳态和增殖的作用,一些lncRNA可能在细胞凋亡中发挥调节作用[23]。LncRNA调控基因表达,主要包括染色质修饰、转录和转录后处理。LncRNA与肿瘤的相关证据主要来源于表达差异。仅有少量lncRNA的机制是清楚的,可能是由于缺乏大量的临床研究样本。
近年来,尽管诊断方法和外科技术的发展显著改善了HCC患者的预后,但晚期HCC患者的5年生存率仍然较差。诊断性生物标志物缺乏导致了HCC早期检测的延迟。迄今为止,很多HCC检测的非侵入性血液标志物都是可用的,如甲胎蛋白、γ-谷氨酸转移酶、去羧基凝血酶原、肝细胞生长因子、热休克蛋白70和白介素-6,目前被用于检测HCC[24]。尽管如此,由于诊断准确性相对较低,这些生物标志物用于确诊HCC并不是很理想。另一方面,病理诊断是HCC检测最可靠的诊断方法,但是会造成不同程度的损伤,并且手术费用较高。因此,找出HCC筛查的新型诊断生物标志物是必需的。目前,lncRNA在很多肿瘤(包括HCC)中表现出诊断价值。此外,很多学者有兴趣通过meta分析对肿瘤诊断和预后的生物标志物间的关系进行全面综合研究。已有研究通过meta分析的方法比较碱性磷酸酶与骨肉瘤预后的关系[25]。
本研究发现血液中整体lncRNA的不一致表达水平在HCC患者和健康对照者之间差异有统计学意义。通过筛选有13篇文献被纳入。因为没有详细描述病理和血液中检测lncRNA之间的时间间隔,可以忽略由于疾病过程而导致的检测结果的偏差。异质性是meta分析中一个重要的参考因素,阈值效应是诊断性meta分析异质性中最重要的因素。该研究采用Spearman模型的相关系数来明确13个研究的不同阈值是否是由异质性引起的,森林图和I2值表明存在异质性。因此,使用meta回归分析来确定异质性的可能原因。Meta回归分析显示,在所有指定的协变量中,P>0.1,表明未找到异质性的来源。因此,采用随机效应模型合并分析其诊断价值。纳入lncRNA在HCC诊断价值中的联合指标,如灵敏度、特异度、阳性似然率、阴性似然率和诊断比值比。ROC曲线的AUC显示,lncRNA在HCC中具有良好的诊断价值。在分析HCC患者和健康对照者之后,lncRNA的ROC曲线的AUC为0.91,灵敏度为83%,特异度为82%,表明其具有潜在的HCC的非侵入性诊断价值。诊断比值比作为诊断效率与病例之间的紧密指标,具有极高的检测价值。本研究中,诊断比值比为30.38,表明其诊断HCC具有更好的诊断准确性。
综上所述,本研究表明,lncRNA表达谱显示出区分HCC患者和无肿瘤人群更好的诊断准确性。LncRNA分析在HCC管理中显示出很高的价值。如果在大规模研究中得到验证,lncRNA有望成为一个HCC临床实践的非侵入性筛查工具。当然,需要更多的研究来明确lncRNA作为HCC发生、发展补充检测标志物的价值。
[参考文献]
[1] MILLER K D, SIEGEL R L, LIN C C, et al. Cancer treatment and survivorship statistics, 2016 [J]. CA Cancer J Clin, 2016,66(4): 271-289.
[2] 李强, 陈良, 黄玉仙. 肝细胞癌治疗新策略—表观遗传治疗 [J]. 临床肝胆病杂志, 2015, 31(6): 870-875.
[3] HUANG J L, ZHENG L, HU Y W. Characteristics of long noncoding RNA and its relation to hepatocellular carcinoma [J].Carcinogenesis, 2014, 35(3): 507-514.
[4] 洪陈亮. 长链非编码RNA [J]. 中南医学科学杂志,2016(5): 598-598.
[5] CHANG Y, ZHANG K, HU Z, et al. Hypoxia-regulated lncRNAs in cancer [J]. Gene, 2016, 575(1): 1-8.
[6] SERGHIOUS S, KYRIAKOPOULOU A, IOANNIDIS J P.Long noncoding RNAs as novel predictors of survival in human cancer: a systematic review and meta-analysis [J]. Mol Cancer, 2016, 15(1): 50.
[7] WHITING P F, RUTJES A W, WESTWOOD M E, et al.QUADAS-2: a revised tool for the quality assessment of diagnostic accuracy studies [J]. Ann Intern Med, 2011,155(8): 529-536.
[8] MA W, WANG H, JING W, et al. Downregulation of long non-coding RNAs JPX and XIST is associated with the prognosis of hepatocellular carcinoma [J]. Clin Res Hepatol Gastroenterol, 2017, 41(2): 163-170.
[9] EL-TAWDI A H, MATBOLI M, EL-NAKEEP S, et al.Association of long noncoding RNA and c-JUN expression in hepatocellular carcinoma [J]. Expert Rev Gastroenterol Hepatol, 2016, 10(7): 869-877.
[10] JING W, GAO S, ZHU M, et al. Potential diagnostic value of lncRNA SPRY4-IT1 in hepatocellular carcinoma [J]. Oncol Rep, 2016, 36(2): 1085-1092.
[11] KONISHI H, ICHIKAWA D, YAMAMOTO Y, et al. Plasma level of metastasis-associated lung adenocarcinoma transcript 1 is associated with liver damage and predicts development of hepatocellular carcinoma [J]. Cancer Sci, 2016, 107(2): 149-154.
[12] 景伟, 罗萍, 朱满, 等. 血浆长链非编码RNA GAS5在肝细胞癌中表达水平及治疗的评估 [J]. 中华检验医学杂志, 2016, 39(8): 599-603.
[13] 孙一奎, 赵枫, 王峰, 等. 血浆长链非编码RNA H19在原发性肝癌诊断中的价值 [J]. 中国卫生检验杂志, 2016,26(3): 305-307.
[14] KAMEL M M, MATBOLI M, SALLAM M, et al. Investigation of long noncoding RNAs expression profile as potential serum biomarkers in patients with hepatocellular carcinoma [J].Transl Res, 2016, 168: 134-145.
[15] LI J, WANG X, TANG J, et al. HULC and linc00152 act as novel biomarkers in predicting diagnosis of hepatocellular carcinoma [J]. Cell Physiol Biochem, 2015, 37(2): 687-696.[16] LU J, XIE F, GENG L, et al. Investigation of serum lncRNAuc003wbd and lncRNA-AF085935 expression profile in patients with hepatocellular carcinoma and HBV [J].Tumour Biol, 2015, 36(5): 3231-3236.
[17] TANG J, JIANG R, DENG L, et al. Circulation long noncoding RNAs act as biomarkers for predicting tumorigenesis and metastasis in hepatocellular carcinoma[J]. Oncotarget, 2015,6(6): 4505-4515.
[18] WANG K, GUO W X, LI N, et al. Serum lncRNAs profiles serve as novel potential biomarkers for the diagnosis of HBV-positive hepatocellular carcinoma[J]. PLoS ONE, 2015, 10(12):e144934.
[19] 杨哲峰, 李娟, 马志元, 等. 血浆lncRNA-HEIH表达水平与肝癌发病风险的关联分析 [J]. 现代肿瘤医学, 2015,23(1): 80-84.
[20] XIE H, MA H, ZHOU D. Plasma HULC as a promising novel biomarker for the detection of hepatocellular carcinoma [J].Biomed Res Int, 2013, 2013: 136106.
[21] LI C H, CHEN Y. Targeting long non-coding RNAs in cancers:progress and prospects [J]. Int J Biochem Cell Biol, 2013,45(8): 1895-1910.
[22] ESPOSTI D D, HERNANDEZ-VARGAS H, VOEGELE C, et al. Identification of novel long non-coding RNAs deregulated in hepatocellular carcinoma using RNA-sequencing [J].Oncotarget, 2016, 7(22): 31862-31877.
[23] CHAUHAN R, LAHIRI N. Tissue- and serum-associated biomarkers of hepatocellular carcinoma [J]. Biomark Cancer,2016, 8(Suppl 1): 37-55.
[24] LI Z, YOU K, LI J, et al. Madecassoside suppresses proliferation and invasiveness of HGF-induced human hepatocellular carcinoma cells via PKC-cMET-ERK1/2-COX-2-PGE2 pathway [J]. Int Immunopharmacol, 2016, 33: 24-32.
[25] HAO H, CHEN L, HUANG D, et al. Meta-analysis of alkaline phosphatase and prognosis for osteosarcoma [J]. Eur J Cancer Care (Engl), 2016, 26(5).

