不同基因型戊型肝炎病毒株宿主选择性差异的基因突变分析
王晨 付红伟 杨桂芳
戊型肝炎病毒(hepatitis E virus,HEV)是一种单链无包膜的小RNA病毒,在我国卫生环境较差地区是急性病毒性肝炎的最主要致病因素[1]。随着对戊型肝炎(戊肝)研究的不断深入,戊肝是人畜共患病的事实已为科研工作者所公认,然而相关报道显示,不同基因型HEV病毒株存在着明显的宿主选择性及致病性强弱的差异[2]。即基因3型和4型HEV病毒株既能感染人又能感染猪、兔、鼠等哺乳类动物,而基因1型和2型HEV病毒株却仅能感染人和非人灵长类动物,不能跨物种传播[3]。据其致病性强弱特点分析,基因1型和2型的致病性较基因3型和4型强[4]。在近年来对HEV衣壳蛋白的晶体结构、其所编码蛋白的功能分析、病毒组装与进入宿主细胞的过程致病性影响因素的研究取得诸多进展的基础上,本研究通过对现有已知HEV全基因组序列的比对分析,寻找影响两大类HEV病毒株间宿主选择性及致病性差异的可疑基因突变决定子。
1 资料与方法
从DNA序列GenBank数据库获取87株背景较为资料清楚的HEV病毒株核苷酸序列,包含基因1~4型人源类或人畜共患类HEV病毒株,见表1。鉴于兔HEV尚不确定是否能够跨物种传播,因此未纳入本次研究分析。
上述87株HEV病毒株分为两大类,一类为仅感染人的人源病毒类(human,H类),包含基因1型和2型HEV病毒株;另一类为广泛分离自人、猪、鹿等多种哺乳类动物,且可跨物种感染人的人畜共患类(zoonosis,Z类),包含基因3型和4型HEV病毒株。Z类HEV病毒株可引起急性散发性戊肝,已被广泛认为是一种人畜共患病的病原体[5]。
利用AlingX软件(Vector NTI软件包)对上述两类HEV核苷酸或编码氨基酸进行序列比对分析,寻找两类HEV病毒株间特异性的基因突变位点,并结合HEV病毒株基因组和编码蛋白的结构特点及功能特点进行综合分析,寻找可能影响不同基因型HEV的宿主选择性及致病性强弱的位点。
2 结果
2.1 5'UTR区的分析 5'UTR区长度为26 nt左右,相对较为保守。两类HEV病毒株间在此区域内仅发现G11A/C(位点参照GU11961)一个特异的核苷酸突变位点,即Z类HEV基因组中第11核苷酸位点均为G,而H类HEV基因组中该位点核苷酸为A或C。见图1。

图1 不同基因型HEV病毒株5'UTR区基因序列比对分析
2.2 ORF1区的分析 在ORF1区,Z类HEV病毒株编码1705-1707位氨基酸(aa),H类HEV病毒株编码1692-1693位氨基酸(aa)。两类病毒株编码氨基酸片段的长短不同主要是由其高变区(711-798aa)的长度不同引起的。在ORF1区域内共发现16个氨基酸置换位点,其中应关注的是位于高变区5'端的9个连续氨基酸位点的置换,其在Z类HEV病毒株中均为 SGFSSD/CFSP(708-716aa),而在 H 类HEV病毒株中相应的位点则分别为VDAVS(基因1型)或ITDTP(基因2型)。见表2。
2.3 ORF2区的分析 基因4型HEV病毒株的ORF2区编码约674个氨基酸,较基因1~3型多编码约14个氨基酸。ORF2区较ORF1区更为保守,有7个氨基酸置换位点在ORF2区域内被发现,见表3。
2.4 ORF3区的分析 基因4型HEV病毒株的ORF3区编码约114个氨基酸,较基因1~3型少编码约9个氨基酸。在ORF3区域内,仅发现E78D一个特异的氨基酸位点突变。
2.5 3'UTR区的分析 去除poly(A)尾后,3'UTR区由65~72个核苷酸组成,该区域有诸多核苷酸缺失或插入,变异性较大。在3'UTR区,所有Z类病毒株在poly(A)尾前较H类病毒株多一个核苷酸G。

表1 87株已知全基因组序列的HEV病毒株的分类和基因分型

表2 ORF1区H类和Z类HEV病毒株16个氨基酸位点置换情况
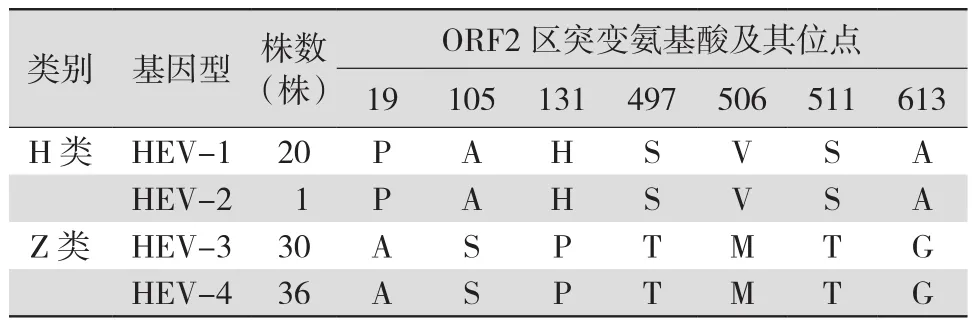
表3 ORF2区H类和Z类HEV病毒株7个氨基酸位点置换情况
3 讨论
本研究利用ALIGN软件比对分析87株基因1~4型HEV病毒株的基因组序列或氨基酸序列,以寻找影响H类和Z类HEV病毒株宿主选择性差异的突变位点。结果显示,发现26个特异的突变位点可能参与HEV宿主选择,其中在5'UTR区发现了G11A/C突变位点。先前报道显示,5'UTR区和ORF1区5'端58个核苷酸一起组成了保守的二级结构(颈环和发卡结构),该二级结构可通过与ORF2编码蛋白的结合,在HEV病毒衣壳的组装过程中发挥重要作用[6]。本研究发现的G11A/C突变位点可能会影响到其HEV基因组5'端的二级结构发生变化,进而可能影响到病毒衣壳的组装,并由此影响到宿主选择性的不同。另外,通过与甲型肝炎病毒(hepatitis A virus,HAV)核苷酸序列的类比可以发现类似现象,即HAV 5'UTR区的基因位点突变与甲型肝炎疾病的严重程度密切相关[7]。而本研究中发现的5'UTR区的G11A/C突变位点是否影响到两类HEV病毒株间的致病性差异亦值得进一步分析研究。
HEV病毒株的ORF1区编码了一个包含多个功能区的非结构蛋白,如甲基转移酶、木瓜样蛋 白 酶(apain-like protease,PLP)、RNA 解 旋 酶(RNA helicase)和RNA依赖的RNA聚合酶(RNA-dependent RNA polymerase,RdRp)等[8]。 本 研 究在ORF1区共发现16个氨基酸置换位点。这些氨基酸位点的突变可能会引起蛋白质功能的改变,而各个位点的突变对病毒株究竟会引起什么样的改变则取决于它们所在功能区的具体功能。例如甲基转移酶区核苷酸编码的蛋白与甲基帽结构(m7GpppX)的形成有关,具体负责将m7GTP添加到 mRNA 的 5'端[9],而本研究发现的 R40H、L62F和A83F位于甲基转移酶区,可能会导致两类病毒株添加甲基化帽的能力强弱有所不同,而甲基化帽结构又是HEV传染力的必要基础[10],从而可以推测发生在甲基转移酶区三个突变可能通过影响甲基化帽的形成对HEV病毒株传染力产生作用。HEV基因组在ORF1区存在一个高变区,既往研究结果提示,将部分或整个ORF1区内高变区完全删除的HEV诱导突变株毒力会减弱[11]。本研究在此高变区5'端发现,708-716位点处Z类病毒株氨基酸均为SGFSSD/CFSP,而在H类病毒株为VDAVS(基因1型)或ITDTP(基因2型),这是发生在高变区的连续9个氨基酸位点的突变,有必要建立删除9个氨基酸位点的HEV突变株,以进一步证实这9个连续氨基酸位点置换对HEV病毒株毒力强弱的影响。此外,在ORF1区发现的16个氨基酸突变位点中,有8个坐落在RdRp区,而RdRp区在单股正链RNA病毒间是十分保守的区域,常被作为遗传进化标志。因此,如此高频率的特异性氨基酸突变发生在RdRp区,可以间接提示,Z类和H类HEV病毒株可能是各自相对独立进化而来的。
HEV病毒株ORF2区编码病毒衣壳蛋白,HEV衣壳负责与宿主细胞结合从而引发感染过程[12-13]。Li等[14]报道了 HEV 衣壳蛋白 E2s区域(氨基酸455-602)的三维晶体结构,并对其功能进行了研究,该报道显示,HEV中和抗体的识别位点也在E2s区域,且E2s蛋白是与宿主细胞相互作用的基础。本研究在ORF2区发现了7个特异的氨基酸置换位点,其中有4个(T497S、M506V、T511S和G613A)位于E2s区。此外,本研究还发现,T511S核苷酸位点突变与郭清顺等[15]研究报道的ORF2蛋白第511位点的差异会影响基因1型和基因4型HEV病毒株的宿主选择的观点是一致的。鉴于以上分析,很有必要通过基因位点敲除突变或重组定点突变的方式,进一步研究证实这4个发生在E2s区的突变是否真正影响了H类和Z类HEV病毒株间的宿主选择差异。
HEV病毒株ORF3区含编码114(基因4型)或编码123(基因1~3型)氨基酸的蛋白(pORF3),pORF3通过激活细胞信号通路(ERK通路),能发挥延长内膜信号进而减弱宿主细胞的凋亡途径等作用,促进宿主细胞存活;同时也可减少急性时相反应蛋白的表达和提高α1-微球蛋白的分泌,降低宿主对HEV病毒株的天然免疫反应,在HEV病毒株侵入宿主的致病过程中发挥了重要影响作用[16]。本研究在此区域仅发现E78D这一个氨基酸位点的置换,该突变是否能够通过影响pORF3而触发强弱不同的细胞信号通路反应或改变宿主细胞的天然免疫反应状态,最终影响两类HEV病毒株间的致病性差异,值得我们进一步研究。
HEV病毒株3'UTR区是高突变区,Agrawal等[17]报道显示,3'UTR与和它相邻的ORF2的3'末端共同组成二级结构(颈环状),该二级结构既负责与HEV RdRp酶结合,也参与宿主蛋白的反应。鉴于此报道结果,3'UTR区可能会参与HEV病毒株与宿主细胞的结合过程。然而Graff等[18]报道结果却显示,将来源于猪HEV病毒株的3'UTR区重组到基因1型人源HEV病毒株形成新的突变镶嵌病毒体,依然不能使该人源HEV镶嵌病毒体成功感染猪。该报道结果说明,HEV的3'UTR区对HEV病毒株的宿主选择起不到决定性作用。本研究发现,Z类HEV病毒株的3'UTR区较H类病毒株多一个核苷酸G,据前期研究报道来看,该突变对HEV宿主选择所发挥的影响作用可能较为有限,不能作为未来研究的重点。
综上所述,本研究发现了26个基因突变位点可能参与影响Z类和H类病毒株的宿主选择,由于目前对HEV进入宿主细胞机制的研究报道甚少,因此很难确定究竟是哪几个基因位点的置换对HEV的宿主选择及致病性差异起到了决定性作用。但是本研究为确定两类病毒株间宿主选择及致病性差异提供了重要的线索,有利于后续实验研究方案的设计。
1 张海明.肝移植患者的戊型肝炎病毒感染[J/CD].实用器官移植电子杂志,2015,3(4):241-245.
2 付红伟,杨桂芳,李昕.我国戊型肝炎流行病学新特点分析[J].实用检验医师杂志,2012,4(3):184-188.
3 Aggarwal R,Jameel S. Hepatitis E[J]. Hepatology, 2011,54(6):2218-2226.
4 Purcell RH,Emerson SU. Hepatitis E: an emerging awareness of an old disease[J]. J Hepatol, 2008,48(3):494-503.
5 付红伟,杨桂芳,李昕.两株猪HEV病毒株全基因组序列分析[J].实用检验医师杂志,2012,4(2):84-91.
6 Surjit M,Jameel S,Lal SK. The ORF2 protein of hepatitis E virus binds the 5' region of viral RNA[J]. J Virol, 2004,78(1):320-328.
7 Fujiwara K,Yokosuka O,Ehata T,et al. Association between severity of type A hepatitis and nucleotide variations in the 5' non-translated region of hepatitis A virus RNA: strains from fulminant hepatitis have fewer nucleotide substitutions[J]. Gut, 2002,51(1):82-88.
8 Koonin EV,Gorbalenya AE,Purdy MA,et al. Computer-assisted assignment of functional domains in the nonstructural polyprotein of hepatitis E virus: delineation of an additional group of positive-strand RNA plant and animal viruses[J]. Proc Natl Acad Sci U S A, 1992,89(17):8259-8263.
9 Emerson SU,Zhang M,Meng XJ,et al. Recombinant hepatitis E virus genomes infectious for primates: importance of capping and discovery of a cis-reactive element[J]. Proc Natl Acad Sci USA,2001,98(26):15270-15275.
10 Magden J,Takeda N,Li T,et al. Virus-specific mRNA capping enzyme encoded by hepatitis E virus[J]. J Virol, 2001,75(14):6249-6255.
11 Pudupakam RS,Huang YW,Opriessnig T,et al. Deletions of the hypervariable region (HVR) in open reading frame 1 of hepatitis E virus do not abolish virus infectivity: evidence for attenuation of HVR deletion mutants in vivo[J]. J Virol, 2009,83(1):384-395.
12 Mushahwar IK. Hepatitis E virus: molecular virology, clinical features,diagnosis, transmission, epidemiology, and prevention[J]. J Med Virol, 2008,80(4):646-658.
13 Graff J,Zhou YH,Torian U,et al. Mutations within potential glycosylation sites in the capsid protein of hepatitis E virus prevent the formation of infectious virus particles[J]. J Virol, 2008,82(3):1185-1194.
14 Li S,Tang X,Seetharaman J,et al. Dimerization of hepatitis E virus capsid protein E2s domain is essential for virus-host interaction[J].PLoS Pathog, 2009,5(8):e1000537.
15 郭清顺,葛胜祥,熊君辉,等.戊型肝炎病毒基因1型和基因4型中和表位区域分子差异研究[J].病毒学报,2007,23(6):454-458.
16 Chandra V,Taneja S,Kalia M,et al. Molecular biology and pathogenesis of hepatitis E virus[J]. J Biosci, 2008,33(4):451-464.
17 Agrawal S,Gupta D,Panda SK. The 3' end of hepatitis E virus(HEV) genome binds specifically to the viral RNA-dependent RNA polymerase (RdRp)[J]. Virology, 2001,282(1):87-101.
18 Graff J,Nguyen H,Kasorndorkbua C,et al. In vitro and in vivo mutational analysis of the 3'-terminal regions of hepatitis e virus genomes and replicons[J]. J Virol, 2005,79(2):1017-1026.

