小鼠粪便细菌基因组DNA提取方法比较
张 杰, 张晓鹏, 李鹏高 , 赵志强
(1. 首都医科大学 公共卫生学院临床流行病学北京市重点实验室, 北京 100069;2. 首都医科大学 信息中心, 北京 100069)
小鼠粪便细菌基因组DNA提取方法比较
张 杰1, 张晓鹏1, 李鹏高1, 赵志强2
(1. 首都医科大学 公共卫生学院临床流行病学北京市重点实验室, 北京 100069;2. 首都医科大学 信息中心, 北京 100069)
分别将研磨、水煮两种预处理方法和改良CTAB法、试剂盒法两种提取方法进行组合,比较提取小鼠粪便细菌基因组DNA的效果。结果发现,研磨预处理过的样品提取到的细菌基因组DNA的浓度和纯度均高于水煮预处理过的样品,研磨结合改良CTAB法提取到的DNA完整性好于其他方法,研磨结合改良CTAB法是一种提取粪便细菌基因组DNA较为实用可靠的方法。
粪便; 细菌基因组DNA; 提取方法
肠道是哺乳动物体内最大的消化器官,其中含有数量巨大的细菌菌群,形成了极其复杂的细菌微生态系统,肠道微生态系统中的菌群结构、数量和种类的变化与机体的健康和疾病有着密切的关系[1-2]。
粪便的主要成分是微生物的菌体和食物残渣,其中包含着重要的肠道微生物信息,可以间接地反映肠道微生物的变化情况。近年来随着分子生物学研究和技术的发展,利用分子生物学技术和方法从粪便中提取肠道微生物总DNA,并对相关疾病进行研究取得了许多突破[3-4]。但是粪便中含有的成分非常复杂,其中含有的色素、多糖和纤维素等物质对TaqDNA聚合酶有抑制的作用;脂类、酸类、多酚等有机物在DNA提取过程中很难去除,这些杂质不但易导致目标DNA的降解 ,还直接影响到后续研究的准确性。因此,以粪便为标本提取细菌基因组DNA进行肠道微生态研究的关键是能否提取到高质量的粪便细菌基因组DNA[5]。
本研究是在已有报道的基础上比较几种小鼠粪便细菌基因组DNA提取方法,旨在保证提取效率的同时得到高质量的细菌基因组DNA,为肠道微生态系的研究提供实验基础。
1 实验材料及仪器
样品的采集与储存:BALB/C清洁级小鼠由首都医科大学动物中心提供,3周龄,体重18~22 g,直接从小鼠肛门处收集粪便于1.5 mL的无菌管中,放入-80 ℃保存,备用。
试剂:三羟甲基氨基甲烷-乙二胺四乙酸(TE,国产);十二烷基磺酸钠(SDS,国产);核糖核酸酶A(RNase A,Promega公司);蛋白酶 K(Promega公司);氯化钠(NaCL,国产);十六烷基三甲基溴化铵(CTAB,国产);氯仿(国产分析纯);异丙醇(国产分析纯);Tris酚(国产);异戊醇(国产分析纯);无水乙醇(国产分析纯);λDNA/HindⅢ DNA Marker(TIANGEN公司);100bp DNA ladder Marker(TIANGEN公司);Gel-Red 核酸染料(Sigma公司);琼脂糖(Sigma公司);荧光定量PCR试剂盒(ABI公司);引物(上海生工生物工程技术服务有限公司合成);粪便细菌基因组 DNA 提取试剂盒(离心柱型TIANamp Stool DNA Kit,TIANGEN公司)。
仪器:Q5 Real-time PCR扩增仪(ABI公司);电泳仪(Bio-Rad公司);高速冷冻离心机(Eppendorf公司);NanoDrop 2000紫外分光光度仪(Thermo公司);凝胶成像分析系统(VILBER LOURMA公司);漩涡混合器(国产);电热恒温水浴锅(国产)。
2 实验方法
2.1 粪便预处理方法
方法1——研磨:称取0.1 g小鼠粪便放入灭菌研钵中,在冰上彻底研碎,然后加入1 mL无菌双蒸水彻底重悬,10 000 r/min、离心10 min ,取上清待用。
方法2——水煮:在0.1 g小鼠粪便中加入1 mL无菌双蒸水于1.5 mL的EP管中,浸泡10 min后充分振荡摇匀15 s;将管放置100 ℃水浴10 min;冷却后10 000 r/min、离心10 min ,取上清待用。
2.2 小鼠粪便细菌基因组DNA提取方法
(1) 改良CTAB方法:参照文献[6](有改动),分别在预处理过的500 μL样品中加入30 μL、10%SDS,3 μL蛋白酶K(20 g/L),4 μL RNaseA后混匀,37 ℃水浴1 h;每管中加入100 μL NaCL(5 mol/L),颠倒混匀后加入80 μL的CTAB/NaCL(10% CTAB,0.7 mol/L NaCL),轻轻混匀;放入65 ℃水浴10 min;加入等体积酚/氯仿/异戊醇(比例为25∶24∶1)混合液,混匀后12 000 r/min、离心10 min,取上清;上清液中加入0.6倍体积的异丙醇,轻轻混匀,12 000 r/min、离心10 min,弃上清;沉淀用1 mL预冷的75%乙醇洗涤后,7 500 r/min离心5 min,弃乙醇,在洁净工作台中稍加干燥,溶于30 μL TE缓冲液中。
(2) 试剂盒方法:将预处理好的样品,分别按照粪便细菌基因组 DNA 提取试剂盒(离心柱型)说明书的步骤进行提取。
2.3 小鼠粪便细菌基因组DNA质量分析
2.3.1 细菌基因组DNA纯度和浓度测定
分别取2 μL提取到的细菌基因组DNA,在NanoDrop 2000紫外分光光度仪上检测DNA的质量浓度及DNA的A260/A280比值。
2.3.2 细菌基因组DNA完整性测定
分别取5 μL提取到的DNA于含Gel-Red核酸染料的0.8%琼脂糖凝胶中电泳,工作电压120 V,50 min后置于凝胶成像分析系统中观察、分析结果。
2.3.3 荧光定量PCR反应
使用细菌16S rDNA基因V3区特异性引物338-F:5’-GCTGCCTCCCGTAGGAGT-3’,548-R:5’-AATACGGAGGGTGCAAGCGT-3’,以提取到的小鼠粪便细菌基因组DNA为模板,进行细菌16S rDNA基因荧光定量PCR扩增,每个样本设置3次重复,同时设置不添加模板的阴性对照组。
根据Power SYBR Green master mix说明书配置荧光定量PCR反应体系20 μL。反应条件:95 ℃、10 min ,95 ℃、15 s,58 ℃、30 s 、72 ℃,30 s,40个循环;反应完成后利用熔解曲线分析产物的特异性,观察其扩增曲线以及记录Ct值(反应管内的荧光信号到达设定值时所经历的循环数);扩增产物在含Gel-Red核酸染料的1%琼脂糖凝胶中电泳(电压100 V,50 min),结果于凝胶成像分析系统中观察、分析。
2.4 统计分析
采用SPSS22.0 统计软件,对数据进行单因素方差分析。
3 实验结果
3.1 不同方法提取到的小鼠粪便细菌基因组DNA质量浓度和纯度
结果见表1。由表1可知:采用相同预处理方法的样本,改良CTAB法提取到的DNA质量浓度c(DNA)明显高于试剂盒法提取到的;对于采用相同的提取方法、不同预处理方法的样本提取到的DNA质量浓度不同,与水煮法相比,研磨法能显著提高小鼠粪便细菌基因组的c(DNA);不同方法提取到的细菌基因组DNA质量浓度之间的差异有统计学意义(P<0.01)。

表1 不同方法提取到的小鼠粪便细菌基因组DNA的纯度和质量浓度
注: a:与研磨+改良CTAB法相比P<0.01;b:与水煮+改良CTAB法相比P<0.01。
A260/A280比值是判断核酸纯度的常用指示值,A260代表核酸的吸光度值,A280代表蛋白质的吸光度值。A260/A280的比值在1.6~1.8之间表示DNA的纯度较好,A260/A280<1.6说明样品中有蛋白质污染,A260/A280>1.8说明样品中有RNA污染。理论上纯化的DNA的A260/A280比值在1.8左右。测定结果表明:相同提取方法,经过研磨预处理后提取到的细菌基因组的DNA纯度好于水煮预处理的样本;采用相同预处理方法,试剂盒法提到的细菌基因组DNA纯度高于改良CTAB法提取到的,但是两者之间的差异没有统计学意义(P≥0.05)。
3.2 细菌基因组DNA完整性测定
通过琼脂糖凝胶电泳图(见图1)可见:不同方法提取到的DNA均在23 kb左右处有清晰条带;相同预处理方法的样品,改良CTAB法提取到的小鼠粪便细菌基因组DNA条带较亮;采用相同DNA提取方法,水煮法预处理过的样本提取到的细菌基因组DNA有明显的拖带,说明提取到的DNA发生了降解,DNA分子的完整性变差。

图1 不同方法提取到的小鼠粪便细菌基因组DNA
3.3 荧光定量PCR检测细菌基因组DNA扩增效果及扩增产物的有效性分析
利用不同方法提取到的粪便细菌基因组DNA为模板,采用相同DNA模板量对细菌16S rDNA基因进行荧光定量PCR扩增。PCR扩增的溶解曲线上显示每个样品扩增的16S rDNA基因均为单一峰,特异性较好(见图2)。图3的PCR扩增曲线显示:以研磨结合改良CTAB法提取到的DNA为模板扩增细菌16S rDNA基因得到的Ct值最低,与水煮结合改良CTAB法提取到的DNA扩增得到的Ct值相比有统计学意义(P<0.05),具体结果见表2。
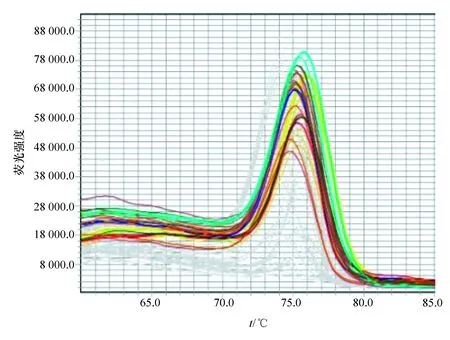
图2 不同方法提取到的小鼠粪便细菌基因DNA荧光定量PCR溶解曲线
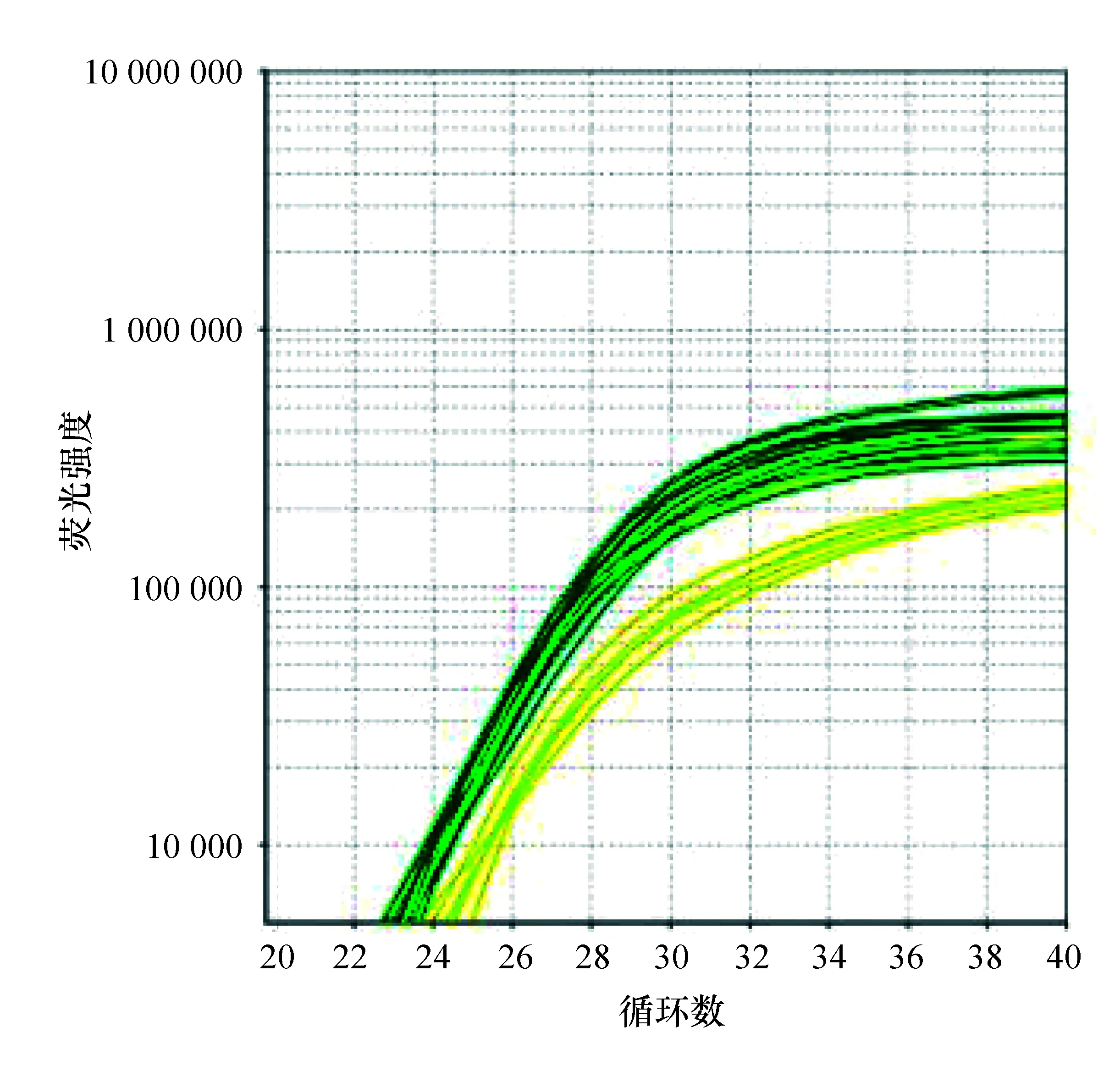
图3 不同方法提取到的小鼠粪便细菌基因组DNA荧光定量PCR扩增曲线

预处理方法提取方法样本数 Ct值研磨水煮改良CTAB法523.9427±0.2791525.0210±0.1327A研磨水煮试剂盒法524.4320±0.2291524.6947±0.9099
注:A:与研磨+改良CTAB法相比P<0.05
对荧光定量 PCR的扩增产物进行1%琼脂糖凝胶电泳检测表明:扩增产物均可以在200 bp左右看到细菌16S rDNA基因的特异性扩增片段,且无干扰性杂带,重复性好(见图4);研磨结合CTAB法获得的DNA扩增产物条带最亮;经过水煮法预处理的样品,不同提取方法提取到的DNA扩增后的产物条带均较暗,说明经过水煮预处理的样品提取到的细菌基因组DNA浓度较低,质量较差。
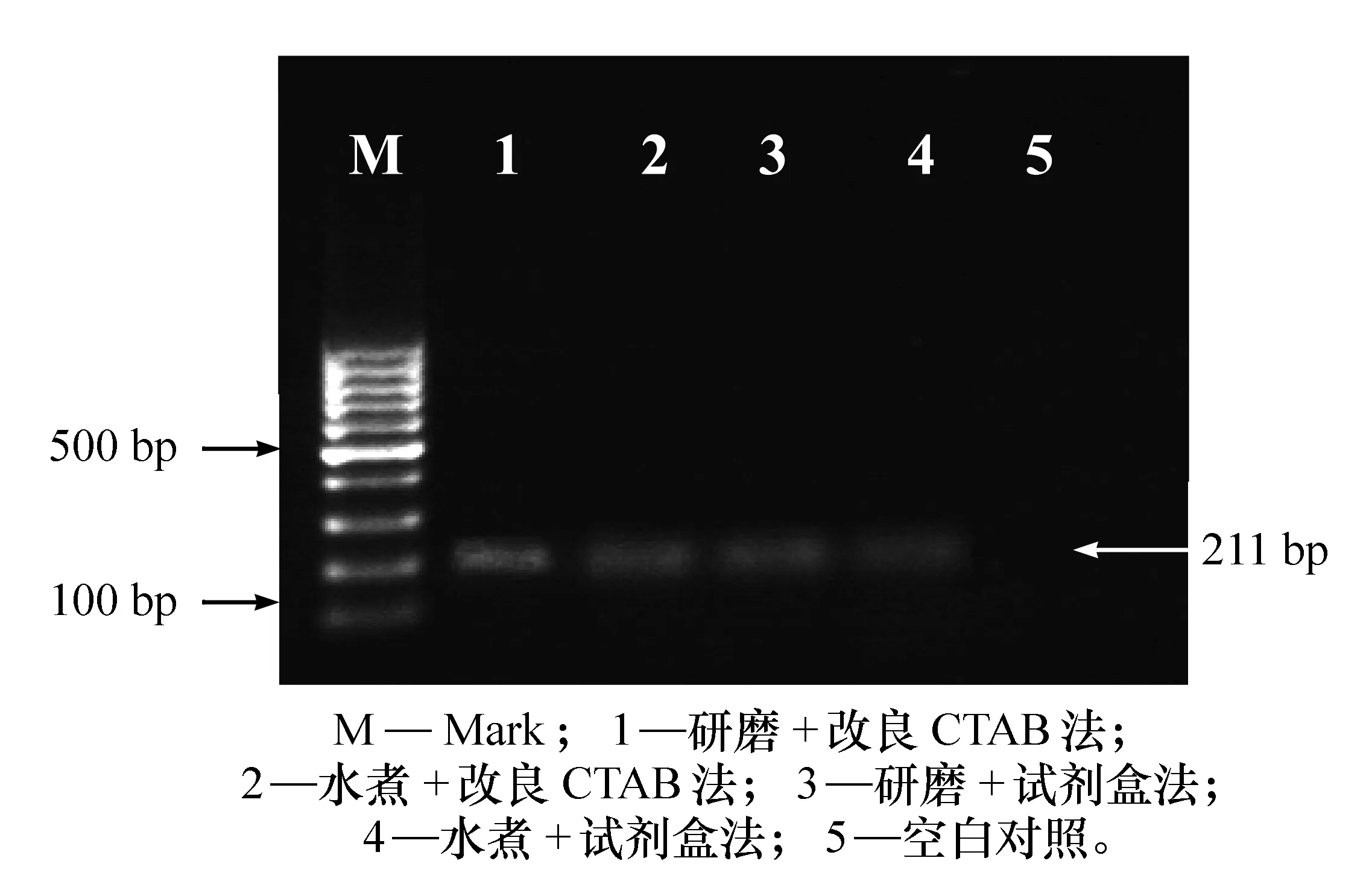
图4 小鼠粪便细菌16S rDNA基因荧光定量PCR扩增产物
4 讨论
从粪便中有效地提取到高质量的细菌基因组DNA是研究肠道微生态的基础和前提。目前,从粪便标本中提取细菌DNA的方法众多,由于提取机理和步骤的差别,获得 DNA 的浓度和纯度各不相同,导致在后续实验,如PCR扩增、DNA 测序等的应用效果不同[5-7]。理想的基因组DAN提取方法既要好的DNA的获得率,又要尽可能地减少DNA的降解[8]。高效稳定的细菌基因组DNA提取方法对后续分子生物学的研究有着重要的意义[9]。
本实验的实验结果显示:经过不同预处理的样本,采用相同的细菌基因组DNA提取方法提取到的DNA质量浓度不同,研磨预处理后提取到的细菌基因组DNA质量浓度明显高于经水煮后提取到的DNA质量浓度。有文献报道机械破壁对厚壁菌门的破壁效果要优于传统的酶解作用[10],研磨可以较为彻底地破坏细菌细胞壁,使细胞充分裂解,使更多的DNA游离出来。
水煮法是利用物理方法处理蛋白质,使其与核酸分离,但是在加热过程中菌体破裂的比例较小,而且煮沸过程中部分DNA发生降解[11-12],所以提取到的DNA质量浓度较低。因此研磨法与水煮法相比,研磨法是一种更为有效的提高粪便细菌基因组DNA浓度的方法。
此外细菌16SrDNA 基因荧光定量PCR扩增结果显示,经过水煮法预处理过的样品,提取到的DNA完整性较研磨预处理后提取到的DNA差。已有的研究法发现[13],采用水煮法虽然促使了细胞膜破裂,释放出DNA,同时高温使部分DNA断裂成小片段。由水煮法处理过的DNA样品不适用于限制性内切酶切分析的实验,只适用于对DNA要求不高的PCR和RAPD等实验。由此可知小鼠粪便细菌基因组DNA提取时研磨预处理方法优于水煮预处理方法。
把经过相同预处理的样本,采用不同提取方法提取到的细菌基因组DNA进行综合比较发现:改良CTAB方法提取到的DNA质量浓度明显高于试剂盒法提取到的DNA质量浓度;改良CTAB方法的DNA纯度低于试剂盒法提取到的DNA纯度。试剂盒的方法操作简单、用时短,提取到的DNA质量稳定,但是荧光定量PCR的扩增效果比改良CTAB法的差,而且试剂盒价格较贵,使用次数有限,试剂盒的质量因不同厂家和批次也会有所差异,不适合大量样本的粪便细菌基因组DNA的提取。
改良的CTAB方法提取到的细菌基因组DNA条带完整、较亮,没有明显的降解和蛋白质污染;以此为模板进行细菌16SrDNA 基因荧光定量PCR扩增,扩增产物条带清晰,说明改良的CTAB方法提取到的小鼠粪便细菌基因组DNA能够满足下游分子生物学研究的需要。而且改良CTAB方法在原有的方法上,简化了操作步骤,减少了有机试剂的使用,缩短了实验时间,实验时不需要特殊的仪器设备,成本较低,适用于大样本量的粪便细菌基因组DNA的提取。
综上所述,研磨预处理方法结合改良CTAB法是一种提取粪便细菌基因组DNA较为实用可靠的方法,可以为肠道微生态的大样本量研究提供帮助。
References)
[1] 骞宇,赵欣.大鼠粪便中细菌基金组DNA提取方法的比较[J]. 食品工业科技,2014,35(4):166-169.
[2] 郭政宏,舒邦杰,严亨秀,等.藏山羊粪便总DNA不同提取方法的比较[J]. 黑龙江畜牧兽医, 2016(7):93-96.
[3] Gerritsen J,Smidt H,Rijkers G E.et al. Intestinal microbiota in human health and disease:The impact of probiotics[J]. Genes & Nutrition,2011,6(3):209-240.
[4] 吴敏娜,武亚琦,屈艳,等. 四种小鼠肠道微生物 DNA提取方法比较[J]. 生态学杂志,2015,34(4):1183-1188.
[5] 郑吉顺,周翔天,陈萌萌,等.介绍一种改良的粪便细菌基因组DNA提取试验方法[J].安徽医药,2014,18(1):52-55.
[6] 宫强,关道明,王耀兵,等. 大肠杆菌总DNA 快速提取方法的比较研究[J].海洋环境科学,2005,24(4):63-66.
[7] 程实,刘文丽,舒鹏,等.难提取细菌基因组DNA提取方法的比较与优化[J]. 生物技术通讯,2016,27(3):412-415.
[8] Chen H,Rangasamy M,Tan S Y,et al. Evaluation 0f five methods for total DNA extraction from western corn rootworm beetles[J].PLoS One,2010,5(8):e11963.
[9] 庞建, 刘占英, 郝敏,等. 革兰氏阳性细菌基因组DNA提取方法的比较及优化[J]. 微生物学通报, 2015, 42(12):2482-2486.
[10] Biesbroek G, Sanders E A, Roeselers G, et a1. Deep sequencing analyses of low densitymicrobialcommunities:working at the boundary of accurate microbiota detection[J].PLoS One,2012,7(3):e32942.
[11] 郝敏, 刘占英, 杨天舒,等. 细菌基因组DNA提取方法概述[J]. 生物学通报,2014,49(3):4-6.
[12] 王国泽, 唐仁勇, 左福元,等. 瘤胃细菌DNA提取方法的研究与优化[J]. 黑龙江畜牧兽医, 2016(16):125-128.
[13] 李战军, 孔杰, 孟宪红,等. 3种DNA提取方法对WSSV检测结果的影响[J]. 渔业科学进展,2011,32(3):92-97.
Comparison of methods for extracting bacterial genomic DNA from mouse feces
Zhang Jie1, Zhang Xiaopeng1, Li Penggao1, Zhao Zhiqiang2
(1.Beijing Municipal Key Laboratory of Clinical Epidemiology, School of Public Health , Capital Medical University, Beijing 100069, China; 2. Center of Network Information, Capital Medical University,Beijing 100069,China)
Two pretreatment methods of grinding and boiling and two extraction methods such as the improved CTAB method and the kit method are combined respectively to compare the effect of extracting bacterial genomic DNA from the mouse feces. The results show that the concentration and purity of the genomic DNA extracted from the milled pretreated samples are higher than those obtained by the boiling pretreated samples, the integrity of the DNA extracted by the grinding combined with the modified CTAB method is better than that by other methods, and the grinding combined with the modified CTAB method is a practical and reliable method for extracting the genomic DNA from the fecal bacteria.
feces; bacterial genomic DNA; extraction method
10.16791/j.cnki.sjg.2017.12.012
2017-09-06
国家自然科学基金项目(81573128)资助
张杰(1968—),女,北京, 学士, 副主任技师, 主要研究方向为分子流行病学
E-mail:zhangjie@ccmu.edu.cn
赵志强(1979—),男,北京,博士,研究员,研究方向为生物医学工程.
E-mail:zzqmr@ccmu.edu.cn
Q933
B
1002-4956(2017)12-0050-04

