CT图像中肝脏自动分割算法研究
张辉++闫谦时

摘要:由于全自动分割方法容易受噪声、场偏移效应等影响,医用上全自动的分割方法尚且无法完全代替手工操作参与的半自动化方法。该文使用一种基于改良的区域生长算法用于对CT图像的肝脏部分进行剥离,以期能尽可能减少图像分割时人工参与,解决现阶段临床上半自动化交互式分割效率低的问题,以助于计算机辅助诊断、病变组织定位、三维重建、治疗方案设计等。方法:改进的区域生长法。该方法对两套肝脏CT切片分割,准确率均在90%以上,鲁棒性强。
关键词:肝脏自动分割;区域生长法;CT图像
中图分类号:TP751 文献标识码:A 文章编号:1009-3044(2017)30-0184-04
The Algorithm Research of Liver CT Image Automatic Segmentation
ZHANG Hui,YAN Qian-shi
(School of Computer Science and Engineering, Xian Technological University, Xian 710021, China)
Abstract: Because of that the automatic segmentation method is easy to be affected by the noise and the field offset effect, the fully automatic segmentation method can not completely replace the artificial semi-automatic segmentation method. This paper proposes an improved algorithm for liver section of CT image segmentation Based on region rowing algorithm, in order to minimize the image segmentation of artificial participation, to solve the present clinical semi automated interactive segmentation problem of low efficiency, and help computer aided diagnosis, lesion location, 3D reconstruction, treatment design. Methods: Based on the improved region growing method, the liver CT image was segmented. The proposed method was applied to the segmentation of two sets of liver CT slices, and the accuracy was above 90%.
Key words: automatic liver segmentation; region growing; CT image
醫学领域的图像分割的发展大致经历了三个阶段的发展,第一阶段完全依赖于工作人员手工勾勒出感兴趣的器官,即手工时期。尤其对于CT图像数以百计的切片来说,依靠工作人员手动去分割的方式不仅耗时耗力,而且分割是否准确很大程度依赖于医生的解剖知识和临床经验,针对不同患者分割结果不可复用,因此很不理想。第二阶段指随着技术的进步和设备的优化,慢慢出现了人工和机器结合的半自动化的分割方法。该方法使医生根据自己的诊断经验、医学知识利用先进的成像设备进行图形分割(例如,文献[6]中提到的自适应区域生长算法需要医者根据经验选取起始种子点,其后则可实现自动分割),采用交互式的半自动化方法完成对医学图像的分割。和手动分割比较,交互式分割方法很大程度降低了手工参与,而且分割效率高、准确度高。第三阶段,近年来,因为人工智能、模糊数学等学科得到广泛推广,出现了一些全自动的分割方法,几乎脱离了手工操作。但是因为各种医学成像技术拥有的成像特征各不相同,没有万能的分割方法,再加之在噪声的影响下,使获取到的图像很不理想,例如:图像灰度不均匀、存在伪影和噪声、对比度低、器官形状结构和微细结构分布复杂、不同软组织之间相互重叠造成边界模糊等特性。所以医用上全自动的分割方法尚无法普及。
现阶段基于CT图像的肝脏分割面临的挑战有:设备扫描范围很大,生成的CT断层图像数量多;肝脏与周围组织器官有大面重叠,且其灰度非常接近,导致肝脏轮廓往往无法用肉眼识别。如下图所示。
图1 肝脏CT切片示意图
CT图像分割,往往要依据切片灰度的两个特点:不连续性和相似性。常用的方法如下:
(1) 边缘检测法:边缘检测的核心思想是检测每个像素和其紧邻域的状态,边缘往往是图像上的突变点,即如果该像素点和其紧邻域的像素灰度差较大,则可以认为其是边缘像素点。最常用的方式是借助图像一二阶导数来计算。
(2) 阈值分割法:阈值分割原理是利用设置的阈值把图像中的像素分成若干类。其特点是分割速度快,操作简单,适用于分割对象和背景灰度差较大的图像。但对于一些灰度分布不均匀,目标与背景差异不明显的图像,只使用阈值分割是不可行的。阈值分割的关键是阈值如何选择,常用的方法有两种,一种是使用滑动条控件,用户可拖拽滑动条通过视觉观察选取合适阈值。还有一种方法是通过计算,例如常见的otsu最佳全局阈值法,通过最计算大类间方差确定最佳阈值。
(3) 区域分割法:其中最普遍的是区域生长法。区域生长是指通过观察或者根据经验在目标区域内选取一个或者多个种子点,依据提前指定的生长规则,判断种子点周围的像素是否合并进来,若合并则以刚合并的点为目标继续判断其周围点是否合并,直到满足预定义的停止规则为止,生长区域的扩张才算结束。生长规则的制定可以是灰度值、纹理、颜色等多种图像信息。
(4) 分水岭分割法:分水岭分割方法是将整个图像视为地貌,借助于地理学的启发,假设地貌的低洼处有孔洞,将整个地貌模型放入水缸模拟浸水过程,当有两个积水区即将融合时通过柞水坝阻止积水相会。直到整个地貌模型完全融入水缸,则水坝就将图形划分成了不同区域。在分水岭分割方法执行过程中借助阈值分割,边缘检测来得到分水岭标记图。其可视为对经典分割方法的有效结合,既保留了经典方法的优点,也克服了其过分依赖于灰度特征的缺陷。
近年来不断有国内外学者对传统分割方法改良,Kumar提出先利用腐蚀图像的最大连通区域的中心坐标轴作为生长区域的种子节点,再利用高斯模型对阈值范围进行确定,然后使用区域生长法进行分割[10]。Pohle R提出了自适应区域生长法,但其算法过于依赖于组织中灰度分布的均匀性[9]。吸收前人的经验,本文对传统区域生长算法做出改良,用于对CT图像的肝脏部分进行分割,以期能尽可能减少图像分割时人工参与,解决现阶段临床上半自动化交互式分割效率低的问题,为临床诊断和手术计划提供有效的个性化数据。
1 区域生长算法
区域生长算法:原理是先从图像上选取一个或多个种子点,遍历与种子点邻接的像素点,将其灰度值与种子点逐个进行比较,依据生长规则判别是否将其合并进生长区域内,最后按照提前拟定好的遏制条件判断是否停止區域扩张。
1.1 基于区域灰度差的生长规则
(1) 自左向右一行一行扫描图像,寻找没有隶属的像素;
(2) 以选定的像素(种子点,也是生长区域的起始点)为对象,遍历与它邻接的像素点,求邻接像素点与种子点的灰度差,要是比提前设置的阈值小,则可以将此邻接像素融合进来;
(3) 用刚刚归并的点作为研究目标,再次进行第2步,当满足停止规则时停止扩张;
(4) 重复进行步骤1,当没有新的像素点加入,算法结束。
1.2 三个关键点
若要使用区域生长法,需面对以下三个关键点:1)种子点的选取;2)确定生长准则;3)确定停止的条件或规则。
1.3 算法存在缺陷
(1) 用户得凭借自身经验手动选种子点,分割速度慢。
(2) 容易出现过分割现象。
2 改进的区域生长算法
图2 改进的区域生长法流程图
2.1 种子点(种子区域)自动选取
1) 在整套 CT中挑选出其中一张含有肾脏区域较大的断层切片作为起始层,医者需要临床诊断经验选择一个种子点,以该点周围5×5(暂定,种子区域宽度作为参数之一)个像素区域作为种子区域。
2) 根据拟定的合并准则(区域生长准则后文介绍)判断周围像素点是否合并,以此让种子区域逐步向外生长,直到没有新的像素点加入,则起始层分割完毕;
3) 考虑相邻层之间的肝脏区域不会出现太大的位置偏移,故可计算出起始层生长结束后的区域中心(可使用matlab提供的centroids函数),并以此中心作为相邻两层的种子点,以该点周围5×5个像素区域作为种子区域。
4) 重复以上方法获取后续层数的种子区域直到CT断层图像上不存在肾脏区域为止。
2.2 改进的区域生长规则
本文利用种子点灰度值与邻接像素的灰度差判断邻域像素是否合并进生长区域内,但缺点是这对种子点的选择有较大的依赖性,其改进方法如下:
1) 假定灰度差的阈值为0,依据以上的区域生长法,归并灰度值相等的点;
2) 首先对每个紧邻的区域之间的灰度差求均值,然后找最小的紧邻区域进行归并;
3) 不断执行第2步中的操作,让生长区域不断扩张,直到没有新的无归属的点加入,区域生长过程结束。
4) 针对一些灰度近似的区域以上方法仍存在一定缺陷,即:可能会将不属于分割目标的像素归并进来。想要完善此缺点,考虑不求解当前点与紧邻点的灰度差,用生长区域的灰度均值代替当前点去与各个紧邻点的求灰度差。用公式:[X=1MQf(x,y)]表示CT断层切片中M个点的Q区域的灰度均值。即:假设当前元素灰度值为y,若满足:|y-[X]|≤T[y-X≤T]则合并,否则不合并。其中T表示提前设置的阈值。
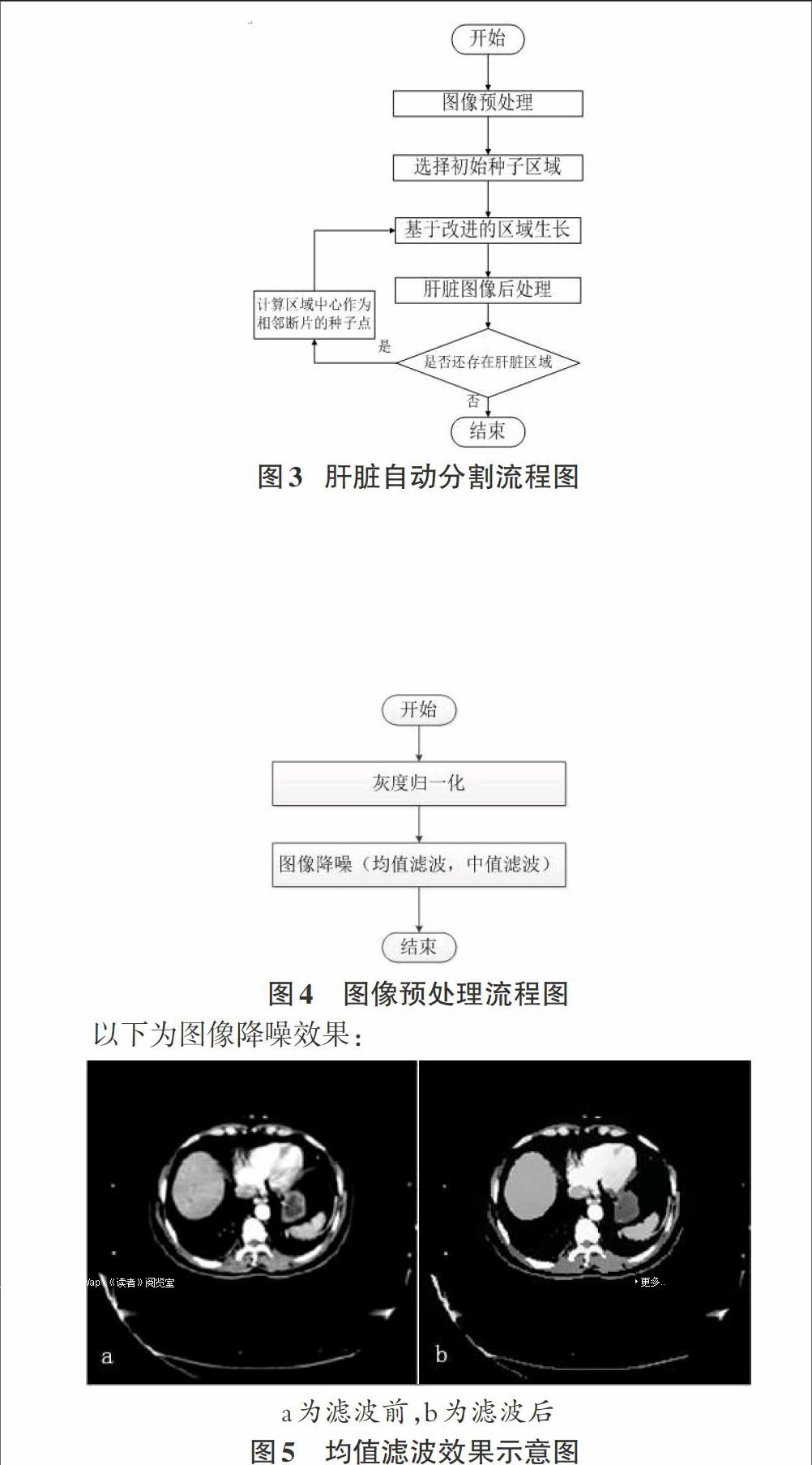
3 实现流程
3.1 图像预处理
读入CT断层图像,先对图像进行灰度归一化处理,然后采用均值滤波法对原始图像进行降噪(基于matlab实现),如图为图像预处理实现流程:
3.2 选择初始种子区域
选取含有肝脏面积较大的切片,根据灰度范围选取种子区域,例如,取种子点为(x,y)=(205,207)。以该为中心的5×5个像素区域作为种子区域。
3.3 基于改进的区域生长算法
肝脏灰度阈值的计算公式如下:
[125i=-22j=-22(f(x+r,y+c)-mean(x,y))]
以(x,y)表示刚兴趣区域的中心点坐标,f(x,y)表示像素点(x,y)的灰度值,
mean(x,y) = [125i=-22j=-22f(x+r,y+c)]
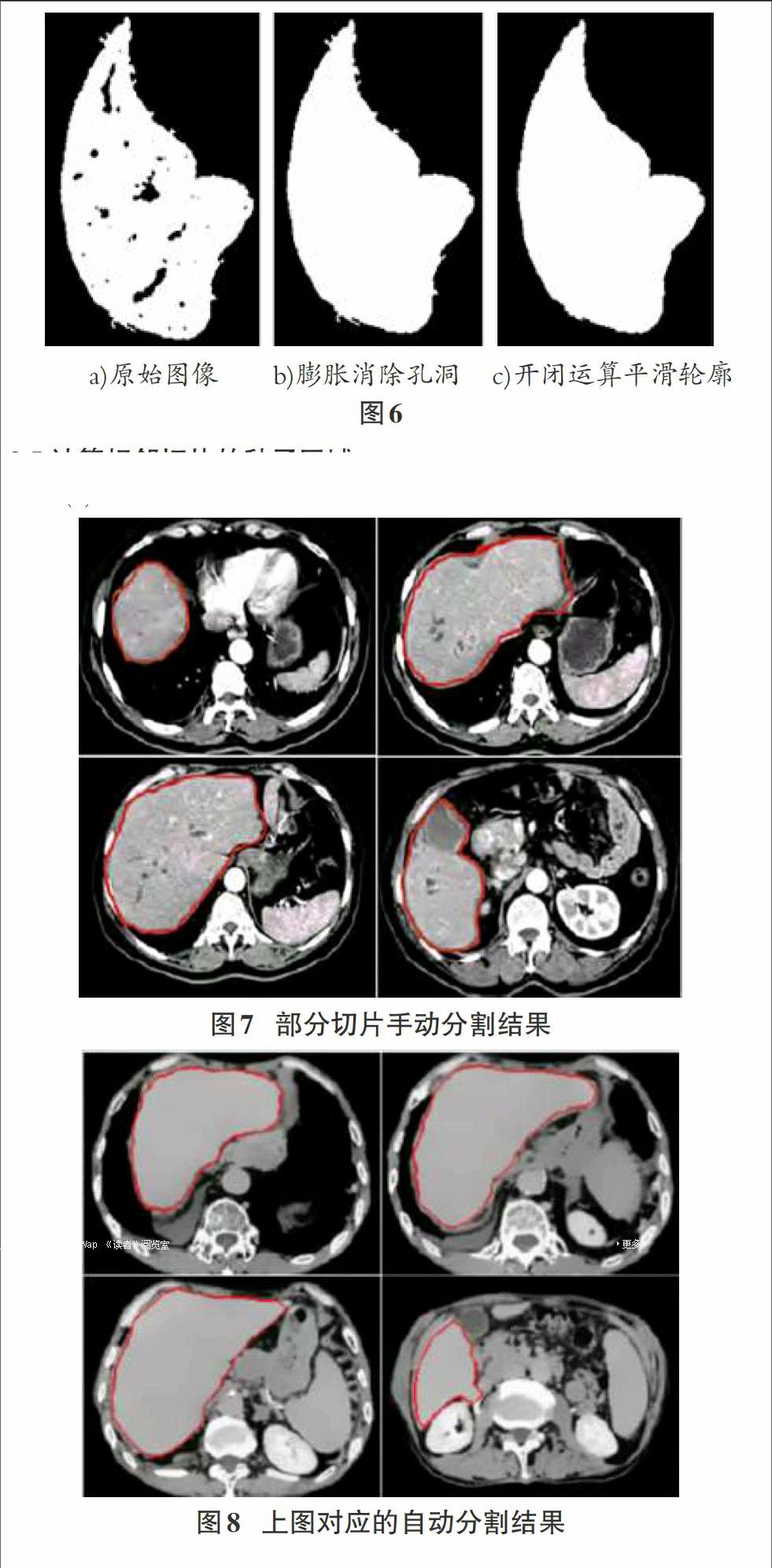
3.4 图像后处理
利用形态学上的膨胀、腐蚀、开运算以及闭运算就图像进行后处理,消除图像上的孔洞,如下图:
3.5 計算相邻切片的种子区域
使用matlab 的centroids函数计算当前切片的几何中心,由于相邻切片之间偏移量很小,可利用相邻切片相似度高的特性,以当前切片的几何中心作为下一切片的种子点,例如,种子点为(x,y)=(206,208)。则相邻切片的种子区域为该点周围5×5个像素区域。
4 结果与分析
(1) 实验数据
本文所用数据源中CT图像的像素间距是0.733594mm,断层图像厚度是1.0mm,断层图像之间的间隔是0.4mm。CT断层图像尺寸为512×512像素。
(2) 实验环境
算法于matlab7.0上仿真,于64位 windows 7系统上运行,处理器为i76700 HQ,其主频为2.8 GHz,内存为16 G DDR42133 MHz,显卡为 NVIDIA GeForce GTX960m,2 G独显。
(3) 实验结果分析
由于相关试验涉及的CT断层切片数量较多,本文只展示部分试验结果,第一组CT图像共有85张断层切片上包含肝脏,有73张分割效果较好,与目标边缘吻合大于92%,然而还有一些 CT切片分割效果仍不理想,73张CT切片自动分割共用时约29.5 s。第二组CT切片共有80张断层切上包含肝脏,有69张分割结果较好,与目标边缘吻合率大于94%,80张CT切片自动分割用时约28.7s。经测试,一个经验较为丰富的医护人员手动勾画CT切片中肝脏部分平均需要8s/张,勾画出第一套CT切片的肝脏部分平均需要11.3分钟,第二套10.7分钟。本文所用方法剥离CT切片肝脏区域只用400 ms~800 ms/张,第一套 CT切片分割用时29.5s,大约仅占医护人员手动勾画耗时的1/25,第二套 CT切片分割用时28.7s,大约仅占医生手动勾画耗时的1/23,速度大幅度增长。
5 结论
本方法是凭借相邻切片偏移量小、近似度高的特点,改良传统的区域生长法,完成肝脏部分的自动分割,解决人工手动分割效率低的问题,实验以两套CT切片为基础数据,通过对比医生手工分割,不难发现该方法比之手工分割效率明显提高,两套实验切片均能在30秒之内完成分割。该方法除了初始阶段需要用户根据先验性经验选取第一个种子区域,在其他分割过程中均无人工参与,两套实验切片肝脏部分的剥离正确率在大于90%,算法拥有较强的精确性和稳定性。
参考文献:
[1] 潘家辉,朱玲利,鲍苏苏.基于区域生长算法的CT序列图像分割[J].洛阳师范学院学报,2015,05:58-61.
[2] 王华松.图像区域生长分割算法研究[J].科技创新导报,2015,18:66+120.
[3] 宋红,王勇,黄小川,李佳佳,等.基于动态自适应区域生长的肝脏CT图像肿瘤分割算法[J].北京理工大学学报,2014,01:72-76.
[4] Adams R, Bischof L. Seeded region growing[J].Pattern Analysis and Machine Intelligence, IEEE Transactions on, 1994, 16(6):641-647.
[5] 高升,葛云. DICOM格式医学图像及其图像信息的显示[J].中国医学物理学杂志,2010,03:1885-1888.
[6] 彭丰平,鲍苏苏,曾碧卿.基于自适应区域生长算法的肝脏分割[J].计算机工程与应用,2010,33:198-200.
[7] 周光华,李岳峰,孟群.医学图像处理技术与应用分析[J].中国卫生信息管理杂志,2011,06:44-47.
[8] Lim S J, Jeong Y Y, Ho Y S. Automatic liver segmentation for volume measurement in CT Images[J].Journal of Visual Communication and Image Representation, 2006, 17(4):860-875.
[9] Pohle R, Toennies K D. Segmentation of medical images using adaptive region growing[C]//Medical Imaging 2001. International Society for Optics and Photonics, 2001: 1337-1346.
[10] Kumar S S, Moni R S, Rajeesh J. Automatic liver and lesion segmentation: a primary step in diagnosis of liver diseases[J]. Signal, Image and Video Processing, 2013, 7(1):163-172.endprint

