基于COI条形码的根口水母科Rhizostomatidae水母分子系统进化研究
李玉龙,王彬,孙明,王小林,于旭光,李轶平,付杰,王文波,董婧
(辽宁省海洋水产科学研究院,辽宁省海洋生物资源与生态学重点实验室,辽宁 大连 116023)
基于COI条形码的根口水母科Rhizostomatidae水母分子系统进化研究
李玉龙,王彬,孙明,王小林,于旭光,李轶平,付杰,王文波,董婧
(辽宁省海洋水产科学研究院,辽宁省海洋生物资源与生态学重点实验室,辽宁 大连 116023)
为探讨DNA条形码基因COI序列在根口水母科Rhizostomatidae水母物种鉴定中的有效性,分析了根口水母科3属6种水母及口冠水母科1属1种共计265条线粒体COI基因同源序列,利用MEGA 3.0计算根口水母科种内与种间的遗传距离,并基于邻接法(Neibour-Joining,NJ)、最大似然法(Maximum Likelihood,ML)和贝叶斯法(Bayesian,BI)构建了其分子系统进化树。结果表明:4个属7种水母种类COI基因同源序列长度为482 bp,种间遗传距离为0.065~0.235,平均遗传距离为0.198,种内遗传距离为0~0.032,平均为0.006,其中种间平均遗传距离(0.198)显著大于种内平均遗传距离(0.006),种间遗传距离是种内遗传距离的33倍;根口水母科水母种间遗传距离均大于Hebert推荐区分物种的最小种间遗传距离(0.02);采用NJ法、ML法和BI法构建的分子系统树拓扑结构基本一致,同一物种不同个体间均能聚成独立的单系群,表明COI基因可作为根口水母科水母种类准确鉴定的有效条形码基因;系统进化树显示,海蜇属为单系群,系统进化分析结果与形态分类学的观点并不十分一致。研究表明,COI基因序列在根口水母科不同阶元间变异较大,适合于根口水母科水母种类鉴定及群体水平的分子标记。
DNA条形码;COI基因;根口水母科;系统分析
水母是一类海洋胶质类浮游生物,具有水螅体、水母体世代交替出现的生活史,不同时期形态变异较大且样品易碎、不易保存,再加上水母生物地理学研究的缺乏导致一些水母种类尚未被描述,因此,有关水母类的形态分类鉴定尚比较混乱和困难,存在诸多争议[1-5]。根口水母科Rhizostoidae水母在分类学上隶属于刺胞动物亚门Cnidaria、钵水母纲 Scyphomedusae、根口水母目Rhizostomeae。目前,已记录的根口水母科水母包括真水母属Eupilema1种、越前水母属1种、海蜇属4种、根口水母属3种,共4属9种,全为大型水母,其中既有经济价值较高的海蜇属种类如海蜇Rhopilemaesculentum、黄斑海蜇R.hispidum,也有近年来在西北太平洋沿海暴发的越前水母Nemopilemanomurai以及在大西洋和地中海海域暴发的根口水母属种类如根口水母Rhizostomaluteum、褐色根口水母Rhizostomapulmo、巨型桶水母Rhizostomaoctopus[4-6],这些灾害水母的暴发致使暴发区内渔业资源锐减并造成严重的生态灾难[6-11]。有关根口水母科水母形态分类及其系统发育关系尚存在较多争议,如很多学者怀疑真水母属的存在,认为其很可能是海蜇属受损坏、不完整的标本;越前水母此前曾被认为是口冠水母Stomolophusmeleagris的同种异名[3,5];海蜇属面蜇Rhopilemaasamusi曾被认为是海蜇R.esculentum的同种异名,但日本学者已证实其为独立的有效种[12],海蜇属实际包括5种。
随着分子生物技术的快速发展,DNA条形码在水母种类鉴定及系统发生关系研究中的应用越来越广泛,一些学者已利用线粒体基因(如16S rRNA、COI)和核基因(如18S、28S rDNA及ITS序列)对根口水母科相关种类进行了分子鉴定及系统相关研究,成功解决了一些有争议的分类问题[12-17]。然而由于水母分布广泛且样品难以采集,上述研究大多集中在某一个种或少数几个种的分子鉴定及群体遗传研究中,对水母类系统发育关系方面的研究仍比较缺乏,仅Dawson等[15]利用DNA条形码COI、ITS1对钵水母纲不同种间的分类进行了研究;Bayha等[16]利用18S和28S rDNA序列分析了钵水母类48个种的系统关系;李玉龙等[17]基于钵水母纲18个种类分析了ITS-5.8S rDNA序列在钵水母纲系统分析中的应用。但这些研究都是对整个钵水母纲水母进行的分析,尚无专门针对根口水母科水母系统发育关系进行分析的报道。此外,上述研究虽然分析的水母种类较多,但所分析的水母种每一种所包含的样品数量较少且采集地较为单一,这可能会导致由于“隐蔽种”的存在致使种类鉴定出现偏差。线粒体COI基因易于扩增且具有丰富的信息变异位点,是进行水母种类鉴定及分子系统分析最常用的分子标记,可以揭示长时间跨度从种、属乃至科不同分类阶元(taxon)的系统发生信息,弥补传统形态分类鉴定的局限。因此,本研究中对采自辽宁沿海的越前水母、海蜇进行了COI基因测定及序列分析,结合GenBank数据库中已有的来自不同海域的根口水母科6种水母以口冠水母S.meleagris为外群对根口水母科水母的系统发育关系进行分析,并探讨DNA条形码在根口水母科物种分子鉴定中的适用性,以期为根口水母科水母物种的有效鉴定、分类及系统发生等提供基础资料。
1 材料与方法
1.1样品采集及基因片段扩增
本研究中所采集的根口水母科样品包括海蜇和越前水母2种,每种样品各20个,都采自辽东湾北部附近海域。样品采集后取伞部及肩板组织于95%酒精中保存备用。采用CTAB法提取基因组DNA,PCR扩增在Eppendorf AG型PCR仪上进行,引物序列为:FFDL 5′TTTCAACTAACCAYAAAGAYATWGG 3′ 和FRDL2 5′TANACTTCWGGRTGNCCRAAGAATCA 3′[18]。反应体积(共25 μL)包括:10×PCR反应缓冲液2.5 μL,DNA模板1 μL,Taq酶(2 U/μL )0.5 μL,dNTPs(10 mmol/L)0.5 μL,每种引物(10 mmol/L )各1 μL,MgCl2(10 mmol/L )1.5 μL,用灭菌超纯水补足剩余体系。反应程序:95 ℃下预变性3 min;95 ℃下变性30 s,50~55 ℃下退火30 s,72 ℃下延伸45 s,共运行35个循环;最后在72 ℃下再延伸5 min。扩增产物纯化后由生工生物工程(上海)股份有限公司进行双向测序。
1.2序列下载
从GenBank数据库中下载根口水母科3属6种及口冠水母科1属1种共7种水母种类175条线粒体COI基因序列,与本研究中的海蜇、越前水母的同源序列共同进行数据分析。所分析水母种类的线粒体COI序列片段及其相关信息见表1。
1.3数据分析
对7种水母共215条序列进行系统分析,利用MEGA 3.0软件[19]基于K2P双参数模型计算单倍型间、种间、属间和种内个数间的遗传距离,并构建NJ(Neighbour-Joining)系统树,采用Bootstrap1000检验分子系统树各分支的置信度。利用Modeltest 3.7[20]选择最佳替代模型,基于Hierarchical Likelihod Ratio Tests (hLRTs)标准,最适替代模型(best-fit evolutionary models)为TVM+I+G。选择最佳替代模型后分别通过MrBayes 3.1.2[21]和PAUP* 4.0b10[22]软件构建贝叶斯(Bayesian,BI)一致树和ML(Maximum Likelihood)系统发育树。贝叶斯树参数设置如下:起始树设为随机树(random),马尔科夫链的蒙特卡洛方法(Markov Chain Monte Carlo Process,MCMC)设置为4条链,3条热链和1条冷链,同时运行1000万代,每1000代取样一次,其节点支持率为后验概率(posterior probabilities,PP),贝叶斯推断同时进行3次以确保MCMC的收敛;ML树采用启发式搜索(heuristic searches)和树二等分再连接(tree-bisection-reconnection,TBR)的分支交换法进行构树,并采用自展值分析(Bootstrap=20)进行检验。
2 结果与分析
2.1序列特征和条形码间隙
PCR扩增得到长度约650 bp的海蜇、越前水母COI基因同源序列,经比对去掉两端引物,本研究中分析得到:20条海蜇COI基因同源序列长度为624 bp,共检测到21个多态位点,定义了17种单倍型,单倍型间遗传距离为0.002~0.015,平均遗传距离为0.007;20条越前水母COI基因同源序列长度为620 bp,共检测到12个多态位点,定义了10种单倍型,单倍型间遗传距离为0.002~0.011,平均遗传距离为0.006。联合来自GenBank中的其他175条COI基因序列比对后,获得长度为482 bp 的共有序列进行分析。T、C、A、G 4种碱基的平均含量分别为36.5%、18.2%、25.3%、20.0%,A+T(61.8%)含量明显高于G+C(38.2%)含量,与其他水母类COI基因A+T含量高的组成特征相似[13]。除去外类群,在长度为482 bp的COI基因序列中,保守位点307 个(63.7%),变异位点175个(36.3%),简约性信息位点162个(33.6%)。
基于线粒体COI序列片段计算了根口水母科各阶元的遗传距离(表2~表4)。口冠水母科口冠水母属与根口水母科3属间的遗传距离平均值为0.211,口冠水母属与根口水母属Rhizostoma间的遗传距离最大值为0.219,口冠水母属与越前属Nemopilema间的最小值为0.203;根口水母科不同属间遗传差异从0.214到0.233,平均值为0.221,该范围与口冠水母科与根口水母科不同属间的分布范围重叠(表2)。根口水母科6个种两两间的遗传距离见表3,最小值出现在巨型桶水母种和褐色根口水母种之间(0.065),最大值出现在巨型桶水母种和越前水母种之间(0.235),种间遗传距离平均值为0.198。根口水母科水母COI种内遗传差异为0~0.032,平均值为0.006,种内遗传差异最大的是褐色根口水母,来自以色列海法湾和地中海西部的不同个体遗传差异可达0.032,而来自地中海的不同褐色根口水母个体间的遗传差异仅为0~0.013,揭示了褐色根口水母可能存在种群遗传结构(表4)。根口水母科水母种内、种间遗传距离存在差异,即存在条形码间隙(barcode gap),种间遗传距离是种内遗传距离的33倍,有利于种的鉴定。
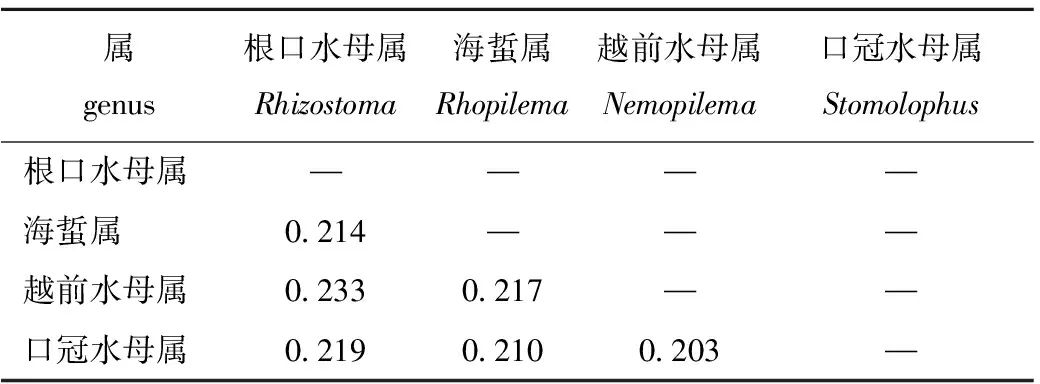
表2 根口水母科COI序列的属间遗传距离Tab.2 Genetic distance between genera of inter-family of Rhizostoidae based on the COI sequences
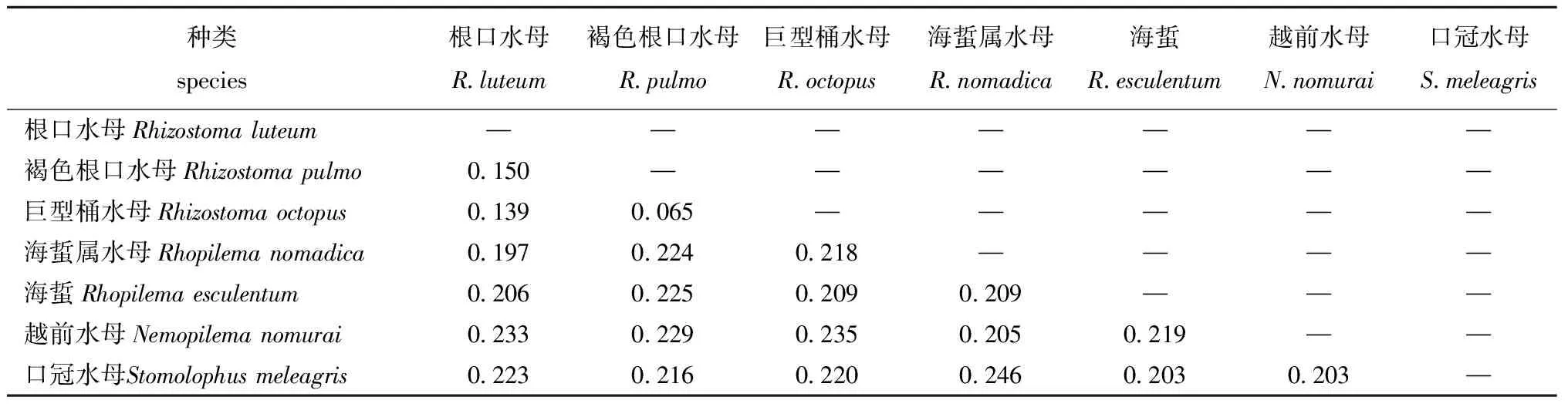
表3 根口水母科COI序列的种间遗传距离Tab.3 Genetic distance between species of inter-family of Rhizostoidae based on the COI sequences
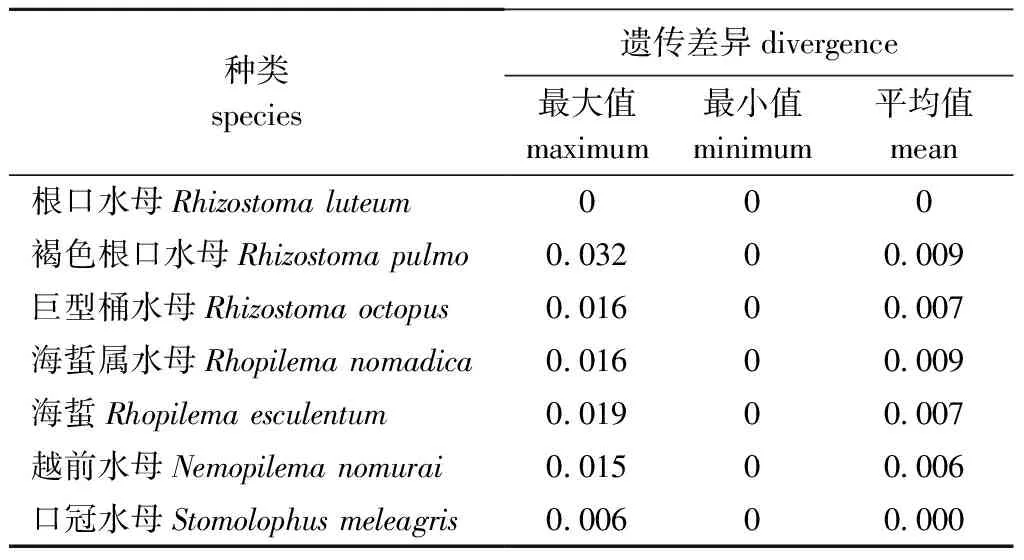
表4 根口水母科水母COI序列的种内个体间遗传距离Tab.4 Genetic distance of intra-species in Rhizostoidae based on the COI sequences
2.2系统分析
为得到可靠的结果,本研究中采用邻接法(Neighbor-Joining,NJ)、最大似然法(Maximum Likelihood,ML)和贝叶斯法(Bayesian,BI),以口冠水母为外群构建了7种水母的分子进化树(图1)。树上各节点上的数值分别为统计分析后对该分支NJ/BI/ML的支持率。结果显示,3种方法构建的根口水母科水母分子系统树的拓扑结构图基本一致,同种不同个体均各自聚支形成单系群且节点支持率均较高。根口水母属3个近缘种聚为一个单系群并得到较高的分支节点支持率,海蜇属不同物种之间未形成单系群,而是在进化树上形成了2个小分支,然后再与越前属聚支,且分支节点支持率都较低,这与Prieto 等[6]利用相同基因片段以及Funahashi 等[23]利用线粒体全基因序列对根口水母科水母构建的NJ或ML系统树的结果类似。此外,分子进化树揭示了地理分布相近的根口水母科种类优先聚支,譬如分布于地中海及大西洋的根口水母属种类优先与分布于地中海的海蜇属种类聚在一起,然后再与分布于西北太平洋的海蜇以及越前水母属聚支,分布于西北太平洋的越前水母属与同域分布的海蜇属种类表现出的亲缘关系更近。

图1 基于COI片段以口冠水母为外群构建的根口水母科水母NJ/BI/ML系统树Fig.1 Phylogenetic tree of the family Rhizostoidae inferred from NJ/BI/ML methods using COI gene sequences
3 讨论
3.1COI在根口水母科水母物种鉴定中的适用性
对许多物种而言,线粒体COI基因片段易于获得且具有良好的系统发育信息,是目前用于物种鉴定及系统进化研究最常用的分子标记[6,12-13,15,18]。自Hebert 等[24-25]提出以COI基因片段作为物种鉴定的DNA 条形码以来,其在物种鉴定中的有效性已在不同生物的研究中得到越来越多的验证[12-13],其在水母物种鉴定中的有效性也已得到有效检验[12-14]。但之前的研究或者是针对较高分类阶元如科及以上水母种类的物种鉴定,物种种类较多且亲缘关系较远;或者所分析的样品数量较少,容易导致误差的产生。GenBank数据库中的水母样品虽不知具体地理来源,但仍可将其用于种内遗传距离计算及系统分析。因此,本研究中分析了海蜇、越前水母各20个样品并联合来自GenBank中的其他175条线粒体COI基因同源序列对水母类科以下分类阶元根口水母科3属6种及口冠水母科1属1种进行分析,结果表明,其种间遗传距离范围为0.065~0.235,平均值为0.198;种内遗传范围为0~0.032,平均值为0.006,种间遗传距离是种内的33倍,且各物种间遗传距离均大于Hebert[24-25]推荐区分物种的最小种间遗传距离0.02,表明COI基因序列在较小的分类阶元如科以下仍可对水母进行有效的物种DNA鉴定。
3.2根口水母科水母的系统进化关系
基于分子标记构建水母的分子系统关系方面的研究,已有Bayha等[16]和李玉龙等[17]等分别利用18S、28S、ITS-5.8S rDNA序列分析了48种和18种水母的系统关系,但上述研究所用的分子标记都为核基因标记,且主要针对钵水母纲的分子系统关系。程方平等[13]利用COI基因片段对中国北方常见水母类的DNA条形码进行了分析,但其研究的对象是科以上及更高的分类阶元,由于较高分类阶元不同水平遗传距离存在重叠,其分子系统关系由于基因突变饱和而无法得到正确解析,而且程方平等[13]的研究中包含的根口水母科的种类较少。
根口水母是钵水母纲中进化最为高等的类群,大多分布于热带至亚热带海域,早在5 亿多年前的寒武纪就已出现[5,26-27]。因其具有漫长的进化历史且仍有很多种类未被发现[5],有关根口水母科水母的系统分类一直存在争议。 1961年Kramp等[1]在前人分类的基础上根据口腕有无附属物、肩板大小和垂管长短等将其分为真水母属、根口水母属和海蜇属3属,但Kramp等[1]怀疑真水母属的存在,认为其是海蜇属受损坏、不完整的标本,且将越前水母认为是口冠水母的同种异名,造成了分类上的混乱。2004年,Omori等[3]将两个物种重新对比后认为,越前水母与口冠水母系不同属、种,应将其回归为原来的越前水母属,随后分子生物学的证据[28-29]也表明,越前水母与口冠水母为不同的种,形成目前较普遍认可的根口水母科4属9种的观点[4-5]。Hanzawa等[12]根据COI条形码将曾被认为是海蜇同种异名的Rhopilemaasamushi提升为有效种,至此根口水母科水母已增加至4属10种。
线粒体COI基因不仅是进行物种鉴定的DNA条形码,而且是分子系统进化研究有效的分子标记。因此,本研究中基于COI条形码序列以口冠水母为外群分析了根口水母科3个属间的系统关系,结果表明,用3种方法构建的系统树拓扑结构基本一致,但与形态分类的结果并不一致。基于COI序列的系统树中,同种不同个体各自聚支形成单系群,各物种所代表的节点支持率均较高。在属内种间分类关系中,根口水母属的水母种类聚在一起形成单系群并得到较高的节点支持率(>0.9),而海蜇属水母在进化树上形成2个分散的独立分支(Ⅰ:地中海,Ⅱ:西北太平洋),且两个分支分别与同域分布的根口水母属及越前水母属水母亲缘关系更近,但各分支的节点支持率都较低。该结果与近几年利用相同基因片段以及利用线粒体全基因序列对根口水母科水母进行系统分析的研究报道一致[6,12]。基于线粒体COI基因片段计算根口水母科各阶元的遗传距离也表现出相同的趋势,同域分布水母种类间遗传距离差异更小,如分布于地中海的根口水母属水母Rhizostomaluteum与地中海水母Rhopilemanomadica间的遗传距离(0.197)甚至小于其与同属海蜇间的遗传距离(0.209,表3)。
此外,本研究中还发现,与其他海洋动物相比基于线粒体COI基因片段计算的根口水母科水母种间的遗传差异较大(0.065~0.235),这可能与其漫长的进化历史有关,在进化过程中由于地理隔离逐渐演变成不同的水母种类且产生较大的分化。本研究结果表明,COI基因序列即使在较小的分类阶元如种、属水平上可能也并不适合解析水母种类的系统发育关系,想要更加全面准确地了解根口水母科水母物种分类及系统进化关系,尚需采集更多的样品,利用多个分子标记序列进行分析并结合形态学与解剖学的数据才能得以有效解决。
[1] Kramp P L.Synopsis of the medusae of the world[J].J Mar Biol Ass U K,1961,40:7-382.
[2] Appeltans W,Ahyong S T,Anderson G,et al.The magnitude of global marine species diversity[J].Curr Biol,2012,22(23):2189-2202.
[3] Omori M,Kitamura M.Taxonomic review of three Japanese species of edible jellyfish (Scyphozoa:Rhizostomeae)[J].Plankton Biology and Ecology,2004,51(1):36-51.
[4] 董婧,姜连新,孙明,等.渤海与黄海北部大型水母生物学研究[M].北京:海洋出版社,2013.
[5] 洪惠馨.中国海域钵水母生物学及其与人类的关系[M].北京:海洋出版社,2014.
[6] Prieto L,Armani A,Macías D.Recent strandings of the giant jellyfishRhizostomaluteumQuoy and Gaimard,1827 (Cnidaria:Scyphozoa:Rhizostomeae) on the Atlantic and Mediterranean coasts[J].Marine Biology,2013,160(12):3241-3247.
[7] Uye S,Ueta Y.Recent increase of jellyfish populations and their nuisance to fisheries in the inland sea of Japan[J].Bulletin of the Japanese Society of Fisheries Oceanography,2004,68(1):9-19.
[8] Yoon W D,Lee H E,Han C,et al.Abundance and distribution ofNemopilemanomurai(Scyphozoa,Rhizostomeae),in Korean waters in 2005-2013[J].Ocean Science Journal,2014,49(3):183-192.
[9] Sun Song,Zhang Fang,Li Chaolun,et al.Breeding places,population dynamics,and distribution of the giant jellyfishNemopilemanomurai(Scyphozoa:Rhizostomeae) in the Yellow Sea and the East China Sea[J].Hydrobiologia,2015,754(1):59-74.
[10] 孙松.水母暴发研究所面临的挑战[J].地球科学进展,2012,27(3):257-261.
[11] 孙松.对黄、东海水母暴发机理的新认知[J].海洋与湖沼,2012,43(3):406-410.
[12] Hanzawa N,Gotoh R O,Kawamura M,et al.Rhopilemaasamushiuchida (Cnidaria,Scyphozoa,Rhizostomidae) newly occurred in Tanabe Bay,Wakayama Prefecture,Japan[J].Biogeography,2015,17:125-127.
[13] 程方平,王敏晓,王彦涛,等.中国北方习见水母类的DNA条形码分析[J].海洋与湖沼,2012,43(3):451-459.
[14] 张珰妮,郑连明,何劲儒,等.基于线粒体COI和16S片段序列的北部湾北部水螅水母DNA条形码分析[J].生物多样性,2015,23(1):50-60.
[15] Dawson M N,Jacobs D K.Molecular evidence for cryptic species ofAureliaaurita(Cnidaria,Scyphozoa)[J].The Biological Bulletin,2001,200(1):92-96.
[16] Bayha K M,Dawson M N,Collins A G,et al.Evolutionary relationships among scyphozoan jellyfish families based on complete taxon sampling and phylogenetic analyses of 18S and 28S ribosomal DNA[J].Integrative and Comparative Biology,2010,50(3):436-455.
[17] 李玉龙,董婧,孙明,等.4种大型水母类ITS-5.8S rDNA序列分析及其在钵水母类系统分析中的应用[J].中国水产科学,2015,22(6):1278-1288.
[18] Armani A,Tinacci L,Giusti A,et al.What is inside the jar? Forensically informative nucleotide sequencing (FINS) of a short mitochondrial COI gene fragment reveals a high percentage of mislabeling in jellyfish food products[J].Food Research International,2013,54(2):1383-1393.
[19] Kumar S,Tamura K,Nei M.MEGA 3:integrated software for molecular evolutionary genetics analysis and sequence alignment[J].Brief Bioinform,2004,5(2):150-163.
[20] Posada D,Crandall K A.MODELTEST:testing the model of DNA substitution[J].Bioinformatics,1998,14(9):817-818.
[21] Ronquist F,Huelsenbeck J P.MrBayes 3:bayesian phylogenetic inference under mixed models[J].Bioinformatics,2003,19(12):1572-1574.
[22] Swofford D L.PAUP*:phylogenetic analysis using parsimony (and other methods),version 4[M].Sunderland,Massachusetts:Sinauer Associates,2003.
[23] Funahashi A,Kadono T,Gotoh R O,et al.Analysis of mitochondrial genome ofN.nomuraiand development of new gene markers from the data[C]//The 11th China-Japan-Korea International Jellyfish Workshop.Xi’an,China,2014.
[24] Hebert P D N,Ratnasingham S,de Waard J R.Barcoding animal life:cytochromecoxidase subunit 1 divergences among closely related species[J].Proc Royal Soc B,2003,270(S1):S96-S99.
[25] Hebert P D N,Cywinska A,Ball S L,et al.Biological identifications through DNA barcodes[J].Proc Royal Soc B,2003,270(1512):313-321.
[26] Hagadorn J W,Dott R H Jr,Damrow D.Stranded on a Late Cambrian shoreline:medusae from central Wisconsin[J].Geology,2002,30(2):147-150.
[27] Chen Junyuan,Schopf J W,Bottjer D J,et al.Raman spectra of a Lower Cambrian ctenophore embryo from southwestern Shaanxi,China[J].Proc Natl Acad Sci,2007,104(15):6289-6292.
[28] 张姝,张芳,刘媛,等.我国海域两种大型水母的分子鉴定[J].海洋与湖沼,2009,40(1):94-101.
[29] 刘敏,马凌波,凌建忠,等.东黄海沙海蜇与口冠水母分类关系的辨析——基于核糖体18S rDNA基因序列[J].海洋渔业,2011,33(2):131-137.
PhylogeneticrelationshipofRhizostomatidaebasedonCOIbarcodingmarker
LI Yu-long, WANG Bin, SUN Ming, WANG Xiao-lin, YU Xu-guang, LI Yi-ping, FU Jie, WANG Wen-bo, DONG Jing
(Key Laboratory of Marine Biological Resources and Ecology in Liaoning Province, Liaoning Ocean and Fisheries Science Research Institute, Dalian 116023, China)
The 482 bp COI was sequenced in 265 individuals sampled from 7 species in 4 genera in Rhizostomatidae, the genetic distances in inter-and intra-species were calculated by MEGA 3.0, and the molecular phylogenetic trees were constructed using Neighbor- Joining, Bayesian inference and Maximum Likelihood methods to investigate the feasibility of COI sequences as DNA barcodes to identify species in Rhizostomatidae. The results showed that the inter-species genetic distance was changed from 0.065 to 0.235, with average of 0.198, while the intra-species genetic distance was varied from 0 to 0.032, with average of 0.006, 32 times higher than that in intra-species. Moreover, each inter-species genetic distance value is higher than the minimum species identification value 0.02 proposed by Hebert. The phylogenic analysis showed that the topology of the Neighbor-Joining tree, Maximum Likelihood tree and the Bayesian tree was consistent each other and individuals of each species formed an independent monophyletic group, indicating that COI was of an effective barcode gene for accurate identification of species among Rhizostomatidae. Meanwhile, the phylogenetic trees showed thatRhopilemajellyfishes are non-monophyletic and different from morphological taxonomy. In conclusion, COI is highly divergent among different jellyfishes in Rhizostomatidae and is an appropriate marker for species identification and population studies at the species level in Rhizostomatidae.
DNA barcode; COI gene; Rhizostomatidae; phylogeny
10.16535/j.cnki.dlhyxb.2017.05.004
2095-1388(2017)05-0520-06
S917
A
2017-03-09
国家自然科学基金资助项目(31770458,31400406);国家重点研发计划项目(2017YFC1404400)
李玉龙(1981—),男,副研究员。E-mail:liyudragon@126.com
董婧(1966—),女,研究员。E-mail:1024470248@qq.com

