两种木腐真菌的分离及鉴定
孙婧,戚大伟*,池玉杰,牟洪波
(1.东北林业大学 理学院,哈尔滨 150040;2.东北林业大学 林学院,哈尔滨 150040)
两种木腐真菌的分离及鉴定
孙婧1,戚大伟1*,池玉杰2,牟洪波1
(1.东北林业大学 理学院,哈尔滨 150040;2.东北林业大学 林学院,哈尔滨 150040)
在影响木材质量的众多因素中,木腐真菌侵染木材造成的木材腐朽现象对木材细胞壁破坏最为严重。为了鉴定侵染木材腐朽的病原菌,提高木材质量,本文建立一种有效鉴定木腐真菌的分子生物学方法。以采集木腐真菌子实体为研究对象,通过分离和选择培养基筛选出目的菌株,并判断木腐真菌的木材腐朽类型。运用基于PCR技术的分子生物方法,通过提取基因组DNA,primer5软件设计特异性引物对序列扩增,MEGA5.0软件构建系统发育树,对木腐真菌进行分子水平的鉴定。并结合以往研究者运用的传统形态学鉴定方法,对木腐真菌形态等特征进行观察。研究结果表明,木腐真菌S1为红缘拟层孔菌(Fomitopsispinicola),木腐真菌S2为木蹄层孔菌(Fomesfomentarius)。将分子生物学方法与传统形态学方法相结合,能够更加准确和可靠的对木腐真菌进行鉴定。
木腐真菌;分离纯化;分子生物学鉴定;形态学鉴定
E-mail:qidw9806@126.com
0 引言
人类生活和生产中离不开对木材的使用,近年来,随着我国经济的发展,森林资源出现供不应求的现象[1]。而在自然界中,木材很容易受到木材腐朽病原菌的侵染,使木材发生腐朽[2-4]。木腐真菌,即木材腐朽菌,能够分解木材细胞壁的大型真菌,是导致木材解体和腐朽的重要因素[5-6]。木材腐朽发生频率高,分布范围广,在适宜环境条件下,木腐真菌常常侵染林区的活立木、倒木、枯立木和木材生产使用等场所,降低木材的质量,甚至使其失去使用价值,对森林资源的保护和利用造成了巨大的经济损失[7-8]。
为更加合理的使用木材,降低木腐真菌对木材质量的影响,有必要对木腐真菌进行鉴定。目前,大多数采用传统形态学观察法对木腐真菌进行鉴定,但对于形态极其相似的子实体,有时难以进行区分。而且不同研究者对同一菌落形态和菌丝特征描述不同,产生的鉴定结果可能不同,此时该方法不具备准确性。本文运用基于PCR技术的分子生物学手段,并结合形态学方法,对侵染木材的木腐真菌进行了更加可靠的鉴定[9-10]。
1 材料与方法
1.1 材料
1.1.1 样品采集
从小兴安岭凉水自然保护区白桦林和大兴安岭五营保护区的红松林采集木腐真菌子实体,用灭菌的小刀从活立木材树干部切割,放到无菌袋中备用。
1.1.2 培养基
基础培养基:马铃薯去皮200 g,葡萄糖20 g,琼脂18 g,加蒸馏水定容至1 L,pH自然。
液体培养基:马铃薯去皮200 g,葡萄糖20 g,加蒸馏水定容至1 L,pH自然。
真菌分离培养基:马铃薯去皮200 g,葡萄糖20 g,琼脂18 g,蛋白胨5 g,KH2PO43 g,MgSO41.5 g,VB110 mg,蒸馏水1 000 mL,pH 6.0[11]。
1.1.3 实验仪器和主要试剂
仪器:YQH-400F人工气候培养箱、超净无菌操作台、(GR85DA致微)高压蒸汽灭菌锅、TechnePrimeQ实时荧光定量PCR仪、紫外可见分光光度计、恒温震荡水浴摇床、电子分析天平等。
试剂:生工SK8259 DNA提取试剂盒、愈创木酚、琼脂糖(巴西)、GoldView、buffer、5×PCR Buffer、dNTP、Taq DNA聚合酶购自上海生工生物工程技术服务有限公司。
1.2 方法
1.2.1 木腐真菌的分离与纯化
用蒸馏水和棉球将子实体表面擦拭干净,在无菌操作台中,用灭菌的镊子挑取子实体内部有活力的组织块,取4块接种于真菌分离培养基中,在25℃恒温箱中培养。挑取菌落边缘菌丝,接种于液体培养基中培养7 d,涂布平板,反复划线纯化直至获得纯菌落,接种于斜面培养,存放于4℃冰箱中,保存备用[12-13]。
菌株复筛:用0.5 g的愈创木酚溶于30 mL 95%的乙醇中,滴定菌落边缘,观察菌落滴定区域变化。根据Bavendamm反应,产生显色反应说明该菌株具有降解木质素的能力,即产生显色圈的菌株为木材白腐菌,由此可以判断木材腐朽菌的木材腐朽类型[14]。
1.2.2 形态学观察法
野外采集木腐真菌的子实体,对其外部形态进行拍照观察,并制作临时载玻片,在光学显微镜下观察菌落形态和菌丝特征[15-16]。
1.2.3 菌种基因组DNA提取与PCR体系的建立
采用上海生工SK8259 DNA试剂盒提取基因组DNA,以ITS1和ITS4为通用引物扩增DNA序列[16-18]。
PCR反应体系(25 uL):基因组DNA 0.5 uL,5×PCR Buffer(含MgCl2)2.5 uL,dNTP(2.5 mmol/L)1 uL,F Primer 0.5 uL,R Primer 0.5uL,Taq DNA聚合酶0.2 uL,最后加dd H2O补至总体积为25 uL。
PCR反应条件:94℃预变性4 min,94℃变性45 s,55℃退火45 s,72℃延伸1 min,共35个循环,再72℃延伸10 min,4℃保存[19-20]。
1.2.4 PCR产物纯化和测序
PCR扩增产物用1%的琼脂糖凝胶电泳检测,用TIANGEN琼脂糖回收试剂盒进行胶回收,将目的片段送检上海生工公司测序。DNA纯度用紫外可见分光光度计检测,获得OD260和OD280的比值,验证提取DNA的纯度。
1.2.5 系统发育树的构建和特异性引物的设计
登陆NCBI数据库,将扩增序列在BLAST中比对,选取与鉴定菌种相似度在99%以上序列,用MEGA5.0构建系统发育树[21],分析亲缘关系。
从genbank下载红缘拟层孔菌和木蹄层孔菌的ITS序列,序列号分别为:FJ608588、 KU171405、JX470538.1和JX163102.1。用primer5软件设计特异性引物,对ITS序列进行PCR扩增(表1)。

表1 特异性引物的设计Tab.1 Primers for PCR
2 结果与分析
2.1 形态学鉴定
子实体S1,无柄,菌盖表面光滑且坚硬,具有褐色胶质的皮壳。子实体边缘钝圆,菌肉木质并呈淡黄色。菌落生长中度较快,3~5周内长满覆盖平皿,菌落正反面均为白色,菌丝紧贴培养基内部均匀生长,边缘轻微升起呈亚毡状。具有轻微蘑菇香味,5周后菌落正面略变褐色,背面培养基略变黄色(图1)。生长新区的菌丝无色,菌丝较粗,有节状分隔和小囊状体,壁薄,孢子卵形至椭圆形(图2)。
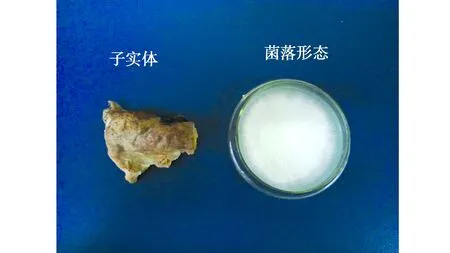
图1 木腐真菌S1的子实体和菌落形态Fig.1 Fruit body and colony morphology of wood-rotting fungi S1
子实体S2,马蹄形,无柄,菌盖表面光滑且坚硬,具有灰色角质皮壳和明显同心环棱。具有木栓质的褐色菌肉,子实体背面呈深褐色。菌落生长快速,2~3周内长满覆盖平皿,菌落中心褐色,周围白色,平伏贴生于培养基表面生长。菌落正反面颜色一致,无气味(图3)。生长新区菌丝无色,有节状分隔和分支,纤维菌丝较多,具有加厚的壁,无分生孢子(图4)。
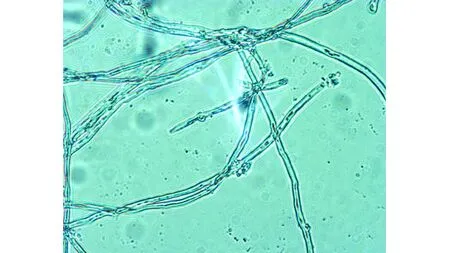
图2 光学显微镜下木腐真菌S1的菌丝照片Fig.2 The mycelium photos of wood-rotting fungi S1 under optical microscope

图3 木腐真菌S2的子实体和菌落形态Fig.3 Fruit body and colony morphology of wood-rotting fungi S2

图4 光学显微镜下木腐真菌S2的菌丝照片Fig.4 The mycelium photos of wood-rotting fungi S2 under optical microscope
根据《中国真菌志》,初步判断菌株S1为红缘拟层孔菌(Fomitopsispinicola),菌株S2为木蹄层孔菌(Fomesfomentarius)。传统形态学鉴定方法,主要根据子实体的形态,如菌盖、菌肉、味道等宏观和菌丝观察等微观特征,但一些极其相似的子实体,通过这种方法,有时难以准确的进行识别。而且不同研究者对同一种菌落、菌丝等观察和形态描述可能不同,或者子实的形态等发生改变,鉴定结果可能不准确。因此本实验将运用分子生物学方法,并结合形态学方法进行鉴定。
2.2 分子生物学鉴定
对木腐真菌分离纯化,筛选出两株目的菌株。根据滴定愈创木酚的实验判断,S1无显色圈出现,腐朽类型为木材褐腐菌,S2有显色圈出现,腐朽类型为木材白腐菌。
紫外分光光度计检测DNA纯度,S1:OD260/OD280=1.91,S2:OD260/OD280=1.95,结果表明DNA纯度较高,图5为提取总DNA电泳检测结果和PCR扩增产物片段,S1和S2长度分别为650bp和600bp左右,目的条带完整,无明显降解。
在NCBI数据库中BLAST比对,初步鉴定菌株S1为红缘层孔菌(Fomitopsispinicola),菌株S2为木蹄层孔菌(Fomesfomentarius)。图6为构建系统发育树分析,结果显示S1与NCBI登录号HM584810.1
序列相似性99%,S2与NCBI登录号KM584810.1序列相似性99%。
所设计的特异性引物FNX2和FNX6分别能且仅能扩增出红缘拟层孔菌和木蹄层孔菌的特异性目的条带,并可成功扩增出与鉴定菌种相似率95%以上的DNA序列,说明FNX2和FNX6引物具有良好的适用性和特异性,可以在两种菌种鉴定过程中进行应用。
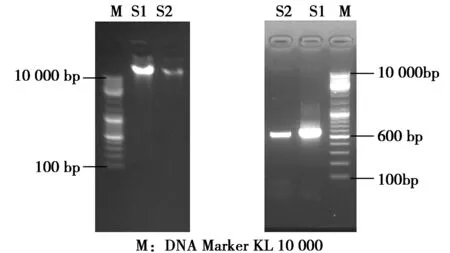
图5 供试菌株基因组DNA和ITS扩增序列 在1%琼脂糖凝胶电泳检测结果Fig.5 Results of agarose(1%)electrophoresis of genomic DNA on tested product
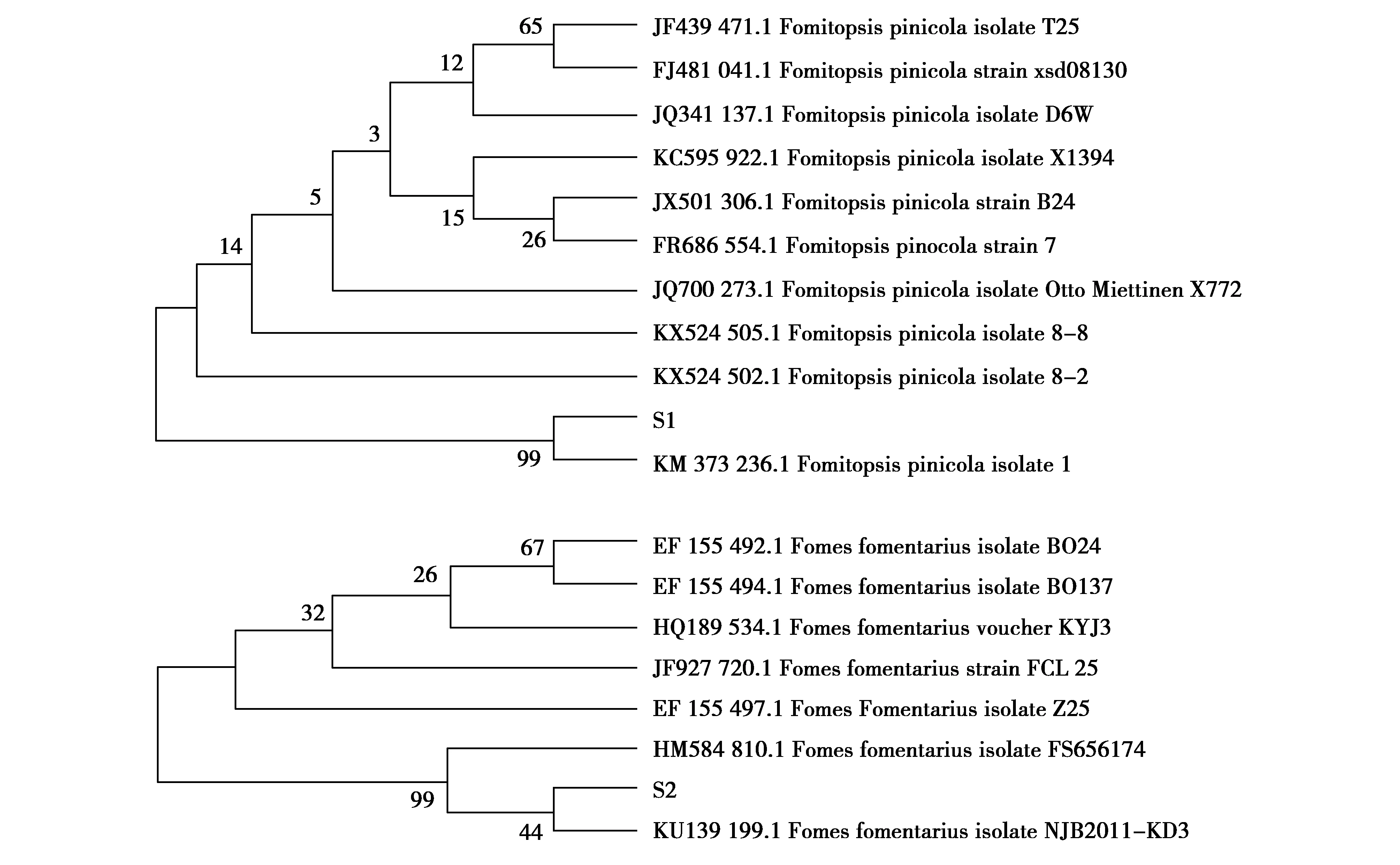
图6 木腐真菌S1和S2的系统发育树构建Fig.6 The phylogenetic tree for wood-decaying fungi S1 and S2
3 结论
本文通过分离和选择培养对木腐真菌子实体进行分离纯化,最终获得两种木腐真菌。通过腐朽类型实验判断,得出S1为木材褐腐菌,S2为木材白腐菌。通过基于PCR技术的分子生物学鉴定方法,构建系统发育树并设计特异性引物,初步鉴定木腐真菌S1为红缘拟层孔菌(Fomitopsispinicola),S2为木蹄层孔菌(Fomesfomentarius)。与形态学鉴定结果一致,但由于形态学方法具有不准确性,因此可将两种鉴定方法相结合,能够更加准确、可靠的对木腐真菌进行鉴定。
[1] 冯璐,戚大伟.基于PCR技术的木材腐朽菌鉴定方法的研究[J].森林工程,2016,32(5):35-39.
[2] Zhou L W,Dai Y C.Progress report on the study of wood-decaying fungi in China[J].Science Bulletin,2012,57(33):4328-4335.
[3] 别格坚巴哈提,邓玉侠.樟子松苗木促生长高效外生菌根菌的筛选[J].林业科技,2017,42(3);31-33.
[4] 庞丽杰,范晓旭.樟子松凋落物中可培养真菌多样性的研究[J].林业科技,2016,41(2);48-50.
[5] 戴玉成.中国木本植物病原木材腐朽菌研究[J].菌物学报,2012,31(4):493-509.
[6] Bednarz J C,Huss M J,Benson T J,et al.The efficacy of fungal inoculation of live trees to create wood decay and wildlife-use trees in managed forests of western Washington,USA[J].Forest Ecology and Management,2013,307(6):186-195.
[7] 司静,戴玉成.东喜马拉雅山地区木材腐朽菌研究5多孔菌的多样性[J].菌物学报,2016,35(3):252-278.
[8] Jang Y,Jang S,Lee J,et al.Wood decay fungi in South Korea:polypores from seoul[J].Mycobiology,2014,42(2):140-6.
[9] Schmidt O,Gaiser O,Dujesiefken D.Molecular identification of decay fungi in the wood of urban trees[J].European Journal of Forest Research,2012,131(3):885-891.
[10] Riley R,Salamov A A,Brown D W,et al.Extensive sampling of basidiomycete genomes demonstrates inadequacy of the white-rot/brown-rot paradigm for wood decay fungi[J].Proceedings of the National Academy of Sciences of USA,2014,111(27):9923-8.
[11] 宋贤冲,唐健,邓小军,等.产纤维素酶真菌的分离筛选、鉴定及其酶学性质分析[J].基因组学与应用生物学,2013,32(3):372-378.
[12] Vantamuri A B,Kaliwal B B.Isolation,screening and identification of laccase producing fungi[M].Understanding Employment Relations.Mcgraw-HillPubl.Comp.2015.
[13] 穆春雷,武晓森,李术娜,等.低温产纤维素酶菌株的筛选、鉴定及纤维素酶学性质[J].微生物学通报,2013,40(7):1193-1201.
[14] 龚国利,陈志宣,陈松,等.高产漆酶白腐真菌的分离与鉴别[J].陕西科技大学学报,2012,30(6):79-83.
[15] 杨立霞,马欣然,王玉英,等.三种白腐菌生物学特性与木质纤维素酶基因遗传多样性[J].生态学报,2016,36(7):2034-2043.
[16] Beales P,Lane C,Hughes K J D.Fungal plant pathogens[M].CABL,2012.
[17] Rangno N,Jacobs K,Langensiepen P.Effective DNA extraction methods for molecular diagnostics of wood decay fungi[J].Mycoses,2014,57:43-43.
[18] Umesha S,Manukumar H M,Sri R.A rapid method for isolation of genomic DNA from food-borne fungal pathogens [J].Biotech,2016,6(2):123.
[19] Khot P D,Fredricks D N.PCR-based diagnosis of human fungal infections[J].Expert Review of Anti-infective Therapy,2009,7(10):1201-1221.
[20] 尹立伟,池玉杰.猴头菌MnP1基因全长cDNA克隆及生物信息学分析[J].林业科学,2015,51(5):68-77.
[21] Escapa I H,Catalano S A.Phylogenetic Analysis of Araucariaceae:Integrating molecules,morphology,and fossils[J].International Journal of Plant Sciences,2013,174(8):1153-1170.
IsolationandIdentificationoftwoWood-rottingFungi
Sun Jing1,Qi Dawei1*,Chi Yujie2,Mu hongbo1
(1.College of Science,Northeast Forestry University,Harbin 150040; 2.College of Forestry,Northeast Forestry University,Harbin 150040)
Among all the many factors that affect the quality of wood,wood decay caused by wood-rotting fungi is the most serious damage to wood cell wall.In order to identify the pathogens causing wood decay and improve the quality of wood,a molecular biological method for the effective identification of wood-rotting fungi was established.Using collected wood-rotting fungi bodies as studying objects,the target strains were screened by separation and selection of culture medium,then the type of decayed wood-rooting fungi were determined.The DNA was extracted,and Primer5 software was used to design specific primers for sequence amplification.MEGA5.0 software was used to construct phylogenetic tree.And then molecular identification of wood-rotting fungi was carried out at molecular level.Combining with the traditional morphological identification methods used by previous researchers,the morphological characteristics of wood-rotting fungi were observed.The results showed that the wood-rotting fungi S1 wasFomitopsispinicola,and the wood-rotting fungi S2 wasFomesfomentarius.Combining molecular biology methods with traditional morphological methods,wood-rooting fungi can be identified more accurately and reliably.
Wood-rotting fungi;isolation and purification;molecular biology identification;molecular identification
S 782.33
:A
:1001-005X(2017)05-0045-05
2017-04-01
国家自然科学基金项目(31570712);黑龙江省教育厅科学技术研究项目(12543019);黑龙江省自然科学基金项目(C201338);高校科研基金项目(2572014CB30);中央高校基本科研业务费专项资金资助项目(2572016AB26)
孙婧,硕士研究生。研究方向:生物物理。
戚大伟,博士,教授。研究方向:生物物理。
孙婧,戚大伟,池玉杰,等.两种木腐真菌的分离及鉴定[J].森林工程,2017,33(5):45-49.

