拟南芥仅含START结构域亚家族基因特性与功能分析
闫青地,赵亚林,张昊,高静,高梦烛,王倩,王蕊,王凤茹,董金皋
拟南芥仅含START结构域亚家族基因特性与功能分析
闫青地,赵亚林,张昊,高静,高梦烛,王倩,王蕊,王凤茹,董金皋
(河北农业大学生命科学学院/河北省植物生理和分子病理学重点实验室,河北保定071001)
【目的】分析拟南芥中仅含START结构域(the lipid/sterol-binding StAR-related lipid transfer protein domains)亚家族成员的启动子元件及表达特性,阐明启动子元件与表达特性的相互关系,为明确仅含START结构域亚家族的生物学功能,解析植物生长发育机理、更好地促控植物生长提供理论依据。【方法】利用生物信息学方法分析仅含START结构域亚家族6个成员的亲缘关系及各自启动子区所包含的所有元件,对启动子元件的功能进行分析和分类;利用real-time PCR方法研究亚家族成员的表达特性;用突变体分析确认基因的功能;分析通过启动子元件分析预测的基因功能与通过突变体分析确认的基因功能间的相关性,为基因的功能和作用机制分析提供科研思路和方法。【结果】生物信息学分析表明,拟南芥中仅含START结构域亚家族成员共6个,分为3个分支,每个分支一对基因,每一对基因中均有一个基因的启动子中含有高转录水平元件5′ UTR Py-rich stretch,6个亚家族成员的启动子区均含有多个光、激素和逆境响应元件,也存在各自特异的响应元件;用real-time PCR分析基因的表达量,结果与生物信息学预测结果一致:At3g13062的表达量明显高于同分支的At1g55960,At3g23080的表达量明显高于同分支的At4g14500,At1g64720的表达量明显高于同分支的At5g54170。组织定位分析表明亚家族成员有特异的时空表达特性:At3g13062和At1g55960在幼苗期就有明显表达,但At3g13062主要在子叶、下胚轴和根中表达,而At1g55960主要在根中表达量较多;在成苗期拟南芥中,At3g13062主要在叶片中表达,在根中表达量较少;而At1g55960在成苗期拟南芥的叶片和根系的表达量均较多;At3g23080和At4g14500在幼苗期表达量较少,而在成苗期表达量增加,At3g23080主要分布于莲座叶中,At4g14500主要分布于莲座叶叶柄部。上述结果表明仅含START结构域亚家族成员可能在拟南芥不同时期和不同的部位分别行使着功能。利用生物信息学对启动子元件分析发现,各个基因均具有胚乳特意表达的Skn-1 motif元件,暗示着该家族基因可能参与调控胚乳的发育,进而影响种子的活力和萌发;通过对At3g23080和At4g14500的T-DNA插入突变体种子的分析表明,At3g23080和At4g14500表达量下降均会导致种子活力和萌发率的下降,这与生物信息学预测结果一致。【结论】拟南芥中仅含START结构域的亚家族有6个成员;6个亚家族成员可能在光、激素和逆境调控的发育过程中具有重要的作用;含START结构域的亚家族6个成员虽然同源关系较近,但具有各自的时空表达特性,在拟南芥的不同组织和不同发育时期执行着各自的功能。基因启动子元件与基因功能有直接关系,启动子区具有胚乳特意表达Skn-1 motif元件的At3g23080和At4g14500表达量下降均会导致种子活力和萌发率下降。
拟南芥;START结构域;启动子元件;表达特性;基因功能
0 引言
【研究意义】动物中,含START(the lipid/sterol- binding StAR-related lipid transfer protein domains)结构域的蛋白质参与磷脂运输和脂质代谢,与疾病的发生密切相关。但植物中START结构域的功能还不十分清楚。以编码仅含START结构域蛋白质的基因为研究对象,检测其表达特性、分析START结构域在进化中的地位,对解析植物生长发育机理、更好地促控植物生长发育具有重要的理论价值。【前人研究进展】START结构域最早在哺乳动物中发现,可以和脂类或甾醇类物质结合转运脂类物质[1]。哺乳动物中START结构域的晶体结构形成一个疏水腔,可结合一个单一配体[2]。结合的配体有固醇、胆固醇、羟基胆固醇、磷脂、鞘脂等[3],最近研究发现,START5蛋白还可以结合胆汁酸[4],因此,START结构域在这些配体的转运过程中至关重要。人类基因组编码15个含START结构域的蛋白质,一些与疾病的发生密切相关,如STARD1决定遗传疾病先天性类脂性肾上腺增生,研究发现,先天性类脂性肾上腺增生病人是START1蛋白的氨基酸发生了变化影响了START结构域的功能造成的[5];START2(磷脂酰胆碱转运蛋白,phosphalidylcholine transfer protein,PCTP)是可转运胆固醇到线粒体内膜的类固醇类调节蛋白[6];START11(ceramide transfer protein,CERT)神经酰胺转运蛋白,可以通过其START结构域来运输神经酰胺[7]。植物中存在大量的含START结构域的蛋白质,大部分成员属于植物特有的HD-ZIP(homeodomain leucine zipper)转录因子家族[8]。拟南芥()中含21个HD-ZIP START结构域的转录因子,在维管束发育、分生组织形成和极性建成等过程中起着重要的作用(如GL2[9]、ANL2[10]、PDF2[11]、ATML1[11]、FWA[12]、PHV[13]、PHB[14]、ATHB-8[15-16]、REV[17]等)。START结构域在这些转录因子中的作用类似于动物中的甾醇类激素受体,通过结合脂类或甾醇类物质来调控转录过程[18]。在HD-ZIP START家族成员GL2的功能研究中,发现START结构域对GL2的转录因子活性是必需的[4]。用老鼠和拟南芥中ATML1的START结构域替代GL2的START结构域后,可以恢复GL2的功能,说明START结构域的功能是保守的[4];用动物和植物中的START结构域替代酵母中的START结构域,均可刺激转录因子的活性,START虽然能激活转录因子,但与传统的转录激活域不同,其激活活性只有在转录因子的激活域也存在时才发挥作用[4]。虽然用小鼠的START结构域和拟南芥ATML1中的START结构域替代拟南芥GL2中的START结构域,功能可以互补替代,但用EDR2和REV中的START结构域替代GL2中START结构域则不能回复GL2的表型,说明START结构域的功能依赖于其编码结构域的序列[19]。研究发现,在动物、植物和微生物中,START结构域均通过与配体的结合调控转录活性[20]。【本研究切入点】植物中含START结构域的家族除了定位于细胞核中的HD-ZIP START家族外,还有只含有START结构域的含跨膜片段的成员,如At3g13062、At1g55960、At3g23080、At4g14500、At5g54170、At1g64720编码的蛋白,这些蛋白仅含START结构域,其功能未知。START结构域具有磷脂酰胆碱结合位点,磷脂酰胆碱是细胞膜的主要成分,推测其在细胞膜稳定性方面具有重要作用。【拟解决的关键问题】通过解析编码仅含START结构域蛋白亚家族基因的顺式作用元件,预测蛋白质的功能;利用遗传学方法确定顺式作用元件启动的START结构域的功能;阐明顺式作用元件和基因功能间的关系,为基因功能研究提供理论依据。
1 材料与方法
试验于2014年3月至2017年4月在河北省植物生理和分子病理学重点实验室完成。
1.1 试验材料
哥伦比亚野生型拟南芥(Col-0)由河北省植物生理和分子病理学重点实验室保存。
培养条件:光周期为16 h光照/8 h黑暗,光照强度为150 μmol·m-2·s-1,温度为22℃,相对湿度为50%—60%。
大肠杆菌()感受态DH5、T载体PMD19-T Vector均购自北京全式金生物技术有限公司(http://www.transgen.com.cn/);根癌农杆菌()GV3101,pCAMBIA 1381z植物组织定位载体由河北农业大学分子和植物病理学实验室保存。
1.2 生物信息学分析
基因的启动子作用元件、保守域及结合位点通过Plant CARE、NCBI(http://www.ncbi.nlm.nih.gov)及TAIR网站(http://www.arabidopsis.org)进行分析。
1.3 基因表达分析
参照北京全式金生物技术有限公司SYBR Green Real-time PCR Super Mix(AQ141)步骤进行。使用荧光定量PCR仪(伯乐IQ5M)进行PCR反应。反应程序为94℃30 s;94℃5 s,55℃15 s,72℃10 s,40个循环;72℃7 min。所用引物见表1。
1.4 基因组织定位分析
在TAIR网站(http://www.arabidopsis.org/)获得目的基因起始密码子ATG上游约1 500 bp序列,根据组织定位载体pCAMBIA 1381z酶切位点及目的基因启动子序列的可用酶切位点,用Primer 5.0设计目的基因启动子引物。以野生型拟南芥DNA为模板扩增目的基因的启动子序列;将其与组织定位载体pCAMBIA 1381z连接;连接后用花絮侵染转化拟南芥,经GUS染色、无水乙醇脱色,显微镜下观察目的基因的组织定位情况。
1.5 花絮侵染法转化野生型拟南芥及纯合株系的筛选
接种含有组织定位载体的农杆菌于60 mL YEB液体培养基(含100 g·mL-1Kan,100 g·mL-1Rif)中,28℃,150 r/min,振荡培养至对数生长晚期;按1﹕50的比例转接到200 mL YEB培养基中,28℃,150 r/min,振荡培养至OD600≈0.6;5 000 r/min,离心15 min,收集菌体;重悬于转化介质(5%蔗糖,0.02% silwet L-77),待OD600≈0.6时,将拟南芥花蕾在渗透缓冲液中浸泡30 s,平放,遮光保湿培养24 h,然后置拟南芥于培养室中继续正常培养;成熟后,收获拟南芥种子;将拟南芥种子播种于潮霉素抗性筛选平板上,筛选3代,获得纯合株系。
1.6 基因的组织定位分析
选取目的基因启动子-GUS载体转基因幼苗和成熟植株浸入提前配制好的GUS染色液中,于37℃染色20 h(幼苗的染色时间相对较短,约3 h),无水乙醇脱色,显微镜下观察。
1.7 种子活力测定
采用氯化三苯基四氮唑(TTC)法测定种子活力。取待测拟南芥种子用30℃左右的温水浸泡2—6 h,使种子吸水膨胀;随机取100粒吸胀种子放入培养皿中,加入TTC溶液,以浸没种子为度;放入30—35℃的恒温箱内保温6 h;倒出药液,用蒸馏水冲洗2—3次,立即观察种胚着色情况,判断种子有无活力。判断标准:活力较强的种子染成鲜红色;活力较弱的种子胚染成粉红色或淡红色。

表1 PCR扩增和基因表达所用引物序列
2 结果
2.1 拟南芥中编码仅含START结构域家族基因启动子分析
拟南芥中仅含START结构域的蛋白质进化关系如图1,仅含START结构域亚家族成员共6个,分为2个大分支:At1g55960和At3g13062在一个大分支,同源关系较近;在另一个大分支的其他4个基因又分为2个分支,At1g64720和At5g54170在一个分支,At4g14500和At3g23080在另一个分支。
通过拟南芥信息资源网站(TAIR)获得了目的基因上游约1 500 bp的启动子片段序列,对目的基因启动子所包含的顺式作用元件进行了系统分析。同源性关系较近的每一对基因都有一个基因的启动子区有高转录水平作用元件(表2,序号1),说明每一对基因中可能其中一个起主要作用;这6个编码仅含START结构域蛋白质的基因的启动子区域均有skn-1-motif和Sp1(表2,序号2、3),说明这些基因均可能与胚乳发育、光的响应有关;一些启动子元件仅存在于1对或2对基因中,而在另外2对或1对基因中不存在(表2,序号4、9、10、11、23、27、32和33),说明每一对基因具有功能的相似性,而与另一对基因的功能明显不同;大部分启动子元件只在某一对基因中的1个中存在(表2,序号5、6、7、8、12、13、14、15、16、17、18、19、20、21、22、24、25、26、28、30、31、34、35、36、37、38、39、40、41和42),说明同一对基因的2个基因功能不尽相同;还有一些启动子元件仅在6个基因中的1个基因启动子中存在(表2,序号31、35、36、37、38、39、40、41和42),说明这些基因可能执行着某些特异的功能,如At4g14500可能是某种激发子的响应基因、控制着细胞周期和叶片的发育,At5g54170可能在地上部特异表达。
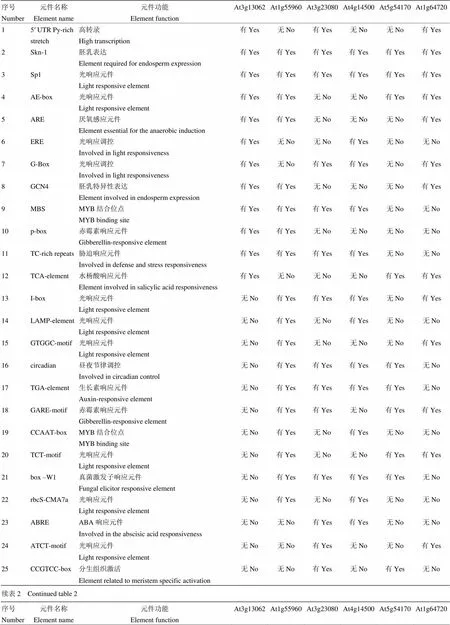
表2 拟南芥中编码仅含START结构域蛋白家族的基因启动子中作用元件分析
2.2 拟南芥中编码仅含START结构域家族基因的表达分析
实时定量PCR分析表明,在进化关系较近的每一对基因中,At3g13062、At3g23080和At1g64720相对表达量较高(图2),同这3个基因的启动子中均含有高转录水平的顺式作用元件响应一致(表2,序号1);所分析的基因均在茎中表达量最高(图2);所有基因均在幼苗期表达量较少,成苗期表达量增加,衰老后表达量下降(图3)。在生长的各个阶段, At1g64720表达量最多(图4)。
利用目的基因启动GUS的转基因拟南芥染色分析目的基因的组织定位情况,结果表明,不同基因具有明显的时空表达特性,At3g13062和At1g55960在幼苗期就有明显的表达,主要分布于根系、茎、子叶和真叶中,At3g13062的表达量较多(可能与该基因的启动子区有高转录水平的顺式作用元件有关,图5-A、5-B,表2),At3g23080和At4g14500在幼苗期表达量很少(图5-C、5-D),到成苗期表达量增加,但主要在莲座叶中表达,At3g23080的表达量多于At4g14500(图5-G、5-H,可能与At3g23080的启动子区有高转录水平的顺式作用元件有关,表2);成苗期At3g13062在根中的表达明显降低,At1g55960在根系中的表达量较多(图5-E、5-F);不同在花器官中的表达部位也有明显差异,At3g13062在萼片、柱头和花丝中表达,但在花药中不表达(图5-I、5-M、5-N),At1g55960在萼片、花瓣、柱头、花丝和花药中表达(图5-J、5-O、5-P);At3g23080和At4g14500只在萼片和花瓣中略有表达(图5-K、5-L、5-Q、5-R)。At3g13062在叶片中主要分布在表皮毛和输导组织中(图5-S),At1g55960主要分布于叶片输导组织中,表皮毛中没有表达(图5-T),At3g23080在整个叶片中均匀表达,但表皮毛中没有表达(图5-U),At4g14500在叶片中表达量很少,主要分布于输导组织(图5-V)。
2.3 含胚乳表达元件的START亚家族成员的功能分析
拟南芥中编码仅含START结构域蛋白家族基因的启动子作用元件中均含有Skn-1——胚乳表达特异性元件(表1),预示这些基因可能与胚乳的发育密切相关。为验证此功能,从美国ABRC(Arabidopsis biological Resource Center)突变体库中购买At4g14500及At3g23080基因T-DNA插入突变体,分别为Salk-014746和Salk-053261c。经Tair网站(http://www.arabidopsis.org/)分析,Salk-053261c在At4g14500的5′ UTR位置插入(图6-a),Salk-014746在At3g23080基因启动子前插入(图7-a),通过三引物法和real-time PCR验证得到其纯合突变体(图6-b、6-c和图7-b、7-c):在DNA水平上,用基因的两端引物LP和RP及通用中间引物BP同时扩增,At4g14500纯合突变体扩增出约为600 bp条带,At3g23080纯合突变体扩增出约为500 bp条带,而野生型扩增出1 000—2 000 bp条带。
通过对At4g14500突变体及野生型拟南芥种子活力及大小进行测定。野生型种子略大于At4g14500突变体,且野生型种子的千粒重为0.0145 g,At4g14500突变体14500-T1、14500-T2种子千粒重分别为0.0134和0.0136 g;野生型种子被明显染色,说明活力要高于At4g14500突变体(图8)。
At3g23080种子略小于WT,野生型种子千粒重为0.0145 g,At3g23080突变体23080-T1、23080-T2、23080-T3种子千粒重分别为0.0128、0.0130和0.0127 g(图9-a);TTC种子活力分析发现,野生型拟南芥种子被明显染色,而At3g23080突变体种子染色均较浅,说明At3g23080突变体种子活力降低(图9-b)。
通过比较野生型拟南芥和At3g23080及At4g14500突变体种子的萌发率,结果表明野生型、At4g14500的2个T-DNA插入突变体、At3g23080的3个T-DNA插入突变体种子在1/2 MS培养基上的萌发率分别为96%、43%、34%、30%、29%和27%(图10)。说明At3g23080和At4g14500表达量降低均能造成种子活力下降,直接导致萌发率的降低。
3 讨论
在拟南芥中含START结构域的蛋白家族共有35个[18]成员,其中21个被融合在同源异型结构域中,此现象表明START结构域在植物的生长发育过程中起到了重要的作用[21]。仅含START结构域成员有6个,分别为At3g13062、At1g55960、At3g23080、At4g14500、At5g54170、At1g64720编码的蛋白质,这些蛋白存在的意义及功能未知,START结构域具有磷脂酰胆碱结合位点,磷脂酰胆碱是细胞膜的主要成分,因此推测其在细胞膜稳定性方面具有重要的作用,因此,本研究利用基因启动子解析了这些基因的表达模式,有利于深入阐明单独START结构域的功能及在生物进化中的地位,对阐明START结构域调控植物生长发育的机理具有重要的理论价值。研究发现仅含START结构域的这6个成员均具有跨膜结构,而且生物信息学分析表明这个亚家族成员的4个成员(At3g13062、At1g55960、At3g23080、At4g14500)上游启动子序列包含响应抗逆防御相关的元件(TC-rich repeats)及MYB转录因子结合位点(MBS),这些元件的存在均提示这些基因可能与抗逆防御反应相关。这些预测结果与已发表的仅含START结构域的蛋白在植物抗逆过程中的报道一致,SSMP即是仅含START结构域并且参与拟南芥抗逆过程的跨膜蛋白[22-23],因此本结果对进一步分析START结构域的功能具有指导意义。
根据生物信息学分析结果,本研究还发现编码仅含START结构域蛋白家族基因的启动子作用元件中均含有Skn-1——胚乳表达特异性元件,这预示着这些基因可能与胚乳的发育密切相关,而胚乳是种子萌发中的营养提供者,因此与种子活力相关,为验证这一猜测,利用T-DNA插入突变体来验证基因缺失对种子活力的影响,结果表明启动子中含有胚乳表达特异性元件Skn-1的At4g14500和At3g23080表达量降低,确实导致了种子活力下降(图8、图9)。而且已有研究表明,种子活力涉及到蛋白[24]、糖[25]、核酸[26]、脂肪酸[27]、挥发性物质乙醛[28]、膜透性[29]、酶活性[30]、呼吸强度[31]、脂质过氧化[32]、机制修复[33]等方面的变化。因此欲明确At4g14500及At3g23080影响种子活力的原因需进一步分析其对种子中糖、脂肪酸、酶活性、呼吸强度的影响。
4 结论
拟南芥中仅含START结构域的亚家族有6个成员;含START结构域的亚家族6个成员虽然同源关系较近,但具有各自的时空表达特性,在拟南芥的不同组织和不同发育时期执行着各自的功能。基因启动子元件与基因功能有直接关系,启动子区具有胚乳特意表达Skn-1元件的At3g23080和At4g14500表达量下降均会导致种子活力和萌发率下降。
References
[1] Tsujishita Y, Hurley J H. Structure and lipid transport mechanism of a StAR-related domain., 2000, 7(5): 408-414.
[2] Roderick S L, Chan W W, Agate D S, Olsen L R, Vetting M W, Rajashankar K R, Cohen David E. Structure of human phosphatidylcholine transfer protein in complex with its ligand., 2002, 9(7): 507-511.
[3] Soccio R E, Breslow J L. StAR-related lipid transfer (START) proteins: mediators of intracellular lipid metabolism., 2003, 278(25): 22183-22186.
[4] Schrick K, Bruno M, Khosla A, Cox P N, Marlatt S A, Roque R A, Nguyen H C, He C w, Snyder M P, Singh D, Yadav G. Shared functions of plant and mammalian StAR-related lipid transfer (START) domains in modulating transcription factor activity., 2014, 12(1): 70-90.
[5] CLARK B J. The mammalian START domain protein family in lipid transport in health and disease., 2012, 212(3): 257-275.
[6] ALPY F, TOMASETTO C. Give lipids a START: the StAR-related lipid transfer(START) domain in mammals., 2005, 118(13): 2791-2801.
[7] KUDO N, KUMAGAI K, TOMISHIGE N, YAMAJI T, WAKATSUKI S, NISHIJIMA M, HANADA K, KATO R. Structural basis for specific lipid recognition by CERT responsible for nonvesicular trafficking of ceramide., 2008, 105(2): 488-493.
[8] NAKAMURA M, KATSUMATA H, ABE M, YABE N, KOMEDA Y, YAMAMOTO KT, TAKAHASHIi T. Characterization of the class IV homeodomain-leucine zipper gene family in., 2006, 141(4): 1363-1375.
[9] SZYMANSKI D B, JILK R A, POLLOCK S M, MARKS M D. Control of GL2 expression inleaves and trichomes., 1998, 125(7): 1161-1171.
[10] HIROYOSHI K, ANTON J M, PEETER S, MARK G M A, ANDY P, MAARTEN K., a homeobox gene affecting anthocyanin distribution and root development in., 1999, 11(7): 1217-1226.
[11] ABE M, KATSUMATA H, KOMEDA Y, TAKAHASHI T. Regulation of shoot epidermal cell differentiation by a pair of homeodomain proteins in., 2003, 130(4): 635-643.
[12] RYO F, YUKI K, AKIRA K, TETSU K, KAZUYA T, MAGNUS N, MIKHAIL E, NASRALLAH A, KENTARO K S, HIROSHI K, TETSUJI K. Evolution and control of imprinted FWA genes in the., 2008, 4(4): e1000048.
[13] ELHITI M, STASOLLA C. Structure and function of homodomain- leucine zipper (HD-Zip) proteins., 2009, 4(2): 86-89.
[14] EMERY J F, FLOYD S K, ALVAREZ J, ESHED Y, HAWKER N P, IZHAKI A, BAUM S F, BOWMAN J L. Radial patterning ofshoots by class III HD-ZIP and KANADI genes., 2003, 13(20): 1768-1774.
[15] MCCONNELL J R, EMERY J, ESHED Y, BAO N, BOWMAN J, BARTON M K. Role of PHABULOSA and PHAVOLUTA in determining radial patterning in shoots., 2001, 411(6838): 709-713.
[16] BAIMA S, POSSENTI M, MATTEUCCI A, WISMAN E, ALTAMURA M M, RUBERTI I, MORELLI G. TheATHB-8 HD-zip protein acts as a differentiation-promoting transcription factor of the vascular meristems., 2001, 126(2): 643-655.
[17] TALBERT P B, AALER H T, PARKS D W, COMAI L. The REVOLUTA gene is necessary for apical meristem development and for limiting cell divisions in the leaves and ste1/2MS of., 1995, 121(9): 2723-2735.
[18] SCHRICK K, NGUYEN D, KARLOWSKI W M, MAYER K F. START lipid/sterol-binding domains are amplified in plants and are predominantly associated with homeodomain transcription factors., 2004, 5(6): 50-67.
[19] KHOSLA A, PAPER J M, BOEHLER A P, BRADLEY A M, NEUMANN T R, SCHRICK K. HD-Zip proteins GL2 and HDG11 have redundant functions intrichomes, and GL2 activates a positive feedback loop via MYB23., 2014, 26(5): 2184-2200.
[20] MURCIA M, FARALDO-GOMEZ J D, MAXFIELD F R, ROUX B. Modeling the structure of the StART domains of MLN64 and StAR proteins in complex with cholesterol., 2006, 47(12): 2614-2630.
[21] PONTING C P, ARAVIND L. START: a lipid-binding domain in StAR, HD-ZIP and signalling proteins., 1999, 24(4): 130-132.
[22] 张海丽, 由诗东, 张昊, 高静, 李生辉, 张利辉, 邢继红, 王凤茹, 董金皋. SSMP的结构解析及在拟南芥抗盐过程中的作用. 中国农业科学, 2015, 48(4): 804-812.
ZHANG H L, YOU S D, ZHANG H, GAO J, LI S H, ZHANG L H, XING J H, WANG F R, DONG J G. Structure analysis of SSMP and its function in salt tolerance in.,2015, 48(4): 804-812. (in Chinese)
[23] 张昊, 由诗东, 高静, 张海丽, 李生辉, 邢继红, 王凤茹, 董金皋. 拟南芥PMRP的表达特性及功能分析. 中国农业科学, 2014, 47(15): 3094-3102.
ZHANG H, YOU S D, GAO J, ZHANG H L, LI S H, XING J H, WANG F R, DONG J G. PMRP expression characteristics and analysis of the function in.,2014, 47(15): 3094-3102. (in Chinese)
[24] KALPANA R, RAO K. Protein metabolism of seeds of pigeonpea ((L.)Millsp) cultivars during accelerated ageing., 1996, 24(3): 475-483.
[25] SUNG J M, JENG T L. Lipid peroxidation and peroxide-scavenging enzymes associated with accelerated aging of peanut seed., 1994, 91(1): 51-55.
[26] RAO K V M, KALPANA R. Carbohydrates and the ageing process in seeds of pigeonpea ((L.)Millsp.) cultivars., 1994, 22(3): 495-501.
[27] THAPLIYAL R C, CONNOR K F. Effects of accelerated ageing on viability, leachate exudation, and fatty acid content ofRoxbseeds., 1997, 25(2): 311-319.
[28] PEREZ M A, ARGÜELLO J A. Deterioration in peanut (L.) seeds under natural and accelerated aging., 1995, 23(2): 439-445.
[29] GOLOVINA E A, WOLKERS W F, HOEKSTRA F A. Behaviour of membranes and proteins during natural seed ageing., 1997, 30: 787-796.
[30] AUNG U T, MCDONALD M B. Changes in esterase activity associated with peanut (L.) seed deterioration., 1995, 23(1): 101-111.
[31] FERGUSON J M, TEKRONY D M, EGLI D B. Changes during early seed and axes deterioration., 1990, 30(1): 175-179.
[32] TRAWATHA S E, KRONY D M, HILDEBRAND D F. Relationship of soybean seed quality to fatty acid and C6-aldehydelevels during storage., 1995, 35(5): 1415-1422.
[33] SIVRITEPE H O, DOURADO A M. The effects of humidification treatments on viability and the accumulation of chromosomal aberrations in pea seeds., 1994, 22(22): 337-348.
(责任编辑 李莉,岳梅)
analysis on gene feature and function of the subfamily members Containing START domain only in
YAN QingDi, ZHAO YaLin, Zhang Hao, GAO Jing, GAO MengZhu, WANG Qian,WANG Rui, WANG FengRu, DONG JinGao
(College of life science, Agricultural University of Hebei/Key Laboratory ofHebeiProvincefor plant physiology and molecular plant pathology, Baoding 071001, Hebei)
【Objective】 The objective of this study is to analyze the promoter elements and expression feature of the genes coding the subfamily containing the START domain (the lipid/sterol-binding StAR-related lipid transfer protein domains) only in, expound the relationship between the cis-acting element and the gene function, and the results of study will provide an important theoretical basis for clarifying the biological function of the genes, analyzing the plant growth and development mechanism and promoting or suppressing plant growth better. 【Method】 Using bioinformatics method to analyze the genetic relationship of 6 members and all the elements contained in their respective promoter regions. The function of the promoter elements of subfamily were analyzed and classified, and the expression features and the regulation factors of these subfamily members were predicted. Real-time PCR method was employed to determine the expression characteristics of subfamily members, clarify the relationship between the test results and the conclusions of promoter analysis, thus providing scientific research ideas and direction for analysis of the function and mechanism of genes. The relationship between the gene promoter element and function was confirmed after analysis of the gene mutation type surface. 【Result】 There are 6 members belong to the subfamily containing START domain only in, divided into three branches, every branch has a pair of genes, each pair of genes has a promoter containing high transcription element 5′ UTR Py-rich stretch. The promoter regions of the 6 subfamily members contain multiple common response elements, such as light, hormone and stress, and also have specific response elements for their own. The real-time PCR results of the genes expression level showed that the expression level of At3g13062 was significantly higher than the same branch gene At1g55960, At3g23080 was significantly higher than the same branch gene At4g14500, At1g64720 was significantly higher than the same branch gene At5g54170. All the subfamily members have higher expression levels at seedling stage, but less expression levels at ageing stage, and have its own specific expression of space and time characteristics. In the gene promoter analysis of the family containing START domain structure only, it was found that each gene promoter has a cis-acting element Skn-1 motif which was specially expressed in endosperm, implied that the family genes may be involved in regulation of endosperm development. Seeds analysis of At3g23080 and At4g14500 T-DNA insertion mutant showed that At3g23080 and At4g14500 expression decreased and caused a decline in the seed vigor. 【Conclusion】 There are 6 members in the subfamily containing START domain only. Six members of the family may be play an important role in the response process to light, hormones and stress. the 6 family members have a close kinship, but their expression characteristics of time and space are different and carry out their respective functions in different development periods of. There is a direct relationship between gene promoter elements and the gene function, and the promoter region with endosperm expressed specially element Skn1 motif in At3g23080 and At4g14500, so the decreased expression of At3g23080 or At4g14500 caused a decline in seed vigor.
; START domain; promoter elements;expression feature; gene function
2017-03-03;接受日期:2017-04-18
河北省自然科学基金(C2017204060)
闫青地,E-mail:1073886780@qq.com;赵亚林,E-mail:1520469399@qq.com。闫青地和赵亚林为同等贡献作者。通信作者王凤茹,E-mail:wfr15931945160@126.com。通信作者董金皋,E-mail:dongjingao@126.com

