大雁、斑头雁和天鹅源沙门菌的耐药性分析
葛润洲,罗 薇,杨怀珍,孔雪英
(西南民族大学生命科学与技术学院,四川成都 610041)
大雁、斑头雁和天鹅源沙门菌的耐药性分析
葛润洲,罗 薇*,杨怀珍,孔雪英
(西南民族大学生命科学与技术学院,四川成都 610041)
研究大雁、斑头雁和天鹅源沙门菌的耐药性及其耐药基因的携带情况。采用K-B纸片法对33株大雁、斑头雁和天鹅源沙门菌进行20种抗菌药物敏感性测定,应用PCR对12种耐药基因的携带情况进行检测。结果表明,33株沙门菌分离株对阿莫西林、萘啶酸耐药性最高,分别为93.94%、87.88%,对氨苄西林(54.55%)、四环素(48.49%)、多西环素(48.49%)、卡那霉素(60.61%)、庆大霉素(48.49%)、链霉素(66.67%)耐药性比较严重,对氧氟沙星、培氟沙星、诺氟沙星敏感。共检测到9种耐药基因,其中blaCMY-2阳性率为87.88%,qnr、aac(6′)-Ib-cr阳性率分别为17.24%、 86.21%,tetA、tetB阳性率分别为45%、20%,aph(3)Ⅱ、aac(3)Ⅱ、aadA1阳性率分别为100%、10%、20%, sul2阳性率为100%,未检出blaTEM-1、tetC、tetG基因。说明大雁、斑头雁和天鹅源沙门菌严重耐药,且携带多种耐药基因。
沙门菌;耐药性;耐药基因
沙门菌(Salmonella)是一种常见的病原菌,被认为是全球第1种最常见的引起人类疾病的食源性细菌,不仅可直接感染人类,还可以通过动物性食品,如鸡肉、牛肉、猪肉、鸡蛋、牛奶、海鲜等食源性感染人类,从而引起疾病[1-2]。沙门菌病是最常见的细菌性食源性疾病之一, 是影响全球食品安全和公共卫生问题[3]。据估计,2000年沙门菌造成约2 160万人患病,导致约20万人死亡[4];在美国,每年大约有120万人因沙门菌感染患病,约400人死亡,造成大约365亿美元的经济损失[5-6];中国约有22.2%食源性疾病是由沙门菌引起的[7]。在人类临床治疗、畜牧业生产中, 抗菌药物仍然是治疗各种病原菌的首选药物,但在过去的几十年中,由于抗菌药物的广泛使用及滥用,导致沙门菌耐药率逐年上升, 耐药谱型迅速增宽, 给食品安全和公共卫生安全造成了极大危害[8-10]。本研究对成都邛崃、双流等地大雁、天鹅人工驯养基地分离的33株大雁、斑头雁和天鹅源沙门菌进行了药物敏感性试验,并对相关耐药基因进行检测,了解大雁、斑头雁和天鹅源沙门菌耐药情况,为临床治疗沙门菌病的合理用药及沙门菌多重耐药机制的进一步研究提供依据。
1 材料与方法
1.1 材料
1.1.1 菌株 质控菌株大肠埃希菌ATCC25922,西南民族大学动物医学实验室提供;试验用33株沙门菌由采自成都邛崃、双流等地大雁、天鹅人工驯养基地的病料,经西南民族大学动物医学实验室分离并保存。
1.1.2 主要试剂和培养基 MH琼脂、MH肉汤,北京陆桥技术有限责任公司产品;2×TaqPCR Master-Mix、Plus DNA Marker DL 1 200,上海生工生物工程技术服务有限公司产品。
1.1.3 药敏纸片 20种抗菌药物包括氨苄西林(ampicillin)、阿莫西林(amoxicillin)、头孢吡肟(cefepime)、头孢噻肟(cefotaxime )、头孢曲松(ceftriaxone)、头孢拉定(cephradine)、环丙沙星(ciprofloxacin)、恩诺沙星(enrofloxacin)、氧氟沙星(ofloxacin)、培氟沙星(pefloxacin)、诺氟沙星(norfloxacin)、萘啶酸(nalidixic acid)、四环素(tetracycline)、多西环素(doxycycline)、丁胺卡那(amikacin)、庆大霉素 (gentamicin)、卡那霉素(kanamycin)、链霉素 (streptomycin)、甲氧苄氨嘧啶(trimethoprim)、氟苯尼考(florfenicol),杭州微生物股份有限公司产品。
1.2 方法
1.2.1 药敏试验 根据世界卫生组织(WHO)推荐的K-B纸片扩散法进行药敏试验。以大肠埃希菌ATCC25922质控菌株,测定分离株的药物敏感性。
1.2.2 细菌DNA的提取 参照文献[11]的方法,采用微波加热法提取细菌DNA。具体方法如下:取1.5 mL菌液12 000 r/min离心2 min,收集菌体,加入灭菌生理盐水洗涤菌体3次后,再加入TE缓冲液洗涤菌体2次,离心弃上清,加入500 μL TE缓冲溶液重悬菌体,取100 μL悬液于PCR管中,微波分别加热40、 50、60、70 s,离心取上清, 置-20℃保存备用。
1.2.3 耐药基因PCR引物设计、扩增及耐药基因的序列分析 参照GenBank中登录的耐药基因序列及相关文献[12-14]设计了11对引物(表1),引物由上海生工生物工程技术服务有限公司合成。分别用于扩增β-内酰胺类耐药基因blaCMY-2、blaTEM-1,四环素类耐药基因tetA、tetB、tetC、tetG,磺胺类耐药基因sul2,氨基糖苷类耐药基因aph(3)Ⅱ、aac(3)Ⅱ、aadA1,氟喹诺酮类耐药基因qnr。
PCR反应体系:2×TaqPCR Master-Mix 10 μL,上、下游引物(10 pmol/L)各1 μL,模板2 μL,灭菌双蒸水加至20 μL。PCR反应参数:95 ℃ 10 min;95 ℃ 1 min,55 ℃~60 ℃ 30 s,72℃ 45 s,35个循环;72 ℃ 10 min。PCR产物使用15 g/L 琼脂糖凝胶电泳,电泳后回收PCR产物,测序后在GenBank数据库进行序列分析。
2 结果
2.1 药敏试验结果
33株沙门菌分离株对20种抗菌药物的敏感性测定结果见表2。 33株沙门菌对阿莫西林、萘啶酸耐药性最高,分别为93.94%、87.88%,对氨苄西林的耐药率为54.55%、四环素为48.49%、多西环素为48.49%、卡那霉素为60.61%、庆大霉素为48.49%、链霉素为66.67%,耐药性比较严重,仅对氧氟沙星、培氟沙星、诺氟沙星敏感。三重以上耐药的菌株高达93.94%,七重以上耐药的菌株为27.27%,耐药最多的菌株可耐受16种抗菌药物。
2.2 抗菌药物耐药基因检测结果与序列分析
33株沙门菌耐药基因检测结果见表3,部分耐药基因检测结果图见图1~图9。其中,β-内酰胺类耐药菌株有29株检出了blaCMY-2基因,检出率达87.88%,未检出检出blaTEM-1;20株四环素类耐药菌株中9株检出tetA基因、4株检出tetB基因,检出率分别为45%和20%,未检出tetC、tetG基因;6株磺胺类耐药菌株中均检测到sul2基因;30株氨基糖胺类耐药菌株中aph(3)Ⅱ 基因均有检出, 3株检出aac(3)Ⅱ基因、6株检出aadA1基因,检出率分别为10%、20%,29株喹诺酮类耐药菌株中仅5株检出qnr基因,检出率为17.24%,25株检测出aac(6′)-Ib-cr基因,检出率为86.21%。测序结果显示各耐药基因序列与预期序列高度相似。

表1 耐药基因引物序列
3 讨论
抗菌药物用于治疗细菌感染为人类及动物带来了诸多好处,但大量的抗菌药物被同时用于生长促进剂,以及不适当地滥用于动物养殖中细菌感染的预防和治疗,使得抗菌药物的耐药性日益增加,甚至产生了多重耐药[15-19]。不断增加的抗菌药物耐药性已成为全球范围内的一个重要问题。Rahmani M等[20]对伊朗2007年—2011年分离的沙门菌进行药敏试验,结果显示沙门菌分离株都对两种抗菌药物耐药,且对萘啶酸和环丙沙星的耐药率达94%;Li B等[21]对美国东南部水中分离的沙门菌进行药敏试验,结果所有菌株对庆大霉素、环丙沙星、萘啶酸、甲氧苄氨嘧啶耐药;Ke B等[22]对中国广东2007年—2012年分离的沙门菌研究结果表明,沙门菌对复方新诺明、四环素、萘啶酸、氨苄青霉素和链霉素都有抗性。
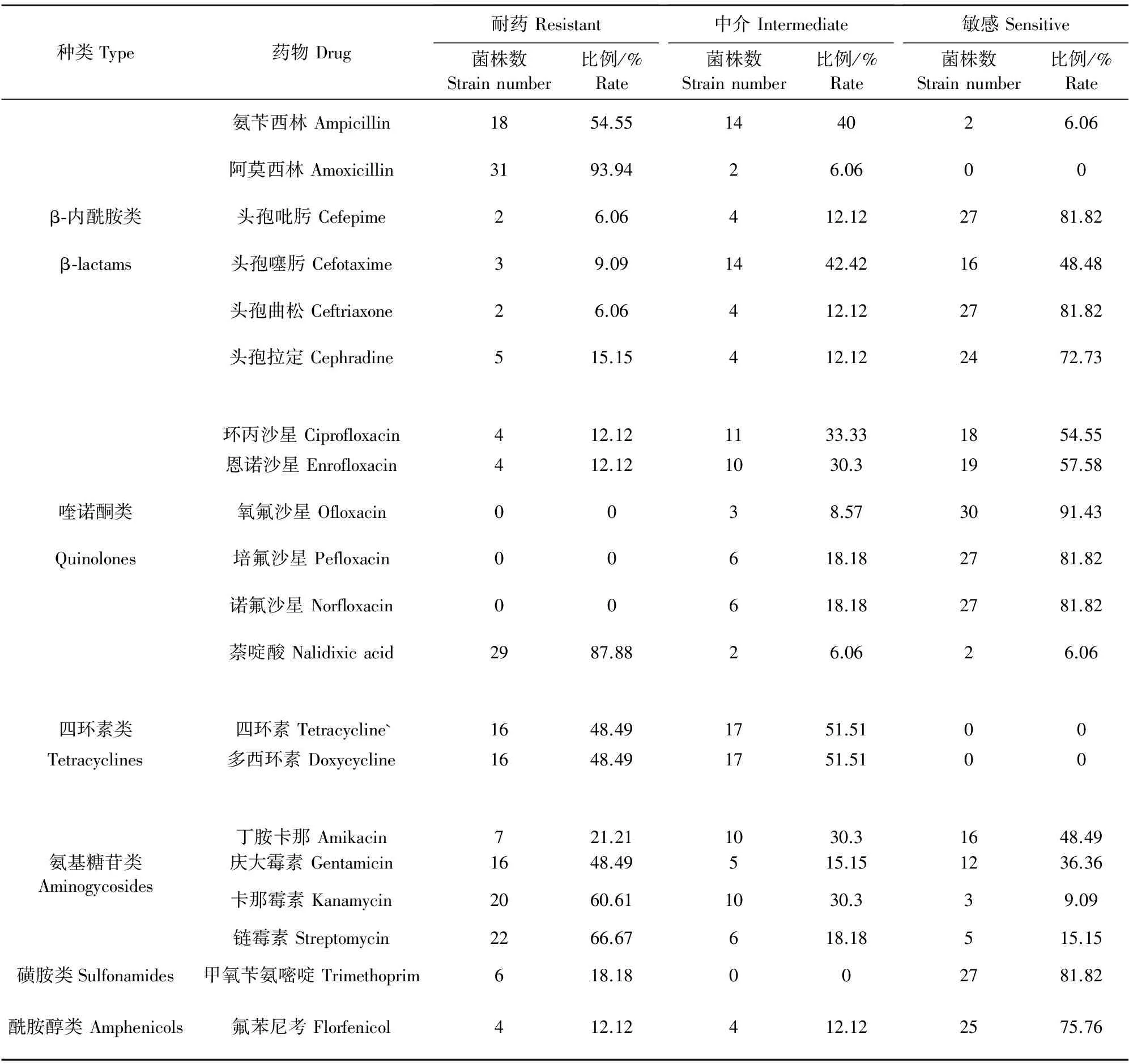
表2 33株沙门菌对抗菌药物的耐药率和敏感率
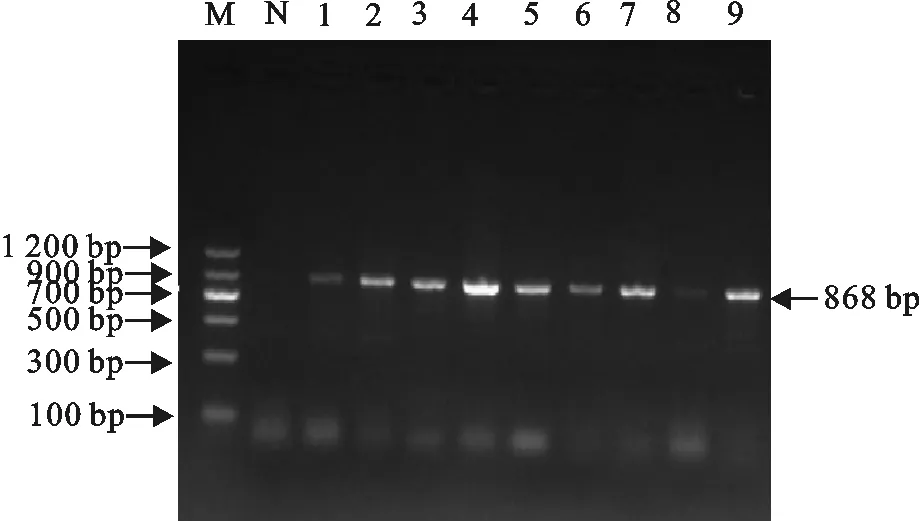
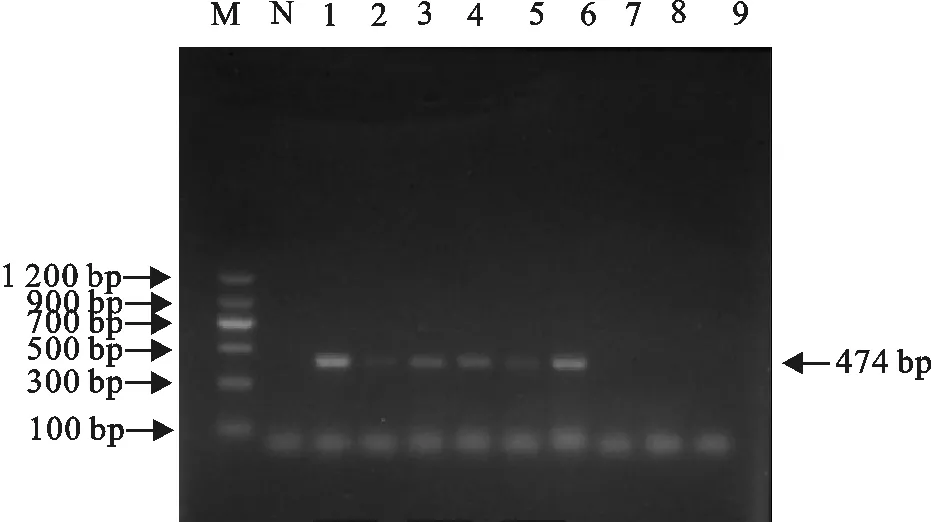
M.DNA 标准DL 1 200;N.阴性对照;1~9.分离株PCR产物
M.DNA Marker DL 1 200;N.Negative control;1-9.PCR products of isolates
图1部分菌株blaCMY-2基因PCR扩增结果
Fig.1 PCR amplification results of blaCMY-2 gene
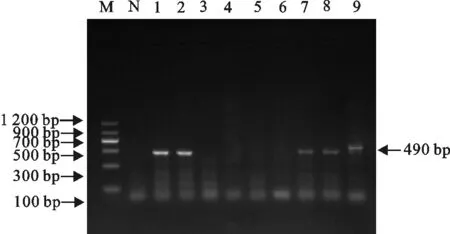
M.DNA 标准DL 1 200;N.阴性对照;1~9.分离株PCR产物
M.DNA Marker DL 1 200;N.Negative control;1-9.PCR products of isolates
图2部分菌株qnr基因PCR扩增结果
Fig.2 PCR amplification result of qnr gene
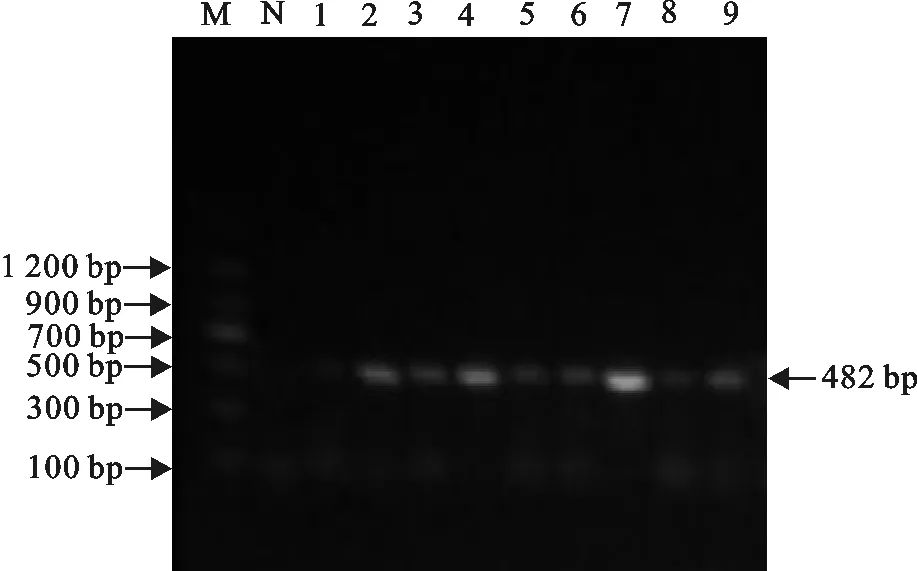
M.DNA 标准DL 1 200;N.阴性对照;1~9.分离株PCR产物
M.DNA Marker DL 1 200;N.Negative control;1-9.PCR products of isolates
图3部分菌株aac(6′)-Ib-cr基因PCR扩增结果
Fig.3 PCR amplification result of aac(6′)-Ib-cr gene
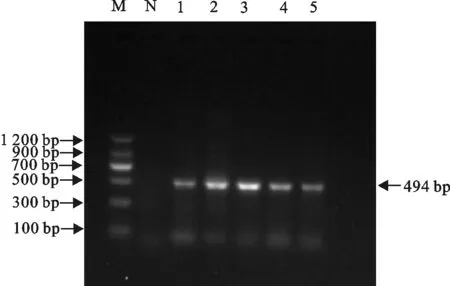
M.DNA 标准DL 1 200;N.阴性对照;1~5.分离株PCR产物
M.DNA Marker DL 1 200;N.Negative control;1-5.PCR products of isolates
图4部分菌株tetA基因PCR扩增结果
Fig.4 PCR amplification result of tetA gene
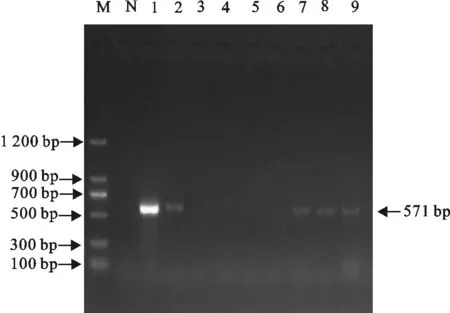
M.DNA 标准DL 1 200;N.阴性对照;1~9.分离株PCR产物
M.DNA Marker DL 1 200;N.Negative control;1-9.PCR products of isolates
图5部分菌株tetB基因PCR扩增结果
Fig.5 PCR amplification result of tetB gene
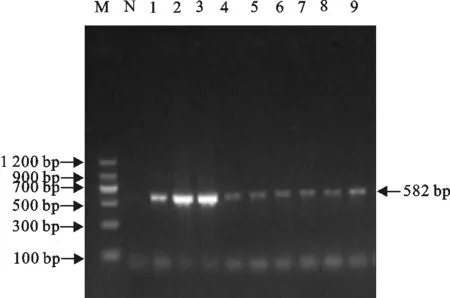
M.DNA 标准DL 1 200;N.阴性对照;1~9.分离株PCR产物
M.DNA Marker DL 1 200;N.Negative control;1-9.PCR products of isolates
图6部分菌株aph(3)Ⅱ基因PCR扩增结果
Fig.6 PCR amplification result of aph(3)Ⅱ gene
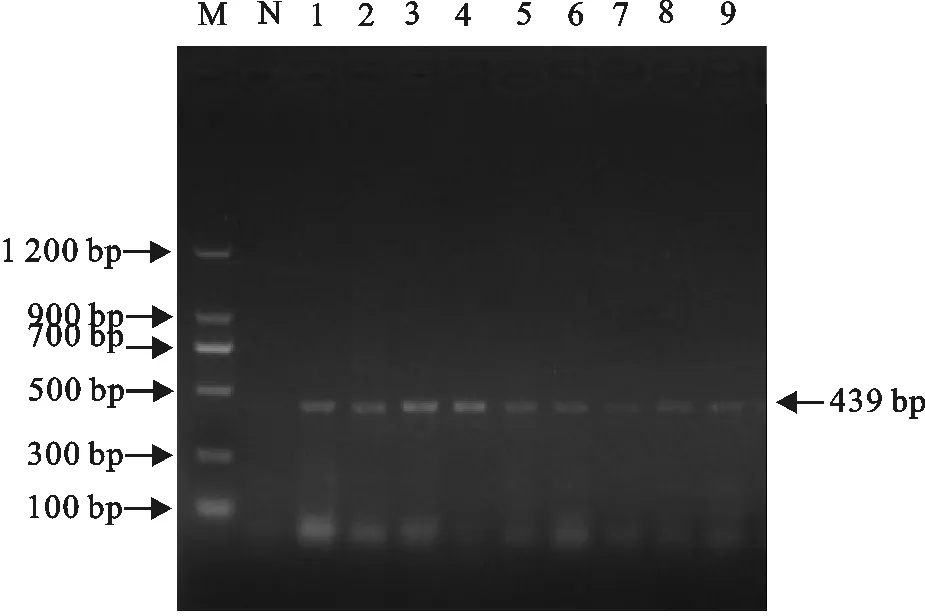
M.DNA 标准DL 1 200;N.阴性对照;1~9.分离株PCR产物
M.DNA Marker DL 1 200;N.Negative control;1-9.PCR products of isolates
图7部分菌株aac(3)Ⅱ基因PCR扩增结果
Fig.7 PCR amplification result of aac(3)Ⅱ gene

M.DNA 标准DL 1 200;N.阴性对照;1~9.分离株PCR产物
M.DNA Marker DL 1 200;N.Negative control;1-9.PCR products of isolates
图8部分菌株aadA1基因PCR扩增结果
Fig.8 PCR amplification result of aadA1 gene
M.DNA 标准DL 1 200;N.阴性对照;1~9.分离株PCR产物
M.DNA Marker DL 1 200;N.Negative control;1-9.PCR products of isolates
图9部分菌株sul2基因PCR扩增结果
Fig.9 PCR amplification result of sul2 gene
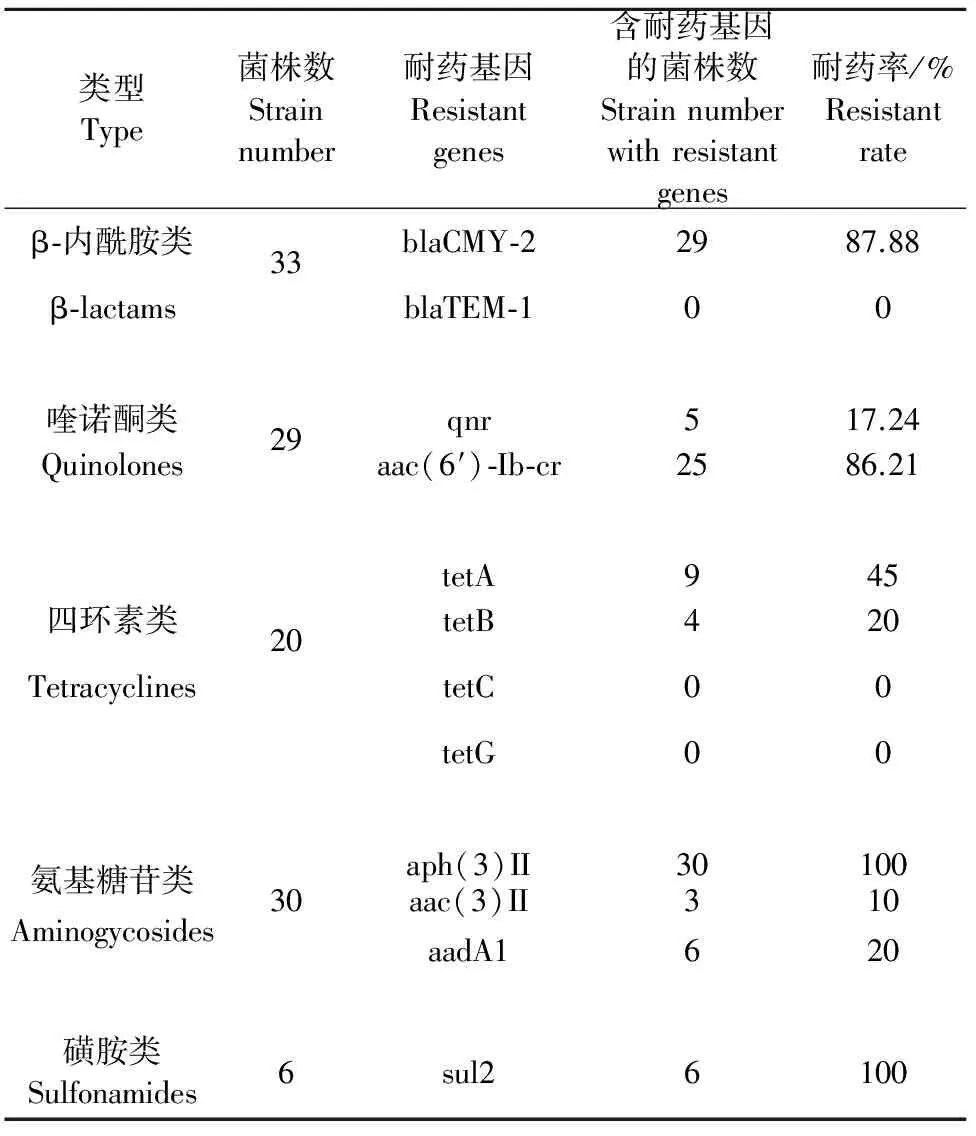
表3 含耐药基因的菌株数与耐药菌株数的比较
本研究对33株大雁、斑头雁和天鹅源沙门菌进行药敏试验,结果显示,沙门菌耐药严重。其中对阿莫西林和萘啶酸的耐药性最高,与陆彦等[23]、赖海梅等[24]研究结果基本一致;对氨苄西林、四环素、多西环素、卡那霉素、庆大霉素、链霉素的耐药率为在45%~70%,临床上都应该谨慎使用。耐药率在20%及以下的头孢吡肟、头孢噻肟、头孢曲松、环丙沙星、恩诺沙星、丁胺卡那、甲氧苄氨嘧啶、氟苯尼考以及氧氟沙星、培氟沙星、诺氟沙星无耐药株产生的这些药物在临床上可以继续使用。所有菌株均呈现多重耐药,三重以上耐药的菌株高达93.94%,七重以上耐药的菌株为27.27%,耐药最多的菌株可耐受16种抗菌药物。
本研究对抗菌药物表型耐药的沙门菌携带相应耐药基因,如β-内酰胺类耐药基因blaCMY-2、blaTEM-1,四环素类耐药基因tetA、tetB、tetC、tetG,磺胺类耐药基因sul2,氨基糖苷类耐药基因aph(3)Ⅱ、aac(3)Ⅱ、aadA1,氟喹诺酮类耐药基因qnr进行了检测,共检测到9种耐药基因。结果表明大雁、斑头雁和天鹅源沙门菌携带多种耐药基因,且总体上耐药基因与耐药表型的检测结果基本一致, blaCMY-2、aac(6′)-Ib-cr、aph(3)Ⅱ、sul2检出率均很高,与药敏试验的结果的结果基本一致;但药敏试验中耐四环素的菌株其四环素类耐药基因检出率较低,这可能与基因的表达程度、不同药物的抗菌活性、对酶的稳定性差异以及基因型的流行有关,还有待进一步研究。
抗菌药物耐药性威胁全球安全,需要迫切解决,本研究对大雁、斑头雁和天鹅源沙门菌的药敏性特征及相关耐药基因的研究,可以更好地了解沙门菌的耐药机理,对有效控制日趋严重的沙门菌耐药性问题提供了依据。
[1] Chiu L H,Chiu C H,Horn Y M,et al.Characterization of 13 multi-drug resistantSalmonellaserovars from different broiler chickens associated with those of human isolates[J].BMC Microbiol,2010,10(10):1-10.
[2] Ceyssens P J,Mattheus W,Vanhoof R,et al.Trends in serotype distribution and antimicrobial susceptibility inSalmonellaentericaisolates from humans in Belgium,2009 to 2013[J].Antimicrob Agents Chemother,2015,59(1):544-552.
[3] Scallan E,Hoekstra R M,Angulo F J,et al.Foodborne illness acquired in the United States-major pathogens[J].Emerg Infect Dis,2011,17(7):1339-1340.
[4] Crump J A,Luby S P,Mintz E D.The global burden of typhoid fever[J].Bull WHO,2004,82(5):346-353.
[5] Scharff R L.Economic burden from health losses due to foodborne illness in the United States[J].J Food Protect,2012,75(1):123-131.
[6] Hoffmann M,Luo Y,Lafon P C,et al.Genome sequences ofSalmonellaentericaserovarheidelbergisolates isolated in the United States from a multistate outbreak of humanSalmonellainfections[J].Genome Announc,2013,1(1):e00004-12.doi:10.1128/genomeA.00004-12.
[7] Chiou C S,Lauderdale T L,Phung D C,et al.Antimicrobial resistance inSalmonellaentericaserovartyphiisolates from Bangladesh,Indonesia,Taiwan,and Vietnam[J].Antimicrob Agents Chemother,2014,58(11):6501-6507.
[8] Afema J A,Byarugaba D K,Shah D H,et al.Potential sources and transmission ofSalmonellaand antimicrobial resistance in Kampala,Uganda[J].PLoS One,2016, 11(3):e0152130.doi:10.1371/journal.pone.0152130.
[9] Solomon S L,Oliver K B.Antibiotic resistance threats in the United States:stepping back from the brink[J].Am Fam Physician,2014,89(12):938-941.
[10] Noda T,Murakami K,Etoh Y,et al.Increase in resistance to extended-spectrum cephalosporins inSalmonellaisolated from retail chicken products in Japan[J].PLoS One,2015,10(7):e0133094.doi:10.1371/journal.pone.0133094.
[11] 王 琼,唐俊妮,汤 承,等. 一种采用微波炉加热快速提取细菌DNA用于PCR扩增的方法[J].西南民族大学学报:自然科学版,2015,41(2):150-155.
[12] De V D,Monno R,Nuccio F,et al.Diffusion and persistence of multidrug resistantSalmonellatyphimuriumstrains phage type DT120 in southern Italy[J].Biomed Res Int,2015,265042.doi:10.1155/2015/265042.
[13] Camarda A,Pupillo A,Pugliese N,et al.Phenotypic and genetic traits ofSalmonellaenterica,subsp.serovartyphimuriumstrains causing salmonellosis foci in rabbit farms from Southern Italy in 1999-2003[J].Res Vet Sci,2013,94(3):394-398.
[14] Folster J P,Tolar B,Pecic G,et al.Characterization of bla(CMY) plasmids and their possible role in source attribution ofSalmonellaentericaserotypetyphimuriuminfections[J].Foodb Pathog Dis,2014,11(4):301-306.
[15] Kariuki S,Gordon M A,Feasey N,et al.Antimicrobial resistance and management of invasiveSalmonella,disease[J].Vaccine,2015,33(1):243-244.
[16] Glenn L M,Lindsey R L,Folster J P,et al.Antimicrobial resistance genes in multidrug-resistantSalmonellaentericaisolated from animals,retail meats,and humans in the United States and Canada[J].Microbial Drug Resist,2013,19(3):175-184.
[17] Pereira R V,Siler J D,Cummings K J,et al.Effect of heifer-raising practices onE.coliantimicrobial resistance andSalmonellaprevalence in heifer raisers[J].Epidemiol Infect,2015,143(15):3256-3265.
[18] Lunguya O,Lejon V,Phoba M F,et al.Antimicrobial resistance in invasive non-typhoid,Salmonella,from the Democratic Republic of the Congo:Emergence of decreased fluoroquinolone susceptibility and extended-spectrum beta lactamases[J].PLoS Negl Trop Dis,2013,7(3):759-767.
[19] Claire V,Sigrid V B,Eva V M,et al.Antimicrobial resistance in the food chain:a review[J].Int J Environ Res Pub Health,2013,10(7):2643-2669.
[20] Rahmani M,Peighambari S M,Svendsen C A,et al.Molecular clonality and antimicrobial resistance inSalmonellaentericaserovarsenteritidisandinfantisfrom broilers in three Northern regions of Iran[J].Bmc Vet Res,2013,9(14):1-9.
[21] Li B,Vellidis G,Liu H,et al.Diversity and antimicrobial resistance ofSalmonellaentericaisolates from surface water in Southeastern United States[J]. Ap Environ Microbiol,2014,80(20):6355-6365.
[22] Ke B,Sun J,He D,et al.Serovar distribution, antimicrobial resistance profiles,and PFGE typing ofSalmonellaenterica,strains isolated from 2007-2012 in Guangdong,China[J].BMC Infect Dis,2014,14(1):1-10.
[23] 陆 彦,吕 安,赵红玉,等.鸡源肠炎沙门氏菌对抗菌药物的耐药性分析[J].中国人兽共患病学报,2014,30(1):17-22.
[24] 赖海梅,邹立扣,刘书亮,等.肉鸡屠宰生产链中沙门氏菌耐药基因检测与耐药相关性分析[J].食品工业科技,2015,36(7):187-191.
AnalysisofAntimicrobialResistanceamongSalmonellaIsolatedfromGeese,Bar-headedGeeseandSwans
GE Run-zhou,LUO Wei,YANG Huai-zhen,KONG Xue-ying
(CollegeofLifeScienceandTechnology,SouthwestUniversityforNationalities,Chengdu,Sichuan,610041,China)
The objective of this study was to investigate the antimicrobial resistance and the resistance genes in 33 pathogenicSalmonellastrains isolated from geese,bar-headed geese and swans with the Kirby-Bauer method to detect antimicrobial resistance and PCR detection for 12 antimicrobial resistant genes.The results indicated that the resistant rate to amoxicillin was 93.94%,nalidixic acid 87.88%,followed by streptomycin 66.67%,kanamycin 60.61%,ampicillin 54.55%,tetracycline 48.49%,doxycycline 48.49%,gentamicin 48.49%,and with no resistance to ofloxacin,pefloxacinand norfloxacin.A total of 12 kinds of resistant genes were amplified with the percentage of blaCMY-2 87.88%,qnr 17.24%, aac (6′) -Ib-cr 86.21%,tetA 45%,tetB 20%,aph (3)Ⅱ 100%,aac (3) Ⅱ 10%,aadA120%,sul2 100%,and tetC,tetG and blaTEM-1 were not detected.These results showed that theSalmonellaisolated from geese,bar-headed geese and swans were seriously resistant to antimicrobials.
Salmonella;antimicrobial resistance;resistance genen
2016-08-20
四川省科技厅应用基础研究项目(2013JY0045)
葛润洲(1992-),男,安徽巢湖人,硕士研究生,主要从事动物传染病的诊断与防控研究。*
S852.612
:A
:1007-5038(2017)09-0049-06

