不同年龄巴山木竹种群克隆结构
余 鸽,龙凤来,刘建军,马青青,康永祥,黄 建,曹 庆
1 西北农林科技大学林学院,杨凌 7121002 杨凌职业技术学院,杨凌 7121003 西北农林科技大学风景园林艺术学院,杨凌 7121004 陕西佛坪国家级自然保护区管理局,佛坪 723400
不同年龄巴山木竹种群克隆结构
余 鸽1,龙凤来2,刘建军3,*,马青青1,康永祥1,黄 建1,曹 庆4
1 西北农林科技大学林学院,杨凌 7121002 杨凌职业技术学院,杨凌 7121003 西北农林科技大学风景园林艺术学院,杨凌 7121004 陕西佛坪国家级自然保护区管理局,佛坪 723400
很多竹类植物是典型的克隆植物,也是大熊猫的食物。研究典型竹子种群克隆结构的形成和发展对竹林的生产和抚育具有理论和实践意义,可为预测该竹林群落的演替趋势和大熊猫保护提供科学依据。利用SSR标记研究不同年龄A(7龄)、B(>30龄)和C(>60龄)巴山木竹种群的克隆结构和多样性,探讨小尺度范围内不同年龄巴山木竹种群的克隆结构及斑块的建立和发展。8对SSR引物共扩增出了118个位点,3个种群样地的256个样本共检测到了49个克隆(基因型),A、B和C种群分别检测出31、10个和8个克隆。随着种群年龄的增长,巴山木竹克隆面积增加,克隆数量减少;A和B样地各克隆分布格局为团块状,而C样地克隆既有团块状又有离散状。这一结果显示出在幼苗定居的初期,基株可能以短距离的克隆延伸为主从而呈现出团块状;而随着年龄的增长,克隆面积不断扩大,当复轴混生型的巴山木竹克隆受到强大的压迫时,基株可能会进行较多的单轴和长距离克隆延伸,呈现出离散状。Mantel 检测和空间自相关分析都支持3个样地在小尺度范围内存在明显的克隆空间遗传结构。3个样地在10 m等级下显著的正相关空间遗传结构距离为3.1、28、48 m,X-轴截距为9.051、30.698和50.536,空间自相关系数的范围分别为0.1—0.167、0.008—0.703和0.006—0.735。由此可推断,随着年龄的增长,巴山木竹克隆斑块的规模在不断地扩大,同一克隆的分株数量增加,在均匀取样情况下,正相关空间遗传结构距离范围内取到具有相同基因型的可能性越大。A、B和C 3样地的基因型比率(G/N)为1、0.14和0.055, Simpson多样性指数(D) 分别为1、0.876和0.744。这说明巴山木竹幼苗期基因型比例远远高于成年的竹林,随着年龄的增长巴山木竹克隆多样性虽有所降低,但由于有性繁殖的作用仍然保持了较高的多样性。聚类和主坐标分析均表明总体上各样地的克隆被聚为一类,但不同样地少数克隆的基因型有重叠和聚集,可推断出不同巴山木竹种群之间可能存在着基因流动和近似的克隆起源。
巴山木竹;克隆结构;克隆多样性
植物克隆结构及斑块的建立和发展与能量分配、生态对策和遗传多样性等生态学热点问题密切相关[1]。克隆植物种群的克隆数量、大小、空间格局及其遗传变异的测定[2-3]是研究克隆植物种群机制和进化的基础。研究克隆植物种群克隆结构和遗传多样性对了解克隆植物种群的形成、维持和衰退有重要意义,同时有助于了解克隆植物的定居、侵殖和演替机理[4]。
植物种群克隆结构和遗传多样性受生物因子和非生物因子共同作用的影响[5- 7],其中克隆植物的种苗补充方式[8- 11]、微生境的异质性[11-12]以及生长策略[4]这几种机理被提出作为影响植物克隆结构和多样性的主要因素。在种群的建立、发展、维持以及衰退的不同阶段,植物种群克隆结构和遗传多样性将会随着各种作用因子的变化而呈现为动态性。然而,由于许多克隆植物的生活史较长,如竹类植物可长达数十年甚至百年[13-14],使得人们很难对同一种群进行连续的长期监测和研究,而通常将种群克隆结构和遗传多样性的研究看成是某一时间点的静态分析[15]。近年来,为研究克隆结构和多样性的时间动态,一些学者通过模型模拟并评估不同时间段的植物特性和各环境条件对克隆植物结构发展的影响[16-17];刘庆等采用“倒逐龄级累加法”研究斑苦竹无性系种群克隆生长格局动态,发现复轴型的斑苦竹克隆种群随时间进程表现为聚集程度逐渐降低的集群分布格局[18]; Lian等用 SSR 标记调查Salixreinii的种群克隆生长动态,发现克隆的面积随着该种群分布斑块的面积增加而扩大[19];Ma等使用AFLP标记证实了两个不同年龄段(小于30岁龄级和大于70岁龄级)的Bashaniafangiana种群都是多克隆的,且克隆多样性水平都较高,说明了该植物的有性繁殖在克隆多样性中起到的重要作用[10]。这些研究方式为研究克隆结构和遗传多样性的动态发展提供了新思路。
巴山木竹为典型的克隆植物,大约每隔70—75年甚至更长时间开一次花[20],开花后,幼苗开始更新;地下根茎为复轴混生型,即通过合轴丛生和单轴散生的生长方式来共同完成新一轮的克隆延伸[21]。巴山木竹是秦岭大熊猫冬春季食物的主要来源[22],主要分布于秦岭南坡、大巴山以及米仓山地区的中山地带。在海拔1600—2000 m处生长良好,常形成大面积纯林,对生态系统的稳定、水土保持及其动物多样性起着至关重要的作用[23]。已有研究表明部分保护区内巴山木竹林老龄化,随时有开花的危险[24],研究不同年龄巴山木竹种群的克隆结构和多样性,有助于理解巴山木竹种群建立、发展和衰退不同阶段的克隆数量、大小、空间格局及其遗传变异情况, 从而为巴山木竹林的复壮更新、抚育实践以及保护寻找出路。目前对于巴山木竹的研究主要集中在生物学特性、分布、生长发育规律、生物量以及无性系种群生态等方面的研究[25],尚未见从遗传角度研究其克隆结构和多样性的报道。在众多分子标记中,SSR 分子标记具有操作简单及共显性变异等优点[26],被运用于多种植物克隆多样性和克隆结构研究中[3,19,27]。与其它分子标记不同之处在于SSR 分子标记需要开发研究对象的专用引物,在前期的研究工作中,作者已经成功开发出了巴山木竹的SSR引物[28]。因此本研究旨在利用SSR分子标记探讨不同年龄巴山木竹种群的克隆结构和多样性及其形成原因,以期为预测竹林群落演替趋势和巴山木竹的竹林抚育实践以及大熊猫保护提供科学依据。
1 材料与方法
1.1 样地设置与采样方法
为研究不同年龄巴山木竹种群的克隆结构,在考虑林分、海拔、坡度、坡向和土壤含水量等生态环境条件较一致的情况下,在2014年共设置A、B和 C 3块不同种群年龄的样地。3块样地大小根据各种群实际大小和生态环境条件而定。A和B 两块样地设置在镇巴县,该县巴山木竹资源丰富,曾在1978—1983年有大面积竹林开花[29]。A样地设置在兴隆镇郑家山一位农户旁边的山上,这片竹林于2007年大面积开花后重新形成稠密的巴山木竹幼苗林,样地大小为20 m×30 m,地理位置坐标为108°01′14.40″E、32°30′31.80″N,海拔1603 m。B样地设置在星子山朱家坪,该片竹林1978—1983年大面积开花后经过幼苗更新形成稠密的巴山木竹成年纯林,竹龄超过30a,样地大小60 m×90 m,地理位置为 107°59′51.04″E、32°31′17.96″N,海拔1610 m左右。C样地设置在佛坪国家级自然保护区,样地大小100 m×120 m, 经过和保护站的工作人员了解,该竹林年龄大于60a,海拔1621 m,到目前为止仍没有开花迹象,此处巴山木竹生长良好形成稠密的纯林。
3块样地均采取均匀(网格法)取样策略,为了得到清晰的克隆结构,对竹林年龄较大的样地C 进行了初步取样间隔检测。在C样地选取20 m×20 m的小样方进行初步取样检测,取样间隔为0—10 m,经过实验最终确定3个样地选择一致的取样策略,即在10 m×10 m为单位的网格交叉点上取样。由于A样地为实生苗,在20 m×3 0m样地中央设置了一块1 m×1 m 的小样地a,采取0—25 cm间隔取样,共取样31个,研究实生苗的克隆生长情况。3个样地共计取样256个,其中样地A 43个,样地B 70个,样地C 143个。所有采样点均用GPS定位记录坐标,取每个分株的新鲜幼嫩叶片装入塑封袋中,并用变色硅胶干燥带回实验室于-80℃保存。
1.2 研究方法
1.2.1 DNA的提取和检测
用植物基因组DNA快速提取试剂盒(北京,百泰克)提取巴山木竹的总DNA。用1%的琼脂糖凝胶电泳检测DNA片段大小,用紫外分光光度计检测DNA的纯度和浓度。所提取的DNA用ddH2O稀释至25 ng/μL,于-20℃保存。
1.2.2 SSR引物和PCR扩增
经过初筛和复筛,决定选用8对巴山木竹SSR引物(loc2—loc8,loc10)来鉴定克隆(基因型)。8对引物的序列信息、退火温度及其PCR扩增条件详见关于发展巴山木竹的微卫星引物这一文献[28]。PCR反应在美国Bio-RAD公司生产的MyCycler Thermal Cycler #170- 9701EDU型PCR仪上进行。所有PCR产物在ABI3730XL DNA 测序仪(Applied Biosystems,USA)上进行毛细管电泳检测,用GeneMapper 4.0 (Applied Biosystems,USA)软件读取扩增片段大小。
1.2.3 克隆的鉴别
样地内所有基因型相同的分株被认为是同一个克隆(基株)[3,19]。使用POLYSAT 软件的assignClones功能对3个样地所有个体的基因型进行分析,基因型相同的个体被分配到同一克隆组[30]。然而,由于巴山木竹为四倍体植物[28,31-32],为了确保克隆划分的准确性,需要确认每个基因片段在该位点上出现的频率。使用MAC-PR (Microsatellite DNA Allele Counting-Peak Ratios)方法[33]来核算每个位点基因片段出现的频率,最终每个样扩增出的基因位点频率将通过微卫星峰图的面积比来获得。利用该方法获得的每个位点在该基因型中出现的频率可以是0、 0.25、0.5、 0.75或者是1, 这些频率被作为特征被记入每个个体[28,34]。仔细比对相同基因型个体每个位点的频率,最终基因型相同且位点基因频率相同的个体被确认为同一个克隆[28,34](如样地中部分个体基因型为ABC, 等位基因C在该基因型中出现的频率为0.5,即为ABCC,最终所有为ABCC的个体被确认为是同一个克隆)。为了方便后续的数据分析,使用POLYSAT软件转化所有具有基因频率特征的基因型数据为0 1 数据[30]。
1.2.4 数据分析
鉴别出所有个体的克隆基因组之后,根据每个采样点的坐标信息绘制出克隆分布图(图1)。为了更清楚的了解克隆的大小和形状,计算了样地B和C克隆分布图(图1)中除去边缘以及单样克隆以外的每个克隆的面积a和最大长宽比率rm。rm的值等于或大于1,rm的值越大,说明该克隆所有分株趋向于离散分布,rm的值越接近1,说明该克隆所有分株趋向于团块分布。
运用不同基因型比率(G/N),Simpson多样性指数(D)以及Fager指数(E)这3个指标来分析3个样地的克隆多样性。这3个指标的计算公式详见参考文献[2,10,35- 37]。D和E的范围为0—1,D为0表示种群内只有1个独克隆,即所有样品的基因型相同,D为1表示种群内每个样品为1个克隆,即每个样品的基因型各不相同;E为0表示种群内所有样品为不同的克隆或者是有一个优势克隆而其它所有样品为不同的克隆,E为1所有克隆的样本是相同的[2,10,35- 37]。
为了说明各种群内和种群间克隆的基因关系,运用NTSYSPC 2.10[38]对所有克隆基因型进行UPGMA非加权算术平均聚类分析(Unweighted pair-group method analysis)。为了进一步说明3个种群间和种群内的克隆基因关系,运用GenAlEx 6.5 对3块样地的克隆基因型进行了PCoA主坐标分析 (Principal Coordinates Analysis)[39]。
运用GenAlEx 6.5[39]软件计算巴山木竹所有分株基因遗传距离矩阵在相应距离等级下的空间自相关系数r,分析其克隆结构。在同一软件下对所有个体遗传距离的线性矩阵和地理距离之间的关系进行Mantel检测[39]。
2 结果与分析
2.1 巴山木竹克隆结构
2.1.1 不同巴山木竹种群克隆大小及其形状
利用8对引物对3个巴山木竹样地的256个样品进行PCR扩增,共检测到118个位点。 A、B和C 3个样地均由多克隆组成,分别为31、10和8个克隆,并且没有相同的基因型(图1)。
A样地中的小样地a 取样31个样,检测到了19个克隆;10 m间隔的A样地取样12个,检测到克隆12个。小样地a(1 m×1 m)中克隆面积均小于1 m2, 且从图1中可以看出克隆内的分株主要进行了短距离的密集型延伸。
B样地取样70个,检测到了10个克隆。除去边缘克隆B1、B4、B6、B7和B10外,B2、B3、 B5、B8和B9克隆面积和形状不同。其中,B2、 B5、B8和B9这4个克隆的面积(a)较为接近在550—650 m2,而B3面积最小为250 m2(表 1和图1)。 同时,这4个克隆的最大长宽比率(rm)值大小也较为接近,分别为2.36、2.76、2.17和2.36;而B3的rm值为4.00则略高于B2、 B5、B8和B9(表1)。结合B样地克隆结构分布图可以得出: B2、B3、B5、B8和B9均趋向于团块分布(图1)。
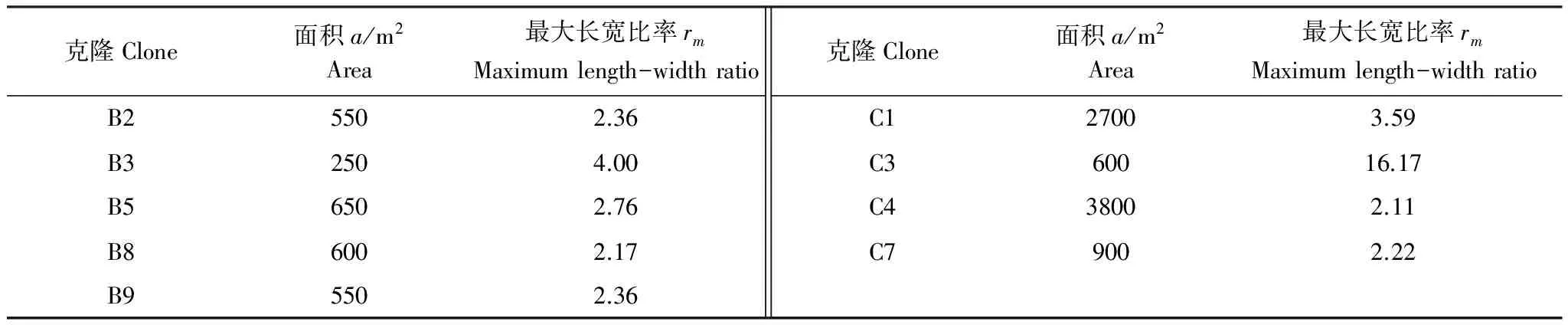
表1 样地B和C 9个克隆参数
B2,B3, B5,B8,B9分别代表的是B样地的5个不同克隆,即克隆B2,B3, B5,B8,B9;面积a的英文全拼: ;最大长宽比率的英文全拼: ;Rxy,xy是R的下标。
C样地共143个样,检测到了8个克隆。除去边缘克隆C2、C5、C6、C7和C8外,C1、C3和C4克隆面积和形状各不相同。其中,C1和 C4克隆面积较大分别为2700 m2和3800 m2,而C3面积则远小于C1和 C4仅有600 m2(表 1和图1)。C1、C3和C4rm值之间的大小差异较大,C1和 C4的rm值分别为3.59和2.11,而C3rm值则远高于C1和 C4为16.17(表1)。结合C样地克隆结构分布图可以得出, C1和 C4趋向于团块分布,而C3趋向于离散分布(图1)。

图1 巴山木竹3个样方的克隆空间分布Fig.1 Clonal structure in the three plots of Bashania fargesii inferred from Simple Sequence Repeat (SSR) fingerprintA:7龄巴山木竹克隆结构图,A1—A12:样地A的12个克隆, 虚线样框内是小样方a的放大,a1—a19: 小样方a的19个克隆; B: ﹥30龄巴山木竹克隆结构图,B1—B10: 样地B的10个克隆;C: ﹥60龄巴山木竹克隆结构图,C1—C8: 样地C的8个克隆
综上所述,随着年龄的增长3个样地的克隆数量减少;从克隆大小来分析(不包括样地边缘的克隆),随着年龄的增长克隆面积增加(表 1和图1);结合rm值和各样地克隆结构分布图分析(不包括样地边缘的克隆),A样地中的小样地a和B样地各克隆趋向于团块状分布,而C样地克隆既有团块状分布又有离散状分布(表1和图1)。
2.1.2 不同巴山木竹种群空间遗传结构
巴山木竹3个样地所有分株的空间自相关分析结果如图2所示。当距离等级间隔为10 m时, 3个样地都存在显著的正相关空间遗传结构。A、B和C 3个样地所有分株在空间距离分别小于3.1、28、48m时存在显著的正相关空间遗传结构,X-轴截距分别为9.051、30.698和50.536,空间自相关系数r的取值范围为0.1—0.167、0.008—0.703和0.006—0.735。 Mantel检测显示 3个样地的不同克隆的基因遗传距离和空间距离之间也存在正相关性 (A,Rxy=0.358,P﹤0.006; B,Rxy=0.529,P﹤0.001; C,Rxy=0.487,P﹤0.001)。因此,这两种分析都支持 3个样地在小尺度范围内存在明显的克隆结构。

图2 A、B和C 3个样地10m 等级下的空间自相关分析图Fig.2 Spatial autocorrelograms from three plots A、B and C in distance class sizes of 10 mr 为空间自相关系数, U和L分别表示不存在显著性空间自相关遗传结构的95%置信区间的上限和下限
2.2 巴山木竹克隆多样性分析
2.2.1 巴山木竹基因型多样性
为了在同一水平上比较3样地克隆多样性指标的变化,仅对10 m间隔取样的样品进行了多样性分析。A、B和C 3样地的基因型比率(G/N)分别为1、0.14和0.055, Simpson多样性指数(D)为1、0.876和0.744, Fager均匀度指数(E)为1、0.945和0.825。比较3个样地各指标可以发现,巴山木竹的基因型比率(G/N)、Simpson多样性 (D) 和Fager均匀度均随着该竹林年龄的增加而下降。
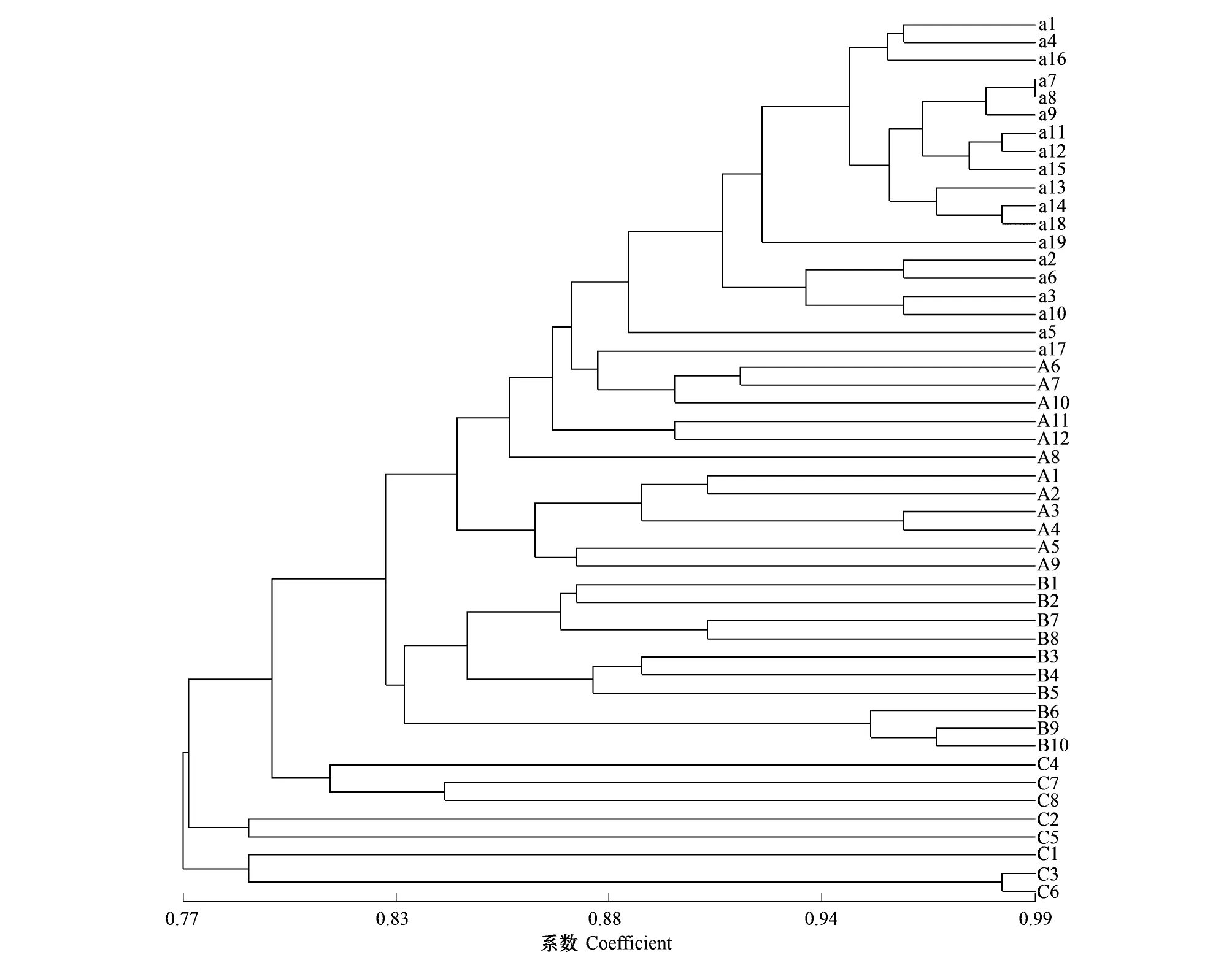
图3 3个样地的49个克隆UPGMA聚类分析图Fig.3 UPGMA clustering analysis of the 49 clones in the three plotsA1—A12:A样地的12个无性系,a1—a19:A样地中小样地a的19个无性系;B1—B10:B 样地的10个无性系; C1—C8:C样地的8个无性系
2.2.2 不同龄级巴山木竹种群聚类和主坐标PCoA分析
3个样地的克隆聚类结果如图3所示,各样地内克隆之间聚类明显,表现出较高的遗传相似性,总体上来自同一样地的克隆被聚为一类,但C样地的C4、C7和C8优先于C样地的其他克隆同样地B的克隆聚在一起。3个样地49个克隆基因型主坐标分析结果显示(图4):3个样地的大多数克隆基因型可以被清楚的分离,仅在C样地的C4、C7和C8和B样地部分区域重合,这与聚类分析有着较为一致的结果。
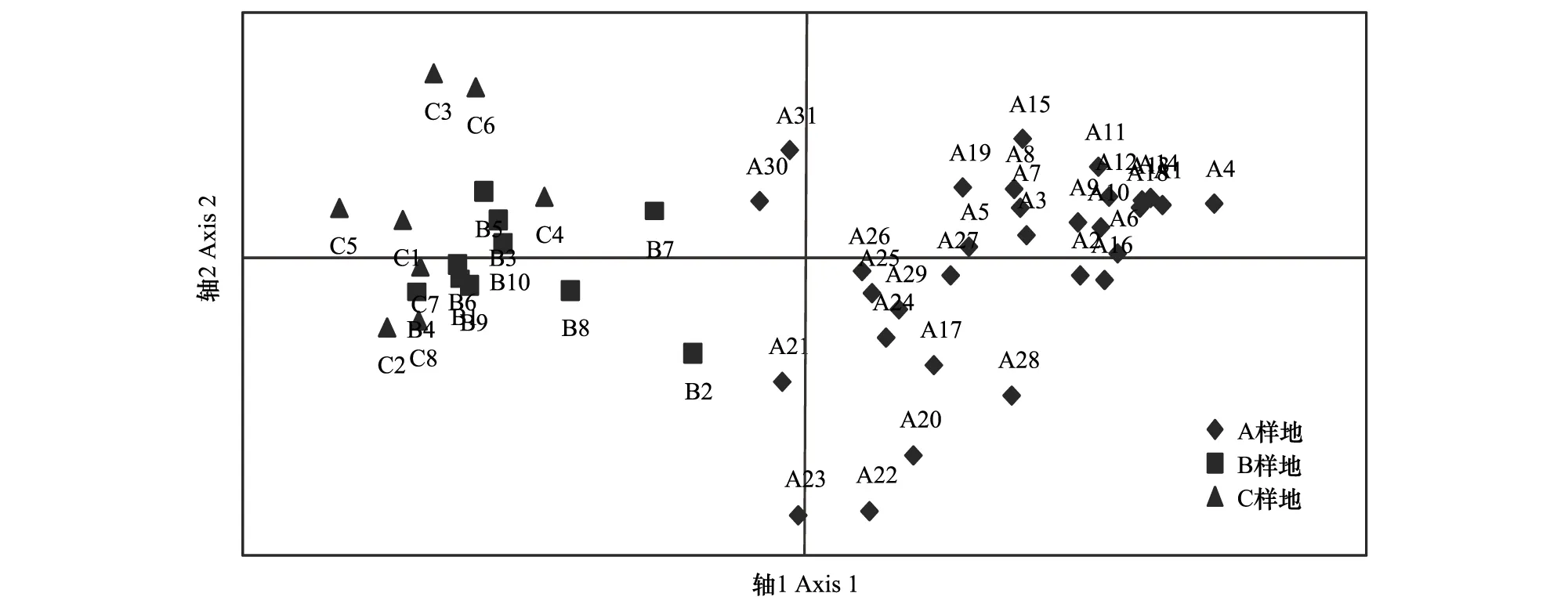
图4 3个样地49个克隆主坐标(pcoA)分析图Fig.4 Principal coordinates analysis (pcoA) of the 49 clones in the three plotsA1—A31: A样地的31个无性系, B1—B10: B样地的10个无性系, C1—C8: C样地的8个无性系
3 讨论
3.1 不同年龄巴山木竹的克隆结构
从3个样地的克隆大小和数量来分析,随着年龄的增长巴山木竹克隆面积逐渐增加,数量逐渐减少。从7龄竹林到﹥30龄的成年竹林克隆数量急剧减少面积急剧增大,而从﹥30龄到﹥60龄竹林克隆数量减少不明显,面积增加的倍数同前者相比也少的多。A样地为实生苗样地,在大尺度取样下(10 m×10 m),未检测到克隆的延伸;在小尺度取样下(0—25 cm), 由于幼苗稠密,基株无法进行更长距离的延伸,克隆内的分株主要进行了短距离的密集型延伸(图1a)。分析B样地的克隆分布图和各克隆的rm值可知,除去样地边缘的克隆外,B2、 B5、B8和B9四个克隆为团块状分布,B3由于处于这4个团块克隆的包围下,有从团块状向离散状发展的趋势。分析C样地的克隆分布图和各克隆的rm值可知,两个较大克隆C1和C4成团块状,而克隆C3成离散状。一般认为,克隆植物会优先占有有利于自己生存的环境,在生长环境良好的情况下,更容易形成较为均匀的密集型克隆[40]。然而,Inghe比较了游击型和密集型克隆植物的竞争力,发现当干扰率低的时候密集型克隆植物占优势,而干扰率较高时游击型植物占优势[41]。因此,B样地中B3处于B2, B5,B8和B9 4个团块克隆包围中,受到了一定的压迫,但由于空间和面积的原因仍然为团块状。C样地中,C3受到了C1和C4两个特大克隆的压迫,呈现为离散状。由于巴山木竹为复轴混生型的竹子,同时具有合轴丛生和单轴散生的两种类型的根茎,可推测出,当克隆基株受到强大的压迫时,可能进行更多的单轴和长距离的克隆延伸,从而呈现为离散状。
3.2 不同年龄巴山木竹的空间遗传结构
从A样地的自相关分析结果看,在3.1 m范围内,所有个体表现为正相关空间遗传结构,但这一相关系数r的范围为0.1—0.167,说明在该范围内进行较长距离延伸的克隆还较少,这同Mental检测结果P﹤0.006时才有相关性是一致的。在小尺度取样下(0—25 cm),检测到了部分相同的克隆分株,这说明巴山木竹在7龄时已经进行了克隆延伸,但仅限于短距离密集型的延伸。由此可见,对于较小年龄的巴山木竹林,应采取较小尺度的取样策略才能清晰地阐明克隆结构。而本研究中B和C样地的10 m间隔取样下的空间分析结果同Mental检测结果一致显示(P<0.001),可以清楚的得到克隆结构,说明取样策略是合理的。
虽然单独的空间自相关分析数据不能揭示真实的克隆结构[42],但是在同一距离10 m等级下比较3块样地的空间自相关分析数据,发现3块样地的正相关空间遗传结构的距离、X-轴截距随着年龄的增加而增加,空间自相关系数r的取值范围随着年龄的增加也在扩大。为了保证数据的可靠性,还做出了5、15、20 m不同距离等级的空间自相关分析图,正如 Peakall 等阐述的那样,改变距离等级X-轴截距也会随之改变[42]。但是,比较各距离等级下3个样地的相关数据发现各指标的变化和10 m距离等级下的改变趋势相同。当一个种群被检测到显著的正相关空间结构时,X-轴截距指的是基因斑块平均最短长度的相对空间距离[42]。由此可见,随着年龄的增长巴山木竹克隆的面积和规模在不断地扩大,同一克隆的分株数量增加,在均匀取样情况下,正相关空间遗传结构距离范围内取到具有相同基因型可能性越大。
3.3 不同年龄巴山木竹克隆多样性的比较及其原因分析
本研究中B和C样地的基因型比率(G/N)远低于其他竹子的值 (Sasasenanensis,G/N=0.43;B.fangiana,G/N=0.9583)[2,10]。采样规模和间隔的大小都会影响巴山木竹的基因型比率[2]。3个样地选择相同的采样间隔10 m, 比之前两种竹子的50 m和30 m都要小,这可能是该值远低于其他竹子的原因。比较3个样地巴山木竹的Simpson多样性指数(D)、基因型比率(G/N)和Fager均匀度指数(E),各指标值随着竹林年龄的增加而降低。其中, A样地与B和C的基因型比率(G/N)差别最大,说明巴山木竹幼苗期基因型比例远远高于成年的竹林。比较样地A、B和C 的Simpson多样性指数(D) 和Fager均匀度(E),发现各指标虽略有降低,但仍然保持了较高的克隆多样性和基因型均匀分布度,说明了巴山木竹成年后随着年龄的增长克隆多样性和基因型均匀分布度仍然较高。这和各样地克隆数量和面积随着年龄变化趋势分析是一致的。
从幼苗更新模式来看,巴山木竹为初始苗补充型(ISR,Initial Seedling Recruitment) 物种[8-9]。巴山木竹大约70—75a甚至更长时间开花结实[20],结实后可产生大量的幼苗,在7龄竹林的调查过程中,我们统计了每平方米的实生苗数可高达130株。从A样地来看,实生苗的克隆多样性很高,只有在0—25 cm更小尺度取样下,才检测到了不同分株属于同一克隆的基因型。关于克隆多样性随时间变化的研究,可以分为两类:一些研究认为,克隆植物基因型多样性会随着年龄的增长而急剧的降低[43-44];相反的,也有一些研究认为不会降低[45-46]。然而,从本实验分析来看,巴山木竹幼苗初始补充量很大,而且基因型多样性很高,随着样地年龄的增长,分株和基株间的竞争排斥或死亡导致克隆多样性略有降低,但仍然维持在一定的水平上,这说明巴山木竹的有性生殖对其克隆基因型多样性贡献很大。此外,Ma等和马青青等发现冷箭竹(Bashaniafangiana)和秦岭箭竹(Fargesiaqinlingensis)的初始补充苗都对克隆多样性有极大贡献[10-11]。
另外,本文在聚类和主坐标分析中,发现了不同样地少数克隆基因型的重叠和聚集,说明不同巴山木竹种群之间存在着基因流动和近似的克隆起源。竹子花粉较轻,无粘性,为风媒传粉植物,其花粉和种子可在外力情况下可以进行长距离扩散[11,47],因此有待进一步开展不同巴山木竹种群之间的基因流动研究。
[1] 董鸣. 克隆植物生态学. 北京: 科学出版社, 2011: 60- 62.
[2] Suyama Y, Obayashi K, Hayashi I. Clonal structure in a dwarf bamboo (Sasasenanensis) population inferred from amplified fragment length polymorphism (AFLP) fingerprints. Molecular Ecology, 2000, 9(7): 901- 906.
[3] Kitamura K, Kawahara T. Clonal identification by microsatellite loci in sporadic flowering of a dwarf bamboo species,Sasacernua. Journal of Plant Research, 2009, 122(3): 299- 304.
[4] 阮成江, 何祯祥, 周长芳. 植物分子生态学. 北京: 化学工业出版社, 2005: 36- 40.
[5] Shabel A B, Peart D R. Effects of competition, herbivory and substrate disturbance on growth and size structure in pin cherry (PrunuspensylvanicaL.) seedlings. Oecologia, 1994, 98(2): 150- 158.
[6] Martins M J F, Vandekerhove J, Adolfsson S, Rossetti G, Namiotko T, Jokela J. Effect of environmental stress on clonal structure ofEucyprisvirens(Crustacea, Ostracoda). Evolutionary Ecology, 2010, 24(4): 911- 922.
[7] De Witte L, Armbruster G F L, Gielly L, Taberlet P, Stöcklin J. AFLP markers reveal high clonal diversity and extreme longevity in four key arctic-alpine species. Molecular Ecology, 2012, 21(5): 1081- 1097.
[8] Eriksson O. Seedling dynamics and life histories in clonal plants. Oikos, 1989, 55(2): 231- 238.
[9] Eriksson O. Dynamics of genets in clonal plants. Trends in Ecology & Evolution, 1993, 8(9): 313- 316.
[10] Ma Q Q, Song H X, Zhou S Q, Yang W Q, Li D S, Chen J S. Genetic structure in Dwarf Bamboo (Bashaniafangiana) clonal populations with different genet ages. PLoS One, 2013, 8(11): 1- 8.
[11] 马青青, 刘建军, 余鸽, 刘伟, 马亦生. 佛坪国家级自然保护区秦岭箭竹克隆结构的SSR分析. 生态学报, 2016, 36(20): 6496-6505.
[12] Antonovics J, Ellstrand N C. Experimental studies of the evolutionary significance of sexual reproduction. I. a test of the frequency dependent selection hypothesis. Evolution, 1984, 38(1): 103- 115.
[13] Janzen D H. Why bamboos wait so long to flower. Annual Review of Ecology and Systematics, 1976, 7(7): 347- 391.
[14] Campbell J J N. Bamboo flowering patterns: a global view with special reference to East Asia. Journal of the American Bamboo Society, 1985, 6(1/4): 17- 35.
[15] 阮成江, 何祯祥, 周长芳. 植物分子生态学. 北京: 化学工业出版社, 2005: 101- 103.
[16] Bittebiere A K, Garbey M, Smaoui-Feki M, Clément B, Mony C. Structural blueprint and ontogeny determine the adaptive value of the plastic response to competition in clonal plants: a modelling approach. Evolutionary Ecology, 2014, 28(3): 573- 590.
[17] Bittebiere A K, Mony C, Clément B, Garbey M. Modeling competition between plants using an individual based model: methods and effects on the growth of two species with contrasted growth forms. Ecological Modelling, 2012, 234(3): 38- 50.
[18] 刘庆, 钟章成. 斑苦竹无性系种群克隆生长格局动态的研究. 应用生态学报, 1996, 7(3): 240- 244.
[19] Lian C L, Oishi R, Miyashita N, Nara K, Nakaya H, Wu B Y, Zhou Z H, Hogetsu T. Genetic structure and reproduction dynamics ofSalixreiniiduring primary succession on Mount Fuji, as revealed by nuclear and chloroplast microsatellite analysis. Molecular Ecology, 2003, 12(3): 609- 618.
[20] 王太鑫. 巴山木竹种群生物学研究[D]. 南京: 南京林业大学, 2005.
[21] 刘志学, 安里宁, 王仕安, 唐建文, 薛纪如. 巴山木竹的林学特性. 西南林学院学报, 1981, (1): 24- 33.
[22] 高新宇, 刘阳, 刘定震, 魏辅文. 秦岭大熊猫冬春季节对巴山木竹竹林生长指标的选择. 动物学研究, 2006, 27(2): 157- 162.
[23] 王太鑫, 丁雨龙, 刘永建, 李继清. 巴山木竹无性系种群的分布格局. 南京林业大学学报: 自然科学版, 2005, 29(3): 37- 40.
[24] 郑晓燕, 何祥博, 刘新玉. 佛坪保护区大熊猫取笋区域内的巴山木竹林现状分析. 陕西林业科技, 2010, (2): 25- 27.
[25] 唐新成, 王逸之. 巴山木竹研究现状及展望. 林业调查规划, 2012, 37(4): 31- 34.
[26] Kalia R K, Rai M K, Kalia S, Singh R, Dhawan A K. Microsatellite markers: an overview of the recent progress in plants. Euphytica, 2011, 177(3): 309- 334.
[27] Rouger R, Jump A S. Fine-scale spatial genetic structure across a strong environmental gradient in the saltmarsh plantPuccinelliamaritima. Evolutionary Ecology, 2015, 29(4): 609- 623.
[28] Yu G, Liu J J, Ma Q Q, Ma Y S. Development polymorphic microsatellite primers for the giant panda′s staple food bambooBashaniafargesiiandB.aristata(Poaceae: Bambusoideae). Conservation Genetics Resources, 2015, 7(3): 739- 741.
[29] 刘颖颖, 傅金和. 大熊猫栖息地竹子及开花现象综述. 世界竹藤通讯, 2007, 5(1): 1- 4.
[30] Clark L V, Jasieniuk M. Polysat: an R package for polyploid microsatellite analysis. Molecular Ecology Resources, 2011, 11(3): 562- 566.
[31] Chen R Y, Zong W X. Chromosome numbers of some scattered bamboos. Acta Phytotaxonomica Sinica, 1991, 29(5): 452- 455.
[32] Zhang Y X, Zeng C X, Li D Z. Complex evolution inArundinarieae(Poaceae: Bambusoideae): incongruence between plastid and nuclear GBSSI gene phylogenies. Molecular Phylogenetics and Evolution, 2012, 63(3): 777- 797.
[33] Esselink G D, Nybom H, Vosman B. Assignment of allelic configuration in polyploids using the MAC-PR (microsatellite DNA allele counting-peak ratios) method. Theoretical and Applied Genetics, 2004, 109(2): 402- 408.
[34] Palop-Esteban M, Segarra-Moragues J G, González-Candelas F. Polyploid origin, genetic diversity and population structure in the tetraploid sea lavenderLimoniumnarbonenseMiller (Plumbaginaceae) from eastern Spain. Genetica, 2011, 139(10): 1309- 1322.
[35] Pielou E C. An Introduction to Mathematical Ecology. New York: Wiley-Interscience, 1969: 219- 220.
[36] Fager E W. Diversity: a sampling study. American Naturalist, 1972, 106(949): 293- 310.
[37] Ellstrand N C, Roose M L. Patterns of genotypic diversity in clonal plant species. American Journal of Botany, 1987, 74(1): 123- 131.
[38] Rohlf F J. NTSYSpc: Numerical Taxonomy and Multivariate Analysis System, Version 2.1. New York: Exeter Software, 2000.
[39] Peakall R, Smouse P E. Genalex 6.5: genetic analysis in Excel. Population genetic software for teaching and research-an update. Bioinformatics, 2012, 28(19): 2537- 2539.
[40] Dale M R T. Spatial Pattern Analysis in Plant Ecology. Cambridge: Cambridge University Press, 1999: 224- 226.
[41] Inghe O. Genet and ramet survivorship under different mortality regimes---A cellular automata model. Journal of Theoretical Biology, 1989, 138(12): 257- 270.
[42] Peakall R, Ruibal M, Lindenmayer D B. Spatial autocorrelation analysis offers new insights into gene flow in the Australian bush rat,Rattusfuscipes. Evolution, 2003, 57(5): 1182- 1195.
[43] Travis S E, Hester M W. A space-for-time substitution reveals the long-term decline in genotypic diversity of a widespread salt marsh plant,Spartinaalterniflora, over a span of 1500 years. Journal of Ecology, 2005, 93(2): 417- 430.
[44] Aarssen L W, Turkington R. Within-species diversity in natural populations ofholcuslanatus,loliumperenneandtrifoliumrepensfrom four different-aged pastures. Journal of Ecology, 1985, 73(3): 869- 886.
[45] Verburg R, Maas J, During H J. Clonal diversity in differently-aged populations of the pseudo-annual clonal plantCircaealutetianaL. Plant Biology, 2000, 2(6): 646- 652.
[46] Hartnett D C, Hartnett B B, Bazzaz F A. Persistence of Ambrosia trifida populations in old fields and responses to successional changes. American Journal of Botany, 1987, 74(8): 1239- 1248.
[47] 林树燕, 石文文, 缪彬彬, 丁雨龙. 竹类植物生殖生物学研究进展. 世界竹藤通讯, 2010, 8(2): 1- 6.
Clonal structure ofBashaniafargesiipopulations at different ages
YU Ge1, LONG Fenglai2, LIU Jianjun3,*, MA Qingqing1, KANG Yongxiang1, HUANG Jian1, CAO Qing4
1CollegeofForestry,NorthwestAgriculture&ForestryUniversity,Yangling712100,China2YangLingVocationalTechnologyCollege,Yangling712100,China3CollegeofLandscapeArchitectureandArts,NorthwestAgriculture&ForestryUniversity,Yangling712100,China4ShaanxiFopingNationalNatureReserve,Foping723400,China
Bamboos are typical clonal plants commonly used as food for giant pandas. Studies of the clonal structure of typical bamboo populations are of both theoretical and practical importance for bamboo forest production and tending. Studies have attempted to predict its population succession for the protection of giant pandas. In this study, simple sequence repeat fingerprints were used to reveal the clonal structure and diversity ofBashaniafargersiipopulations at three different genet ages (A 7 years, B > 30 years and C > 60 years). We described how the clonal structure ofB.fargersiipopulations at different genet ages established and developed at the small scale. We amplified 118 microsatellite locus using 8 selective primer pairs. A total of 49 clones were identified from 256 leaf samples collected from three populations, among which 31 clones were detected in plot A, 10 clones in plot B, and 8 clones in plot C. The size of clones in the three plots increased and the number of clones decreased with population aging. The spatial distribution pattern of clones in plots A and B exhibited a clumped distribution, while plot C showed two different patterns with simultaneously clumped and discrete distributions. The results showed that the genet generally formed a clumped distribution pattern during the seeding stage. The clones may expand into either the more sympodial type of ramets or short-distance clones, indicating that clonal propagation restricts dense seedling growth. However, with increasing clone size and genet age, compound axis mixedB.fargesiimay expand into either the more monopodial type of ramets or long-distance clones representing a discrete distribution pattern when the genet are pressured by other strong clones. In addition, in our study, both the Mantel test and spatial autocorrelation analysis supported the significant presence of positive spatial clonal structures in three plots at the small-scale level. Spatial autocorrelation analysis also showed that the positive spatial genetic structure distance of the three plots in the 10 m distance class were 3.1, 28, 48 m,X-intercepts were 9.051, 30.698, and 50.536, and scope of spatial autocorrelation coefficientsrwere 0.1—0.167, 0.008—0.703, and 0.006—0.735, respectively. Our results showed that the size and scale of clones increased with genetic aging. Additionally, the number of ramets in the same clone increased under uniform sampling conditions, indicating that more samples with the same genotype can be collected with a positive spatial genetic structure distance. In our study, the distinguishable genotypes (G/N) from populations A, B, and C were 1, 0.14, and 0.055 and the Simpson′s indices of diversity (D) were 1, 0.876, and 0.744, respectively. This result revealed that the distinguishable genotypes at the seeding stage (A 7 years) was much greater than that at the adult stage (B 30 years and C 60 years). Although the genotypic diversity of clonal populations reduced with genet aging, because of initial seedling recruitment, the value remained high. The unweighted pair-group method and principal component analysis demonstrated that clones in the same plot were always classified into the same clade. However, a few clones from different plots exhibited aggregation and overlap during analysis. In conclusion, we demonstrated that gene flow and an approximate clone origin might exist in different populations.
Bashaniafargesii; clonal structure; clonal diversity
国家“十二五”农村领域科技计划课题(2015BAD07B0203)
2016- 04- 19; 网络出版日期:2017- 03- 02
10.5846/stxb201604190733
*通讯作者Corresponding author.E-mail: ljj@nwsuaf.edu.cn
余鸽,龙凤来,刘建军,马青青,康永祥,黄建,曹庆.不同年龄巴山木竹种群克隆结构.生态学报,2017,37(14):4743- 4753.
Yu G, Long F L, Liu J J, Ma Q Q, Kang Y X, Huang J, Cao Q.Clonal structure ofBashaniafargesiipopulations at different ages.Acta Ecologica Sinica,2017,37(14):4743- 4753.

