新型拟除虫菊酯降解酶的分子改造及降解特性
刘孝龙,刘玉焕,范新炯
(1.安徽医科大学基础医学院,安徽 合肥 230032;2. 中山大学生命科学学院,广东 广州 510275 )
新型拟除虫菊酯降解酶的分子改造及降解特性
刘孝龙1,2,刘玉焕2,范新炯1,2
(1.安徽医科大学基础医学院,安徽 合肥 230032;2. 中山大学生命科学学院,广东 广州 510275 )
为了获得稳定性和酶活性提高的拟除虫菊酯类降解酶,利用易错PCR技术对来源于海底泥宏基因组文库的新型酯酶基因(est825)进行体外定向进化,获得了一个酶活性和热稳定性均提高的突变酶(EstM46)。与野生型相比其酶活力提高1.5倍;最适反应温度提高了5 ℃,同时热稳定性明显增强。此外,EstM46对氯氟氰菊酯、氯氰菊酯、氰戊菊酯和溴氰菊酯的降解率分别提升至92.21%、99.75%、93.21%和89.48%。这对降低酶的生产成本、促进工业化发展和环境保护有重要意义。
拟除虫菊酯;农药降解酶;定向进化;生物降解
拟除虫菊酯作为高效、低毒,环境友好型农药,在世界范围内得到了广泛的应用,但其对生态环境和人类健康带来的危害也逐渐显现出来。因而,菊酯类农药的残留问题越来越受到人们的重视。生物法尤其是生物酶类,在处理拟除虫菊酯类农药的残留时,具有操作简单、安全高效、应用范围广且无二次污染等优点,逐渐成为菊酯类农药残留生物修复的研究热点。国内外的科学研究者们利用基因工程手段从环境中筛选到各种菊酯类农药降解酶基因,并构建了异源表达工程菌,获得具有降解谱广、降解能力强等优点的降解酶,为微生物降解农药开辟了新的途径[1-5]。但这些已开发的工程酶一般存在稳定性差和非天然底物或环境下的催化能力低等限制条件,不能满足工业生产需求。定向进化技术无需对蛋白质的结构、结构与功能之间的关系、催化机制等因素进行研究,直接对酶基因进行突变,再结合高通量筛选方法获得新功能酶,成为天然酶突破自身因素限制的重要工具[6-8]。为获得稳定、高度专一性以及对非天然底物催化能力强的新功能酶指明了新的方向。
当前,在核酸分子水平利用分子定向改造手段对宏基因组学技术来源的新型菊酯类降解酶基因进行改造的研究报道还很少见。因而,本研究利用易错PCR技术对本实验室从海底泥宏基因组文库中筛选到的酯酶基因est825进行体外定向进化,结合高通量的筛选方法,以期获得稳定性好、酶活性高的突变酶,为生产低成本、高质量的菊酯类农药降解酶制剂奠定基础。
1 材料和方法
1.1 材料
1.1.1 菌株和质粒用卡 宿主菌E.coliBL21(DE3), (Novagen);载体pET-28a(+), (Novagen)和含酯酶基因的质粒pUC118-est825由本实验室保存;
1.1.2 培养基 LB培养基:胰蛋白胨10 g,酵母提取物5 g,氯化钠10 g,用蒸馏水定容至1 L,121 ℃灭菌20 min。固体培养基即加入w=1.5%~2%的琼脂粉;
SOC培养基:胰蛋白胨2 g,酵母提取物0.5 g,氯化钠0.05 g,氯化钾0.018 6 g,氯化镁0.095 g,蒸馏水溶解并定容至80 mL,葡萄糖0.36 g,蒸馏水溶解并定容至20 mL,115 ℃灭菌25 min,将两种溶液混合,按900 μL/管分装,-20 ℃保存备用。
1.1.3 酶和试剂 胰蛋白胨、酵母提取物购于OXOID公司(英国),硫酸卡那霉素(Kana)、异丙基-β-D-硫代半乳糖苷(IPTG)、5-溴-4-氯-3-吲哚-β-D-半乳糖苷(X-gal)、5-溴-4-氯-3-吲哚辛酯(5-Bromo-4-chloro-3-indolyl caprylate, X-caprylate)均为进口分装,购于北京普博欣生物科技有限公司;引物合成和基因测序由上海英骏生物技术有限公司广州实验室完成;气相色谱法测定残留农药降解率由东莞市农业科学研究中心完成。各种限制性内切酶、T4连接酶、低相对分子质量蛋白质标准购于宝生物(大连)工程有限公司;质粒提取试剂盒,核酸定量标准购于广州东盛生物科技有限公司;蛋白纯化试剂盒(His·Bind®Purification Kit)购于Novagen公司(德国);易错PCR突变试剂盒(GeneMorh®II Random Mutagenesis Kit)购于Stratagene公司(美国);凝胶回收试剂盒和连接产物回收试剂盒购于Omega公司(美国)。
1.2 方法
1.2.1 易错PCR 扩增与突变文库的构建 根据est825基因序列设计引物Mut-F:5′- GCCATGGCTGATATCGGATCC ATGAGTGAGCTGACATCAATCTCCG -3′ (下划线部分为BamHⅠ酶切位点);Mut-R:
5′-TTCAAGTTCAGACT CAAGCTTTCAGGGGGCCAGCAACTC -3′ (下划线部分为HindⅢ酶切位点)。以含pUC18-est825质粒为模板,用GeneMorh®Ⅱ Random Mutagenesis Kit进行易错PCR 扩增。每100 μL 反应体系为:10 μL 10×易错PCR 缓冲液;2 μL 10 × dNTP 混合物(40 mmol/L);引物Mut-F和Mut-R各5 ng;500 ng(以目的基因计)质粒DNA模板;2 μL Mutazyme Ⅱ DNA Polymerase (2.5 U/μL),再加入灭菌的超纯水至总体积为100 μL。PCR 程序为:95 ℃ 2 min;95 ℃ 1 min,65 ℃ 1 min,72 ℃ 1 min,30 个循环;72 ℃ 10 min。
将PCR 产物以w=1.0%的琼脂糖凝胶电泳,用PCR 纯化回收试剂盒进行纯化回收。纯化后的易错PCR 产物酶切后与经同样酶切处理的pET-28a 载体连接,转化大肠杆菌BL21(DE3)超级感受态细胞,涂布固体LB(含50 μg/mL 的Kana)平板,挑选具有插入片段的克隆,组成突变体库。
1.2.2 阳性克隆的筛选和鉴定 首先将所有转化子点种于种子板(含Kana 50 μg/mL)和诱导板(含50 μg/mL Kana、24 μg/mL IPTG)中,37 ℃培养48 h,取出诱导板,在菌落周围滴加底物X-caprylate测试液进行初筛,酯酶可将该测试液从无色水解为蓝色化合物,若变蓝则为阳性克隆子,酶活性越高,颜色越深;然后将初筛时显色深的突变克隆子,接种于LB液体培养基(含50 μg/mL Kana、1 mmol/L IPTG)中进行诱导培养,收集菌体并超声波破碎,获得粗酶液;用ρNPE法[9]检测其酶活性,同时做野生型酯酶对照,将酶活性提高的突变子送交测序,对测序结果与原序列进行生物信息学分析。
1.2.3 蛋白质含量测定及突变酶的酶学性质研究 诱导的菌体经破碎后用蛋白纯化试剂盒进行纯化。纯化后的蛋白质含量采用Bradford 法[10]进行测定并用于酶学性质的测定。
1.2.4 突变酶的结构预测和突变位点分析 将突变酯酶和野生型酯酶的氨基酸序列提交至SWISS-MODEL在线分析服务器(https:∥www.swissmodel.expasy.org/interactive),将反馈的模拟结果用DeepWIEW软件分析结构,推测氨基酸突变在酶蛋白空间结构中的位置以及对酶蛋白功能可能造成的影响。
1.2.5 拟除虫菊酯农药的降解能力测定 选取农产品中易超标的氯氟氰菊酯、氯氰菊酯、氰戊菊酯和溴氰菊酯为降解对象,用GC-2010气相色谱仪(日本岛津公司)进行定量分析,测定突变酶EstM46对拟除虫菊酯类农药的降解能力。具体方法:吸取1 mL标准农药(4 mg/mL)溶液,加入3 mL酯酶酶液,每种农药做5个平行,以灭活酶液为空白对照;37 ℃水浴反应60 min,按1∶2(V/V)的比例在待测样品中加入正己烷,并加入4 g无水硫酸钠,在恒温摇床上振荡20 min (200 r/min),8 000 r/min离心5 min,准确吸取1 μL用GC-2010气相色谱仪(日本岛津公司)进行定量分析。检测条件:GC-2010气相色谱仪:ECD检测器,RestekRTX-5色谱柱(30 m×0.25 mm×0.25 μm)。分流进样,分流比60∶1;进样量1.0 μL;进样口温度:250 ℃;柱流量:2.0 mL/ min;恒压方式,柱前压为50 kPa;载气为高纯氮气(纯度>99.999%),流速40 mL/ min;柱箱程序升温:150 ℃,保持1 min,以30 ℃/ min的速度上升至270 ℃,保持10 min;ECD检测器温度300 ℃,电流1 nA;尾吹流量30 mL/ min。农药降解率计算:
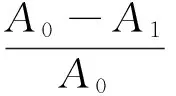
式中,A0为对照组中农药浓度;A1为实验组中农药浓度。
2 结 果
2.1 突变酯酶基因的易错PCR产物检测
利用突变引物Mut-F和Mut-R扩增得到突变酯酶基因片段,w=1%的琼脂糖凝胶电泳分析其大小约825 bp (见图1)。
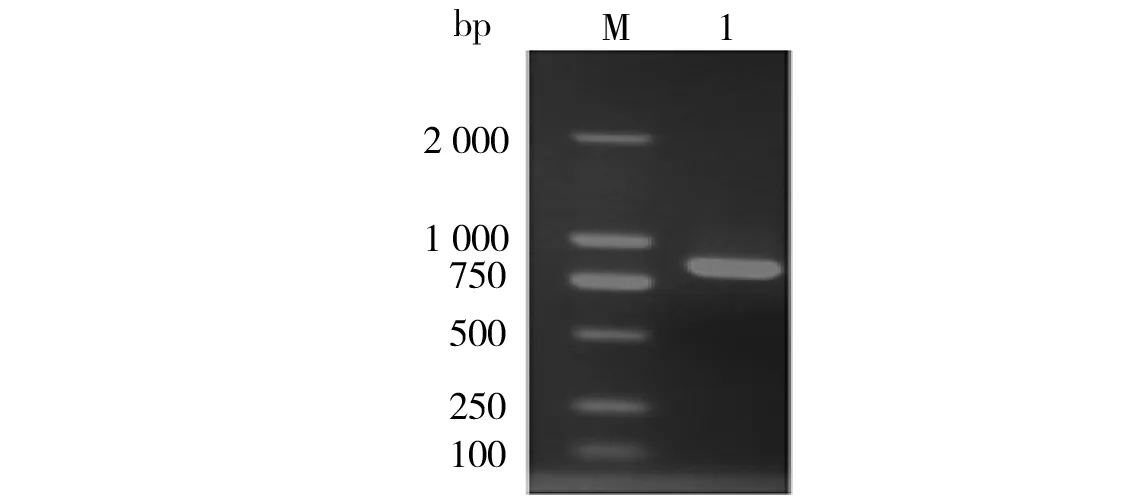
图1 酯酶基因est825易错PCR产物电泳分析Fig.1 The electrophoresis analysis of ep-PCR products of esterase gene est825M: DNA maker DL2000; Lane 1: est825的易错PCR产物
2.2 随机突变文库的构建
经转化E.coliBL21(DE3),生长出约10 000个转化子。随机挑取16株阳性突变株,提取质粒并用HindⅢ和BamHⅠ双酶切,凝胶电泳分析如图2,93.75%的转化子都插入了随机突变的酯酶基因。
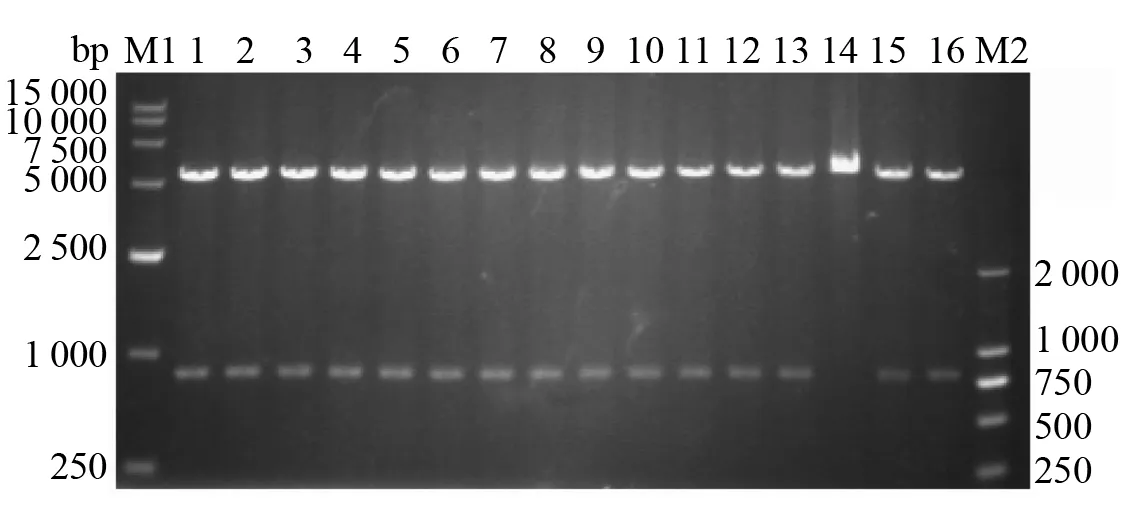
图2 质粒酶切验证随机突变库阳性转化子Fig.2 The identification to recombinant plasmid from random mutation libraries by restriction digestionM1: DL 15 000; M2:DL 2 000; Line 1-16: 阳性克隆子质粒的酶切产物
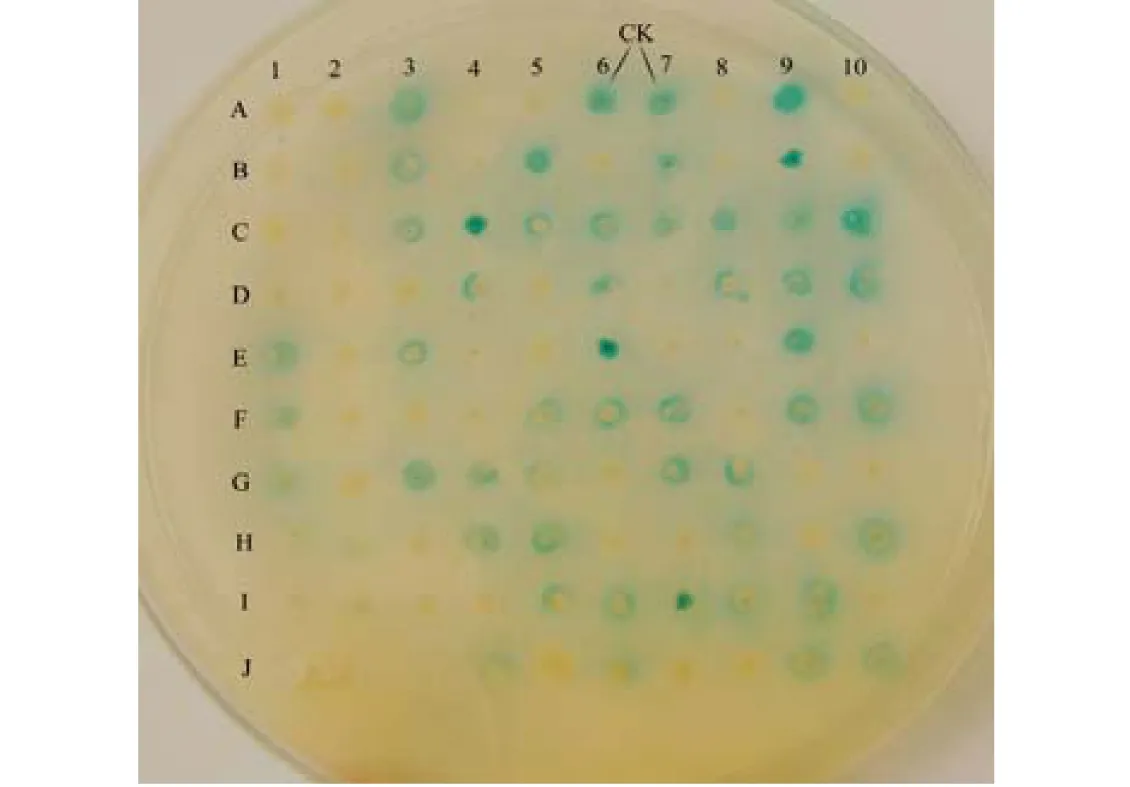
图3 易错PCR文库转化子的初筛Fig.3 The primary screening for transformants from EP-PCR
2.3 随机突变子的筛选
2.3.1 初筛 插入随机突变酯酶基因片段的阳性克隆子,经诱导后滴加X-caprylate测试液,结果见图3。经统计 (见图4),只有约5%的突变子产生正突变,其余突变子表现为酶活性未发生变化(20%)、酶活性降低(25%)和无酶活性(50%)。
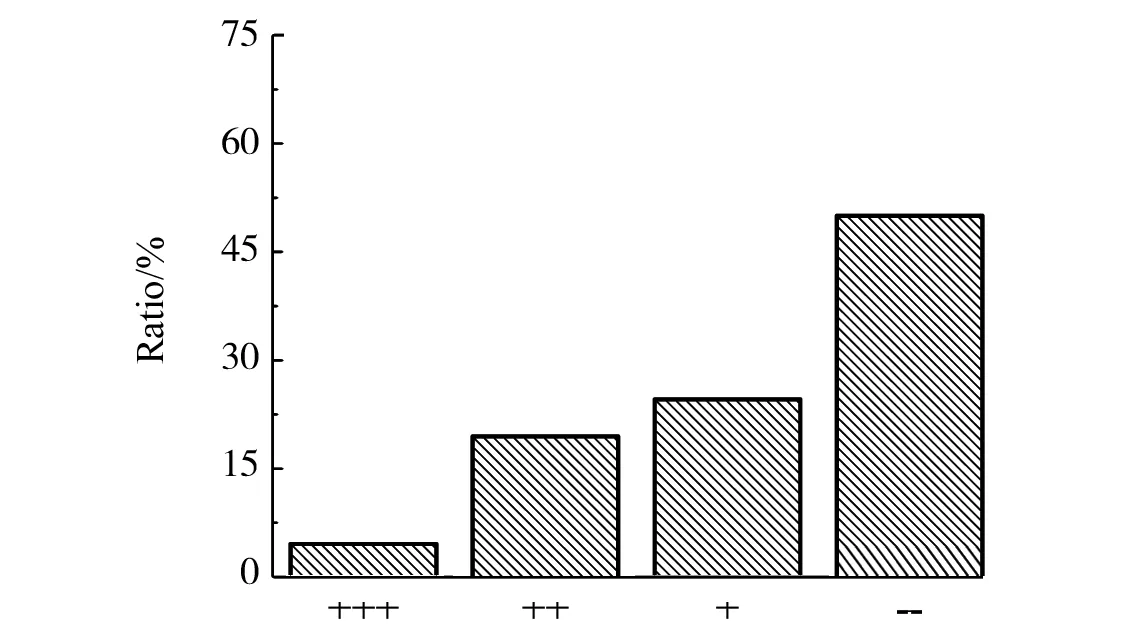
图4 阳性转化子突变结果统计Fig.4 Statistics profile of the mutants from EP-PCR+++: 酶活性较高; ++: 酶活性正常; +:酶活性较低; -: 无酶活性
2.3.2 随机突变子的复筛 挑取初筛得到的酯酶活性提高的突变子,诱导培养后超声波破碎获得粗酶液,用ρNPE法检测其酯酶活性,同时以野生酯酶为对照。最终筛选得到一株酶活性提高的1.5倍突变子M46,经SDS-PAGE蛋白质电泳分析,该突变子所表达的酯酶蛋白相对分子质量和表达量分别与野生型酶相同和相当(见图5)。
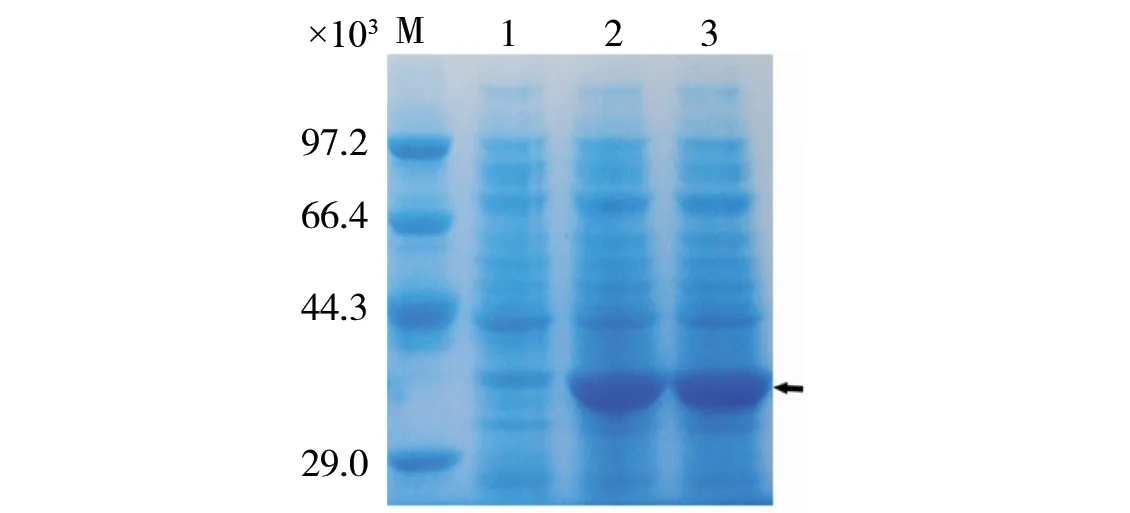
图5 SDS-PAGE电泳分析突变酶和野生酯酶Fig.5 SDS-PAGE of wild type and mutant esteraseM:低相对分子质量蛋白标准 (TaKaRa); Lane 1:无插入片段载体对照; Lane 2:野生型酯酶Est825蛋白; Lane 3:突变酶EstM46蛋白
2.4 突变酶的诱导表达和分离纯化
重组菌株经30 ℃,1.0 mmol/LIPTG诱导8 h,离心收集菌体,用缓冲液洗涤菌体并超声波破碎细胞,收集上清液即为粗酶液。用His·Bind®Purification Kit (Novagen)纯化粗酶液,所得产物进行SDS-PAGE蛋白电泳检测,结果见图6,其相对分子质量约为34 100(其中包含4 000的融合标签)。用Bradford法定量显示其最高表达量可达200 mg/L。使用Quantity One软件(Bio-Rad公司,美国)对蛋白电泳结果进行分析,结果表明重组蛋白含量占细胞内全部可溶性蛋白的45%。
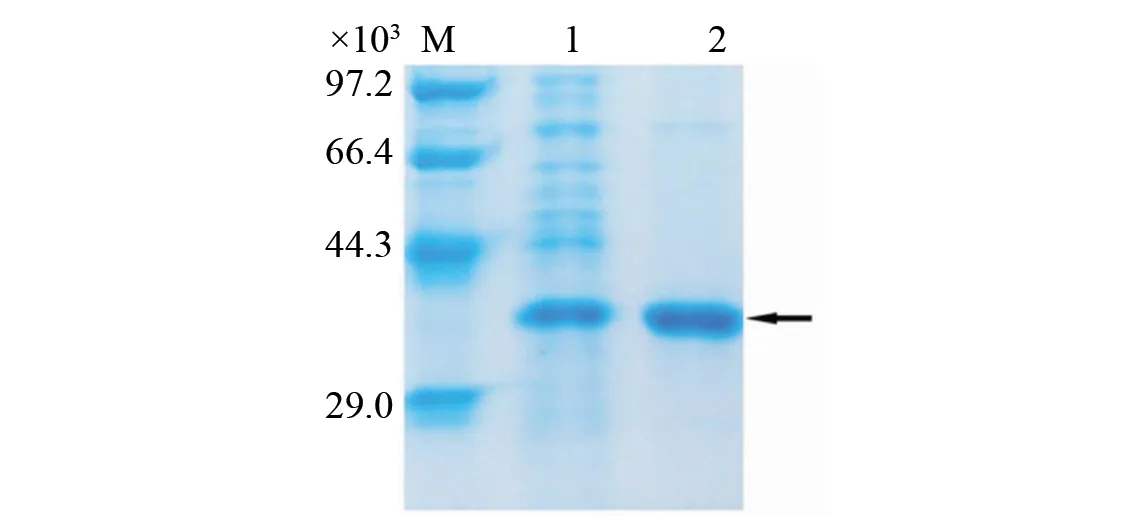
图6 重组酯酶EstM46的SDS-PAGE电泳图Fig.6 SDS-PAGE analysis of recombinant estase EstM46 M:低相对分子质量标准蛋白(TaKaRa); Lane 1:纯化前的重组蛋白EstM46; Lane 2:纯化后的重组蛋白EstM46
2.5 突变酶的酶学性质分析
2.5.1 突变酯酶EstM46的最适反应温度 在不同温度下对突变酶EstM6的酶活性进行测定,以酶活性最高为100%,计算相对酶活,结果如图7所示,突变酶的最适反应温度为45 ℃,与野生酶的酶活性比较,提高5 ℃,且突变酶温度>45 ℃时具有较好的水解能力。
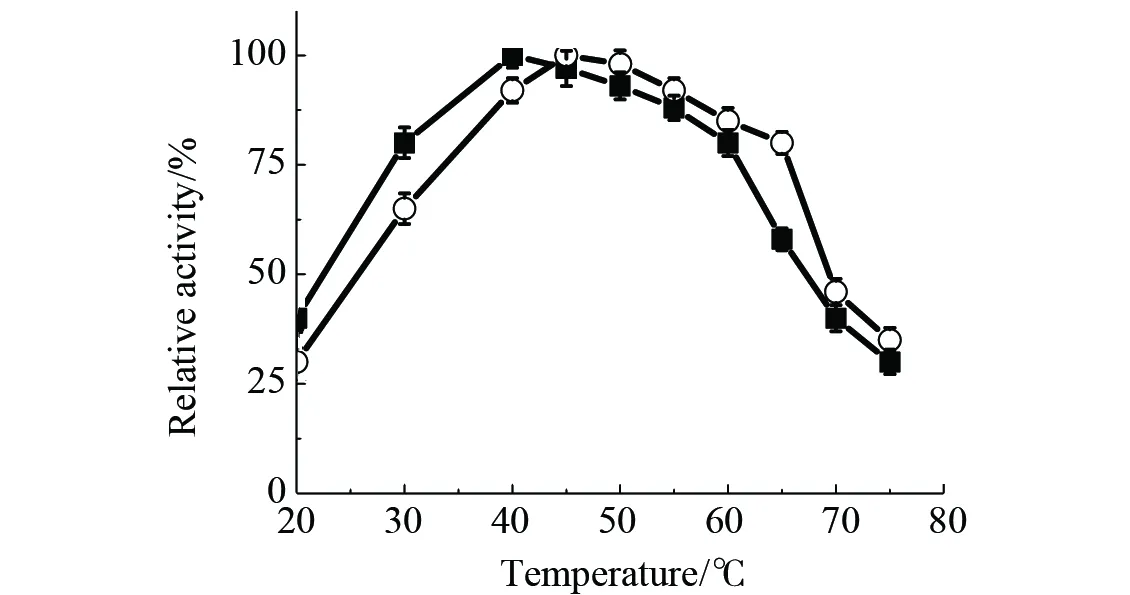
图7 温度对野生酯酶(■)和突变酯酶(○)酶活性的影响Fig.7 Effect of pH on the activity of wild type and mutated esterases
2.5.2 突变酯酶EstM46的温度稳定性 将突变酶EstM46置于不同温度下保温5 h,每隔1 h取样,45 ℃下测定残余酶活性,结果如图8所示,该酶在60 ℃保温5 h后剩余75%以上的酶活性,且在70 ℃保温2 h后仍剩余约80%左右的酶活性。因此,突变酶的热稳定性明显提高。
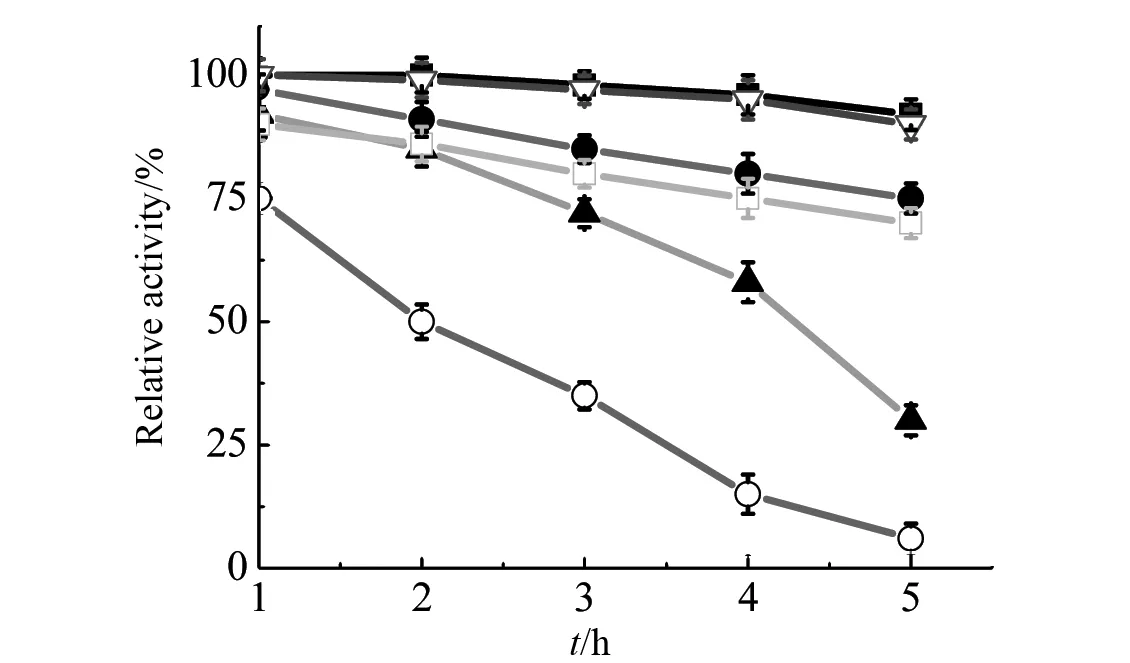
图8 温度对野生酯酶和突变酯酶稳定性的影响Fig.8 Effect of temperature on the stability of wild type and mutated esterases■:野生酶, 45 ℃; □:野生酶, 60 ℃; ○:野生酶, 70 ℃;●:突变酶, 60 ℃; ▽:突变酶, 45 ℃; ▲:突变酶, 70 ℃
2.5.3 突变酯酶EstM46的最适反应pH值及pH稳定性 取等量的突变酶EstM6置于不同缓冲体系中,45 ℃反应4 min,以对硝基苯酚乙酸酯为底物,酶活性最高为100%,测定突变酶的最适反应pH值。如图9所示,突变酶和野生酶相比,在pH值介于7.0~8.5之间时具有较好的催化活力。
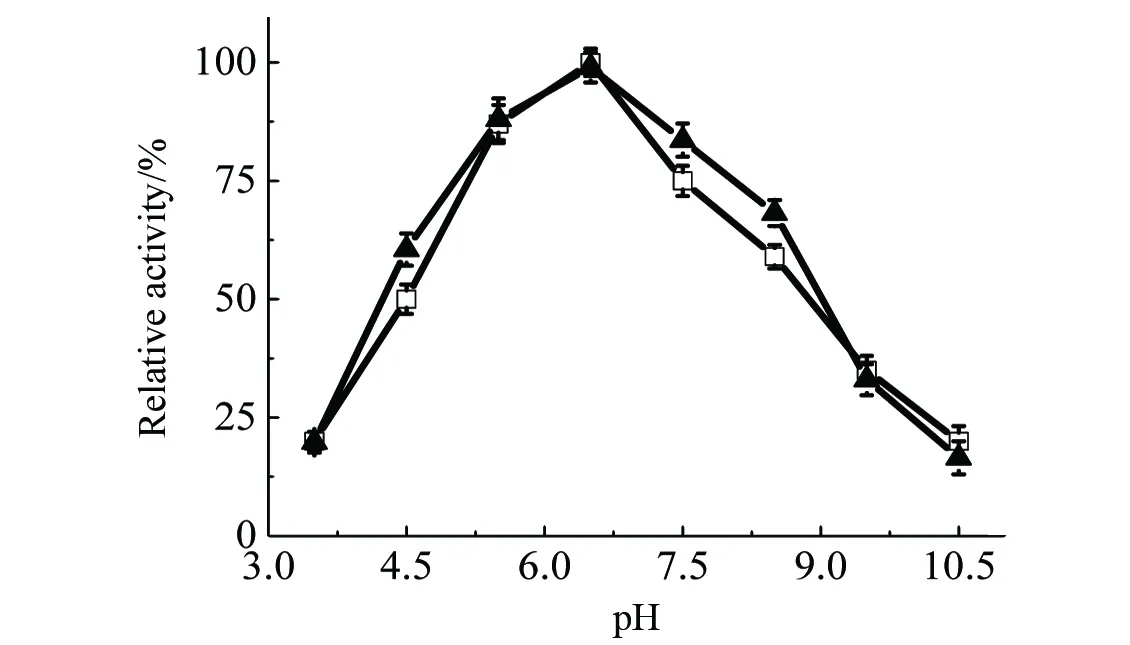
图9 pH对野生酯酶(□)和突变酯酶(▲)酶活性的影响Fig.9 Effect of pH on the activity of wild type and mutant esterases
取等量突变酶置于不同pH值的缓冲液体系中,室温下放置2 h,用对硝基苯酚乙酸酯为底物,45 ℃反应4 min,以酶活性最高为100%,测定其pH稳定性。图10所示,与野生酶相比,突变酶的pH稳定性无明显变化。
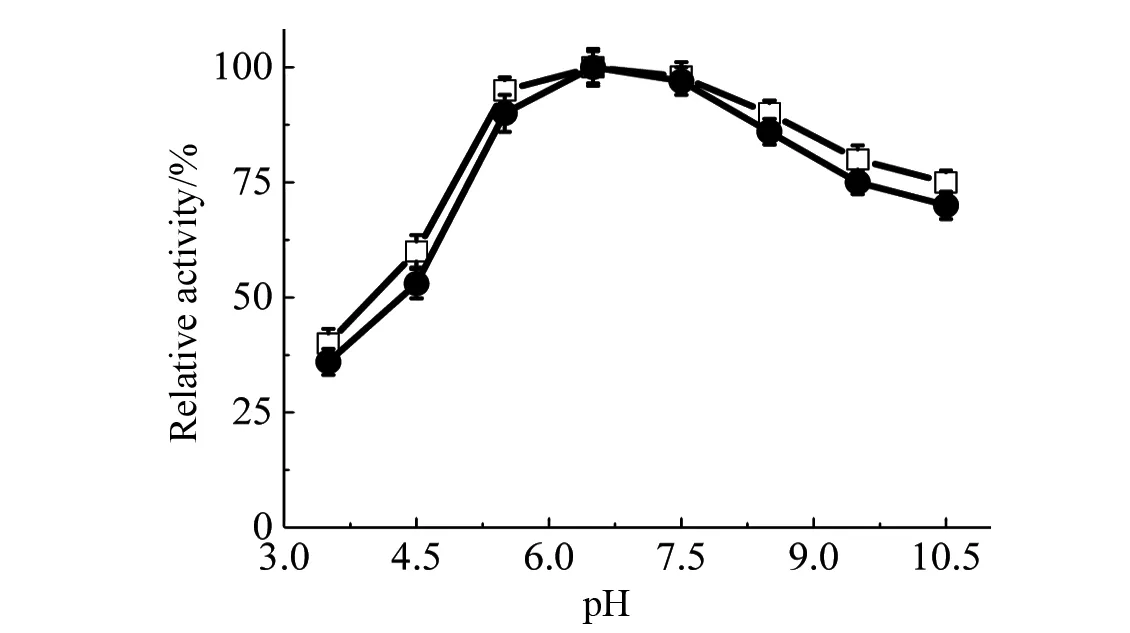
图10 pH对野生酯酶(●)和突变酯酶(□)稳定性的影响Fig.10 Effect of pH on the stability of wild type and mutant esterases
2.5.4 突变酯酶EstM46拟除虫菊酯农药的降解能力测定 将3 mL突变酶EstM46与1 mL 4 mg/L的农药标准液混合,37 ℃反应60 min,用正己烷提取剩余农药,取1 μL测定气相色谱分析,根据测定色谱峰(图11)计算回收率和降解率(如表1),每组5个平行。结果表明突变酶EstM46对4种拟除虫菊酯类农药的降解率均有不同程度的提高,分别达到92.21%、99.75%、93.21%和89.48%。
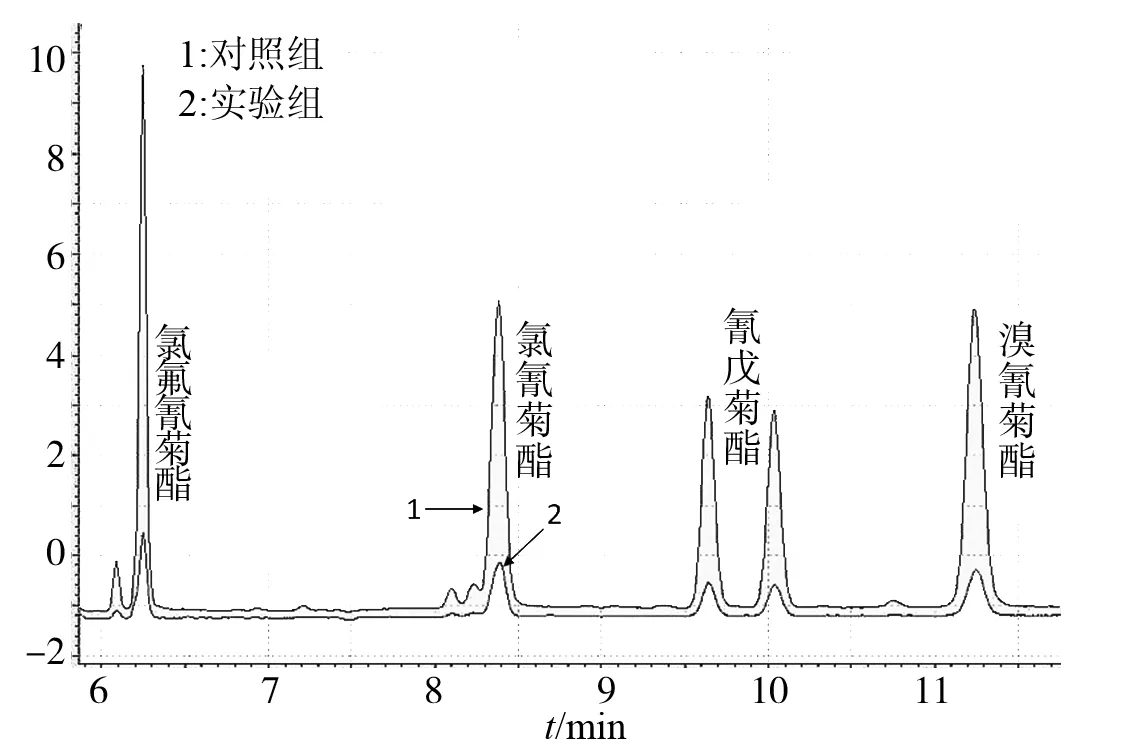
图11 气相色谱分析突变酯酶EstM46对降解拟除虫菊酯的降解能力Fig.11 Pyrethroids degradation ability analysis of mutant esterase EstM46 by gas chromatography
2.6 突变子M46的生物信息学分析
2.6.1 突变子M46碱基序列突变分析 NCBI的Blast软件(http:∥blast.ncbi.nlm.nih.gov/Blast.cgi)将M46测序结果和野生酶的碱基序列进行同源性比对,结果显示在estm46基因825 bp的碱基序列内部共发生2个碱基突变(T105A, C232G),符合低突变率的要求。
2.6.2 突变酯酶EstM46的氨基酸序列分析、结构预测和突变位点分析 利用Clustal X软件将得到的突变酶EstM46的氨基酸序列与Est825的氨基酸序列进行同源性比对,结果如图12所示,突变酯酶和野生型酯酶相比,只在78位上发生了一个氨基酸突变(由P变为A,▲所示),并未发生在Est825中保守的GXSXG五联体(方框中)氨基酸残基中。
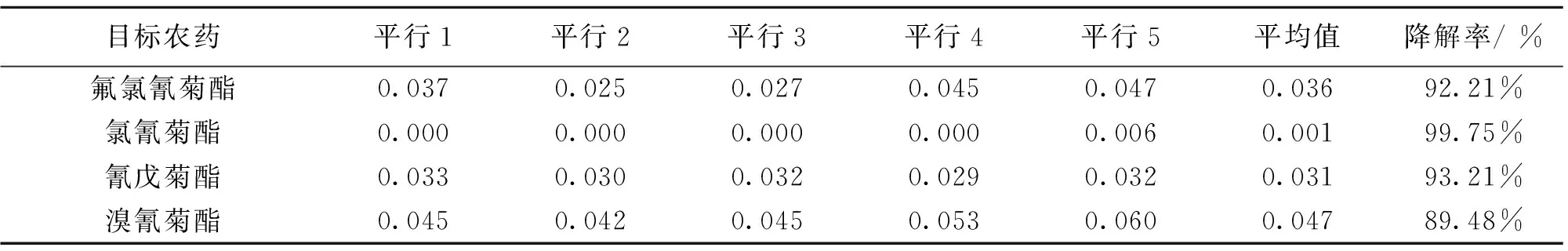
表1 突变酯酶EstM46对拟除虫菊酯的降解率Table 1 Pyrethroids degradation rate of EstM46 mg·L-1

图12 突变酯酶EstM46氨基酸序列分析Fig.12 The amino acid sequence analysis of mutant esterase EstM46
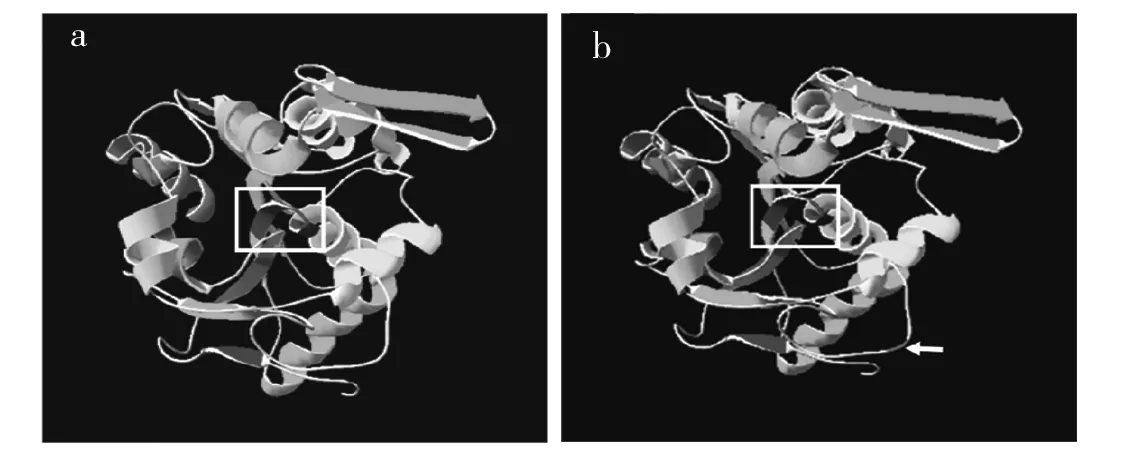
图13 野生酶与突变酶蛋白的三维结构计算机模拟图Fig.13 Ribbon representation of wild and mutanted esterase 3D structure modeled by computer modeling, with the mutated residue highlighted in red
2.6.3 突变酯酶EstM46蛋白结构的预测 采用DeepVIEW软件分析服务器反馈结果,突变酶EstM46(图13b)以单亚基蛋白的形式存在,其突变位点发生于表面的无规则卷曲中(箭头所示),与其活性中心(方框内)相距较远,且突变后的EstM46蛋白与Est825(图13a)蛋白的三维结构基本一致,主要原因可能是突变的位点并非发生在构成蛋白骨架的关键氨基酸残基上,另外,Gly是唯一不含手性碳原子的氨基酸,不具有旋光性,在蛋白质中提供的空间位阻最小,容易形成灵活的转角,而Pro的R基的环状结构,不易转动,产生转角方式的灵活性较差。因此,我们推测由Pro突变成Ala赋予了蛋白更加灵活的构象变化能力,进而导致氢键、疏水性、底物亲和力等一系列的变化,使得酶活性及热稳定性的提高。
3 讨 论
3.1 易错PCR
易错PCR技术以其相对简单、快速、廉价等优点,广泛应用于小片段基因的定向进化中,其通过不断积累小的有益突变,达到较大的突变效益,在提高酶活性,改善酶学性质和研究酶分子结构和功能的关系中,发挥重要作用[11]。本研究利用变异并缺乏修复功能的Taq酶,有效地减少突变中的热点变异,只需控制模板中酯酶基因est825量为500 ng,经过30个循环,得到的突变碱基个数介于2~4之间,并得到一个酶活性提高1.5倍的突变酶,其发生2个碱基突变,并导致一个氨基酸残基的变化,是一种行之有效的随机突变方法。
3.2 氨基酸突变对酶分子的影响机制
Morley等[12]认为随机突变中大多数酶学性质如立体选择性、底物特异性和新催化活性的变化与发生突变的氨基酸位点关系密切,突变位点与活性中心距离的远近,在酶活性、热稳定性和有机溶剂耐受性的变化中,具有同等作用。Voigt等[13]认为,低突变率的随机突变较易发生蛋白表面和环状区域上,本研究中获得酶活性提高1.5倍的突变酶,其碱基序列中发生了两个碱基转换,导致第78位氨基酸突变,对突变酶EstM46的三维结构进行模拟,突变发生在蛋白表面的环状区域中,远离活性中心位点,该突变位点并未影响酶的空间结构,这可能是由于该位点并不参与维持酶蛋白空间骨架结构。此外,突变酶的最适反应温度和热稳定性均得到提高,可能是由于突变导致酶分子表面的氢键或疏水性改变,使其在高温下保持良好的催化能力[14]。另外,本研究中采用平板显色法,在滴加X-caprylate测试液后,只需通过肉眼观察颜色变化,即可简单快速地完成对突变文库的高通量初筛;复筛时采用标准诱导培养和酶活性检测方法,提高了筛选结果的可信性。该方法不仅快速、简单、准确的完成对突变文库筛选,还减少筛选的工作量和工作强度,可以实现大规模筛选。
[1] 周刚, 丁伟. 农产品中菊酯类农药残留降解动态研究进展[J]. 安徽农业科学, 2008, 36(5):1939-1941. ZHOU Gang, DING Wei. Research progress on the pyrethroid pesticide residue degradation dynamics in farm produce [J]. Journal of Anhui Agriculture Sciences, 2008, 36( 5) : 1939- 1941.
[2] 王兆守, 刘丽花, 陈小兰, 等. 拟除虫菊酯类农药降解菌及降解酶的研究概况[J]. 微生物学通报, 2008, 35(5): 825-829. WANG Zhaoshou, LIU Lihua, CHEN Xiaolan, et al. The research survey of the degrading-bacteria and degrading-enzyme of synthetic pyrethroid insecticides [J]. Microbiology, 2008, 35(5): 825-829.
[3] LAN W, GU J, ZHANG J. Coexpression of two detoxifying pesticide-degrading enzymes in a genetically engineered bacterium [J]. International Biodeterioration & Biodegradation, 2006, 58(2): 70-76.
[4] ZHAI Y, LI K, SONG J L. Molecular cloning, purification and biochemical characterization of a novel pyrethroid-hydrolyzing carboxylesterase gene fromOchrobactrumanthropiYZ-1 [J]. Journal of Hazardous Materials, 2012, 221/222: 206-212.
[5] CORINNA D, JOACHIM W, REGINA H W. Characterization of a cypermethrin-degradingMethylobacteriumsp. strain A-1 and molecular cloning of its carboxylesterase gene [J]. Journal of Basic Microbiology, 2015, 55(11):1245-1254.
[6] MOORE J C, ARNOLD F H. Directed evolution of a para-nitrobenzyl esterase for aqueous-organic solvents [J]. Nature Biotechnology, 1996, 14 (4): 458-467.
[7] YAMADA R, HIGO T, YOSHIKAWA C, et al. Improvement of the stability and activity of the BPO-A1 haloperoxidase from Streptomyces aureofaciens by directed evolution [J]. Journal of Biotechnology, 2014, 192: 248-254.
[8] YU X W, WANG R, ZHANG M, et al. Enhanced thermostability of aRhizopuschinensislipase byinvivorecombination inPichiapastoris[J]. Microbial Cell Factories, 2012, 11: 102.
[9] FAN X J LIU X L, HUANG R, et al. Identification and characterization of a novel thermostable pyrethroid-hydrolyzing enzyme isolated through metagenomic approach [J]. Microbial Cell Factories, 2012, 11(1):1-11.
[10] 杨安钢, 毛积芳, 药立波. 生物化学与分子生物学实验技术[M]. 北京: 高等教育出版社, 2001: 255-257.
[11] PRITCHARD L, CORNE D, KELL D, et al. A general model of error-prone PCR[J]. Journal of Theoretical Biology, 2005, 4:497-509.
[12] MORLEY, KRISTA L, KAZLAUSKA S, et al. Improving enzyme properties: when are closer mutations better?[J]. Trends in Biotechnology, 2005, 23(5):231-237.
[13] VOIGT C A, MAYO S L, ARNOLD F H, et al. Computational method to reduce the search space for directed protein evolution [J]. Proceedings of the National Academy of Sciences(USA), 2001, 7:3378-3383.
[14] 胡长浩, 邹有土, 刘燕雅, 等. 随机突变对扩展青霉脂肪酶最适作用温度的改善[J]. 饲料工业, 2009, 30(6):30-32. HU Changhao, ZOU Youtu, LIU Yanya, et al. Improvement of the optimum temperature ofPenicilliumsp. lipase by random mutation [J]. Feed Industry, 2009, 30(6):30-32.
Molecular modification and degradation characteristics of a novel marine mud-derived pyrethroid hydrolase
LIU Xiaolong1,2, LIU Yuhuan2, FAN Xinjiong1,2
(1. School of Basic Medical Sciences, Anhui Medical University, Hefei 230032, China; 2. School of Life Sciences, Sun Yat-sen University, Guangzhou 510275, China)
In order to enhance the activity of pyrethroid degrading enzyme, error prone PCR technology was used to modify a novel esterase gene (est825) which was derived from marine sediment metagenomic library. A mutant enzyme EstM46 was cloned and characterized. The activity of EstM46 was 1.5-fold higher than wild type (Est825), and the optimum temperature was 5 degrees higher than that of Est825. Furthermore, the mutant enzyme stayed stable up to 70 ℃, remaining 80% (50% with Est825) of its activity after incubated at 70 ℃ for 2 h. The hydrolysis rates of cyhalothrin, cypermethrin, sumicidin and deltamethrin were 92.21%, 99.75%, 93.21% and 89.48%, respectively. A broad-spectrum pyrethroid-degrading enzyme with higher hydrolytic activity and more thermostability will play a better role in practical biodegradation.
pyrethroids; pesticide degradation enzyme; directed evolution; biological degradation
10.13471/j.cnki.acta.snus.2017.04.018
2016-09-05 基金项目:国家自然科学基金(31400680);安徽医科大学博士启动基金(XJ201322);广东省科技计划项目(2012B010300021, 2013B010404044);广东省教育项目(2013KJCX0107)
刘孝龙(1985年生),男;研究方向:生物化学与分子生物学;E-mail:liuxiaolong85@126.com
范新炯(1985年生),女;研究方向:微生物学;E-mail:fanxinjiong@126.com
Q556+.1
A
0529-6579(2017)04-0111-07

