微卫星标记分析3个耐寒鲤品种的遗传多样性
桑 滨, 鲁翠云, 李 超, 孙效文, 李丹彤
(1. 大连海洋大学 水产与生命学院, 大连 116023; 2. 中国水产科学研究院 黑龙江水产研究所, 哈尔滨 150070)
微卫星标记分析3个耐寒鲤品种的遗传多样性
桑 滨1,2, 鲁翠云2, 李 超2, 孙效文2, 李丹彤1
(1. 大连海洋大学 水产与生命学院, 大连 116023; 2. 中国水产科学研究院 黑龙江水产研究所, 哈尔滨 150070)
耐寒品种的培育与养殖对于三北地区的鲤养殖业非常重要,对育成品种进行遗传分析有助于种质资源的可持续利用。研究采用30个微卫星标记分析了3个遗传背景相近的耐寒鲤品种松荷鲤(GG,CyprinuscarpioL.)、荷包红鲤抗寒品系(HH,Cyprinuscarpiovar.wuyuanensis)与松浦鲤(SS,Cyprinuscarpiovar.Songpu)的遗传多样性。结果显示:1)3个耐寒鲤品种GG、HH和SS平均有效等位基因数为4.392、4.501和4.518,平均观测杂合度为0.801、0.815和0.809,平均多态信息含量为0.720、0.717和0.720,GG、HH和SS均属高度多态水平(PIC>0.5),3个群体偏离Hardy-Weinberg平衡的位点较少,种群结构合理。2)计算3个耐寒鲤品种间的遗传距离并绘制聚类图,结果GG和HH先聚为一个分支,再与SS聚在一起。基于Structure亚种群数检验的分析结果显示,当K=2时,HH群体从3个群体中分离出来,GG和SS群体主要遗传组分相同;当K=3时,GG、HH和SS群体相互分离开,并且各自形成自己的遗传组分。3)在26个标记中检测出品种特异等位基因73个,其中41个品种特异等位基因的频率超过10%,进而获得基因型频率大于10%的品种特异基因型161个,可用于3个耐寒鲤品种的种质鉴定。
鲤;微卫星;种质鉴定;遗传多样性
鲤(CyprinuscarpioL.)是我国具有重要经济价值的淡水性鱼类,主要分布于东北、西北和华北的“三北”地区[1]。三北地区的年平均温度低、冰封期长等地域特点决定了该地区的鲤必须要耐高寒,以东北地区的黑龙江野鲤为代表,具有极强的抗寒和抗病能力,但生长速度慢的特点限制了黑龙江野鲤的养殖和推广。为开发适合三北地区大面积推广养殖的鲤品种,黑龙江水产研究所利用杂交育种技术,将黑龙江鲤耐寒、荷包红鲤配合力高、镜鲤生长速度快等特点集中于一身,培育出3个北方鲤养殖的当家品种:荷包红鲤抗寒品系、松浦鲤和松荷鲤(高寒鲤)[2]。由于3个鲤品种种源相近,仅通过体型以及个体性状难以对其进行区分,因此,开展鲤品种的分子鉴定极为重要。另外,用于鲤品种苗种生产的亲本多采用群体中有较好经济性状的个体,可能对其他生存相关的基因型产生负向效应。已有研究报道松荷鲤和荷包红鲤抗寒品系在人工繁育过程中遗传多样性降低[3-4],松浦鲤养殖群体由于人工选择力度较大经历了瓶颈效应,环境适应能力降低[5-6],鲤养殖生产中出现的抗病力逐渐减弱和优势性状不断衰退的现象严重制约着鲤养殖业的发展。评估鲤品种的种质资源好坏最直接的方法就是群体遗传多样性分析,因此,为保证鲤养殖品种优良性状的稳定遗传,开展鲤养殖品种遗传多样性研究十分必要。
微卫星标记作为多态性丰富、重复性好的共显性分子标记,适用于亲缘关系较近的生物群体间的遗传关系研究。尽管用微卫星标记分析荷包红鲤抗寒品系、松浦鲤和高寒鲤的遗传多样性的研究较多[7-9],但是由于研究中使用标记的随机性,尚不足以揭示3个品种的遗传差异。因此,本研究从鲤微卫星标记库中(http://www.carpbase.org)筛选了30个多态性高、条带清晰并具有差异扩增条带的微卫星标记,对此3个鲤养殖品种进行了分子鉴定和遗传多样性分析,以期为鲤养殖品种的种质鉴定、科学育种和资源保护提供可靠的参考依据。
1 材料与方法
1.1 材料
实验用鱼均采自黑龙江水产研究所呼兰实验站,从保种群体中随机采集松荷鲤30尾(GG)、荷包红鲤抗寒品系30尾(HH)以及松浦鲤30尾(SS),剪取部分鳍条组织。将组织置于滤纸干燥以备提取基因组DNA[10]。
1.2 方法
1.2.1 基因组DNA的提取
用组织基因组DNA提取试剂盒(天根生物)提取基因组总DNA,紫外分光光度计(260/280)测定浓度和纯度。将提取的DNA稀释至100 ng/μL,-20℃储存备用。
1.2.2 PCR扩增与检测
从鲤微卫星标记资源库中,选择多态性较高、扩增稳定的152个微卫星标记[11-13],用6个个体进行筛选,最终选择了30个多态性较高、品种间有差异、条带清晰且扩增结果稳定的标记,用于3个鲤养殖品种的PCR扩增。微卫星引物的信息和扩增情况如表1所示。PCR反应体系为15 μL,包括终浓度为10 mmol/L Tris-Cl (pH 8.0)、1.5 mmol/L MgCl2、50 mmol/L KCl、0.3 μmol/L引物与200 μmol/L dNTP、100 ng DNA模板和1 UTaqDNA聚合酶。反应程序是94℃预变性3 min;94℃变性30 s,56℃复性30 s,72℃延伸30 s,循环25次;最后72℃延伸5 min。8%Native-PAGE对PCR扩增产物电泳检测,0.1%的AgNO3对其染色,拍照给出凝胶电泳结果。
1.2.3 数据统计与分析
采用Gel-Pro Analazer4.5软件分析每个微卫星标记电泳条带的长度,获得等位基因数据,汇总至Excel。利用Popgen32[14]软件计算等位基因数(Observed number of alleles,N)、有效等位基因数(Effective number of alleles,Ne)、观测杂合度(Observed heterozygosity,Ho)和期望杂合度(Expected heterozygosity,He)。BOTSTEIN等[15]公式计算多态信息含量(Polymorphism information content, PIC):
式中Pj、Pi分别是群体的第j和第i个等位基因的频率,n是某一个基因座位上的等位基因数。

图1 松荷鲤(GG)、荷包红鲤抗寒品系(HH)和松浦鲤(SS)在标记HLJ1148的PCR扩增结果
Hardy-Weinberg平衡偏离通过χ2检验估计,3个鲤养殖品种的Nei氏遗传距离、遗传相似系数(Genetic similarity index,I)、基因流(Nm)和群体间近交系数(FST)等遗传分化情况由PopGen32软件给出。
将Popgen32软件生成的Nei氏遗传距离带入MEGA4.0软件,利用UPGMA法构建3个鲤养殖品种的遗传关系聚类图[16]。Structure2.3.4软件[17]分析3个耐寒鲤品种的潜在亚种群数,借助遗传结构数据划分最可能亚种群数。
2 结果与分析
2.1 PCR扩增结果分析
30个微卫星标记对3个耐寒鲤品种松荷鲤(GG)、荷包红鲤抗寒品系(HH)和松浦鲤(SS)120尾样本进行PCR扩增,结果30个微卫星标记在3个群体中共扩增出724条稳定、清晰的条带,片段长度在113~396 bp,每个标记检测出等位基因数5~11个(表1)。部分Native-PAGE的结果如图1所示。26个标记检测到品种特异等位基因73个,其中41个特异等位基因频率超过10%。HLJ1093、HLJ1119、HLJ1148等6个标记扩增出GG群体特异等位基因7个,其中HLJ1148扩增出的片段大小为172 bp和160 bp、HLJ1337的220 bp的等位基因频率超过10%;HLJ385、HLJ1119、HLJ1120等6个标记扩增出HH群体特异等位基因7个,其中HLJ1120的236 bp和HLJ1148的208 bp等位基因频率超过10%;HLJ385、HLJ1113和HLJ3938扩增出SS群体特异等位基因3个,其中HLJ1113的164 bp等位基因的频率达到10%。进而获得基因型频率大于10%的品种特异基因型161个,可用于3个耐寒鲤品种的种质鉴定。各标记等位基因、特异基因型及其频率见表2,等位基因按照从大到小排列,依次以A、B、C等字母表示。

表1 30对鲤微卫星引物和PCR扩增结果

表2 3个耐寒鲤品种在30个微卫星标记的等位基因频率及特异基因型频率

(续表2Continuedtable2)标记Marker等位基因编号No.ofalleles等位基因/bpAllelesize等位基因频率Allelefrequency特异基因型Specificgenotype基因型频率GenotypefrequencyB2540.01670.18330.0834CE0.1000/0.0667C2420.21670.31670.2333CF0.10000.0667/D2320.13330.05000.2500BI/0.1667/E2280.18330.03330.1167FI0.1000//F2260.13330.0667/G2180.06660.0167/H2040.01670.01670.0333I1980.21670.28330.1833HLJ1093A2260.03330.21670.0334AA/0.1000/B2180.06670.0167/CC0.3000/0.1333C2140.56670.21670.3833AD/0.20000.0667D2100.18330.28330.3500DF/0.06670.1333E206/0.03330.0333F1940.10000.23330.1667G1900.0333/0.0333H1780.0167//HLJ1098A244/0.03330.1667BB/0.13330.0667B2240.21670.35000.2333AC//0.2000C2120.50000.21670.2333CC0.16670.0333/D2080.28330.11670.1667DE/0.1333/E200/0.13330.0833F192/0.15000.1167HLJ1113A1960.03330.16670.0333CC0.10000.0667/B1920.08330.13330.3500AD0.03330.2667/C1880.43330.23330.0833BD0.0333/0.2000D1800.21670.40000.2167DD0.03330.2000/E1720.06670.01670.0667DF//0.1000F164//0.1000G1600.16670.05000.1500HLJ1115A2320.1333/0.1500AD0.1000//B2240.0166/0.0500DE/0.13330.0667C2120.1333/0.0167CI0.1667//D2080.25000.17240.1333FI//0.1333E2040.01670.15520.1500DJ0.1000//F2000.01670.01720.1500IJ0.03330.1000/G1920.01670.05170.0334H1880.01670.0691/I1800.26670.18970.1833J1760.10000.1379/K1720.03330.10340.0833HLJ1116A3960.1167/0.1500CC0.1667/0.0333B3720.08330.18330.1167AE0.1000/0.0667C3600.36670.11670.1833BE/0.1667/D3440.13330.25000.3333EF/0.1333/E3360.25000.35000.2000F3240.05000.10000.0167HLJ1119A2060.05000.01670.0834BG0.10000.0333/B2020.10000.01670.0834EG/0.13330.0333C1900.0833/0.0333IK/0.1000/D1820.0667/0.1500E1700.10000.15000.0833F1660.0500//G1580.30000.25000.2000H1540.16670.28330.1000I150/0.10000.0833J142/0.0500/K1380.08330.13330.1833HLJ1120A3760.01670.06670.0667EI0.1667/0.1000B2960.06660.10000.0333FI/0.1000/C2760.0333/0.0833AJ/0.03330.1000D2680.0167/0.1333BK/0.1333/E2480.1833/0.1000F240/0.0667/G236/0.1000/H2120.01670.1000/I2080.60000.23330.1500J2040.06670.18330.4167K200/0.15000.0167
注:加下划线的数字为群体特有的片段大小
2.2 3个耐寒鲤品种的遗传参数分析
用Popgen32软件计算3个耐寒鲤品种松荷鲤(GG)、荷包红鲤抗寒品系(HH)和松浦鲤(SS)的遗传参数(表3)。结果发现群体平均等位基因数(N)为6.633(HH)~6.733(GG),平均有效等位基因数(Ne)为4.392(GG)~4.518(SS),平均观测杂合度(Ho)为0.801(GG)~0.815(HH),平均期望杂合度(He)为0.762(HH)~0.768(SS),平均多态信息含量(PIC)分别为0.717(HH)和0.720(GG和SS),3个品种保种群体均属于高度多态水平(PIC>0.5)[15]。对群体间遗传参数进行成对样本t检验,结果显示Ne在3个鲤群体间的差异性不显著(P>0.05);N在3个群体间的差异达到显著水平,而GG和HH、HH和SS在30个微卫星标记的等位基因数差异极显著(P<0.01);除此之外,HH和SS群体在He和PIC的差异均达到显著水平,P分别为0.000和0.017;而GG和SS群体在Ho和PIC的差异达到显著水平,P分别为0.04和0.05。
Hardy-Weinberg平衡检验结果显示,GG群体在4个标记显著偏离平衡,其中HLJ1116、HLJ1119、HLJ1120表现为杂合子极显著缺失;HH群体在10个标记显著偏离,其中HLJ1120和HLJ3938表现为杂合子显著不足,HLJ1093、HLJ1120等8个标记表现为杂合子显著过剩;SS群体在4个标记显著偏离平衡,其中HLJ1120和HLJ3415表现为杂合子不足,HLJ3473和HLJE547表现为杂合子过剩。HLJ1120在3个群体均极显著偏离平衡,表现为杂合子不足;标记HLJ3291在GG和HH群体显著偏离平衡,表现为杂合子过剩。3个群体的详细统计参数见表3。

表3 3个鲤品种在30个标记上的遗传多样性参数
(续表3 Continued table 3)

标记MarkerGGNNeHoHePICPhweHHNNeHoHePICPhweSSNNeHoHePICPhweHLJ235285.6780.7330.8380.8010.58775.590.9330.8350.7970.06785.3250.7670.8260.7880.124HLJ265665.0000.8330.8140.7710.23865.0700.8670.8160.7740.049*64.6750.9330.7990.7540.224HLJ327663.4950.7330.7260.6810.12052.3870.6000.5910.5380.000**62.4930.7330.6090.5380.954HLJ329173.7980.7670.7490.7050.016*64.3580.9670.7840.7410.040*75.1430.8670.8190.7780.121HLJ335965.1140.9330.8180.7770.15863.0880.6330.6880.6360.25853.2470.7590.7040.6390.503HLJ341074.9450.7000.8110.7720.25575.7880.8330.8410.8050.06974.8000.9330.8050.7610.601HLJ341553.8300.9330.7510.6920.41985.6430.9000.8370.7990.20285.8070.80.8420.8050.001**HLJ346653.1200.7670.6910.6260.21641.9170.6000.4860.4470.52453.9300.8670.7580.7010.677HLJ347374.1480.8330.7720.7320.51976.3000.9310.8560.8210.28184.0050.8620.7640.7200.014*HLJ359776.0810.9000.850.8140.25576.0610.8670.8490.8150.05964.8000.9330.8050.7590.269HLJ363384.5460.7670.7930.7500.86353.1920.8670.6980.6220.57064.4260.8280.7880.7400.947HLJ363963.1320.7590.6930.6430.67563.7580.8000.7460.6900.001**62.1830.4070.5520.5090.303HLJ365675.8070.9670.8420.8040.11084.6390.7330.7980.7560.57674.3800.7500.7860.7380.738HLJ366253.1970.6670.6990.6300.63552.6050.9670.6270.5420.000**52.5530.6670.6190.5360.405HLJ393885.2630.8670.8240.7860.75185.5730.8330.8350.7970.002**95.3570.8670.8270.7910.261HLJE54762.6950.8670.6400.5950.33152.2840.7330.5720.5050.20942.1430.6670.5420.4800.000**平均Mean6.7334.3920.8010.7660.7200.3946.6334.5010.8150.7620.7170.2506.74.5180.8090.7680.7200.419
注:*表示显著偏离Hardy-Weinberg平衡(P<0.05);**表示极显著偏离Hardy-Weinberg平衡(P<0.01)
2.3 3个耐寒鲤品种的遗传分化分析
3个鲤品种的群体间近交系数(FST)平均值为0.0527,表明5.27%的遗传差异来自群体间,其中HH和SS群体的遗传分化较大(FST=0.0418),而GG和HH(FST=0.0391)、GG和SS(FST=0.0394)的遗传分化相对较小。基因流(Nm)的统计结果也得出了相似的结论,HH和SS群体间的基因流动(Nm=5.7375)小于GG和HH(Nm=6.1403)以及GG和SS(Nm=6.0936)。GG、HH和SS的遗传距离(Ds)和遗传相似度(I)见表4,HH和SS间的遗传距离最远(0.300),其次为SS和GG(0.288),GG和HH间遗传距离最近(0.283)。利用UPGMA法构建的群体遗传关系聚类图如图2所示。GG和HH先聚类为一支,再与SS聚在另一支。Structure分析结果显示,当K=2时,HH群体从3个群体中分离出来,GG和SS群体主要遗传组分相同;当K=3时,GG、HH和SS群体相互分离开,并且各自形成自己的遗传组分(图3)。

表4 群体间遗传距离与遗传相似度
注:对角线下方的是遗传相似度;对角线上方的是遗传距离

图2 依据群体间的遗传距离构建的UPGMA聚类图
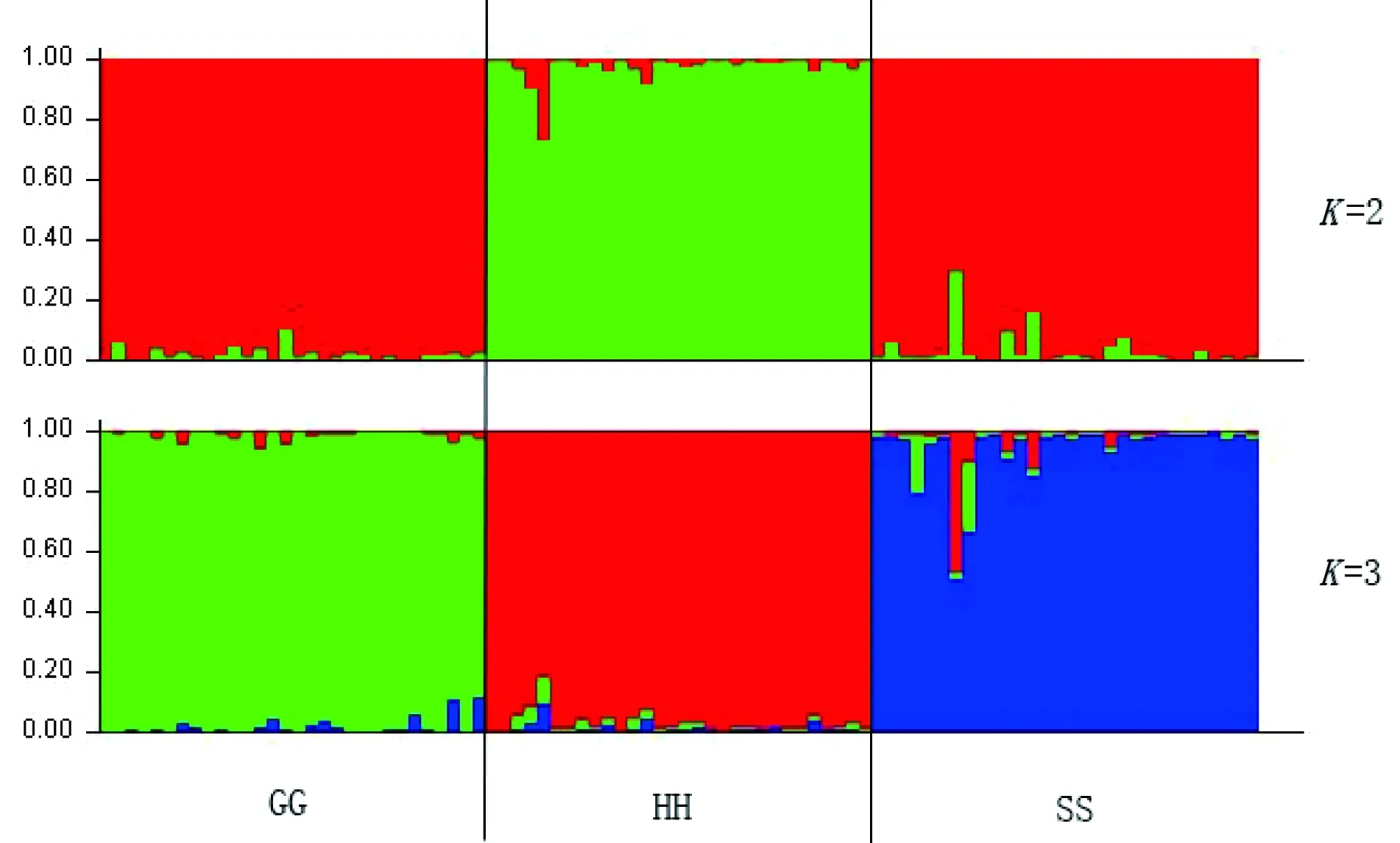
图3 利用Structure构建的耐寒鲤品种结构(显示当潜在亚种群等于2、3时种群的聚类关系)
注:纵坐标为各群体间个体占其祖先成份的百分比;每个垂直条代表一个个体,鲤鱼品种沿横坐标指示
3 讨论
根据Barker[18]的微卫星选择标准,若较准确地对群体遗传多样性进行评估每个微卫星标记的等位基因数至少为4,本研究所使用的微卫星标记等位基因数为5~11,能够较好地用于遗传多样性评估。其中26个标记在3个鲤群体扩增出特异等位基因,GG、HH、SS分别获得了7个、7个和3个特异等位基因,其中3个、2个和1个特异等位基因的频率超过10%;其余56个特异等位基因为2个品种共享而区别于第3个品种。在此基础上,获得了频率大于10%的特异基因型,可用于3个抗寒鲤品种的分子鉴定。此结果表明以相似种源杂交形成的3个鲤耐寒品种均具有了各自独特的等位基因及基因型,也说明3个品种经过独立选育,基本上形成了特异的遗传结构。
平均杂合度是衡量群体遗传多样性的重要参数,可以反映群体遗传多样性水平的高低[19]。本研究中3个耐寒鲤品种GG、HH、SS的平均期望杂合度值(0.766、0.763、0.768)均小于平均观测杂合度值(0.801、0.815、0.809),表现为杂合子过剩。本研究结果中,GG和HH的Ho(0.801、0.815)高于梁利群等[8]的研究结果(0.355、0.305)和杜长斌等[4]的研究结果(0.4、0.38),SS的Ho(0.809)也高于贾志英等[9]的研究结果(0.572),造成此结果的原因可能与筛选到的微卫星标记的高度多态性和较多的标记数量有关。根据Takezaki等[20]的研究,通过微卫星标记计算的Ho平均值应为0.3~0.8,3个耐寒鲤品种的Ho均超出此范围,可能是由两个主要原因造成的,一是3个品种均为杂交选育种,每个品种至少含有2个以上的种源;二是杂合个体通常具有较高的生产性能,在以生长和表型为目标的人工选育群体中,易于造成杂合个体的富集,使杂合度偏高。平均多态信息含量(PIC)是另一个衡量群体遗传多样性常用的参数,Botstein等[15]认为:当PIC≥0.5时,为高度多态水平。3个耐寒鲤品种GG、HH、SS在30个标记上的平均多态信息含量相对较高,依次为0.720、0.717和0.720,与梁利群等[8](GG、HH,遗传多样性丰富)和贾志英等[9](SS,较丰富的遗传多样性)结果相同。所有标记除HLJ3466(在HH群体PIC=0.447)和HLJE547(在SS群体PIC=0.480)外,在3个群体中均属高度多态位点(PIC>0.500)。这说明3个群体遗传多样性水平较高,同时也证明了以上多态位点对于3个耐寒鲤品种属于高度多态位点。本研究结果表明3个耐寒鲤品种松荷鲤、荷包红鲤抗寒品系和松浦鲤群体结构合理,具备适应变化多样环境的能力,在遗传育种和改良方面蕴藏着巨大的潜能,同时说明黑龙江水产研究所呼兰试验站以上3个耐寒鲤品种种质资源良好。
群体间近交系数值是体现群体间的遗传分化程度不可或缺的量度指标。根据Wright[21]对遗传分化指数和种群基因流系数(Nm)的界定,本研究中3个耐寒鲤品种间FST值较小(0.0527)而Nm值较大(4.4902),因此属于轻度遗传分化水平,不同鲤群体之间存在一定的基因流动,但因为存在品种间的隔离,基因流动并不频繁。群体聚类图可以更直接地反应群体间的遗传分化,荷包红鲤抗寒品系和松荷鲤可以聚类为一支,可能是因为均继承了耐高密度养殖的荷包红鲤的基因多于松浦鲤,或者松浦鲤群体选育过程中过多地继承了德国镜鲤的基因导致其单独聚类为一支。而Structure亚种群数检验结果显示,当K=2时,松荷鲤和松浦鲤遗传组分接近,这与2个品种选育过程中和黑龙江鲤回交密切相关,也可能GG、SS群体选育过程中分别与散鳞镜鲤、德国镜鲤杂交而融入镜鲤的遗传组分有关;而K=3时,3个群体均形成了各自独特的遗传组分,形成了具有不同性状优势的新品种。杂种优势的强弱由双亲优势性状是否得到互相补充决定,本研究对鲤品种进行Structure亚种群数检验的目的之一就是通过遗传组分的分析从抗寒鲤材料中筛选遗传组分差异相对较大的个体进行杂交,为抗寒鲤品种的进一步选育提供科学有效的指导。
群体遗传多样性分析作为评价品种环境适应能力的手段,对养殖品种进一步选育的指导具有重要的现实意义。本研究选取了北方地区的抗寒鲤品种(松荷鲤、荷包红鲤抗寒品系和松浦鲤)作为实验对象,用30个微卫星标记分析其遗传多样性并进行种质鉴定,发现26个标记可根据扩增得到的特异性片段大小对以上3个耐寒鲤品种进行分子鉴定,15个标记可通过获得的基因型鉴别3个耐寒鲤品种,其遗传多样性良好,种群能稳定地生存繁衍。鉴于不同微卫星标记在鲤品种间等位基因频率和基因型频率较低的特点,建议在利用特异性扩增片段和基因型进行种质鉴定的工作中可通过增加样本数、不同标记组合或者结合其他鉴定方法来达到降低风险的目的。如果3个抗寒鲤品种中可用于种质鉴定的低频率基因型在选育过程中丢失,品种间不同的优势性状可能会因此丧失,以至于种质发生退化。因此,在鲤品种的选育过程中,除了利用分子标记技术使具优势性状的基因型得到纯合之外,还应该筛选遗传组分差异比较大或者亲缘关系比较疏远的个体进行杂交以达到改善育种群体遗传结构的目的,这样才能有效避免近亲繁殖可能带来的遗传衰退。
[1]蒋高中, 李 群, 明俊超, 等. 中国古代淡水养殖鱼类苗种的来源和培育技术研究[J].南京农业大学学报(社会科学版),2012, 12(3): 88-93.
[2]石连玉, 李池陶, 葛彦龙, 等. 黑龙江水产研究所鲤育种概要[J]. 水产学杂志, 2016, 29(3): 1-8.
[3]闫华超, 高 岚, 贾少波. 鲤野生群体与养殖群体遗传多样性初步分析[J]. 水利渔业, 2006, 26(4): 21-23.
[4]杜长斌, 孙孝文, 楼允东, 等. 应用微卫星技术对野鲤和两种鲤选育品系的遗传多样性分析[J]. 上海水产大学学报, 2000, 9(4): 285-289.
[5]吕耀平,王成辉,胡则辉,等.“全红”瓯江彩鲤不同世代间的遗传结构及遗传分化初步分析[J]. 水生生物学报,2010, 34(1):65-71.
[6]单云晶, 鲁翠云, 李 超, 等. 基于线粒体COI基因序列的5种鲤养殖品种遗传多样性研究[J]. 中国水产科学, 2013, 20(5): 931-938.
[7]全迎春, 孙效文, 梁利群. 应用微卫星多态分析四个鲤鱼群体的遗传多样性[J]. 动物学研究, 2005, 26(6): 595-602.
[8]梁利群, 冯国军, 常玉梅, 等. 鲤野生和养殖群体遗传多样性分析中的微卫星(SSLP)分析[J]. 淡水渔业, 2007, 22(2): 38-40.
[9]贾智英, 李 飞, 李池陶, 等. 松浦鲤保种群体遗传结构研究[J]. 华北农学报, 2011, 26(S1): 29-34.
[10]李 超, 鲁翠云, 郑先虎, 等. 一种保存鱼类鳍条的便捷方法[J]. 水产学杂志, 2014, 27(1): 22-24.
[11]孙效文, 贾智英, 魏东旺, 等. 磁珠富集法与小片段克隆法筛选鲤微卫星的比较研究[J]. 中国水产科学, 2005, 12(2): 126-132.
[12]鲁翠云, 毛瑞鑫, 李 鸥, 等. 鲤鱼三、四核苷酸重复微卫星座位的筛选及特征分析[J]. 农业生物技术学报, 2009, 17(6): 979-987.
[13]ZHANG X, ZHANG Y, ZHENG X, et al. A consensus linkage map provides insights on genome character and evolution in common carp (CyprinuscarpioL.) [J]. Marine Biotechnology, 2013, 15(3): 275-312.
[14]YEH F C, BOYLE T J B. Population genetic analysis of codominant and dominant markers and quantitative traits[J]. Belgian Journal of Botany, 1997, 129: 157.
[15]BOTSTEIN D, WHITE R L, SKOLNICK M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics, 1980, 32(3): 314-331.
[16]SAITOU N, NEI M. The neighbor-joining method: a new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution, 1987, 4(4): 406-425.
[17]PRITCHARD J K, STEPHENS M, DONNELLY P. Inference of population structure using multilocus genotype data[J]. Genetics, 2000, 155(2):945-959.
[18]BARKERJ S F. A global protocol for determining genetic distances among domestic livestock breeds. [C]. //World Congress on Genetics Applied To Livestock Production, 1994, 21: 501-508.
[19]GUO Z H, FU K X, ZHANG X Q, et al. SSRs transferability and genetic diversity of three allogamous ryegrass species.[J].C R Biol, 2016, 339(2): 60-67.
[20]TAKEZAKI N, NEI M. Genetic distances and reconstruction of phylogenetic trees from microsatellite DNA[J]. Genetics, 1996, 144(1): 389-399.
[21]WRIGHT S. Variability within and among natural populations[M]. Chicago: University of Chicago Press, 1978, 4:439-459.
Genetic diversity of three cold-resistant varieties of common carp analyzed by microsatellite markers
SANG Bin1,2, LU Cui-yun2, LI Chao2, SUN Xiao-wen2, LI Dan-tong1
(1. College of Fisheries and Life, Dalian Ocean University, Dalian 116023; 2. Heilongjiang Fisheries Research Institute, Chinese Academy of Fishery Sciences, Harbin 150070, China)
Cultivation and breeding of cold resistant varieties are very important for carp farming in the Three-North Region, meanwhile, genetic analysis of cultivars is helpful to the sustainable utilization of the germplasm resources.Comparative analysis of genetic diversity was conducted using microsatellite markers on three cold-resistant varieties of common carp with similar genetic background,CyprinuscarpioL. (GG),Cyprinuscarpiovar.wuyuanensis(HH), andCyprinuscarpiovar.Songpu(SS).The results showed as following: (1) The average effective number of alleles of three cold-resistant varieties of common carp (GG, HH, SS) were 4.392, 4.501 and 4.518, respectively. Average observed heterozygosities were 0.801, 0.815 and 0.809, respectively. The average polymorphism information contents were 0.720 and 0.717, 0.720, respectively. Three cold-resistant varieties of common carp were of highly polymorphic level. Three populations deviated from the Hardy-Weinberg equilibrium and the population structure were reasonable. (2) A tree diagram was constructed by clustering analysis according to genetic distance of three breeding carp. The results indicated that SS clustering was a separate branch firstly, followed by GG and HH. In accordance with the results of Structure analysis, HH separated as a group alone, GG and SS clustered together for the modelK=2.Whereas GG, HH, and SS were clustered into 3 different groups for the modelK=3.(3)seventy-three specific alleles were detected from 26 markers, of which 41 specific alleles frequency reached more than 10%. There were 161 specific genotypes with genotypic frequencies over 10%, which can be used for germplasm identification of 3 varieties of cold resistant carp. The purpose of this study was to provide a beneficial reference bases for the breeding and germplasm identification of common carp.
common carp; microsatellite; germplasm identification; genetic diversity
2016-05-09;
2016-08-02
农业部“948”项目(2016X15);国家水产种质资源平台项目(2016DKA30470)
桑 滨, 硕士, 主要从事水产生物技术研究,E-mail:BinSang2016@163.com
李丹彤, 教授, 主要从事海洋生物技术的研究,E-mail:lidantong@dlou.edu.cn
Q75;Q959.46+8
A
2095-1736(2017)03-0024-09
doi∶10.3969/j.issn.2095-1736.2017.03.024

