CRISPR/Cas9系统对斑马鱼fhl1a基因敲除有效性的研究
陈思行+陈发+蔡湾湾+吴燕+廖艳伟+邓云+吴秀山+王跃群


摘 要 为研究fhl1a基因在心脏发育中的作用,利用CRISPR/Cas9系统敲除斑马鱼fhl1a基因.在斑马鱼fhl1a基因的三号外显子处选取打靶位点,构建sgRNA表达载体.将体外转录的sgRNA和Cas9 mRNA混合物分别显微注射到斑马鱼单细胞期的受精卵中以期实现对目的靶基因fhl1a的敲除.随后收集部分72 h胚胎进行PCR检测和产物直接测序,确定有无突变.为进一步验证突变是否可稳定遗传,选择具有敲除了的F0与野生型斑马鱼进行外交,对获得的F1进行逐一剪尾,提取DNA进行PCR和测序分析.结果显示F1获得了不同程度的插入、缺失等突变类型.研究结果证明所设计的fhl1a基因CRISPR/Cas9敲除系统能在斑马鱼体内有效地形成基因突变,该突变能稳定地遗传到下一代个体,这为进一步研究fhl1a基因在心脏发育中的具体调控机制打下了基础.
关键词 CRISPR/Cas9;fhl1a基因;斑马鱼;敲除
中图分类号 Q812 文献标识码 A 文章编号 1000-2537(2017)03-0021-06
The Validation Study of Zebrafish Fhl1a Gene Knockout by CRISPR/Cas9 System
CHEN Si-xing, CHEN Fa, CAI Wan-wan, WU Yan, LIAO Yan-wei, DENG Yun, WU Xiu-shan, WANG Yue-qun*
(The Center for Heart Development, Key lab of MOE for Department Biology and Protein Chemistry, Hunan Normal University, Changsha 410081, China)
Abstract To study the function of the fhl1a gene in heart development, we generated the fhl1a gene knockout zebrafish using the CRISPR/Cas9 system. At first, we constructed the sgRNA expression vector targeting zebrafish fhl1a gene at the third exon. After in vitro transcription of sgRNA and Cas9 vectors, we co-injected them into single-cell fertilized zebrafish eggs to generate zebrafish with the targeted mutation. After 72 h of injection, embryos were collected, the genome DNA was extracted, PCR and sequencing were then performed. To further verify the stability of genetic mutations, we selected mutated founder to outcross with wild type zebrafish. The first generation (F1) was then analyzed by PCR and sequencing of the caudal fin. Our results confirmed that insertion and deletion mutations were able to be introduced into F1 using this method. Our study demonstrated that CRISPR/Cas9-induced fhl1a gene mutations can be successfully transmitted through the gremline, which lays the foundation for future studies on gene fhl1as specific regulatory mechanism during heart development.
Key words CRISPR/Cas9;fhl1a gene;zebrafish;knockout
斑馬鱼因其成年鱼个体小、易于饲养、发育快速、性成熟期短、繁殖力强、体外受精、胚胎在体外发育并且透明等特点成为研究生物发育过程非常受青睐的模式生物[1-2].
FHL家族因含有四个半LIM结构域而得名.它们通过LIM结构域与其他蛋白质相互作用,在细胞增殖、肿瘤、骨骼肌疾病以及基因的转录调节中发挥重要作用,但其是否调控心脏的发育还有待研究.fhl1a (four and a half LIM domains 1a)基因编码产物是斑马鱼心脏组织cDNA中的一个LIM结构域蛋白,与人FHL1第五亚型高度同源[3-5],因此建立fhl1a基因敲除斑马鱼,对于研究fhl1a基因与心脏发育的关系具有重要意义.
CRISPR/Cas9 是最近发展起来的一种新型基因定向编辑技术[6-10],CRISPR/Cas9系统是细菌的一套免疫系统,依据基因的多样性,CRISPR/Cas系统被分为1,2和3 型,其中2型来源的Cas9蛋白表现出非常强的DNA裂解活性,可仅在一种非编码RNA(sgRNA)介导下实现定向基因打靶[11-14].本研究利用CRISPR/Cas9系统对斑马鱼的fhl1a基因进行敲除,从而获得能稳定遗传的fhl1a敲除个体.
1 材料与方法
1.1 实验材料
1.1.1 实验动物 AB品系斑马鱼购于武汉水生所,饲养在湖南师范大学心脏发育中心自动循环水养殖系统中,光暗周期比为14 h∶10 h.取健康、性成熟的斑马鱼,于饲养系统关灯前将雌雄按1∶1的比例放入产卵缸内.次日给光后拿掉挡板,让其完成产卵与受精.
1.1.2 实验质粒 gRNA 骨架载体为p42250;Cas9的质粒为PH-Cas9.
1.1.3 实验主要试剂 T4 DNA 连接酶和10 kb Marker购自TaKaRa公司;BsaⅠ及XbaⅠ 内切酶购自Promega 公司; 氨苄青霉素( ampicilin) 及寡核苷酸(oligo)购自上海生工公司; 琼脂糖购自BBI有限公司; PCR 产物纯化试剂盒购自康为世纪有限公司; Gel Extraction kit 和Plasmid Mini Kit 购自Omega公司.
1.2 实验方法
1.2.1 基因靶位点的设计 用Esembl(http://www.ensembl.org/index.html)网站获得斑马鱼目标基因的完整序列、该基因组上的位置、基因的转录本、编码序列、编码蛋白、功能域和结构域等信息;用NCBI(http://www.ncbi.nlm.nih.gov/)网站对目的基因的信息进行核实与补充.CRISPR/Cas9系统中gRNA靶位点设计需要遵循如下原则:靶位点处以18~20个碱基为最佳;PAM区其序列必须严格要求为NGG(N可以为任意碱基);所选靶位点位置一定要在该基因编码蛋白的功能结构域内部或之前的位置所对应的核苷酸序列,或是尽量靠前的位置,一般来讲,靶位点要在该基因CDS区前2/3区域的外显子中,起始密码子之后,同时注意避免首个起始密码子下游还存在其他拥有相同读码框的起始密码子.如果该基因编码多种蛋白,所选靶位点应尽量能同时破坏其所有编码蛋白的结构域.另外,靶位点也可选在外显子和内含子交界处,也可能破坏基因的剪接.靶位点处的碱基序列最好具有一定的复杂性,没有过多的连续重复.符合这些要求的靶位点需要在NCBI中斑马鱼全基因组上进blast, 以确定其单一性和减少脱靶效应.其中PAM区以及靠近PAM区部分的序列较为重要,比较不同靶位点各种参数后择优使用.
1.2.2 gRNA的制备 将质粒p42250 通过BsaⅠ进行酶切( 37 ℃水浴2 h).酶切产物经过琼脂糖凝胶电泳回收后作为PCR的模板.以公司合成的fhl1a-oligo-1,fhl1a-oligo-2引物(见表1)分别作为F端引物,序列(AAGCACCGACTCGGTGCACT)作为R端引物,退火温度54 ℃,延伸时间30 s,高保真酶进行PCR扩增,之后用试剂盒纯化PCR产物作为模板,T7启动子进行体外转录,体外转录产物使用RNA纯化試剂盒进行纯化回收,1%琼脂糖电泳检测以及RNA浓度测定之后置于-80 ℃冰箱待用.
1.2.3 Cas9 mRNA的制备 将质粒h-Cas9 通过XbaⅠ进行酶切( 37 ℃水浴2 h),使之线性化.之后用DNA纯化试剂盒进行回收溶于RNase Free 的ddH2O中作为模板,参照T7 Ultra Kit (Ambion,AM1345) 使用说明书进行体外加帽转录.体外转录产物使用RNA纯化试剂盒进行纯化回收,1%琼脂糖电泳检测以及RNA浓度测定之后置于-80 ℃冰箱待用.
1.2.4 靶位点有效性检验 按照约300 pg Cas9 mRNA和30 pg sgRNA剂量混合注射到斑马鱼胚胎一细胞期的单细胞中,注射后的胚胎培养72 h后,收集部分胚胎进行检测,并用野生型胚胎作为对照组.收集好的胚胎经过裂解提基因组DNA,用引物fhl1a-PCR-F(AATCCACCACTGTTCTGTT)和fh1a-PCR-R(GGAGTACAAGCATAAAGTC)进行PCR扩增,然后T7E1酶进行酶切检测.
1.2.5 突变个体筛选 待有效性检测为阳性的那一批斑马鱼长成至三个月后进行单个个体剪尾提取基因组测序.若测序结果显示有双峰即表明有碱基的插入或缺失,再将测序有双峰的样品与PMD18-T克隆载体进行连接,进行转化后挑取10个单克隆进行PCR后再测序,根据BLAST结果判断靶位点具体碱基突变情况.
2 结果
2.1 gRNA靶位点的确定及打靶载体的构建
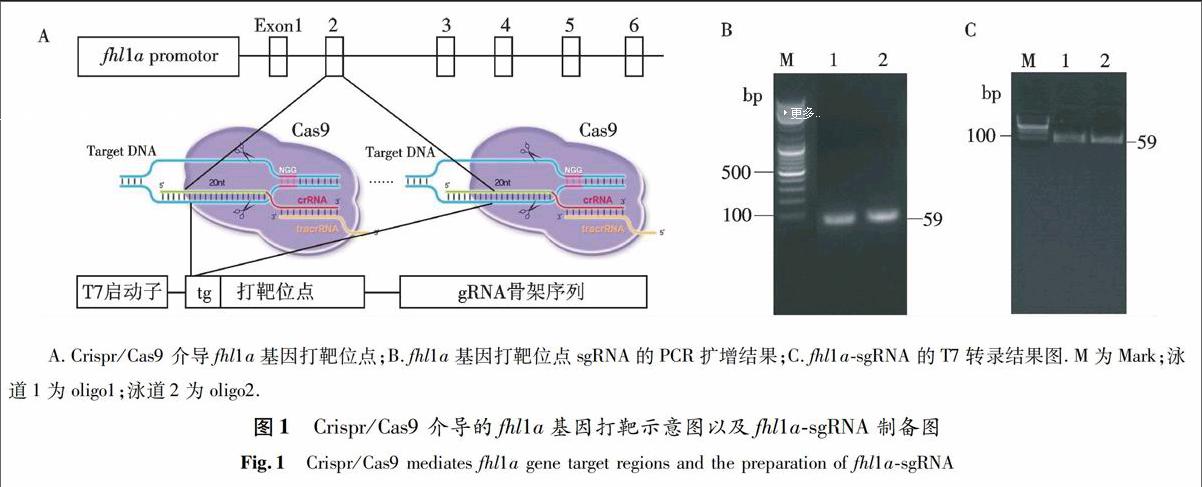
按照1.2.1所述方法择优选取两个靶位点,在设计好的靶位点序列之前加上保护碱基tg和T7启动子,靶位点之后加上gRNA骨架上游序列作为正向引物,gRNA骨架的下游序列为反向引物进行扩增, fhl1a-PRC-F/R 为鉴定基因型所用扩增引物序列(见表1、图1A和1B).
2.2 sgRNA活性检测及突变个体的筛选
按照约300 pg Cas9 mRNA和30 pg sgRNA剂量混合注射到斑马鱼胚胎一细胞期的单细胞中,并对注射情况以及鱼卵存活状态进行统计分析见表2所示.
待注射胚胎培养到72 h,随机提取部分斑马鱼全胚胎基因组,5个胚胎为一组,共6组进行打靶位点的有效性检测.若此打靶位点有效则继续让斑马鱼生长至两个月大剪尾提取基因组筛选突变个体.
按照1.2.4所述方法将6组注射fhl1a-Cas9-sgRNA的72 h胚胎提取基因组标号为1~6号以及1管野生型胚胎作为对照组,之后用T7E1酶进行酶切,结果见图2.
将注射后检测有效的胚胎养至3个月龄左右即可筛选F0代阳性个体,通过剪尾鳍方法进行基因组鉴定(方法见1.2.5),其中fhl1a-Cas9-sgRNA-oligo1和fhl1a-Cas9-sgRNA-oligo1共注射后长大的成鱼46条,分别进行剪尾、提基因组进行扩增,目的条带大小为651 bp,PCR扩增产物测序,部分测序结果见图3.
由图3可知,fhl1a基因F0代胚胎的测序峰图中靶位点以及之后序列的峰图出现套峰,说明fhl1a基因的F0代存在突变.再将测序有双峰的样品与PMD18-T克隆载体进行连接,转化后挑取10个单克隆PCR后再测序,根据BLAST结果判断靶位点具体碱基突变位点,测序结果见图4.黑框表示为靶点;横线为缺失片段(-);N为出现同种突变位点斑马鱼个体数.
2.3 稳定性遗传的检测
为进一步验证该技术造成的缺失是否可以稳定传代,笔者将这些F0代阳性嵌合体斑马鱼与野生型雄鱼杂交得到F1代,并培养至性成熟期.对F1代成鱼剪尾鳍,提取基因组DNA,进行PCR扩增测序,测序结果有双峰的进行TA克隆,将单克隆测序,结果显示有3种突变类型的斑马鱼获得了能稳定遗传的突变(见表3).
综上,本文选择F1-fhl1a-Cas9-sgRNA的1号缺失7 bp个体,F1-fhl1a-Cas9-sgRNA的2号缺失8 bp个体以及F1-fhl1a-Cas9-sgRNA的3号缺失16 bp的这三种突变F1代来建立稳定的纯合子敲除品系,并用这三个fhl1a突变品系斑马鱼进行斑马鱼fhl1a基因在心脏发育中的功能研究.
3 讨论
CRISPR/Cas9系统相较于ZFNs[15]和TALENs[16]两种更简单方便,较短的sgRNA序列也避免了超长、高度重复的TALENs编码载体带来的并发症,而且CRISPRs比TALENs和ZFNs更容易操作,因为每一对TALENs都需要重新合成,而用于 CRISPR的sgRNA只需要替换20个核苷酸就行,因此相较于TALENs和ZFNs具有更好的应用前景.
本研究在sgRNA靶点的筛选中,选择了位于fhl1a第三号外显子区域的位置.笔者选取了两个fhl1a基因的打靶位点并进行了共注射,目的是希望得到大片段缺失的fhl1a基因的突变体.但是后來检测中发现可能只有第二个靶位点发挥了功能,因为笔者得到的突变体都位于2号靶位点附近,1号靶位点在这里并没有起作用.
注射fhl1a-Cas9-sgRNA后的斑马鱼F0代属于嵌合体,这就意味着并不是所有的细胞都发生了基因突变,若突变没有发生在生殖细胞中则不会产生遗传.因此笔者在F0代中筛选到了4种fhl1a敲除类型,而到F1代中只有3种敲除类型得到了遗传,说明10个碱基缺失的斑马鱼的生殖细胞并没有发生突变.F1代为生殖细胞遗传,不存在嵌合体现象,为杂合子,所以可用F1代 fhl1a-Cas9-sgRNA-1,2,3号三个品系来继续建立稳定敲除系.fhl1a基因敲除斑马鱼的成功构建为进一步研究fhl1a基因在心脏发育中的功能提供了研究材料.
参考文献:
[1] 贾顺姬,孟安明.中国斑马鱼研究发展历程及现状[J].遗传,2012,34(9):1082-1088.
[2] GARCIA G R, NOYES P D, TANGUAY R L, et al. Advancements in zebrafish applications for 21st century toxicology[J]. Pharmacol Ther, 2016,161(2):11-21.
[3] PEN A E, NYEGAARD M, FANG M, et al. A novel single nucleotide splice site mutation in FHL1 confirms an Emery-Dreifuss plus phenotype with pulmonary artery hypoplasia and facial dysmorphology[J]. Eur J Med Genet, 2015,58(4):222-229.
[4] DOMENIGHETTI A A, CHU P H, WU T, et al. Loss of FHL1 induces an age-dependent skeletal muscle myopathy associated with myofibrillar and intermyofibrillar disorganization in mice[J]. Hum Mol Genet, 2014,23(1):209-225.
[5] MALFATTI E L, OLIV M, TARATUTO A L, et al. Skeletal muscle biopsy analysis in reducing body myopathy and other FHL1-related disorders[J]. Neuropathol Exp Neurol, 2013,72(9):833-845.
[6] CONG L, RAN F A, COX D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013,339(6121):819-823.
[7] GASIUNAS G, BARRANGOU R, HORVATH P, et al. Cas9-crRNAribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria[J]. Proc Natl Acad Sci, 2012,109(39):E2579-86.
[8] JINEK M, CHYLINSKI K, FONFARA I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptivebacterial immunity[J]. Science, 2012,337(6096):816-821.
[9] MALI P, YANG L, ESVELT K M, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013,339(6121):823-826.
[10] GAGNON J A, VALEN E, THYME S B, et al. Efficient mutagenesis by Cas9 protein-mediated oligonucleotide insertion and large-scale assessment of single-guide RNAs[J]. PLoS One, 2014,9(5):e98186.
[11] BOLOTIN A, QUINQUIS B, SOROKIN A, et al. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin[J]. Microbiology, 2005,151(8):2551-2561.
[12] MOJICA F J, DIEZ-VILLASENOR C, GARCIA-MARTINEZ J, et al. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements[J]. J Mol Evol, 2005,60(2):174-182.
[13] POURCEL C, SALVIGNOL G, VERGNAUD G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies[J]. Microbiology, 2005,151(3):653-663.
[14] BARRANGOU R, FREMAUX C, DEVEAU H, et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science, 2007,315(5819):1709-1712.
[15] CADE L, REYOND D, HWANG W Y, et al. Highly efficient generation of heritable zebrafish gene mutations using homoand heterodimeric TALENs[J]. Nucl Acids Res,2012,40(16):8001-8010.
[16] ZU Y, TONG X J, WANG Z X, et al. TALEN-mediated precise genome modification by homologous recombination in zebrafish[J].Nat Methods, 2013, 10(4):329-331.

