Hox基因及其在软体动物中的研究进展*
王 师, 杨志辉, 李语丽, 程陶然, 刘平平, 米 雪, 包振民,3
(1.中国海洋大学海洋生命学院,海洋生物遗传学与育种教育部重点实验室,山东 青岛 266003;2.海洋生物学与生物技术功能实验室,青岛海洋科学与技术国家实验室,山东 青岛266237;3.海洋渔业科学与食物产出过程功能实验室,青岛海洋科学与技术国家实验室,山东 青岛266237)
综 述
Hox基因及其在软体动物中的研究进展*
王 师1,2, 杨志辉1, 李语丽1, 程陶然1, 刘平平1, 米 雪1, 包振民1,3
(1.中国海洋大学海洋生命学院,海洋生物遗传学与育种教育部重点实验室,山东 青岛 266003;2.海洋生物学与生物技术功能实验室,青岛海洋科学与技术国家实验室,山东 青岛266237;3.海洋渔业科学与食物产出过程功能实验室,青岛海洋科学与技术国家实验室,山东 青岛266237)
Hox基因是动物基因组内高度保守的一类转录因子,在动物体轴形成中起着重要的作用,是一类重要的发育调控基因。动物形态的进化和多样性与Hox基因数目、序列及其表达模式密切相关,Hox基因在模式生物系统中得到了广泛的研究,但对于软体动物中Hox基因及其功能的了解还相对较少。软体动物具有高度的躯体结构多样性,研究软体动物的Hox基因及其功能对于理解和解释其形态多样性的产生具有重要意义。本文主要综述了Hox基因的研究概况、软体动物中Hox基因的分布、功能及其最新研究进展,以期为理解及深入研究软体动物的发育及其分子调控机制提供重要的参考。
Hox基因;软体动物;形态多样性;发育
1 Hox基因研究概况
1.1Hox基因的结构和功能
同源异型盒(Homeobox)最初被描述为一段保守的DNA模序,含有大约180个碱基并编码大约60个氨基酸的同源异型结构域(Homeodomian,HD)[1]。HD蛋白具有螺旋-转角-螺旋(Helix-turn-helix,HTH)的超二级结构。它的3个α-螺旋通过短环结构相连,N-末端的2个螺旋反向平行,C-末端的螺旋则与前2个螺旋所在的轴相互垂直。C-末端的螺旋可通过氢键和疏水作用在DNA的大沟处发生结合反应,N-末端则与DNA的小沟接触起稳定结合的作用[2](见图1)。含有这样的同源异型盒的基因被称为同源异型盒基因(Homeobox gene),大部分的同源异型盒基因能够编码一类转录因子,通过与特异序列的DNA结合和与蛋白辅因子的相互作用来调节其它基因的表达,从而影响细胞行为和活动的改变。同源异形盒基因几乎存在于所有的真核生物中,在动物中同源异形盒基因分为11个组100多个家族,植物中分为10~14个组[3]。其中有几类同源异型盒基因在染色体上成簇存在,并且高度保守,Hox基因就是其中最为大家熟知的一种。
Hox基因的发现最早始于Lewis[4]对果蝇同源异型突变(Homeotic mutation)的研究,果蝇中含有2组同源异型选择基因:ANT-C(Antennapedia complex)和BX-C(Bithorax complex)。ANT-C基因簇包括labial(Lab),proboscipedia(Pb),Deformed(Dfd),Sex combs reduced(Scr),Antennapedia(Antp);BX-C 基因簇包括Ultrabithorax(Ubx),Abdominal-A (Abd-A)和Abdominal-B(Abd-B)等基因。Bürglin[1]将这2个基因簇合称为同源异型基因复合物(Homeotic gene complex ,HOM-C),在果蝇中含有1个这样的基因簇,而在哺乳类动物中含有4个这样的基因簇,每个基因簇都位于不同的染色体上[5]。将具有高度序列相似性的Hox基因分为一组并称作旁系同源组(Paralogy group, PG)。随着对Hox基因的广泛研究,许多不同门类的后生动物的Hox基因得到了鉴定。1984年Carrascco等[6]从蟾蜍中分离得到了第一个脊椎动物Hox基因,这是进化发育生物学中一个里程碑式的发现。
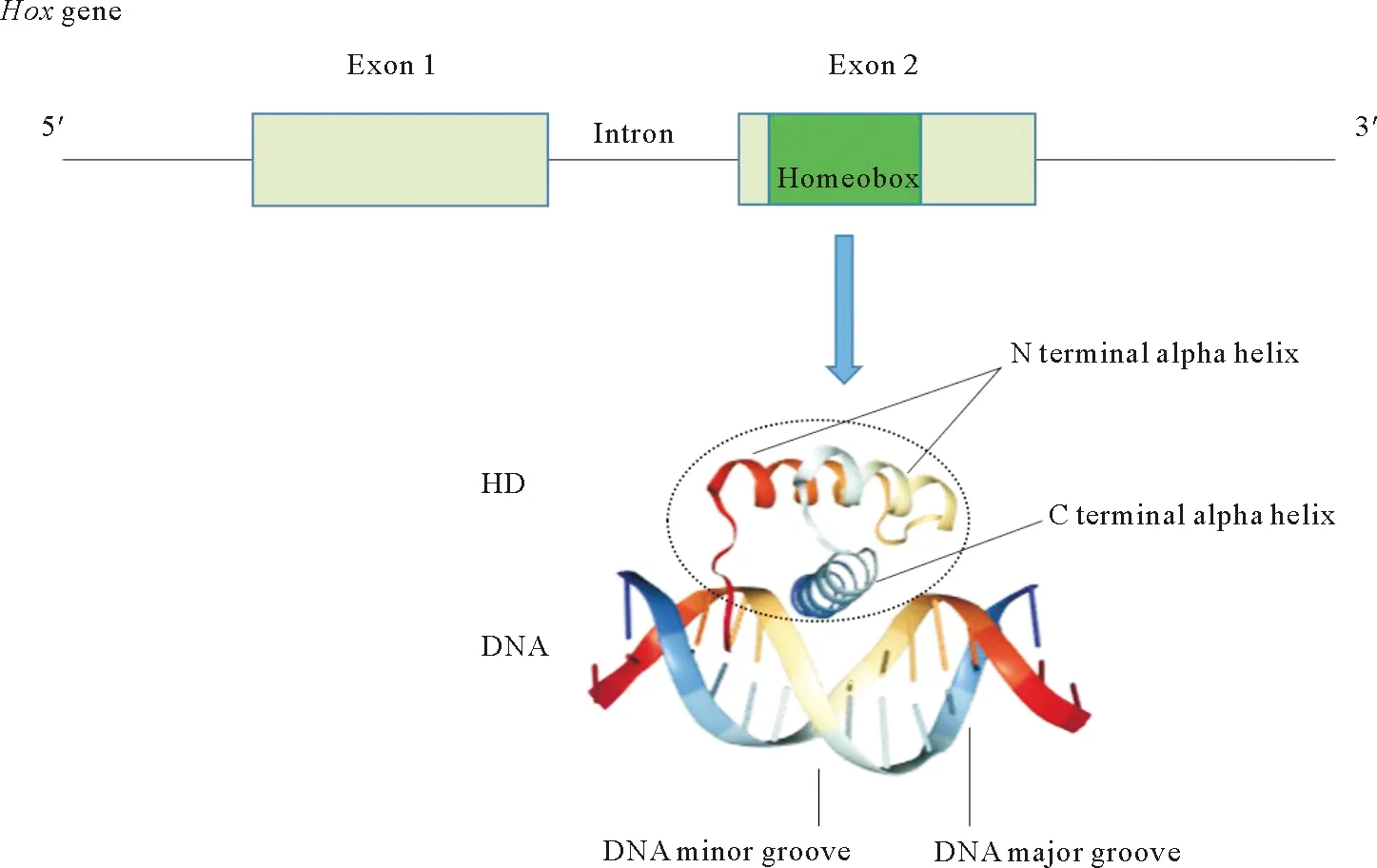
(Exon1:外显子1;Exon2:外显子2;Intron:内含子;DNA major groove:大沟;DNA minor groove:小沟;N terminal alpha helix:氮末端ɑ螺旋;C terminal alpha helix:碳末端ɑ螺旋)
Hox基因是生物体中一类重要的发育调控基因,它参与调控体轴[7]、肢体[8]和神经系统[9]等的发育。胚胎发育时期,Hox基因的表达决定着特定区域细胞将来的发育命运,例如发育成肢体或其他的身体结构。在脊椎动物肢体的发育过程中,HoxA、HoxD和HoxA/HoxD基因簇的的缺失会导致肢体的发育缺陷[8]。而HoxA和HoxD基因簇的同时缺失将导致严重的肢体发育缺陷,即肢体的发育在早期就会停滞[10]。
1.2Hox基因进化假说
Hox基因在动物身体前后轴形成中起着关键性作用,根据Hox基因在基因簇上的位置以及它们沿体轴的表达位置,可将Hox基因分为4组:Anterior,Group3,Central和Posterior[11-13]。虽然Hox基因的起源还不确定,但原口动物和后口动物最后的共同祖先被认为含有7~9个基因组成的单一Hox基因簇。原口动物的几个类群Hox基因簇扩张到8个或9个基因[14],而脊索动物则扩张到14个基因[15]。基因簇的复制以及部分基因的丢失形成了现在哺乳动物中的39个Hox基因,并分布于4个基因簇中。
Hox基因簇的一个姐妹基因簇ParaHox基因簇由Brooke等[16]在文昌鱼中发现并命名。ParaHox基因簇由Gsx,Xlox(Pdx),和Cdx(cad)这3个基因组成。它们的序列与Hox基因相似,是Hox基因的旁系同源基因。虽然ParaHox基因簇在文昌鱼中表现出保守性[16-17],但保守性不如Hox基因簇高[1]。
Mendivil Ramos等[19]认为在海绵动物出现之前Hox/ParaHox基因已经进行了复制,因此所有现存的动物的最后共同祖先拥有2个基因,即1个Hox基因和1个ParaHox基因。而对于双侧对称动物共同的祖先Hox基因簇即ProtoHox基因簇,Brooke[16]等则认为ProtoHox基因簇由一个anterior基因和一个posterior组成(二基因模型),它的复制产生了Hox和ParaHox基因。为了解释基因簇的起源问题,除了二基因模型,Garcia-Fernàndez[18]还讨论了三基因模型(Anterior,Group3和Posterior)和四基因模型(Anterior,Group3,Central和Posterior)(见图2)。
Hox基因和ParaHox基因都属于ANTP基因家族(ANTPclass),该基因家族还包括NK基因、NK-related基因、Dlx基因和其它基因。Garcia-Fernàndez[18]和 Holland[3]认为ANTPclass最初可能以一个单一的基因簇形式存在,即ANTP-class Magacluster,在后生动物进化早期该基因簇中的基础基因ANTP通过串联重复产生了ProtoHox基因和NK基因。
1.3Hox基因的表达特性
1.3.1 时空共线性Hox基因的表达遵循空间共线性[20],Hox基因的表达顺序和它们在基因簇上的前后位置一致:靠近5’端的基因主要调控体轴远端的发育,而靠近3’端的基因则调控体轴近端的发育。在一些动物类群中,主要为脊椎动物,Hox基因的表达还遵循时间共线性:前端基因先于后端基因表达[21-22]。
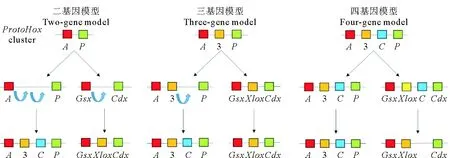
(A:前区;3:3区;C:中区;P:后区。曲箭头表示串联复制;分向箭头表示ProtoHox基因簇的复制。A:Anterior;3:Group 3;C:Central;P:Posterior.Curve arrows indicate cis-duplication events; divergent arrows indicate duplication of the ProtoHox cluster.)
1.3.2 定性调节作用和剂量效应 在果蝇中Hox基因对其体轴发育的调控表现出定性调节作用,例如,在肢体成虫原基中,Ubx和Abd-A基因通过抑制肢体形成相关基因Distalless的功能来阻止果蝇在腹部长出肢体[23]。而在脊椎动物中除了观察到上述定性调节作用外,还有剂量效应[24],例如,Horan[25]等在研究Hox4旁系同源基因突变对脊椎骨的影响时发现,Hox4旁系同源基因突变数越多对脊椎骨形态偏移的影响越大。
1.3.3Hox编码和后端优势模型Hox基因具有调节脊椎动物中轴骨形成的功能。Kessel和Gruss[26]在研究鼠类脊椎骨的同源异型转化现象时提出了“Hoxcode”(Hox编码)模型,该模型认为体节形态是由多个Hox基因的蛋白产物共同作用的结果,某些Hox基因的突变将导致这些Hox基因整个控制区域表型的改变(见图3A、B)。为了解释Hox基因对脊椎动物肢体形成的调控作用,Duboule[27]提出了“posterior prevalence”(后端优势)模型。该模型认为在胚胎特定区域表达的最后端的Hox基因对更前端的Hox基因表现出显性作用,这种作用又叫做Hox基因的表型抑制(见图3C、D)。在大多数情况下,相对于Hox编码模型,后端优势模型能更好的解释Hox基因突变导致的表型变化。

(图A和C用Hox code和Posterior prevalence这2种简单的模型来表示3个Hox基因(Hox I、Hox II、Hox III)在一个假设的体轴上对其表型的影响,每个Hox基因都有各自的表达区域。图B和图D表示Hox II基因功能丧失(基因敲除)后的体轴表型。对于图B的Hox基因编码模型,某区域体轴的表型是由该区域表达的Hox基因的共同作用决定的,Hox II基因的敲除则会导致整个Hox II基因表达区域的体轴表型的改变。对于图D所示后端优势模型,由于Hox III基因对后端体轴的表型决定具有显性作用,Hox II基因的敲除仅导致中间区域表型的改变。(A, C) Expression of three Hox genes (Hox I to Hox III) along a given axial is showed with Hox code or Posterior prevalence model respectively, and each Hox gene has a distinct expression domain. (B, D) The phenotype when Hox II is functionally deleted. (B) The phenotype is a result of combinatorial function of Hox genes,and the deletion of Hox II causes a new phenotype in the whole expression domain.(D) Hox III has a dominant function in the posterior axial, and the
2 软体动物Hox基因的研究进展
2.1 软体动物的分类
软体动物是冠轮动物中的一个类群,具有丰富的躯体结构多样性。从简单如蠕虫一样的外形到复杂如拥有发育完善脑系统的鱿鱼般的形态,现存的软体动物有11万种以上,已知的化石类群有6万种以上,是动物界中仅次于节肢动物的第二大门类[29]。现存的软体动物通常分为8个纲:管腹亚纲(Solenogastres)(新月贝类(Neomeniomorpha))、尾窝纲(Caudofoveata)(毛皮贝类(Chaetodermomorpha))、多板纲(Polyplacophora)、单板纲(Monoplacophora)、头足纲(Cephalopoda)、腹足纲(Gastropoda)、掘足纲(Scaphopoda)和双壳纲(Bivalvia)[30]。Smith[31-32]等采用分子系统生物学手段分析了目前已有软体动物转录组数据及其它分子数据,解决了软体动物的进化关系,结果支持软体动物分为两个分支:有刺亚门和有壳亚门:在上述8纲中,前2纲又合称为无板纲(Aplacophora),前3纲合称为有刺亚门(Aculifera)[31, 33],后5纲合称为有壳亚门(Conchifera)。在进化地位上,有刺亚门中多板纲是两个无板纲(管腹亚纲和尾窝纲)的姐妹群;有壳亚门中单板纲和头足纲是姐妹群。
2.2 软体动物祖先的Hox基因簇组成假说
Winnepenninckx等[34]认为由于多种替换掩盖了分子数据中大量的信息,以及软体动物多样性的爆发式的出现,对于软体动物的起源有了单源和多源假说。如果不考虑软体动物的身体结构的多样性,软体动物通常被认为是单源的。Callaerts等[35]推测软体动物基因组中仅含有一个Hox基因簇。软体动物是原口动物两大分支之一冠轮动物中的一个成员,DE, Rosa等[14]推测Hox基因在进化过程中,数目由原口动物共同祖先中的8个变为冠轮动物共同祖先中的10个。在假设软体动物为单一起源,各同源Hox基因簇并非完全独立进化,且Hox基因不存在水平转移的情况下,Iijima等[30]分离鉴定了7个分属于除单板纲外其它7个软体动物纲相关物种的Hox基因并分析了各族Hox基因在7个物种中的分布情况,并推测软体动物的祖先至少含有12个Hox基因(Hox1,Hox2,Hox3,Hox4,Hox5a,Hox5b,Hox6a,Hox6b,Hox7,Hox8,Post1andPost2)。典型的冠轮动物Hox基因簇由10个Hox基因组成:2个anterior基因、1个PG3、5个central基因和2个posterior基因[14]。冠轮动物和蜕皮动物的中区和后区基因具有明显的区别,前者以Lox5、Lox2、Lox4、Post1、Post2的形式表示,而后者以ftz、Antp、Ubx、Abd-A、Abd-B的形式表示[14, 36]。Lottiagigantea(Gastropoda)是第一个被发现具有完整Hox基因簇的物种,它的Hox基因簇由11个Hox基因组成(PG-1、PG-2、PG-3、PG-4、PG-5、Lox5、Antp、Lox2、Lox4、Post1、Post2)。我们团队近期的研究发现双壳贝类的Hox基因簇也含有11个Hox基因,且Hox基因的转录方向与L.gigantea的一致。根据软体动物进化史中腹足纲和双壳纲的分离节点推测,至少在5亿年前Hox基因簇就以含有11个Hox基因的状态存在[37]。另外3个含有这11个基因的软体动物物种是Pectenmaximus、Pinctadafucata(Bivalvia)和Gibbulavaria(Gastropoda)。Biscotti[38]分析了已报道的软体动物Hox基因的分布情况发现,软体动物中不同物种共有一个相似的Hox基因组成,即如L.gigantea中的11个Hox基因。而且数据表明,在研究最广泛的3个软体动物纲(双壳纲、头足纲、腹足纲)中这11个Hox基因都有出现。据此,Biscotti推测软体动物最后的共同祖先的Hox基因簇可能由11个基因组成。
2.3 软体动物Hox基因的分布与表达
Hox和ParaHox基因在数目、序列、调控以及表达模式上的多样性与身体结构的进化和多样性直接相关[38]。了解成簇的Hox基因的进化史对于理解双侧对称动物身体结构的进化、遗传和形态多样性之间的联系以及后生动物系统发育相关的问题至关重要[14]。软体动物是原口动物中最重要的类群之一,并且具有丰富的形态多样性,因此研究Hox和ParaHox基因在软体动物中的的分布具有重要意义。Degnan和Morse[39]最早在腹足纲Haliotisrufescens中分离鉴定了软体动物的5个Hox基因。目前对软体动物Hox基因的研究涉及了除单板纲以外的其他7个纲,具体为:掘足纲、管腹亚纲、尾窝纲各一个物种[30];多板纲3个物种[30, 40-43];头足纲5个物种[30, 35, 44-46];腹足纲10个物种[14, 30, 39, 47-53];双壳纲13个物种[30, 54-61](见图4)。可见,软体动物Hox基因的研究主要集中于多板纲、头足纲、腹足纲和双壳纲。
Fritsch等[42]首次报道了软体动物多板纲Acanthochitonacrinita中11个Hox基因的表达情况,A.crinita中7个Hox基因(Acr-Hox1~5,Hox7和Post2)沿前后轴呈共线性表达模式,且该种表达模式仅局限于幼虫时期的后端圆环区域,在软体动物的一些特异性结构如壳、足中则没有这种表达模式。接着Fritsch[43]等又进一步研究了A.crinita中Lox5、Lox4和Lox2的表达模式,这3个基因也按前后轴的顺序呈现出共线性的表达模式。
头足纲Hox基因的相关研究涉及Nautiluspompilius[30, 45]、Nautilusmacromphalus[45]、Sepiaofficinalis[45]、Octopusbimaculoides[46]和Euprymnascolopes[35, 44]这5个物种。Pernice等[45]分离鉴定了前3个物种中的Hox基因。Albertin等[46]测定了O.bimaculoides的基因组并发现该物种的Hox基因完全散在分布。Callaerts等[35]发现头足纲中的E.scolopes具有9个Hox基因,并推测软体动物基因组中只含有一个Hox基因簇。Lee等[44]通过原位杂交实验检测了E.scolopes的9个Hox基因中8个的表达情况,结果发现有6个Hox基因在中枢神经系统(Central Ner-vous System,CNS)中呈现共线性表达,与祖先中这些Hox基因一样沿着CNS轴向表达,表达模式与基因组上的排列方式相对应。另外E.colopes中Hox基因在外胚层发育而来的如足、漏斗、触手等一系列组织中特异性表达,且这种特异性表达仅存在于头足纲中。多板纲与头足纲的表达数据表明软体动物Hox基因的功能具有重塑性,也许这也正是软体动物表现出丰富的形态多样性的原因。
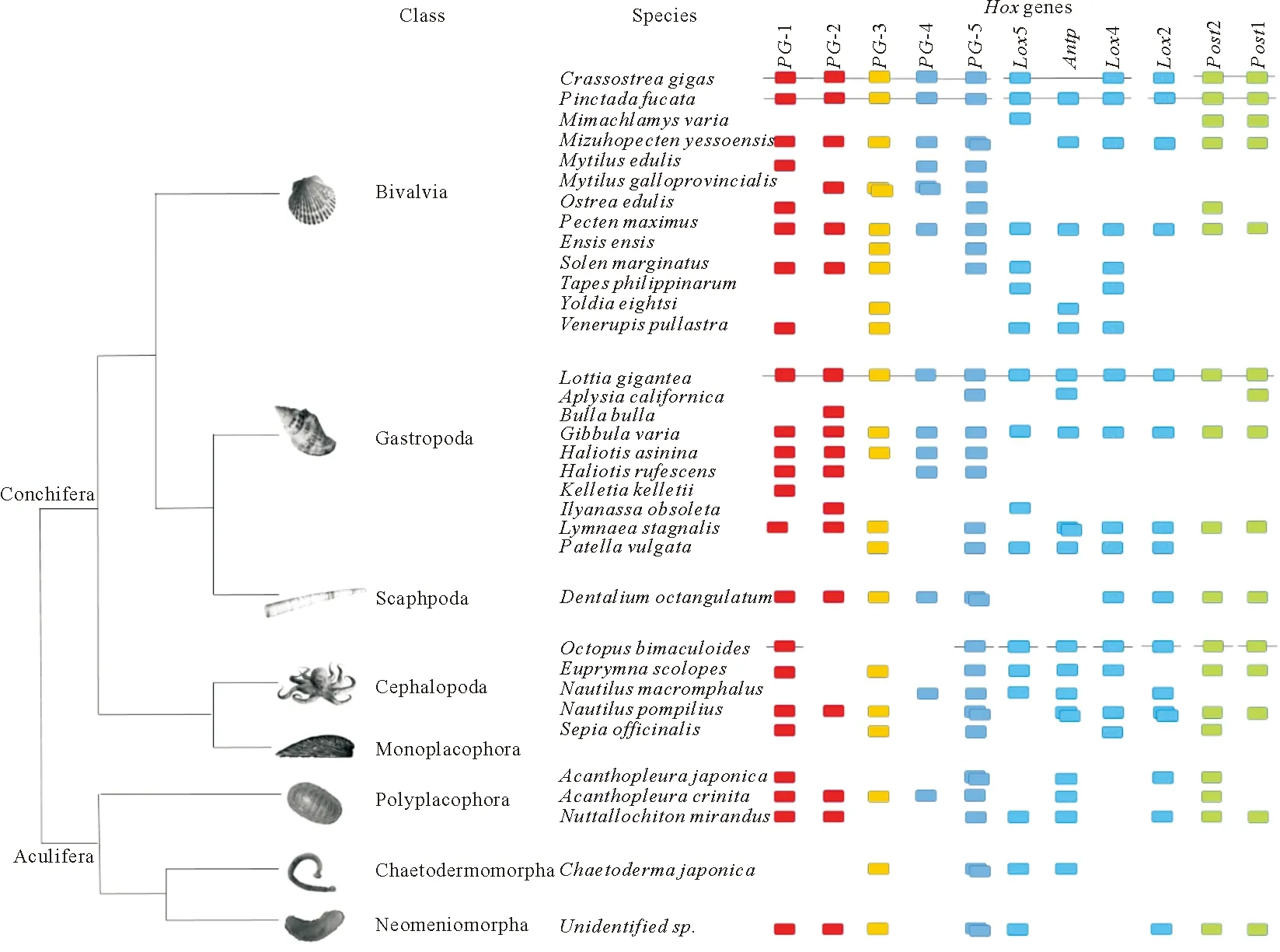
(图中为目前已报道的软体动物Hox基因。数据来源于Barucca等[62]。Hox genes identified in Mollusca are shown.
在腹足纲Hox基因的研究中,继Degnan和Morse分离得到H.rufescens的Hox基因之后,Giusti等[47]通过反转录PCR(Reverse transcription PCR,RT-PCR)分析了Hox5基因在H.rufescens幼虫时期的表达,并且原位杂交结果显示该基因与中枢神经系统的形成有关。Hinman等[49]分析了与H.rufescens同属的物种Haliotisasinina的5个Hox基因Hox1、Hox2、Hox3、Hox4和Hox5在幼虫时期的表达情况。Hox2、Hox3和Hox4在单轮幼虫腹部的神经外胚层细胞中表达,并呈现出两侧对称的表达模式。它们与Hox5在神经节中共线性表达。Hox1和Hox4在面盘幼虫外套膜的边缘表达,这暗示着它们与壳的形成有关。另外Samadi和Steiner[51-52]报道了Gva-Hox1、Gva-Post2和Gva-Post1基因在G.varia的担轮幼虫时期起始表达,且表达区域就是将来成壳的区域。Lox5、Lox4和Lox2基因在早期幼虫的顶器和晚期幼虫的脑神经节中表达,研究结果支持在变态期间顶器感觉神经细胞促进了脑神经节细胞的形成这一假设。
Barucca等[54]和Canapa等[55]采用PCR(Polymerase chain reaction)技术研究了双壳类中Hox基因的分布情况。Carpintero等[56]采用PCR技术分离第一次从双壳贝类P.maximus中分离得到了ParaHox基因caudal和ANT-C中的Pb基因。Zhang等[59]报道了Crassostreagigas中的Hox基因的表达不具有时空共线性:Hox1、Hox4、Lox5、Lox4和Post2基因在发育的早期表达,而Hox2、Hox3、Hox5、Lox2和Post1基因在面盘幼虫时期才表达。
3 结语
Hox基因作为一类重要的发育调控基因在许多物种中得到了广泛研究。软体动物作为原口动物中最重要的门类之一,具有丰富的形态多样性。研究软体动物相关Hox基因的表达及功能将有助于我们深入理解软体动物的发育及其形态高度多样性的分子基础。目前软体动物相关Hox基因的研究还比较缺乏并有待于进一步深入研究。以往对软体动物Hox基因的研究主要局限于相关基因的分离与鉴定,且采用的研究手段也比较单一,主要采用PCR技术分离获得软体动物Hox基因,这并不能代表完整的Hox基因簇的组成状态,同时,虽然对Hox基因在特定区域或组织的表达开展了相关研究[41,43,46,48,50-51],但Hox基因对下游基因的调控机制及其参与的调控网络是否与模式动物相一致等都尚未可知。新技术与新方法的开发与利用必将推动软体动物Hox基因研究的发展,高通量测序技术使我们能够快速获得生物体完整的Hox基因簇,而基因编辑技术如CRISPR/Cas9等作为一种研究基因功能的重要工具,近期已被初步运用于软体动物[63]和节肢动物[64-65]发育相关基因的研究并取得了重要进展。可以预测,飞速发展的基因编辑技术将有效推动非模式生物Hox基因的深入研究。
[1] Bürglin T R. “Homeodomain subtypes and functional diversity”.A handbook of transcription factors[M].Dordrecht: Springer, 2011: 95-122.
[2] Schofield P N. Patterns, puzzles and paradigms: the riddle of the homeobox[J]. Trends in Neurosciences, 1987, 10(1): 3-6.
[3] Holland P W. Evolution of homeobox genes[J]. Wiley Interdisciplinary Reviews: Developmental Biology, 2013, 2(1): 31-45.
[4] Lewis E B. A gene complex controlling segmentation in Drosophila[J]. Nature, 1978, 276(5688): 565-570.
[5] Duboule D, Morata G. Colinearity and functional hierarchy among genes of the homeotic complexes[J]. Trends in Genetics, 1994, 10(10): 358-364.
[6] Carrasco A E, Mcginnis W, Gehring W J, et al. Cloning of an X. laevis gene expressed during early embryogenesis coding for a peptide region homologous to Drosophila homeotic genes[J]. Cell, 1984, 37(2): 409-414.
[7] Wellik D M. Hox patterning of the vertebrate axial skeleton[J]. Developmental Dynamics An Official Publication of the American Association of Anatomists, 2007, 236(9): 2454-2463.
[8] Zakany J, Duboule D. The role of Hox genes during vertebrate limb development[J]. Current Opinion in Genetics & Development, 2007, 17(4): 359-366.
[9] Rogulja Ortmann A, Technau G M. Multiple roles for Hox genes in segment-specific shaping of CNS lineages[J]. Fly, 2008, 2(6): 316-319.
[10] Kmita M, Tarchini B, Zàkàny J, et al. Early developmental arrest of mammalian limbs lacking HoxA/HoxD gene function[J]. Nature, 2005, 435(7045): 1113-1116.
[11] Martinez P, Amemiya C T. Genomics of theHoxgene cluster[J]. Comparative Biochemistry & Physiology Part B Biochemistry & Molecular Biology, 2002, 133(4): 571-580.
[12] Prince V E. The Hox Paradox: More Complex(es) Than Imagined[J]. Developmental Biology, 2002, 249(1): 1-15.
[13] Ferrier D E, Minguillón C. Evolution of theHox/ParaHoxgene clusters[J]. International Journal of Developmental Biology, 2003, 47(7-8): 605-611.
[14] De R R, Grenier J K, Andreeva T, et al.Hoxgenes in brachiopods and priapulids and protostome evolution[J]. Nature, 1999, 399(6738): 772-776.
[15] Powers T P, Amemiya C T. Evidence for aHox14 paralog group in vertebrates[J]. Current Biology Cb, 2004, 14(5): 183-184.
[16] Brooke N M, Garcia-Fernàndez J, Holland P W. TheParaHoxgene cluster is an evolutionary sister of the Hox gene cluster[J]. Nature, 1998, 392(6679): 920-922.
[17] Luke G N, Castro L F, Mclay K, et al. Dispersal of NK homeobox gene clusters in amphioxus and humans[J]. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(9): 5292-5295.
[18] Garcia Fernàndez J. The genesis and evolution of homeobox gene clusters[J]. Nature Reviews Genetics, 2005, 6(12): 881-892.
[19] Ramos O M, Barker D, Ferrier D E. Ghost loci imply Hox and ParaHox existence in the last common ancestor of animals[J]. Current Biology, 2012, 22(20): 1951-1956.
[20] McGinnis W, Krumlauf R. Homeobox genes and axial patterning[J]. Cell, 1992, 68(2): 283-302.
[21] Duboule D. Temporal colinearity and the phylotypic progression: a basis for the stability of a vertebrate Bauplan and the evolution of morphologies through heterochrony[J]. Development, 1994, 1994(Supp.): 135-142.
[22] Kmita M, Duboule D. Organizing axes in time and space; 25 years of colinear tinkering[J]. Science, 2003, 301(5631): 331-333.
[23] Vachon G, Cohen B, Pfeifle C, et al. Homeotic genes of the bithorax complex repress limb development in the abdomen of the Drosophila embryo through the target gene Distal-less[J]. Cell, 1992, 71(3): 437-450.
[24] Greer J M, Puetz J, Thomas K R, et al. Maintenance of functional equivalence during paralogousHoxgene evolution[J]. Nature, 2000, 403(6770): 661-665.
[25] Horan G S, Ramírezsolis R, Featherstone M S, et al. Compound mutants for the paralogous hoxa-4, hoxb-4, and hoxd-4 genes show more complete homeotic transformations and a dose-dependent increase in the number of vertebrae transformed[J]. Genes & Development, 1995, 9(13): 1667-1677.
[26] Kessel M, Gruss P. Homeotic transformation of murine bertebrae and concimitant alteration of Hox codes induced by retinoic acid[J]Cell, 1991, 67(1): 89-104.
[27] Duboule D. Patterning in the vertebrate limb[J]. Current Opinion in Genetics & Development, 1991, 1(2): 211-216.
[28] Iimura T, Pourquié O.Hoxgenes in time and space during vertebrate body formation[J]. Development, growth & differentiation, 2007, 49(4): 265-275.
[29] Brusca R, Brusca G. “Phylum Mollusca”. Invertebrates (2nd ed)[M].Sunderland: Sinauer Associates, 2002: 701-769.
[30] Iijima M, Akiba N, Sarashina I, et al. Evolution ofHoxgenes in molluscs: A comparison among seven morphologically diverse classes[J]. Journal of Molluscan Studies, 2006, 72(3): 259-266.
[31] Smith S A, Wilson N G, Goetz F E, et al. Corrigendum: Resolving the evolutionary relationships of molluscs with phylogenomic tools[J]. Nature, 2013, 493(7434): 708.
[32] Smith S A, Wilson N G, Goetz F E, et al. Resolving the evolutionary relationships of molluscs with phylogenomic tools[J]. Nature, 2011, 480(7377): 364-367.
[33] Scheltema A H. Aplacophora as progenetic aculiferans and the coelomate origin of mollusks as the sister taxon of Sipuncula[J]. The Biological Bulletin, 1993, 184(1): 57-78.
[34] Winnepenninckx B, Backeljau T, De Wachter R. Investigation of molluscan phylogeny on the basis of 18S rRNA sequences[J]. Molecular Biology and Evolution, 1996, 13(10): 1306-1317.
[35] Callaerts P, Lee P N, Hartmann B, et al.Hoxgenes in the sepiolid squid Euprymna scolopes: Implications for the evolution of complex body plans[J]. Proceedings of the National Academy of Sciences, 2002, 99(4): 2088-2093.
[36] Balavoine G, Rosa R D, Adoutte A.Hoxclusters and bilaterian phylogeny[J]. Molecular Phylogenetics & Evolution, 2002, 24(3): 366-373.
[37] Wang S Z J, Jiao W, Li J, et al. Scallop genome provides insights into evolution of bilaterian karyotype and development[J]. Nature Ecology & Evolution, 2017. (In press; doi: 10. 1038/s41559-017-0120).
[38] Biscotti M A, Canapa A, Forconi M, et al. Hox and ParaHox genes: A review on molluscs[J]. Genesis, 2014, 52(12): 935-945.
[39] Degnan B M, Morse D E. Identification of eight homeobox-containing transcripts expressed during larval development and at metamorphosis in the gastropod molluscHaliotisrufescens[J]. Molecular Marine Biology & Biotechnology, 1993, 2(1): 1-9.
[40] Barucca M, Biscotti M A, Olmo E, et al. All the three ParaHoxgenes are present in Nuttallochiton mirandus (Mollusca: polyplacophora): Evolutionary considerations[J]. Journal of Experimental Zoology Part B Molecular & Developmental Evolution, 2006, 306(2): 164-167.
[41] Biscotti M A, Canapa A, Olmo E, et al.Hoxgenes in the antarctic polyplacophoranNuttallochitonmirandus[J]. Journal of Experimental Zoology Part B Molecular & Developmental Evolution, 2007, 308(4): 507-513.
[42] Fritsch M, Wollesen T, de Oliveira A, et al. Unexpected co-linearity of Hox gene expression in an aculiferan mollusk[J]. BMC Evolutionary Biology, 2015, 15(1): 151.
[43] Fritsch M, Wollesen T, Wanninger A.HoxandParaHoxgene expression in early body plan patterning of polyplacophoran mollusks[J]. Journal of Experimental Zoology Part B: Molecular and Developmental Evolution, 2016, 326(2): 89-104.
[44] Lee P N, Callaerts P, de Couet H G, et al. Cephalopod Hox genes and the origin of morphological novelties[J]. Nature, 2003, 424(6952): 1061-1065.
[45] Pernice M, Deutsch J S, Andouche A, et al. Unexpected variation of Hox genes’ homeodomains in cephalopods[J]. Molecular Phylogenetics & Evolution, 2006, 40(3): 872-879.
[46] Albertin C B, Simakov O, Mitros T, et al. The octopus genome and the evolution of cephalopod neural and morphological novelties[J]. Nature, 2015, 524(7564): 220-224.
[47] Giusti A F, Hinman V F, Degnan S M, et al. Expression of a Scr/Hox5 gene in the larval central nervous system of the gastropod Haliotis , a non-segmented spiralian lophotrochozoan[J]. Evolution & Development, 2000, 2(5): 294-302.
[48] Hinman V, Degnan B. Mox homeobox expression in muscle lineage of the gastropod Haliotis asinina: Evidence for a conserved role in bilaterian myogenesis[J]. Development Genes & Evolution, 2002, 212(3): 141-144.
[49] Hinman V F, O'Brien E K, Richards G S, et al. Expression of anterior Hox genes during larval development of the gastropodHaliotisasinina[J]. Evolution & Development, 2003, 5(5): 508-521.
[50] Lee S E, Gates R D, Jacobs D K. Gene fishing: the use of a simple protocol to isolate multiple homeodomain classes from diverse invertebrate taxa[J]. Journal of Molecular Evolution, 2003, 56(4): 509-516.
[51] Samadi L, Steiner G. Involvement ofHoxgenes in shell morphogenesis in the encapsulated development of a top shell gastropod (GibbulavariaL.)[J]. Development Genes and Evolution, 2009, 219(9-10): 523-530.
[52] Samadi L, Steiner G. Expression ofHoxgenes during the larval development of the snail, Gibbula varia (L.)-further evidence of non-colinearity in molluscs[J]. Development Genes & Evolution, 2010, 220(5-6): 161-172.
[53] Simakov O, Marletaz F, Cho S J, et al. Insights into bilaterian evolution from three spiralian genomes[J]. Nature, 2012, 493(7433): 526-531.
[54] Barucca M, Olmo E, Canapa A. Hox and paraHox genes in bivalve molluscs[J]. Gene, 2003, 317(1-2): 97-102.
[55] Canapa A, Biscotti M A, Olmo E, et al. Isolation of Hox and ParaHox genes in the bivalvePectenmaximus[J]. Gene, 2005, 348: 83-88.
[56] Carpintero P, Pazos A J, Abad M, et al. Presence of proboscipedia and caudal gene homologues in a bivalve mollusc[J]. Journal of Biochemistry and Molecular Biology, 2004, 37(5): 625-628.
[57] Mesías Gansbiller C, Sánchez J L, Pazos A J, et al. Conservation ofGbxgenes from EHG homeobox in bivalve molluscs[J]. Molecular Phylogenetics & Evolution, 2012, 63(1): 213-217.
[58] Pérez-Parallé M L, Carpintero P, Pazos A J, et al. TheHoxgene cluster in the bivalve molluscMytilusgalloprovincialis[J]. Biochemical Genetics, 2005, 43(7-8): 417-424.
[59] Zhang G, Fang X, Guo X, et al. The oyster genome reveals stress adaptation and complexity of shell formation[J]. Nature, 2012, 490(7418): 49-54.
[60] Morino Y, Okada K, Niikura M, et al. A genome-wide survey of genes encoding transcription factors in the Japanese pearl oyster, Pinctada fucata: I. homeobox genes[J]. Zoological Science, 2013, 30(10): 851-857.
[61] Paps J, Xu F, Zhang G, et al. Reinforcing the egg-timer: Recruitment of novel lophotrochozoa homeobox genes to early and late development in the pacific oyster[J]. Genome Biology & Evolution, 2015, 7(3):677.
[62] Barucca M, Canapa A, Biscotti M. An overview ofHoxgenes in lophotrochozoa: Evolution and functionality[J]. Journal of Developmental Biology, 2016, 4(1):12.
[63] Perry K J, Henry J Q. CRISPR/Cas9-mediated genome modification in the mollusc,Crepidulafornicata[J]. Genesis, 2015, 53(2): 237.
[64] Martin A, Serano J M, Jarvis E, et al. CRISPR/Cas9 mutagenesis reveals versatile roles ofHoxgenes in crustacean limb specification and evolution[J]. Current Biology Cb, 2015, 26(1): 14-26.
[65] Gui T, Zhang J, Song F, et al. CRISPR/Cas9-mediated genome editing and Mutagenesis of EcChi4 in exopalaemon carinicauda[J]. G3 Genesgenetics 2016, 6(11):3757-3764.
责任编辑 朱宝象
HoxGenes and Their Research Progresses in Molluscs
WANG Shi1, 2, YANG Zhi-Hui1, LI Yu-Li1, CHENG Tao-Ran1, LIU Ping-Ping1, MI Xue1, BAO Zhen-Min1, 3
(1.The Key Laboratory of Marine Genetics and Breeding, Ministry of Education, College of Marine Life Sciences, Ocean University of China, Qingdao 266003, China; 2.Laboratory for Marine Biology and Biotechnology, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266237, China; 3.Laboratory for Marine Fisheries Science and Food Production Processes,Qingdao National Laboratory for Marine Science and Technology, Qingdao 266237, China)
TheHoxgenes are highly conserved and encode a family of transcription factors that play a central role in anterior-posterior axial patterning in animals. Body plan evolution and diversification are affected by variations in the number and sequence ofHoxgenes, as well as by their expression patterns. Although they have been extensively studied in model organisms, relatively few is known aboutHoxgenes and their functions in molluscs. For the reason that Mollusca is one of the most important groups of protostomes, and characterized by a high body plan diversity, investigation of the relatedHoxgene and its function is of great significance for understanding and explaining the emergence of morphological diversity in molluscs. Here, we reviewed recent progresses in the investigation ofHoxgenes and their functions in molluscs, providing an important reference for understanding and further studying the molecular mechanisms of development in molluscs.
Hoxgenes; molluscs; morphological diversity; development
青岛海洋科学与技术国家实验室“鳌山人才”计划项目(2015ASTP-ES02);中央高校基本科研业务费专项(201564009);教育部长江学者奖励计划项目(Q2015119)资助 Supported by AoShan Talents Program Qingdao National Laboratory for Marine Science and Technology (2015ASTP-ES02); Fundamental Research Funds for the Central Universities (201564009); Chang Jiang Scholars Program of Ministry of Education (Q2015119)
2016-10-11;
2017-01-19
王师(1979-),男,教授,主要从事贝类遗传育种研究。E-mail: swang@ouc.edu.cn
Q1;S968.3
A
1672-5174(2017)08-023-08
10.16441/j.cnki.hdxb.20160348
王师, 杨志辉, 李语丽, 等.Hox基因及其在软体动物中的研究进展[J]. 中国海洋大学学报(自然科学版), 2017, 47(8): 23-30.
WANG Shi, YANG Zhi-Hui, LI Yu-Li, et al.Hoxgenes and their research progresses in molluscs[J]. Periodical of Ocean University of China, 2017, 47(8): 23-30.
——紫 苏

