畜禽养殖场附近污水中多粘菌素E耐药性肠杆菌的分离分析及内源质粒检测
钟传青,宗工理,张超,付加芳,姜天翼,曹广祥*
1. 山东建筑大学市政与环境工程学院,山东 济南 250101;2. 山东省医学科学院山东省医药生物技术研究中心,山东 济南 250062
畜禽养殖场附近污水中多粘菌素E耐药性肠杆菌的分离分析及内源质粒检测
钟传青1,宗工理2,张超1,付加芳2,姜天翼1,曹广祥2*
1. 山东建筑大学市政与环境工程学院,山东 济南 250101;2. 山东省医学科学院山东省医药生物技术研究中心,山东 济南 250062
畜禽养殖业大量使用抗生素引起了广泛的微生物耐药性问题,研究抗生素耐药菌株可以了解环境中细菌的耐药性情况和抗性基因的传播机制。采集畜禽养殖场周边的污水,分析了污水中多粘菌素E耐药性细菌和耐药性肠杆菌的丰度;同时以一株具有较高多粘菌素E耐药水平的肠杆菌R11为例,首先进行16S rDNA测序和抗生素耐药谱分析,然后通过质粒提取、酶切电泳以及脉冲场凝胶电泳(PFGE),分析其内源性质粒等情况。结果表明:在畜禽养殖场周边污水环境中,多粘菌素E耐药性细菌占细菌总数的0.14%~0.35%,多粘菌素E耐药性肠杆菌占肠杆菌总数的1.50%~7.40%,而肠杆菌占细菌总数的1.13%~2.45%,说明肠杆菌可能是携带多粘菌素E抗性基因的主要细菌种类,同时也是养殖业污水中细菌的主要组成部分。16S rDNA序列分析确定R11是一株Enterobacter cloacae,抗生素耐药谱分析R11具有多重耐药性,除对林可霉素敏感外,阿米卡星、四环素和环丙沙星的最小抑菌浓度(MICs)为10 μg∙mL-1,阿莫西林的MICs为25 μg∙mL-1,磺胺甲恶唑的MICs为32 μg∙mL-1,氨苄西林的MICs为40 μg∙mL-1,链霉素的MICs为90 μg∙mL-1,而多粘菌素E的MICs最高,达到96 μg∙mL-1。质粒提取酶切和PFGE分离鉴定结果显示,R11菌株可能有多个内源性质粒。综上所述,R11菌株具有较高水平的多粘菌素E耐药性,并且具有多重耐药性和多个内源性质粒,对于研究多粘菌素E耐药性机制具有一定的参考价值。
畜禽养殖场;肠杆菌;多粘菌素E;多重耐药性;质粒
微生物的抗生素耐药性已成为影响病原菌治疗效果的重要问题,严重危及公众健康。抗生素耐药性微生物普遍存在于非人类活动的自然环境中,具有古老的起源(李显志,2013),但是人类活动特别是抗生素滥用极大地加速了抗生素耐药性的进化与传播(Singer et al.,2016)。调查显示,2013年中国抗生素总使用量约为16.2×107kg,而畜禽与水产养殖业抗生素使用量占到总量的50%以上(Zhang et al.,2015)。许多研究证实不论是在医院、动物养殖场、土壤,还是在人体、食品、工业品,甚至早产儿体内都可以分离出耐药性微生物(Giannino et al.,2009;Spellberg et al.,2011;陶宏兵等,2015),说明抗生素耐药性已经发展成为无法忽视和回避的问题。
肠杆菌科(Enterobacteriaceae)细菌是一类与人类关系密切的细菌,在动植物、土壤和水体中广泛存在。研究表明,肠杆菌科细菌在抗生素耐药性的转移和传播中起着重要的媒介作用(Iredell et al.,2016),位于基因组的抗生素耐药性基因可以通过溶原性噬菌体、转座子和整合子进行转移(Stokes et al.,2011),而位于内生质粒的耐药性基因可能通过转化作用进行水平转移(Yanat et al.,2017)。与此同时,许多肠杆菌科的细菌还是条件致病菌,这类医源性耐药菌加大了临床治疗的难度(Perez et al.,2013)。本研究分析了山东省济南市畜禽养殖场周边环境中耐药性细菌的分布,从中分离出一株具有较高水平多粘菌素E耐药性和多重耐药性的肠杆菌科细菌,并研究了该菌株的生物学特征,以期为深入研究肠杆菌科细菌的多粘菌素E耐药机制奠定基础。
1 材料与方法
1.1 样品的采集
样品1、样品2、样品3分别来源于济南市贾庄生猪和云川蛋鸡养殖场的排污沟水。利用无菌吸管采集100 mL水样放入无菌三角瓶,用带滤膜的封口膜密封,贮存于4 ℃环境中。
1.2 培养基
微生物的分离和计数采用LB培养基和mTEC培养基(Brenner et al.,1993),不加或加入8 μg∙mL-1多粘菌素E。mTEC培养基成分为1 L培养基含:蛋白胨5 g,酵母提取物3 g,乳糖10 g,氯化钠7.5 g,磷酸氢二加3.3 g,磷酸二氢钾1 g,十二烷基硫酸钠0.2 g,去氧胆酸钠0.1 g,X-Gluc 0.15 g。
1.3 耐药性细菌的计数与分离筛选
采用稀释平板涂布法计算样品中细菌、肠杆菌科细菌和耐药性细菌的数量。样品用无菌水依次梯度稀释,涂布到加入8 μg∙mL-1多粘菌素E或不加入抗生素的LB固体培养基平板上,28 ℃培养箱内培养24 h,计数细菌菌落的数量。肠杆菌数量的检测采用mTEC固体培养基,将上述稀释样品涂布到入8 μg∙mL-1多粘菌素E或不加入抗生素的mTEC固体培养基平板上,置于44.5 ℃培养箱中2 h杀死部分不耐高温的非肠杆菌细菌,然后转入37 ℃培养箱继续培养24 h,计数蓝色菌落的数量。上述每个样品做3个重复,用空白培养基作为对照。同时挑取单菌落到含8 μg∙mL-1多粘菌素E的固体平板上,多次划线传代分离单菌落,将分离到的菌株进行命名,选取R11菌株进行后续研究。
1.4 R11菌株的16S rDNA鉴定
用LB培养基过夜培养R11菌株,按照QIAamp DNA Mini Kit(Qiagen,Germany)说明书提取基因组总DNA。采用通用引物27F 5′-GAGTTTGA TCCTGGCTCAG-3′和1492R 5′-AGAAAGGAGG TGATCCAGCC-3′进行PCR扩增,DNA测序后利用BLAST(NCBI)数据库进行16S rDNA序列的比对。
1.5 R11菌株的抗生素耐药性研究
本研究选择畜禽养殖场使用率较高的氨苄西林(ampicillin)、阿莫西林(amoxicillin)、链霉素(streptomycin)、阿米卡星(amikacin)、四环素(tetracyclines)、林可霉素(lincomycin)、磺胺甲恶唑(sulfamethoxazole)、环丙沙星(ciprofloxacin)和多粘菌素E(polymyxin E)等作为研究对象,分别配制上述各种抗生素母液并稀释成工作浓度,然后根据文献方法(Nordmann et al.,2016)测定R11菌株对上述各种抗生素的最小抑菌浓度(MICs)。
1.6 R11菌株的质粒分析
采用碱裂解法提取R11菌株的质粒(萨姆布鲁克等,2001),利用限制性内切酶EcoR I酶切后进行常规的琼脂糖电泳分离,检测R11菌株是否含有内源性质粒。同时将培养好的R11菌体包埋到琼脂糖凝胶中,SDS破壁后用蛋白酶K去除菌体蛋白,然后将含DNA的琼脂糖块置于脉冲场凝胶电泳(PFGE)分离胶中,采用PFGE电泳仪(CHEF Mapper,Bio-rad)进行检测,设置分离片段大小为10~600 kb,检测条件采用默认参数,检测R11菌株中内源性质粒。试验采用不含内源性质粒的Escherichia.coli DH5a菌株作为对照。
2 结果与讨论
2.1 畜禽养殖场污水中耐药性细菌的分析
平板菌落计数检测发现污水样品中细菌数量分别达到2.2×106、8.1×106和4.8×105cfu∙mL-1,多粘菌素E耐药性细菌数量分别为3.1×102、2.9×103和1.4×102cfu∙mL-1(表1),结果显示耐药性细菌占总细菌数的0.14%~0.35%,表明养殖场周围污水中存在大量的多粘菌素E耐药性细菌。样品中肠杆菌数量分析显示,肠杆菌占细菌总数的1.13%~2.45%,说明肠杆菌是污水中一类主要的细菌群体。与此同时,多粘菌素E耐药性肠杆菌数量占肠杆菌总数的1.50%~7.40%,明显高于耐药性细菌占细菌总数的比例,说明肠杆菌在细菌耐药性中占据重要的地位,可能是携带抗生素耐药性基因的主要细菌种类。研究表明,医源性耐药菌中肠杆菌占一定比例(Potter et al.,2016),由于肠杆菌是条件致病菌,在养殖场排放污水中检出的大量耐药性肠杆菌可能威胁人类健康。
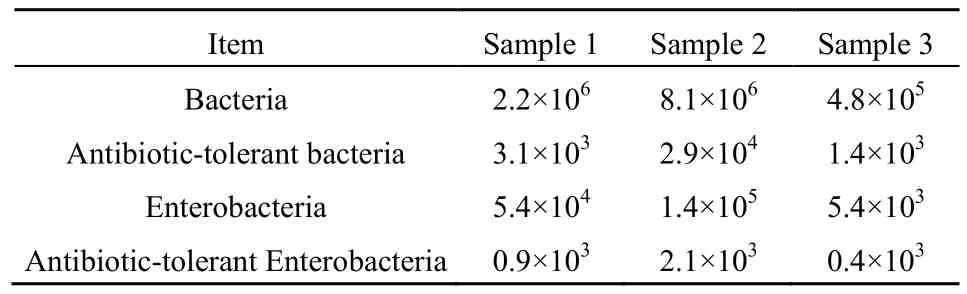
表1 耐药性细菌和肠杆菌数量与比例分析Table 1 Analysis on quantity and proportion of antibiotic-tolerant bacteria and Enterobacteria cfu∙mL-1
2.2 R11菌株的分离与16S rDNA鉴定
从上述含抗生素固体平板上挑取菌落,多次划线传代、分离单菌落并编号,最后得到一株具有稳定的多粘菌素E抗性菌株,命名为R11菌株。R11菌株在LB固体培养基呈湿润、微微凸起、乳白色、边缘光滑的圆形菌落,可以在28~44.5 ℃范围内生长,说明R11可能属于肠杆菌科细菌。
R11菌株的16S rDNA鉴定以基因组总DNA为模板,通过PCR扩增得到16S rDNA片段,构建到中间载体后进行DNA测序。利用NCBI Blast程序对测序结果进行比对,结果显示R11菌株的16S rDNA序列与Enterobacter cloacae DSM 16690的相似度达到99%,结合R11菌株的表型特征,认为R11菌株可能是Enterobacter cloacae。
2.3 R11菌株的抗生素耐药性分析
研究进一步测定了R11的抗生素耐药性抗性谱和MICs,R11菌株的耐药谱和最小抑菌浓度见表2。结果显示,R11菌株具有多重耐药性,除氯霉素外,其对其他供试的抗生素都有明显耐药性,说明R11菌株可能拥有多种抗生素耐药基因。此外,R11菌株具有较高水平的多粘菌素E耐药性。多粘菌素E曾经被认为是临床上治疗抗生素耐药性病原菌感染的最后一条防线(Falagas et al.,2005),但是由于多粘菌素被大量应用于畜禽养殖业中,在世界各地的不同环境甚至在病人感染部位中都已经分离出多粘菌素的耐药菌,引起公众的担忧(Vanduin et al.,2015;Rapoport et al.,2016)。分析显示,R11菌株多粘菌素E的MICs达到96 μg∙mL-1,在从中国不同环境中分离到的耐药菌株中属于较高的水平(Liu et al.,2016),对于研究多粘菌素耐药机制具有一定的参考价值。

表2 R11菌株的多种抗生素MICs分析Table 2 MICs analysis of strain R11 on different antibiotics μg∙mL-1
2.4 R11菌株的内生质粒检测
微生物的大部分抗生素耐药性基因位于基因组上,部分位于质粒中,并可以通过质粒转移到其他微生物或菌株中。试验首先用碱裂解法提取R11菌株的质粒(图1),DNA电泳显示样品存在多条DNA条带。为了判断这些DNA条带是否是提取过程中裂解形成的染色体断裂条带,利用限制性内切酶EcoR I对上述DNA样品进行酶切处理,然后进行DNA电泳分析。结果显示,DNA经酶切后产生多条特异性条带,而细菌基因组由于具有多个EcoR I限制性酶切位点,经酶切后通常会产生数百个DNA片段,从而出现弥散型条带,这说明通过碱裂解法能够从R11菌株中提取内源性质粒,而且质粒可能不止一个。
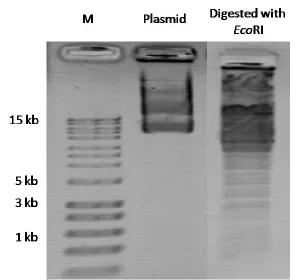
图1 R11菌株中质粒的提取和酶切电泳分析Fig. 1 Analysis on extraction and digestion of plasmids in strain R11
为了进一步鉴定R11菌株的质粒,试验将R11菌体和不含质粒的E. coli DH5a菌体包埋在琼脂糖凝胶中,然后利用蛋白酶K降解去除菌体蛋白,最后用PFGE电泳分离琼脂糖凝胶中的基因组DNA和质粒DNA,结果如图2所示。结果显示,R11菌株和阴性对照E. coli DH5a菌株都有一条大约600 kb的染色体断裂条带;与此同时,R11菌株中有多个比较模糊的特征DNA条带,如图2中箭头所示,而DH5a样品中没有特征性DNA条带,这说明这些特征条带不是由染色体断裂产生的。因此,可以肯定R11菌株存在质粒DNA。另外,由于质粒可能存在多种天然构象,不同构象质粒的电泳迁移速率各不相同,无法根据PFGE结果判断样品中质粒具体数量和大小(Marasini et al.,2014),但是根据PFGE电泳结果可以确定R11菌株具有多个质粒,而且部分质粒属于大质粒。多粘菌素耐药基因最早被发现于基因组上,但是最新研究发现质粒也可能编码多粘菌素耐药基因(Fernández et al.,2013;Srinivasan et al.,2014),说明细菌可能有多种对抗多粘菌素的机制。本研究分离的R11菌株拥有多个质粒,并且具有较高水平的多粘菌素耐药性,因此值得进一步研究其多粘菌素的耐药机制。

图2 R11菌株中质粒的PFGE电泳分析Fig. 2 PFGE analysis of plasmids in strain R11
3 结论
本研究从济南市周边的生猪和蛋鸡养殖场附近的污水沟中取样,研究发现耐药肠杆菌占总细菌数的0.14%~0.35%,占肠杆菌总数的1.50%~7.40%,说明肠杆菌可能在细菌耐药性中占据重要地位,可能是携带抗生素耐药性基因的主要细菌种类。通过分离纯化得到一株肠杆菌属菌株R11,研究表明该菌株具有多重耐药性,并且具有较高水平的多粘菌素E耐药性。采用碱裂解法提取质粒和PFGE分析,结果显示R11菌株具有多个质粒,可能与该菌株具有多种耐药性有关,R11菌株的多重耐药性基因定位在染色体上还是质粒上值得进一步研究。
BRENNER K P, RANKIN C C, ROYBAL Y R, et al. 1993. New medium for the simultaneous detection of total coliforms and Escherichia coli in water [J]. Applied and Environmental Microbiology, 59(11): 3534-3544.
FALAGAS M E, KASIAKOU S K. 2005. Colistin: the revival of polymyxins for the management of multidrug-resistant gram-negative bacterial infections [J]. Clinical Infectious Diseases, 40(9): 1333-1341.
FERNÁNDEZ L, ALVAREZ-ORTEGA C, Wiegand I, et al. 2013. Characterization of the polymyxin B resistome of Pseudomonas aeruginosa [J]. Antimicrob Agents Chemother, 57(1): 110-119.
GIANNINO M L, MARZOTTO M, DELLAGLIO F, et al. 2009. Study of microbial diversity in raw milk and fresh curd used for Fontina cheese production by culture-independent methods [J]. International Journal of Food Microbiology, 130(3): 188-195.
IREDELL J, BROWN J, TAGG K. 2016. Antibiotic resistance in Enterobacteriaceae: mechanisms and clinical implications [J]. British Medical Journal, 352: h6420.
LIU Y Y, WANG Y, WALSH T R, et al. 2016. Emergence of plasmid-mediated colistin resistance mechanism MCR-1 in animals and human beings in China: a microbiological and molecular biological study [J]. The Lancet Infectious diseases, 16(2): 161-168. MARASINI D, FAKHR M K. 2014. Exploring PFGE for detecting large plasmids in Campylobacter jejuni and Campylobacter coli isolated from various retail meats [J]. Pathogens, 3(4): 833-844.
NORDMANN P, JAYOL A, POIREL L. 2016. Rapid detection of polymyxinresistance in Enterobacteriaceae [J]. Emerging infectious diseases, 22(6): 1038-1043.
POTTER R F, DSOUZA A W, DANTAS G. 2016. The rapid spread of carbapenem-resistant Enterobacteriaceae [J]. Drug Resistance Updates, 29: 30-46.
PEREZ F, VAN DUIN D. 2013. Carbapenem-resistant Enterobacteriaceae: a menace to our most vulnerable patients [J]. Cleveland Clinic Journal of Medicine, 80(4): 225-233.
RAPOPORT M, FACCONE D, PASTERAN F, et al. 2016. First description of mcr-1-mediated colistin resistance in human infections caused by Escherichia coli in Latin America [J]. Antimicrobial Agents and Chemotherapy, 60(7): 4412-4413.
STOKES H W, GILLINGS M R. 2011. Gene flow, mobile genetic elements and the recruitment of antibiotic resistance genes into Gram-negative pathogens [J]. FEMS Microbiology Reviews, 35(5): 790-819.
VANDUIN D, DOI Y. 2015. Outbreak of colistin-resistant, carbapenemase-producing Klebsiella pneumoniae: Are we at the end of the road [J]. Journal of Clinical Microbiology, 53(10): 3116-3117.
SINGER A C, SHAW H, RHODES V, et al. 2016. Review of antimicrobial resistance in the environment and its relevance to environmental regulators [J]. Frontiers in Microbiology, 7: 1728.
SPELLBERG B, BLASER M, GUIDOS R J, et al. 2011. IDSA Public Policy: Combating antimicrobial resistance: policy recommendations to save lives [J]. Clinical Infectious Diseases, 52(Suppl 5): S397-428. SRINIVASAN V B, SINGH B B, PRIYADARSHI N, et al. 2014. Role of novel multidrug efflux pump involved in drug resistance in Klebsiella pneumonia [J]. PLoS One, 9(5):e96288.
YANAT B, RODRÍGUEZ-MARTÍNEZ J M, TOUATI A. 2017. Plasmid-mediated quinolone resistance in Enterobacteriaceae: a systematic review with a focus on Mediterranean countries [J].European Journal of Clinical Microbiology &Infectious Diseases, 36(3): 421-435.
ZHANG Q Q, YING G G, PAN C G, et al. 2015. Comprehensive evaluation of antibiotics emission and fate in the river basins of China: Source analysis, multimedia modelling, and linkage to bacterial resistance [J]. Environmental Science and Technology, 49(11): 6772-6782.
萨姆布鲁克J, 拉塞尔D W. 2001. 分子克隆实验指南[M]. 第三版. 北京: 科学出版社: 27-29.
李显志. 2013. 抗生素耐药基因古老起源与现代进化及其警示[J]. 中国抗生素杂志, 38(2): 81-89.
陶宏兵, 邱晓颖, 冯劲, 等. 2015. 被污染的工业产品中常见微生物种类及耐药性分析[J]. 工业微生物, 45(5): 42-45.
Isolation and characterization of the polymyxin E resistant enterobacteria from sewage water near livestock and poultry feedlot [J]. Ecology and Environmental Sciences, 26(4): 671-675.
ZHONG Chuanqing, ZONG Gongli, ZHANG Chao, FU Jiafang, JIANG Tianyi, CAO Guangxiang. 2017.
Isolation and Characterization of the Polymyxin E Resistant Enterobacteria from Sewage Water near Livestock and Poultry Feedlot
ZHONG Chuanqing1, ZONG Gongli2, ZHANG Chao1, FU Jiafang2, JIANG Tianyi1, CAO Guangxiang2*
1. School of Municipal and Environmental Engineering, Shandong Jianzhu University, Jinan 250101, China;
2. Shandong Medicinal Biotechnology Centre, Shandong Academy of Medical Sciences, Jinan 250062, China
Overuse of antibiotics in livestock and poultry feedlot has caused broad antibiotics resistance of microbes. Research on antibiotics-resistant microbes is helpful to reveal the status of antibiotics resistance and the transmission mechanism of antibiotics-resistant genes in the environment. In this study, samples were collected from sewage water near livestock and poultry feedlot, the abundance of both antibiotics-resistant bacteria and antibiotics-resistant Enterobacteria was analyzed, in the meantime, strain R11 with high polymyxin E resistance was isolated and studied. Firstly, 16S rDNA of strain R11was sequenced and antibiotics-resistance was analyzed, plasmids were extracted and digested with restriction enzyme, and then PFGE was used to detect the endogenous plasmids. Results showed that polymyxin E-resistant bacteria accounted for 0.14%~0.35% of all bacteria and polymyxin E-resistant Enterobacteria accounted for 1.50%~7.40% of all Enterobacteria. In addition, Enterobacteria occupied 1.13%~2.45% of all the bacteria, which showed that Enterobacteria may be the vital bacteria with polymyxin E resistance, and also occupied large amounts of bacteria in sewage water near livestock and poultry feedlot. Strain R11 was identified as Enterobacter cloacae by 16S rDNA sequence analysis. Strain R11 has multi-antibiotics resistance, which was only sensitive to lincomycin. The MICs of amikacin, tetracycline and ciprofloxacin of strain R11 were 10 μg∙mL-1, while the MICs of amoxicillin, sulfamethoxazole, ampicillin, streptomycin, polymyxin E was 25 μg∙mL-1, 32 μg∙mL-1, 40 μg∙mL-1, 90 μg∙mL-1and 96 μg∙mL-1, respectively. In addition, strain R11 carried several endogenous plasmids. All the results showed that strain R11 had a high level of polymyxin E resistance, and it was useful to study the mechanism of polymyxin E resistance in microbes.
swine feedlot; Enterobacteria; polymyxin E; multi-antibiotics resistance; plasmid
10.16258/j.cnki.1674-5906.2017.04.018
X171.5
A
1674-5906(2017)04-0671-05
钟传青, 宗工理, 张超, 付加芳, 姜天翼, 曹广祥. 2017. 畜禽养殖场附近污水中多粘菌素E耐药性肠杆菌的分离分析及内源质粒检测[J]. 生态环境学报, 26(4): 671-675.
国家自然科学基金项目(31500094);山东省自然科学基金项目(ZR2015EM018);山东省医学科学院医药卫生科技创新工程项目(201604)
钟传青(1977年生),女,副教授,博士,主要从事环境微生物研究。E-mail: zhongchq@163.com
*通信作者
2017-03-16

