规律有氧运动对大鼠纹状体miRNA207/542及下游靶信号通路的影响
刘文锋 印大中 汤长发 唐利花 陈宗平
1湖南师范大学体适能与运动康复湖南省重点实验室(长沙 410012)
2湖南师范大学蛋白质化学与发育生物学教育部重点实验室(长沙 410081)
规律有氧运动对大鼠纹状体miRNA207/542及下游靶信号通路的影响
刘文锋1,2印大中1,2汤长发1唐利花1陈宗平1
1湖南师范大学体适能与运动康复湖南省重点实验室(长沙 410012)
2湖南师范大学蛋白质化学与发育生物学教育部重点实验室(长沙 410081)
目的:采用规律有氧运动干预不同年龄大鼠,基于miRNA表达谱芯片技术,旨在寻找运动适应性作用对神经退行性病变早期作用的靶标,挖掘关键的可解释运动适应性作用机理的靶标基因的生物功能。方法:选取SPF级健康雄性3月龄(青年,n=20)、13月龄(中年,n=24)和22月龄(老年,n=24)SD大鼠,每个年龄组大鼠按体重随机分为青年对照组(Y-SED,n=10)、青年运动组(Y-EX,n=10),中年对照组(MSED,n=12)、中年运动组(M-EX,n=12),老年对照组(O-SED,n=12)和老年运动组(O-EX,n=12)。三组对照组安静饲养;三组运动组进行10周递增负荷中等强度的规律有氧跑台运动。每周进行体重监控;采用HE染色观察大鼠脑纹状体形态学变化;采用miRNA微阵列芯片miRCURYTM LNA Array(v.18.0)等方法分析miRNAs差异表达谱,使用qRT-PCR等对筛选重要差异miRNA及其相关通路的mRNA表达进行验证与研究。结果:大鼠纹状体的形态学变化明显呈现增龄性变化,实施规律有氧运动后,各年龄组相应地出现线团状的基质部分明显紧凑,之间间隙非常紧密,显微镜下观察细胞核排列有序,数量明显增加。miRNA微阵列芯片数据结果显示,发生2倍及以上变化差异的miRNAs有26个,经生物信息学分析,最终筛查出规律有氧运动上调miRNA207和下调miRNA542的表达,miRNA207和miRNA542所作用的下游基因PI3K、Akt和mTOR mRNA表达呈现增龄性下调,与对照组M-SED和O-SED相比较,M-EX和O-EX组PI3K、Akt和mTOR mRNA表达上调(P<0.05,P<0.01)。Vdac1 mRNA呈现增龄性上升趋势,与各年龄安静组相比较,运动组CaMKIIα和Vdac1 mRNA表达上调显著(P<0.05)。结论:规律有氧运动通过上调miRNA207和下调miRNA542的表达而激活大鼠纹状体的PI3K/Akt/mTOR和CaMKIIα信号通路共同参与调控纹状体的生物学功能和改善神经老化。
有氧运动;纹状体;老化;miRNA207;miRNA542;PI3K;Akt;mTOR;CaMKIIα
microRNAs(miRNAs)是一种小RNA基因,长度约20~24个核苷酸,为内源性非编码基因,miRNA广泛表达并通过多种方式调控转录后的一系列基因,参与生命活动的各项重要过程[1-3]。当miRNA与靶mRNA碱基完全互补配对时,miRNA会引起靶mRNA的降解,而当碱基序列不完全互补时,miRNA在蛋白翻译水平上调控mRNA的表达。研究报道miRNA对脑老化和神经退行性疾病的形成具有关键调节作用,如miRNA-429调控阿尔茨海默病(Alzheimer’s Disease,AD)和亨廷顿病(Huntington’s disease,HD)两种疾病,miRNA-743b-3p调节HD和帕金森病(Parkinson’s disease,PD)两种疾病,miRNA-141-3p,miRNA-200a-3p,miR⁃NA-200a-5p和miRNA-499a-5p参与AD和PD这两种疾病的调控,miRNA-182对这三种疾病都有影响[4-7]。Yin等[8]构建自然增龄性老化模型,取全脑进行miRNA深度测序,进一步采用PANTHER分类系统[9]对靶基因进行了分析,研究发现23种差异表达的miRNAs所调控的24种靶基因,分别对AD,HD或PD有直接的作用。
本研究基于miRNA表达谱芯片技术,旨在寻找运动适应性作用对神经退行性病变早期作用的新的靶标,再经过实时荧光定量PCR验证趋势与miRNA芯片表达结果的一致性,经生物信息学分析试图寻找上调和下调miRNA而调控相关信号通路的mRNA表达,从而挖掘关键的可解释运动适应性作用机理的靶标基因的生物功能。
1 材料与方法
1.1 实验对象与分组
健康雄性SD大鼠3月龄(青年,n=20)、13月龄(中年,n=24)和22月龄(老年,n=24),均为SPF(Specific pathogen Free,SPF)级动物,由长沙市开福区东创实验动物科技服务部提供,动物许可证号:SCXK(湘)2009-0012。以国家标准啮齿类动物饲料(A级)饲养。室温保持20~24℃,相对湿度为45%~55%。实验过程中对动物的处置符合《实验动物管理条例》、《关于善待实验动物的指导性意见》等规定。
每个年龄组大鼠按体重随机分组,分为青年对照组(Y-SED,n=10)、青年运动组(Y-EX,n=10);中年对照组(M-SED,n=12)、中年运动组(M-EX,n=12);老年对照组(O-SED,n=12)和老年运动组(OEX,n=12)。三组对照组静息,不做跑台运动;三组运动组进行10周递增负荷中等强度的规律有氧跑台运动。
1.2 规律有氧运动处方
所有运动组SD大鼠适应性饲养2周后,采用杭州立泰科技有限公司研制的PT动物电动跑台进行3天5~10 min适应性训练,坡度0°,速度10 m/min。中等强度规律有氧运动模型:参考本实验前期动物实验等[10],以及借鉴国内张勇等研究报道[11],运动强度相当于最大摄氧量(VO2max)60%~65%逐渐递增到70%~75%,坡度0°,为期10周。青年、中年和老年大鼠的运动负荷均从每天以15 m/min速度跑台运动15 min开始,第2周速度不变,时间递增5 min,第3周速度增加3 m/ min,时间延长5 min,第4周速度不变,时间递增5 min;根据增龄性因素,第5周从速度和时间两方面考虑递增延续到第6周作为运动负荷固定的过渡运动期,前6周每周运动6天。后4周固定运动强度(随机抽取3只大鼠,尾部采血测试血乳酸浓度监控运动强度):青年组以25 m/min速度运动45 min,中年组以22 m/min速度运动40 min,老年组以20 m/min速度运动35 min,每周5天(见表1)。
1.3 实验取材与样本制备
最后一周运动结束后,腹腔注射10%水合氯醛溶液按0.5 ml/100g麻醉大鼠进行取材。每组随机选取3只大鼠,进行升主动脉灌注[生理盐水(37℃)快速灌注5 min(60 ml)左右以移除血液],然后缓慢点滴4%多聚甲醛0.1 M磷酸缓冲液(pH 7.4)(4℃)400~500 ml灌注固定,直到动物的肝脏发硬与尾巴僵直,取脑,保存在4%多聚甲醛0.1 M磷酸缓冲液中4°冰箱过夜,经石蜡包埋后用于切片制作。其余大鼠直接取脑,参考Glwinski and Iversen[12,13],诸葛启钏主译[14]大鼠脑立体定位图谱,分离出两侧的新鲜纹状体,经液氮速冻后置于-80°冰箱保存,用于miRNA芯片等测试。
1.4 体重监控
所有大鼠采用体重计每周一开始隔天测量体重,将每周测得3次体重求均值进行统计学处理。
1.5 HE染色
按照大鼠脑立体定位图谱定位点:对耳线10 mm左右,前卤0.7 mm左右,进行切片,即可切到纹状体部位。常规步骤:脱腊水化、苏木素染色10~15 min、1%的盐酸酒精分色、5%伊红染色1 min、梯度酒精脱水、二甲苯透明、中性树胶封片、镜检。采用美国Simple PCI Version 6.0生物显微镜系统采集图像。每组至少选片5张,每张切片镜下(×200)随机取5个视野进行分析。
1.6 miRNA基因芯片基因芯片
采用丹麦Exiqon公司研发的第7代产品miRNA表达谱芯片miRCURYTMLNA Array(v.18.0)(Exiqon),包含3100个微探针,覆盖人类、小鼠和大鼠以及除病毒之外的等所有已发现的microRNAs(参考miRBase 18.0),另外还包含25 miRPlusTM人类 miRNAs。
1.6.1 总RNA的提取
称大鼠脑纹状体60 mg左右,采用TRIzol法提取总RNA。采用紫外分光光度计(ND-1000,Nanodrop Technologies)检验RNA样品完整性。采用1.2%的琼脂糖凝胶电泳检测所提取的RNA经甲醛变性胶电泳完整性。由图1可见RNA样品质量符合miRNA芯片实验要求。
图1 增龄大鼠样本抽提RNA的电泳情况
1.6.2 miRNA的芯片技术
使用试剂盒TRIzol(Invitrogen)and miRNeasy mini kit(QIAGEN)和miRCURY™Hy3™/Hy5™Pow⁃er labeling kit等抽提总RNA、量化RNA和进行杂交,处理好的Exiqon’s miRCURY LNA™miRNA Array(miRNA芯片)采用仪器Axon GenePix 4000B microar⁃ray scanner扫描等;最后采用软件miRCURY™LNA Array(v.18.0)、GenePix Pro 6.0 software(Axon)和Ingenuity Pathway Analysis等进行RNA芯片修正、分析和生物信息学分析。经过步骤:抽提RNA;RNA质量检测;总RNA的纯化和浓缩;RNA反转录和扩增;标记miRNA;miRNA芯片杂交;洗片与扫描等。
1.6.3 miRNA芯片的扫描与数据分析
从图2芯片扫描图可见,图像清晰,无背景信号,无划痕,信号点规则,边缘清晰杂交信号均一,表明芯片杂交反应成功。

图2 M-SED组(上)和M-EX组(下)的芯片扫描图
采用中位数据归一化方法(Median Normalization Method)得到标准值(Normalized Data):Normalized Data=修正值(Foreground-Background)/medi⁃an,即为各个miRNA的修正值除以各张芯片的中位数做标准化后的数值,进行样本比较时采用的数值[15,16]。为了检测集合中的miRNAs的差异化表达水平,我们通过 Bioconductor DESeq package[17](http://www.biocon⁃ductor.org/),来对miRDeep2中已知的miRNAs的表达数据进行统计学分析。
1.6.4 miRNAs生物信息学分析:靶点预测和功能分析
采用IPA(Ingenuity Pathway Analysis)分析工具以及靶标预测数据库TargetScan[18]中的所有miRNA靶标数据。通过GO(Gene Ontology)分析可得到分子功能、生物过程和细胞组成三方面信息[19]。蛋白质或者基因可以通过ID对应或者序列注释的方法找到与之对应的GO号,从而得到功能类别或者细胞定位等。
通过miRDB(http://mirdb.org/miRDB/)数据库来预测差异性表达的已知miRNAs的靶点——预测得分≥60[20,21]。借助DAVID(http://david.abcc.ncifcrf.gov/)生物信息学资源(6.7版本)对每个测到的靶点基因进行功能学注释,包括一个整合的生物学知识库和分析工具,可以系统地从大基因/蛋白系列中提取生物学意义[22,23]。通过KEGG(Kyoto Encyclopedia of Genes and Genomes)网站数据库[24,25]了解代谢、遗传信息处理、环境信息处理、细胞过程和人类疾病等信息[26]。
1.7 miRNA207和miRNA542表达的qRT-PCR验证
1.7.1 qRT–PCR miRNA引物

表2 qRT-PCR miRNA引物设计
1.7.2 目的基因miRNA的相对定量
每组随机3个样本,每个样品3个复管进行实时荧光定量PCR,确认实时荧光定量PCR的扩增曲线和熔解曲线,记录各个基因miRNA的Ct(cycle threshold)值。以β-actin作为内参,采用2-△△Ct法,对目的基因miRNA表达进行相对定量。

1.8 下游靶信号通路mRNA的qRT-PCR检测
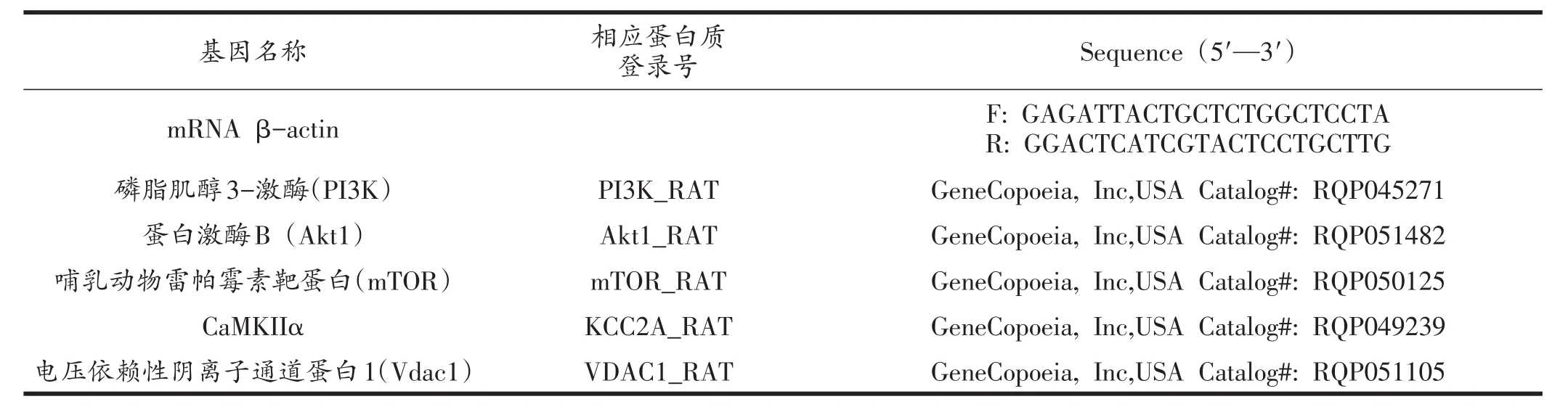
表3 qRT-PCR mRNA引物设计
1.9 统计学分析
所有数据均用SPSS16.0统计学软件进行处理。所有数据均采用平均值 ±标准差(±s)表示;各组间显著性差异采用方差分析、组内显著性差异用双侧t检验;本实验使用LSD法和SNK法进行多重比较;P< 0.05为具有统计学意义。
2 结果
2.1 体重监控情况
从图3可以得出,健康雄性SD大鼠3.0月龄(青年组)体重481.25±22.17 g、13月龄(中年组)体重547.75±21.94 g和23月龄(老年组)体重693.21± 68.85 g。各年龄安静组的大鼠体重随着实验时间而出现比较稳定的缓慢增加趋势,体重增加的变化无统计学意义。而各年龄运动组从第2周开始体重有下降趋势,趋势一直延续到实验结束,其中第3周中年安静组与运动组的大鼠体重相比较,差异有统计学意义(P<0.05);第4周开始到第10周,青年、中年和老年安静组与相应的运动组相比较,体重差异均具有统计学意义(P<0.05)。
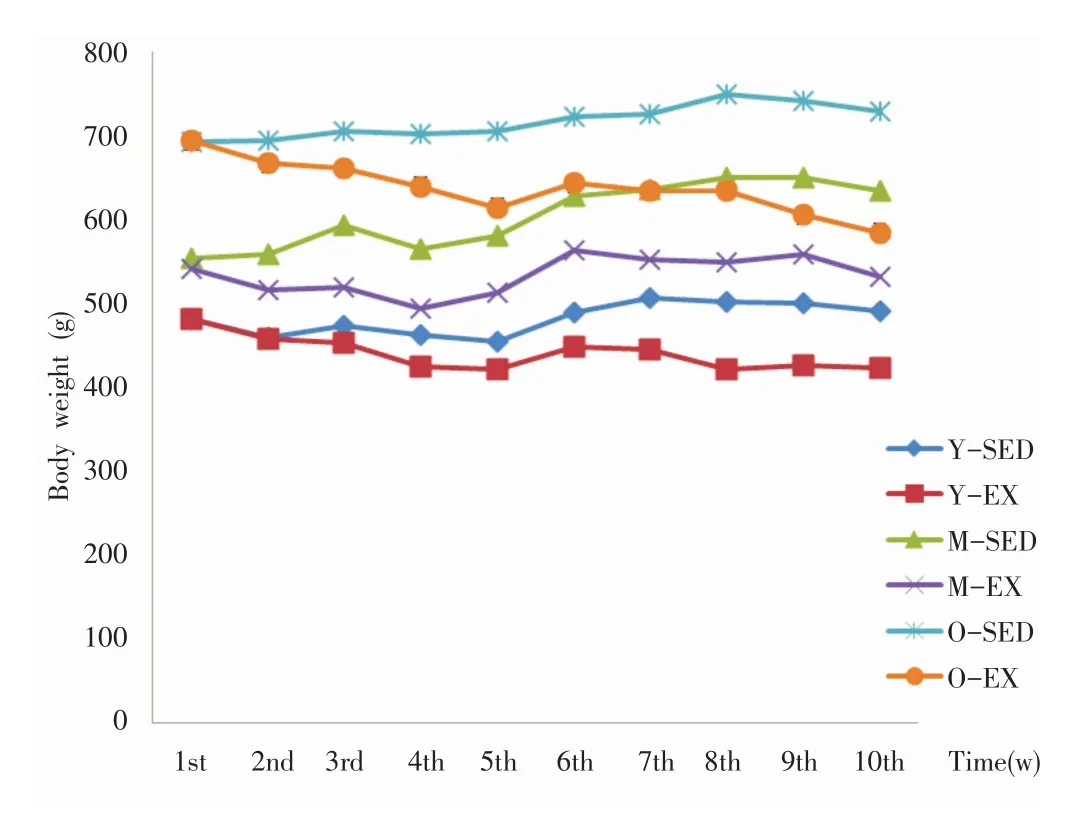
图3 增龄大鼠及运动干预对大鼠体重的监控
2.2 纹状体HE染色结果
从图4可以看出,Y-SED组大鼠纹状体的线团状的基质部分很明显,之间间隙明显,M-SED组则基质部分能明显分别出来,之间间隙比Y-SED组明显减小,而O-SED组难以辨出基质部分,之间间隙非常小。可见安静组明显呈现年龄增龄性变化。实施规律有氧运动后,各年龄组相应的出现线团状的基质部分明显紧凑,之间间隙非常紧密,显微镜下观察细胞核排列有序,数量明显增加,表明规律有氧运动改善了纹状体的形态结构。
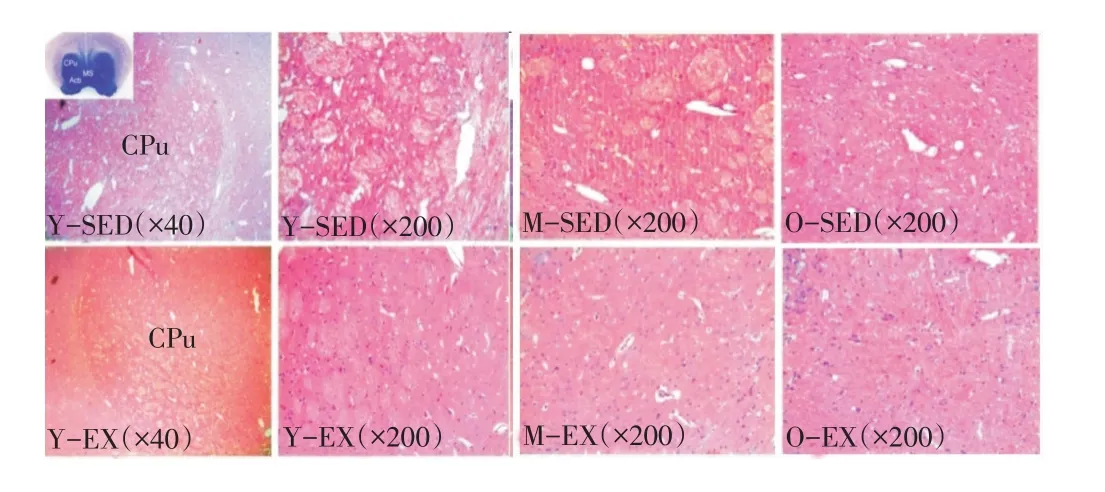
图4 增龄大鼠纹状体HE染色(其中CPu即为纹状体)
2.3 差异miRNAs的结果
本实验通过miRNA微阵列芯片数据筛选出发生2倍及以上变化差异的miRNAs 26个(见表4和5)。其中规律有氧运动上调2倍及以上大鼠纹状体的15个miRNAs:miR-207、miR-3593-3p和miR-106b-3p等;而下调2倍及以上的11个miRNAs:miR-542-3p、miR-363-5p和miR-141-5p等。
本实验对上述26个miRNAs分别进行生物信息学分析:靶点预测及其功能分析。通过表达量趋势配对、亚细胞定位筛选、靶标基因在已知疾病中的作用、通路筛选等提炼可能调控的靶基因对;使用转录因子预测、分子活性预测等模块,从靶标数据库中筛选出最可信的miRNA-mRNA相互作用网络;结果发现miRNA207和miRNA542符合本实验的研究需要,值得进一步研究。
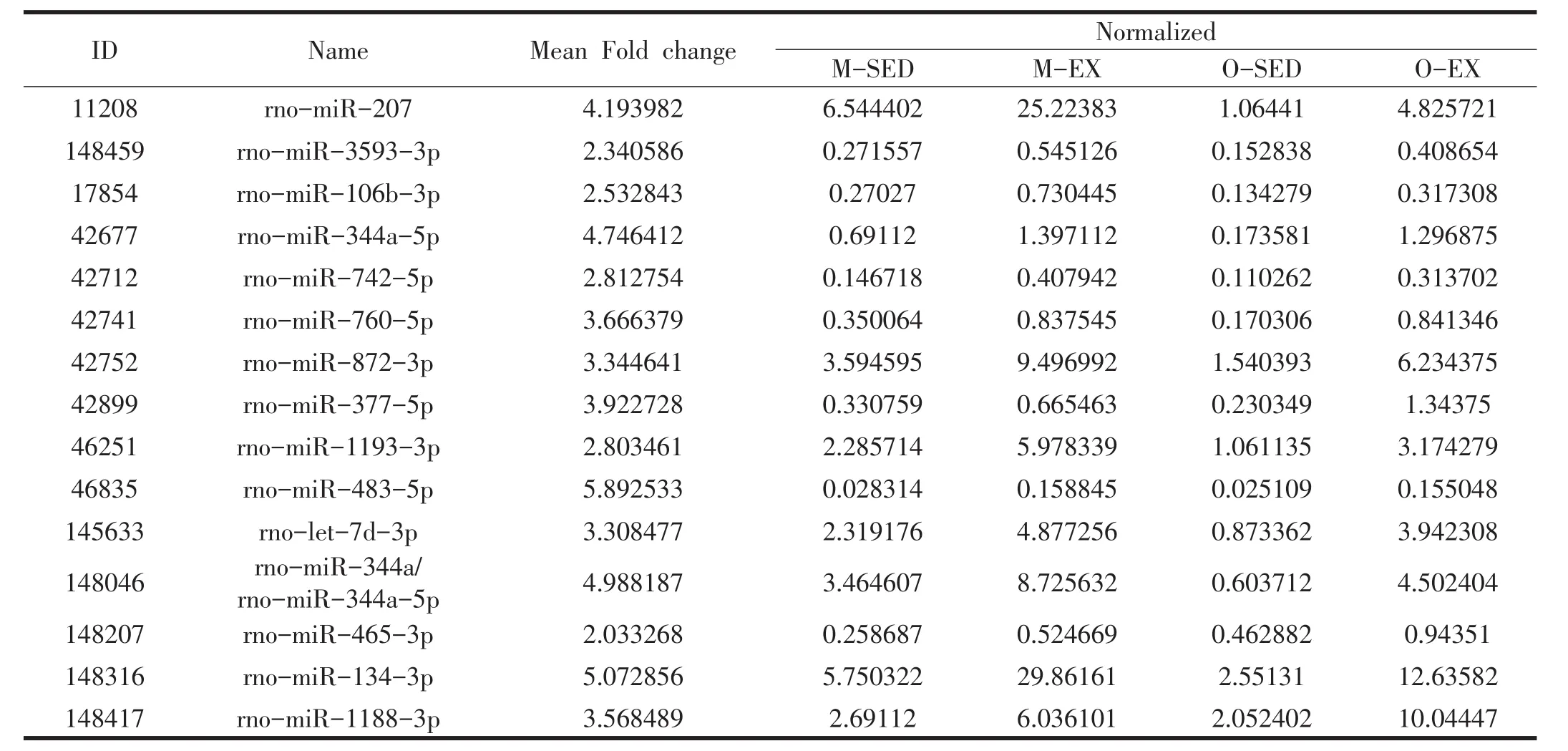
表4 筛选出大鼠纹状体运动组比安静组上调2倍及以上的miRNAs
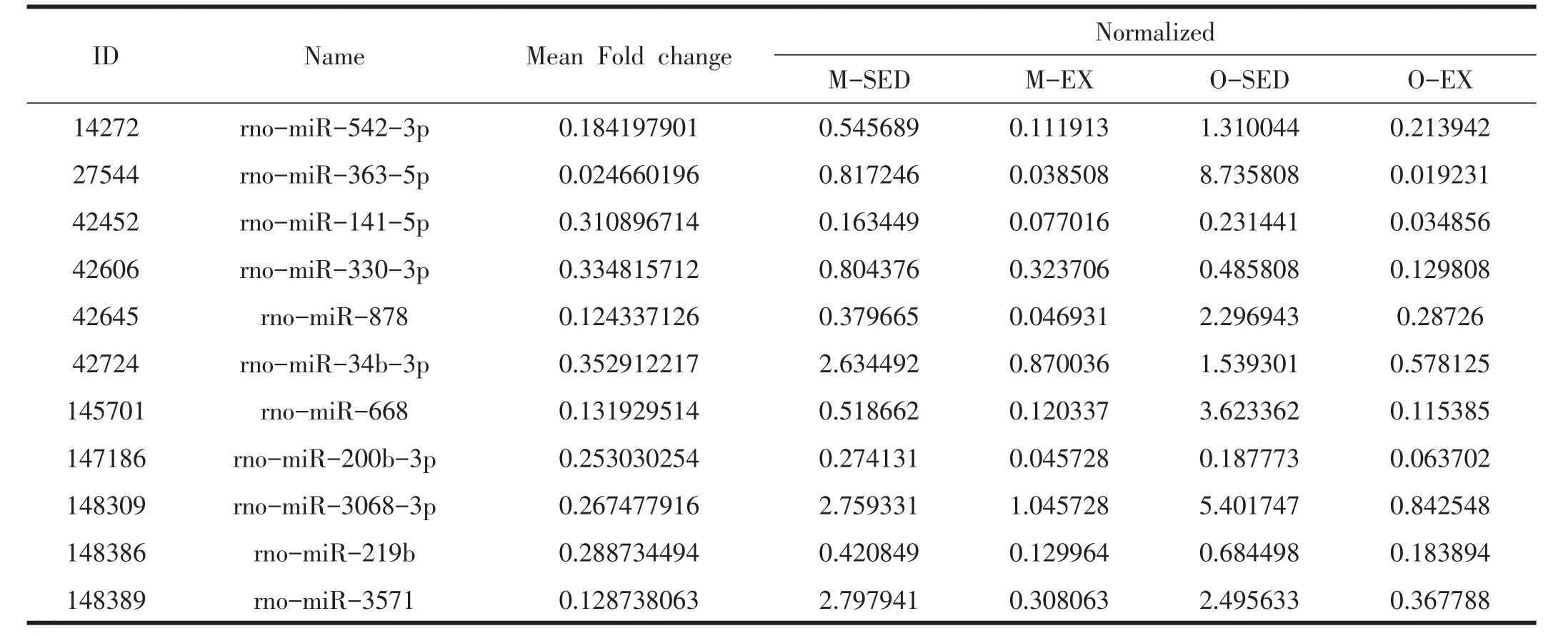
表5 筛选出大鼠纹状体运动组比安静组下调2倍及以上的miRNAs
2.4 miRNA207和miRNA542的生物信息学分析与下游靶基因预测
2.4.1 GO分析
根据GO生物信息学分析,预测miRNA207和miR⁃NA542所作用的靶标相关蛋白的功能等,靶标相关蛋白主要涉及到蛋白结合的蛋白质有122个、催化结合蛋白105个和阳离子结合蛋白61个等,主要参与AMP结合、神经递质转运活动、ATP合成和谷氨酸结合等生物学功能(图5)。96个蛋白参与生物合成、93个蛋白为氮有关活动蛋白、86个蛋白为转运蛋白、84个蛋白参与能量代谢、84个蛋白参与发育过程等。蛋白相关的生物功能涉及嘌呤核苷酸信号、Ca2+依赖的细胞外分泌正调控、细胞凋亡负调控、大脑行为应答和P53参与调控的DNA损坏应答调控等(图6)。
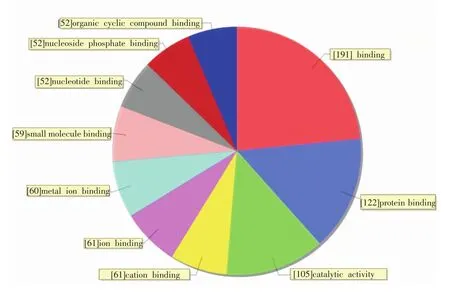
图5 GO分析得出大鼠纹状体miRNA的分子功能分类
2.4.2 KEGG生物学通路分析
由KEGG生物学通路分析可知miRNA207与钙信号依赖苏氨酸激酶(Cask)、突触素、1433蛋白和Bcl-2等蛋白有着密切关系;而miR-542-3p与谷氨酸结合蛋白、ATP结合蛋白和核内不均一核糖蛋白3等有着密切关系。经过KEGG生物学通路分析,对可能富集的生物学通路进行了预测,对符合P值者(EASE-score,Fisher-P value or Hypergeometric-P value)进行相关通路的预测,包括:神经活性配体-受体相互作用信号通 路 (Neuroactive ligand- receptor interaction,rno04080)、钙信号通路(Calcium signaling pathway,rno04020)和γ-氨基丁酸突触信号通路(GABAergic synapse,rno04727)等。这些统计学意义的生物学信号通路可能与神经老化或者神经退行有着密切关系。GO和KEGG生物学通路分析结果显示,运动对增龄大鼠纹状体的细胞死亡、突触结构重塑、氨基酸代谢、糖代谢和DNA损伤等的影响是本研究中miRNAs靶基因生物信息学分析的主要方向。
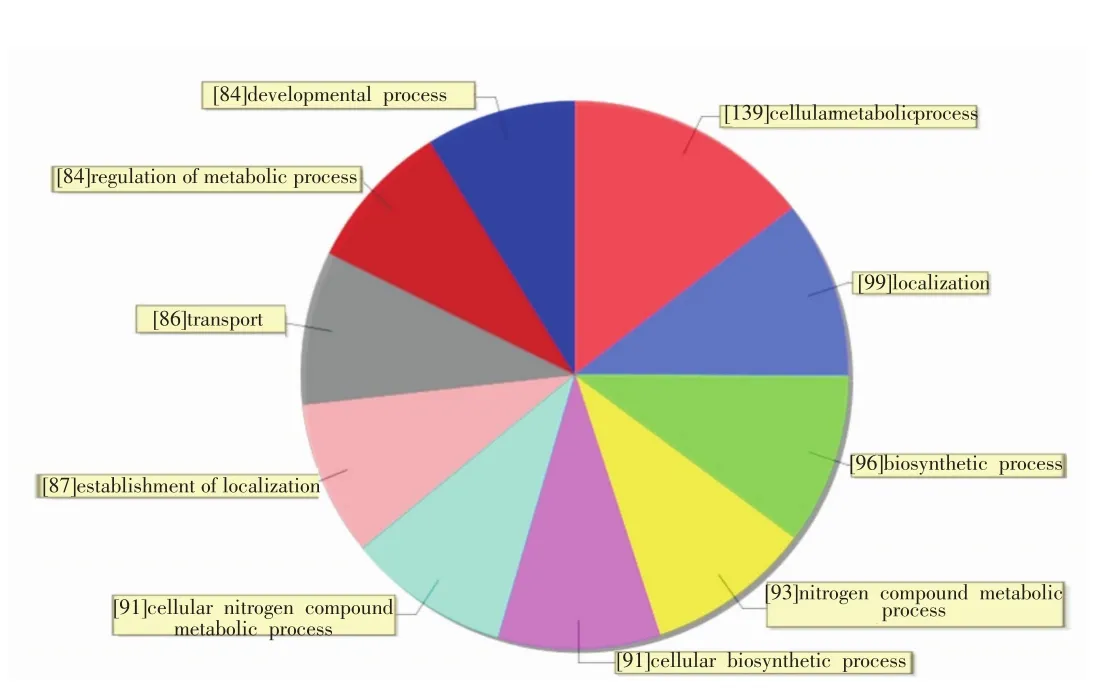
图6 GO分析得出大鼠纹状体miRNA的生物学功能分类
2.5 差异筛选miRNA207和miRNA542的验证
采用qRT-PCR验证差异表达的miRNA,根据前面靶基因预测与分析,选择上调miRNA207和下调miR⁃NA542进行验证,验证结果与芯片结果基本一致。
由图7可以看出,miRNA207表达呈现增龄性上升趋势。与Y-SED相比较,M-SED和O-SED的miR⁃ NA207表达上调显著(P<0.05),但是M-SED和O-SED之间没有统计学意义(P>0.05);各年龄安静组与运动组相比较,miRNA207表达上调显著(P<0.01)。
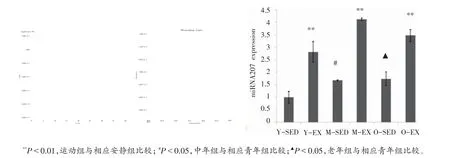
图7 qRT-PCR测定miRNA207扩增曲线和溶解曲线及其表达水平
从图8可以看出,miRNA542表达的变化呈现不明显的增龄性上升或下降趋势。与Y-SED相比,M-SED的miRNA542表达稍上调(P>0.05),而O-SED上调显著(P<0.01);与Y-SED相比,Y-EX的 miRNA542表达略增高(P>0.05);与M-SED相比,M-EX的miRNA542表达有所下降(P>0.05);与O-SED相比较,O-EX的miRNA542表达显著下调(P<0.01)。
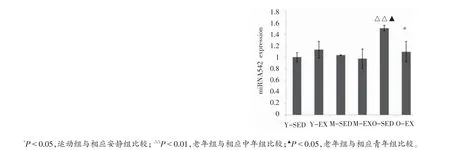
图8 qRT-PCR测定miRNA542扩增曲线和溶解曲线及其表达水平
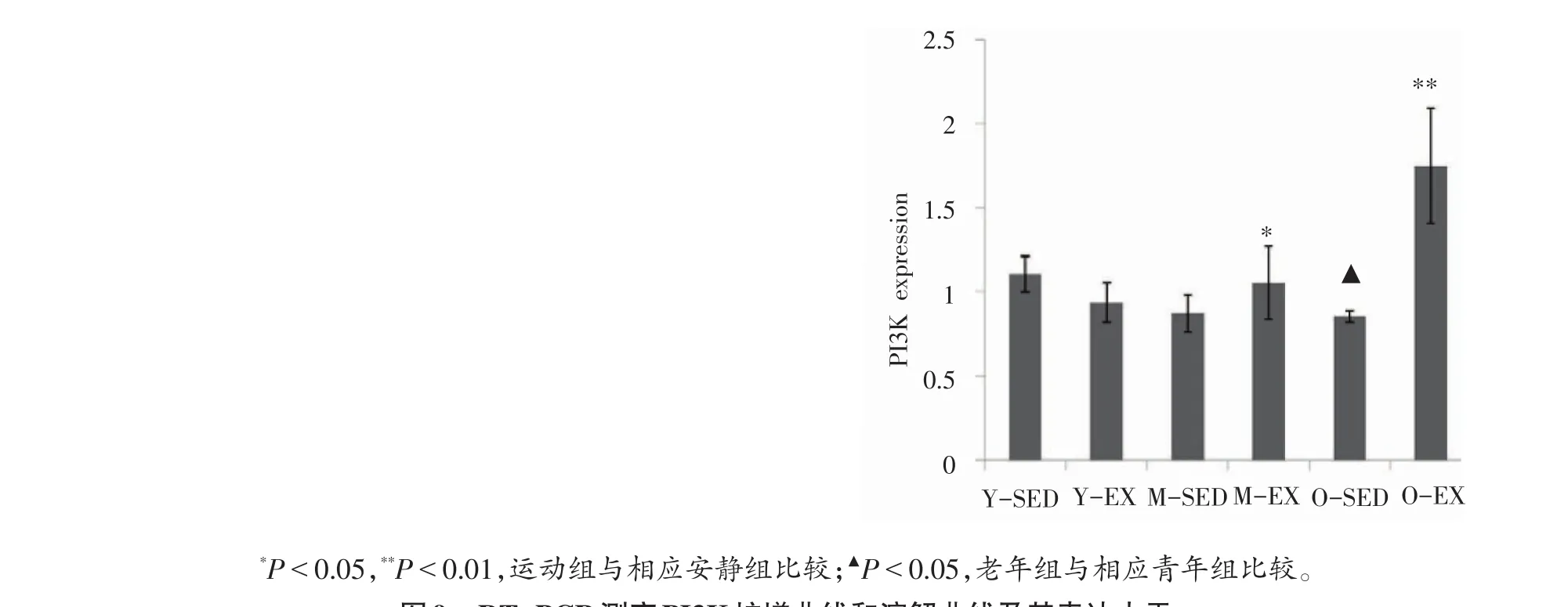
图9 qRT-PCR测定PI3K扩增曲线和溶解曲线及其表达水平
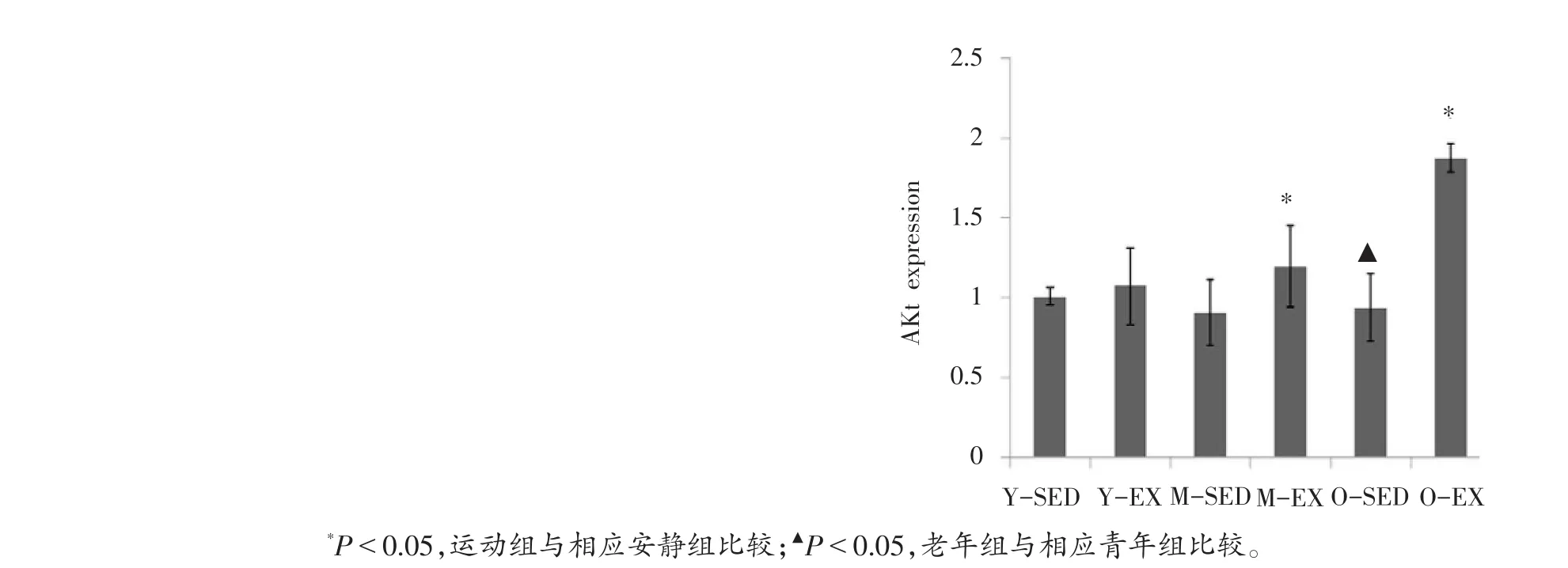
图10 qRT-PCR测定Akt扩增曲线和溶解曲线及其表达水平
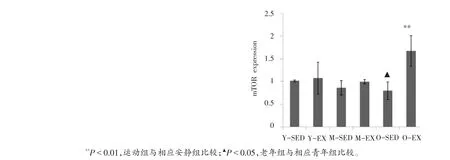
图11 qRT-PCR测定mTOR扩增曲线和溶解曲线及其表达水平
2.5 下游靶信号通路PI3K/Akt/mTOR表达水平
从图9-11可以看出,PI3K、Akt和mTOR mRNA表达呈现增龄性下调,与Y-SED相比较,O-SED的PI3K、Akt和mTOR mRNA表达下调显著(P<0.05)。与Y-SED相比,Y-EX的Akt和mTOR mRNA表达稍上调,而PI3K出现下调趋势,但没有统计学意义(P>0.05);与M-SED相比较,M-EX的PI3K、Akt和mTOR mRNA表达上调(P<0.05);与O-SED相比较,O-EX的PI3K、Akt和mTOR mRNA表达上调显著(P<0.01)。
2.6 下游靶信号通路CaMKIIα的影响
从图12和13可以看出,Vdac1 mRNA呈现增龄性上升趋势,与Y-SED相比较,M-SED和O-SED的CaMKIIα和Vdac1 mRNA表达均显著上调(P<0.05),但M-SED和O-SED之间比较没有统计学意义(P>0.05);各年龄安静组与运动组相比较,CaMKIIα和Vdac1 mRNA表达上调显著(P<0.05),其中与M-SED相比较,M-EX明显上调(P<0.01)。
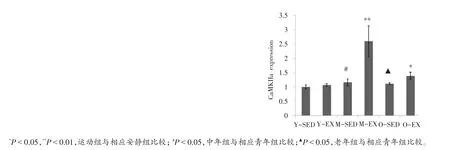
图12 qRT-PCR测定CaMKIIα扩增曲线和溶解曲线及其表达水平

图13 qRT-PCR测定Vdac1扩增曲线和溶解曲线及其表达水平
3 讨论
本实验发现规律运动上调2倍及以上的大鼠纹状体的15个miRNAs,如miR-207等;而下调2倍及以上的11个miRNAs,如miR-542-3p等。Tan等[27]研究发现miRNA207调控下游靶基因Akt3作用于DNA损伤与细胞凋亡的过程。PI3K/Akt/mTOR通路通过影响下游多种效应分子的活化状态,参与调节细胞增殖、凋亡、分化和代谢等一系列重要的生理活动,也与肿瘤细胞的增殖、分化、凋亡、化疗耐药性及血管生成密切相关[28]。本实验结果显示PI3K、Akt和mTOR mRNA表达呈现增龄性下调,表明增龄过程中,生存信号转导通路PI3K/Akt/mTOR随年龄的增长而下调;运动对各年龄大鼠PI3K、Akt和mTOR mRNA表达总体性上调,除青年运动组PI3K出现下调趋势(P>0.05),表明规律有氧运动能激活PI3K/Akt/mTOR信号通路。Fujita和Sato等[29,30]研究发现在抗凋亡途径PI3K/Akt激酶通路中,Akt激酶等一系列激酶均属于HSP90底物蛋白,HSP90及辅助伴侣分子Cdc37与Akt结合,使其活化发挥抗凋亡作用。Tao等[31]报道侧脑室注射miR-207激动剂后,神经功能缺损和脑梗死体积被减弱,同时线粒体棘结构被改善;此外,miR-207模拟物可以减少溶酶体和自噬体数量,增加自噬泡的数量;他们的研究结果提示miR-207可能影响自噬—溶酶体信号通路和线粒体诱导的凋亡。Long等[32]报道miR542-3p抑制癌细胞的增长与扩散并促进其凋亡。说明miR-207和miR542-3p都可能是研究运动改善神经老化的新的靶标分子,亟待进一步研究。
本实验结果表明运动上调增龄大鼠纹状体的miR⁃NA207表达和下调miRNA542表达;miRNA207和miR⁃NA542所作用的下游信号通路主要有:神经老化或者神经退行有着密切关系的神经活性配体—-受体相互作用信号通路、钙信号通路和γ-氨基丁酸突触信号通路等12条通路。同时,本实验通过生物学通路分析,得知miRNA207与钙信号依赖苏氨酸激酶(Cask)、突触素、1433蛋白和Bcl-2等蛋白表达有着密切关系,提示运动有利于改善脑老化,可能与突触蛋白相关联。CaMKII是脑内最为丰富的蛋白激酶之一,是调控钙信号等多条信号转导通道的重要分子,在突触可塑性、学习和记忆中具有关键作用[33,34]。本实验发现CaMKIIα和Vdac1 mRNA呈现增龄性趋势,与各年龄安静组相比较,运动组CaMKIIα和Vdac1 mRNA表达上调显著。已有实验证实CaMKII-α缺乏的转基因小鼠表现出显著的突触可塑性缺陷和记忆形成障碍[35]。CaM⁃KII-α被发现同时存在于突触前和突触后部位,突触前的CaMKII-α主要参与了突触小泡群集和神经递质释放的调控,位于突触后部位的CaMKII-α则参与调节NMDA受体的表达水平及其向突触后膜的定位。Foster等[36]将老化脑中钙调神经素(Calcineurin,CN)活性变化与胞内Ca2+浓度的变化联系了起来,他们证明其在学习记忆中有重要作用,参与了大脑神经元突触效应的去增强、多种不同机制的长时程增强、长时程抑制、认知记忆、短期记忆向长期记忆的转换、脑老化等过程[37]。
4 结论
规律运动通过上调miRNA207和下调miRNA542的表达而激活大鼠纹状体的PI3K/Akt/mTOR和CaM⁃KIIα信号通路共同参与调控纹状体的生物学功能和改善神经老化。
[1] Van RooijE.Theartofmicrornaresearch [J].Circ Res,2011,108(2):219-234.
[2] Wienholds E,Kloosterman WP,Miska E,et al.Micror⁃na expression in zebrafish embryonic development[J]. Science,2005,309(5732):310-311.
[3] Wittmann J,Jack HM.Micrornas in rheumatoid arthri⁃tis:Midget rnas with a giant impact[J].Ann Rheum Dis,2011,70 Suppl 1(i92-96).
[4] Abe M,Naqvi A,Hendriks GJ,et al.Impact of ageassociated increase in 2'-o-methylation of mirnas on ag⁃ing and neurodegeneration in drosophila[J].Genes Dev,2014,28(1):44-57.
[5] Lau P,Bossers K,Janky R,et al.Alteration of the mi⁃crorna network during the progression of alzheimer's dis⁃ease[J].EMBO Mol Med,2013,5(10):1613-1634.
[6] Persengiev SP,Kondova,II,Bontrop RE.The impact of micrornas on brain aging and neurodegeneration[J]. Curr Gerontol Geriatr Res,2012,2012(3):359369.
[7] Van den Hove DL,Kompotis K,Lardenoije R,et al. Epigenetically regulated micrornas in alzheimer's disease [J].Neurobiol Aging,2014,35(4):731-745.
[8] Yin L,Sun Y,Wu J,et al.Discovering novel micror⁃nas and age-related nonlinear changes in rat brains us⁃ing deep sequencing[J].Neurobiol Aging,2015,36(2):1037-1044.
[9] Mi H,Muruganujan A,Casagrande JT,et al.Largescale gene function analysis with the panther classifica⁃tion system[J].Nat Protoc,2013,8(8):1551-1566.
[10]Hu X,Duan Z,Hu H,et al.Proteomic profile of car⁃bonylated proteins in rat liver:Exercise attenuated oxida⁃tive stress may be involved in fatty liver improvement [J].Proteomics,2013,13(10-11):1755-1764.
[11]韩雨梅,刘子泉,常永霞,等.有氧耐力训练对增龄大鼠骨骼肌线粒体生物合成的影响 [J].中国运动医学杂志,2010,29(4):425-429.
[12]Glowinski J,Axelrod J,Iversen LL.Regional studies of catecholamines in the rat brain.Iv.Effects of drugs on the disposition and metabolism of h3-norepinephrine and h3-dopamine[J].J Pharmacol Exp Ther,1966,153(1):30-41.
[13]Glowinski J,Iversen LL.Regional studies of catechol⁃amines in the rat brain.I.The disposition of[3h]norepi⁃nephrine,[3h]dopamine and[3h]dopa in various regions of the brain[J].J Neurochem,1966,13(8):655-669.
[14]Watson原著,诸葛启钏主译 GP.大鼠脑立体定位图谱[m][M].北京:人民卫生出版社(第三版),2005.
[15]Liu Y,Lai L,Chen Q,et al.Microrna-494 is re⁃quired for the accumulation and functions of tumor-ex⁃panded myeloid-derived suppressor cells via targeting of pten[J].J Immunol,2012,188(11):5500-5510.
[16]Wu L,Zhao Q,Zhu X,et al.A novel function of mi⁃crorna let-7d in regulation of galectin-3 expression in at⁃tention deficit hyperactivity disorder rat brain[J].Brain pathology,2010,20(6):1042-1054.
[17]Anders S,Huber W.Differential expression analysis for sequence count data[J].Genome Biol,2010,11(10):R106.
[18]Lewis BP,Shih IH,Jones-Rhoades MW,et al.Predic⁃tion of mammalian microrna targets[J].Cell,2003,115(7):787-798.
[19]Ashburner M,Ball CA,Blake JA,et al.Gene ontolo⁃gy:Tool for the unification of biology.The gene ontolo⁃gy consortium[J].Nature genetics,2000,25(1):25-29.
[20]Wang X.Mirdb:A microrna target prediction and func⁃tional annotation database with a wiki interface[J].Rna,2008,14(6):1012-1017.
[21]Wang X,El Naqa IM.Prediction of both conserved and nonconserved microrna targets in animals[J].Bioinformat⁃ics,2008,24(3):325-332.
[22]Huang da W,Sherman BT,Lempicki RA.Bioinformat⁃icsenrichmenttools:Pathstoward thecomprehensive functional analysis of large gene lists[J].Nucleic Acids Res,2009,37(1):1-13.
[23]Huang da W,Sherman BT,Lempicki RA.Systematic and integrative analysis of large gene lists using david bioinformatics resources[J].Nat Protoc,2009,4(1):44-57.
[24]Blandini F,Nappi G,Tassorelli C,et al.Functional chang⁃es of the basal ganglia circuitry in parkinson's disease [J].Prog Neurobiol,2000,62(1):63-88.
[25]Macaya A,Munell F,Gubits RM,et al.Apoptosis in substantia nigra following developmental striatal excitotox⁃ic injury[J].Proc Natl Acad Sci USA,1994,91(17):8117-8121.
[26]Kanehisa M,Goto S,Hattori M,et al.From genomics to chemical genomics:New developments in kegg[J]. Nucleic Acids Res,2006,34(Database issue):D354-357.
[27]P-x Tan,S-s Du,C Ren,et al.MicroRNA-207 enhances radiation-induced apoptosis by directly targeting akt3 in cochlea hair cells[J].Cell Death Dis,2014,5:1-8
[28]Morgensztern D,McLeod HL.Pi3k/akt/mtor pathway as a target for cancer therapy[J].Anti-cancer drugs,2005,16(8):797-803.
[29]Fujita N,Sato S,Katayama K,et al.Akt-dependent phosphorylation of p27kip1 promotes binding to 14-3-3 and cytoplasmic localization[J].J Biol Chem,2002,277(32):28706-28713.
[30]Sato S,Fujita N,Tsuruo T.Modulation of akt kinase activity by binding to hsp90 [J].Proc Natl Acad Sci USA,2000,97(20):10832-10837.
[31]Tao J,Liu W,Shang G,et al.MiR-207/352 regulate ly⁃sosomal-associated membrane proteins and enzymes fol⁃lowing ischemic stroke[J].Neuroscience,2015,305:1-14.
[32]Long HC,Gao X,Lei CJ,et al.miR-542-3p inhibits the growth and invasion of colorectal cancer cells through targeted regulation of cortactin [J].Int J Mol Med,2016,37(4):1112-1118.
[33]Ghosh A,Greenberg ME.Calcium signaling in neurons:Molecular mechanisms and cellular consequences[J].Sci⁃ence,1995,268(5208):239-247.
[34]Malenka RC,Nicoll RA.Long-term potentiationa de⁃cade of progress?[J].Science,1999,285(5435):1870-1874.
[35]Miller S,Yasuda M,Coats JK,et al.Disruption of den⁃dritic translation of camkiialpha impairs stabilization of synaptic plasticity and memory consolidation[J].Neuron, 2002,36(3):507-519.
[36]Foster TC,Sharrow KM,Masse JR,et al.Calcineurin links ca2+dysregulation with brain aging[J].J Neurosci,2001,21(11):4066-4073.
[37]Sola C,Tusell JM,Serratosa J.Comparative study of the distribution of calmodulin kinase ii and calcineurin in the mouse brain[J].J Neurosci Res,1999,57(5):651-662.
The Effect of Regular Aerobic Exercise on the Expression of miRNA207 and miRNA542 and Downstream Target Signal Transduction in the Striatum of Rats
Liu Wenfeng1,2,Yin Dazhong1,2,Tang Changfa1,Tang Lihua1,Chen Zongping1
1 Hunan Provincial Key Laboratory of Physical Fitness and Sports Rehabilitation,Hunan Normal University,Changsha 410012,China
2 The Key Laboratory of Protein Chemistry and Developmental Biology of Ministry of Education,Hunan Normal University,Changsha 410081,China Corresponding Author:Liu Wenfeng,wfliu@hunnu.edu.cn
ObjectiveTo explore the new targets of the impact of exercise on the striatal in the ear⁃ly neurodegeneration among rats of different ages with regular aerobic exercise by means of the miR⁃NA expression profile chip technology,and explain the key biochemical and molecular regulation mech⁃anism of the new target genes.MethodsThree,13 and 22-month-old specific pathogen free(SPF)male Sprague-Dawley Rat(SD)rats were randomly divided into a young(Y-SED,n=10),a middleaged(M-SED,n=10)and an old-aged(O-SED,n=12)sedentary control group,and the correspond⁃ing Y-EX(n=10),M-EX(n=10)and O-EX(n=12)in the aerobic exercise group.The rats in the sedentary control groups didn’t exercise,while those in the aerobic exercise groups underwent a 10-week regular moderate-intensity aerobic exercise on treadmill.The rats’body weight was measured ev⁃ery week.HE staining was used to present the morphological changes in the striatum.The miRNA mi⁃croarray of miRNA miRCURYTM LNA Array(v.18.0)(Exiqon,Danish)was employed to analyze the expression profile of miRNAs.The qRT-PCR was performed to screen and to verify the related signal pathways for the biochemical mechanisms.ResultsThe morphology of the rats’striatum changed clear⁃ly with ages.The nuclei in the striatum were arranged orderly and the number of nuclei increased sig⁃nificantly in each group undergoing regular aerobic exercise.miRNA microarray data showed that 26 miRNAs increased to no less than twice of the original level.The expression of miRNA207 was found up-regulated while that of miRNA542 was down-regulated after regular aerobic exercise through screen⁃ing the bioinformatics of miRNA.The expression of PI3K,Akt and mTOR mRNA,downstream genes of miRNA207 and miRNA542,was also found down-regulated with the increase of age.However,com⁃pared with the sedentary groups,the expression of PI3K,Akt and mTOR was higher in the exercise groups.The Vdac1 mRNA was observed an increasing tendency with age.Compared with all the seden⁃tary groups,significant upregulation was observed in the expression of CaMKIIα and Vdac1 in the aer⁃obic exercise groups.ConclusionRegular aerobic exercise can regulate the biological function of rats’striatum and alleviate the aging process through upregulating the expression of miRNA207 and downreg⁃ulating that of miRNA542 to activate the PI3K/Akt/mTOR and CaMKIIα signaling pathway.
aerobic exercise,striatum,aging,miRNA207,miRNA542,PI3K,Akt,mTOR,CaM⁃KIIα
2016.08.10
湖南师范大学青年优秀人才培养计划(ET1507);国家自然科学基金(31271257);国家高科技 863计划(2008AA02Z411);湖南省自然科学基金(11JJ6082);湖南教育厅优秀青年基金(12B088)
刘文锋,Email:wfliu@hunnu.edu.cn

