基于等高线的染色体高度曲线提取方法*
马立 李丰甜 刘波 陈瑞华 汝长海
(1.上海大学 机电工程与自动化学院, 上海 20007; 2; 苏州大学 附属第一医院, 江苏 苏州 215001;3.苏州大学 机器人与微系统研究中心, 江苏 苏州 215001)
染色体核型分析是指染色体组在有丝分裂中期的表型分析,该阶段染色体的结构和数目变异可以为染色体异常引起的疾病诊断提供很好的依据,因此,核型分析在遗传学研究及医学诊断中都具有重要的价值[1- 2].
基于原子力显微镜(AFM)的染色体早期研究[3- 5]发现,染色体表面的高度分布并不均匀,波峰与波谷交替分布,且沿染色体分布的峰谷与显带后的染色体显隐带区域之间存在对应关系[6- 7].这一结果表明,显带是染色体的固有结构,显带处理只是使这种结构在光镜下可见[8].因此,在染色体核型分析中,沿染色体中心线的高度曲线最常被用来与光学显微镜(OM)的成像结果进行对比分析.目前许多研究中使用的高度曲线提取及相应的显带分析方法[9- 10]是:首先通过软件对形貌图进行处理,然后基于操作者的视觉,沿染色体中心线方向画一条直线并提取该目标直线上的高度分布,最后将其与对应的显带区段进行对比研究[11].这种高度曲线与OM显带结果之间的对应分析存在两个问题:第一,忽视了染色体样品制备过程中产生的变形,导致所提取的高度曲线不准确;第二,由于受分辨率及其他差异的影响,在基于操作者视觉的高度曲线分析中,虽然很多研究都提到了显隐带与峰谷间的对应关系,但是这种关系并不严谨.
考虑到传统方法的不足,文中参考等高线在地形分析中的应用,以染色体表面峰值点与显带的对应关系为前提,在现有AFM[12- 14]研究的基础上提出一种基于等高线的染色体高度曲线提取方法,并通过实验进行验证.
1 实验样品及设备
液体环境下的染色体表面柔软且结构不稳定,相对而言,空气中干燥的染色体,其表面的结构刚性与稳定性都更为理想.利用AFM对空气中干燥的染色体样品进行成像能够获得更高的分辨率,且样品的显带处理过程通常在空气中完成.为了更好地分析染色体的表面形貌结构并与显带结果形成对应,选用空气中制备的染色体样品作为研究对象,采集健康供体外周静脉血5 mL,抽取肝素0.2 mL抗凝,置于37 ℃恒温培养箱中培养72 h.终止培养前2~4 h加入秋水仙素,使溶液最终质量浓度达0.2 μg/mL.细胞悬液经离心、低渗、固定后,滴2~3滴于冰水浸泡过的载玻片上,吹散,过火后气干.
为了实现染色体目标区域的定位,染色体样品制备结束后用记号笔在载玻片的背面做多个标记点.利用相差显微镜对载玻片进行观察,从中挑选分散较好的染色体区域用于后续的AFM成像.调整分辨率,确定标记点区域后截图,并将目标区域用玻璃刀分别切下5 mm×5 mm~10 mm×10 mm大小的方块.实验系统实物如图1所示,研究中选用了Nanonic公司的多功能型AFM(Multiview 1000),水平和竖直方向的测量分辨率分别为0.005和0.002 nm,该显微镜配备了一个高分辨率的光学显微镜,可对样品表面进行高分辨率的光学成像.在利用AFM对制备的染色体样品进行成像前,将配套光镜的成像结果与之前保存的相差显微镜成像结果进行对比,确定目标区域所处的位置.扫描头用于放置样品并完成扫描过程,集成控制系统负责扫描过程中的运动控制,电脑及相关软件用于完成相关参数的设置以及扫描结果的处理、显示.AFM以轻敲模式进行成像,设定点为探针共振峰值的80%.成像用探针为Nanonic公司的NanoToolKit光纤探针,针头直径为20 nm左右,探针的弹性系数为1 N/m,推荐谐振频率为25~70 Hz.WSxM[15]为扫描探针显微镜配套的图像处理软件,用于完成高分辨率形貌图的处理,文中采用WSxM完成对AFM所得到图像的预处理及辅助完成染色体轮廓的分析测量.

图1 实验系统实物图
2 高度曲线提取方法
等高线是高度相同的点连接所形成的封闭曲线,可以清晰地反映形貌图中的高度变化及波峰波谷的分布情况.基于等高线的高度曲线提取方法的原理为:将等高线描述地貌的原理用于染色体,即通过封闭曲线将染色体表面的三维形貌描述出来,进而得到所研究染色体的形貌走势,并提取所需的峰值点.
图2(a)所示为未经仔细筛选且表面在一定程度上受到细胞浆质干扰的染色体样品.传统的高度曲线提取方法为:沿染色体的中心线画一条直线(如图2(b)所示),然后通过计算直线上各点的坐标及相对位置得到最终的高度曲线(如图2(c)所示).这种方法在染色体样品比较直的情况下能够得到与模式图对应较好的高度曲线.然而在染色体干燥过程中,染色体表面在应力作用下会发生不规则的收缩,收缩后的染色体形状不规则,如果仍采用画直线的方法,所得到的染色体中心线高度曲线不准确.
基于等高线的高度曲线提取方法的原理如图2(d)所示.具体步骤如下:
1)利用WSxM处理扫描结果,经分析得到所研究染色体的等高线图;
2)通过等高线分辨出染色体干燥过程中的收缩方向,同时,利用显微镜配套处理软件中的像素彩色查找方法来反映染色体形貌图中的高度变化,再结合等高线较为方便地确定轮廓,并清晰显示干燥后染色体样品的波峰点;
3)在确定染色体收缩方向的情况下,通过反复调节等高线的高度间隔和等高线数,直到等高线图上出现的波峰个数与相应染色体显带模式图上的波峰个数相近或相等时,停止调整,从而较精确地找到其几何中心点(即峰值点);
4)通过一条折线连接染色体收缩方向上所有的峰值点,将其作为染色单体的中心线;
5)通过提取并分析沿着中心线的高度及坐标信息,得沿染色单体中心线的高度曲线,如图2(e)所示.

图2 基于等高线的高度曲线提取方法原理图
调节等高线的高度间隔和等高线数等参数不一定能够得到理想的峰值点分布图,以图3所示染色体为例对染色体峰值点分布情况进行介绍.图3为经WSxM预处理去除背景干扰后,显示等高线所得到的染色体形貌图,其中由虚线标示出来的区域分别对应3种峰值点分布情况,黑点为等高线轮廓的几何中心,配合显微镜处理软件的像素彩色查找可确定为沿染色体分布的峰值点.区域f内的峰值点分布较为接近且连接线与染色单体的轮廓中心较为匹配,因此可直接连接.区域g内的峰值点分布较为分散,如果将其直接连接,连接线与单体的轮廓中心偏差较大,因此,区域g内峰值点间应依照轮廓边缘的趋势,通过一条折线进行连接.区域h内的峰值点分布虽然较为接近,但是它们的连线与染色单体的轮廓中心偏差较大,这些峰值点由染色体表面所存在的干扰结构所导致,在连接时应当舍去.端点选择在染色单体两端附近的位置,并且使折线的最前面两段与最后面两段在方向上保持一致.以上基于等高线的峰值点判断方法所得到的峰值点数与显带后染色体显隐带的个数基本符合,因此能保证充分且准确的染色体高度信息.
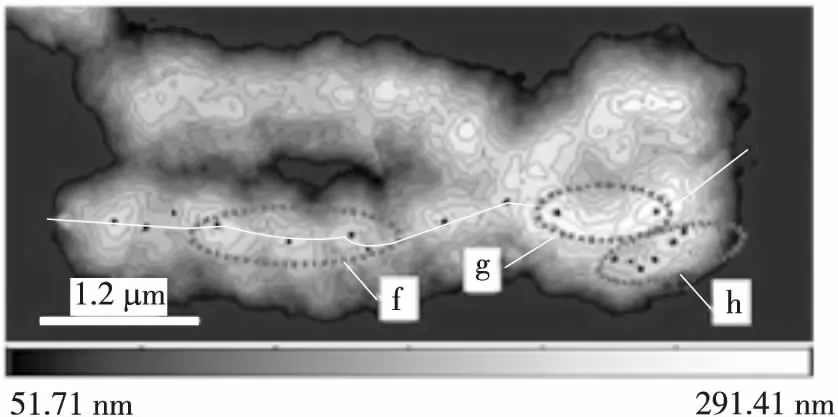
图3 沿染色体表面的峰值点分布
3 实验结果与分析

图4 染色体3D效果图
图4为利用AFM对实验过程中制备的染色体进行闭环扫描成像所得到的形貌图.扫描模式为轻敲模式,每行扫描点数为300,图像行数为300,空间分辨力为X0.005 nm、Y0.005 nm、Z0.002 nm,扫描时间为18 min.观察图像发现,染色体周边分布着大量细小的颗粒状结构,这是样品制备过程中细胞器等杂质及载玻片表面显微结构共同作用的结果.在染色体区域可以较明显地看到高低起伏结构,测量发现染色体的平均高度为150~300 nm,染色体表面分布的波峰与波谷间的平均高度差约为10 nm.
实验过程中,在利用等高线寻找染色体表面的峰值点时,需要多次调整等高线的高度间隔和等高线数等参数,以获得较好的结果.在调节等高线相关参数的过程中,前述图3中区域g与h所示的情况占较大比例,而区域f所示的情况出现频率较小.分析认为,这是由于染色体表面存在的特征峰值点与周边区域的高度差较小而引起.从所制备的染色体样品中挑选10条染色体进行方法有效性的对比,其中较直以及弯曲较为严重的染色体各5条.以图5中所示的姐妹染色体为例,采用常规方法以及文中提出的方法分别提取上下两条单体上的高度曲线.通过WSxM预处理去除形貌图中的背景干扰,并显示出形貌图中的等高线分布,得到如图5(a)所示结果.为得到较为理想的峰值分布图,对等高线的参数进行了相应的调整,最大高度为231.161 nm,最小高度为0 nm,中间共有10层的等高线.图5(a)中的a1、b1代表常规高度曲线提取方法,a2、b2代表基于等高线的高度曲线提取方法.采用上述两种方法提取到的图5(a)中上方与下方染色单体的高度曲线分别标记为a1、a2、b1、b2,如图5(b)和5(c)所示.
如图5(b)所示,a1与a2两条高度曲线呈现出较多相似的特征,但是其中一些特征的位置及细节存在一定差异.分析认为,图5(a)中上方的染色单体相对较直,从而导致两种方法所得到的高度曲线存在较大相似性;而在单体的一些区段染色体出现变形,从而导致高度曲线的特征存在一定差异.从图5(b)可以很明显地看出,虚线框内a1与a2之间的差异最大,a1偏离染色体的中心区域,但其路径仍沿着染色体的中心区域.另外,图5(b)虚线框区域中两高度曲线所表现出来的特征差异明显大于其他区段,其中a2呈现出更多的高度变化,且两曲线所提取到的高度值相差也较大.由图5(a)可以发现,下方的染色单体存在较为严重的变形,因而上述差异在图5(c)中表现得更为明显——b1部分区段完全偏离了染色体的中心区域,而基于等高线提取的高度曲线b2与染色体所发生的变形保持了很好的一致性.另外,从图5(c)中可以明显看出高度曲线b1与b2之间的差异——b1存在明显的特征错误,并且在偏离染色体的区域没有得到有价值的高度信息;而b2与染色体的变形保持较好的一致性,所提取到的高度曲线能很好地反映下方染色单体的高度变化情况.
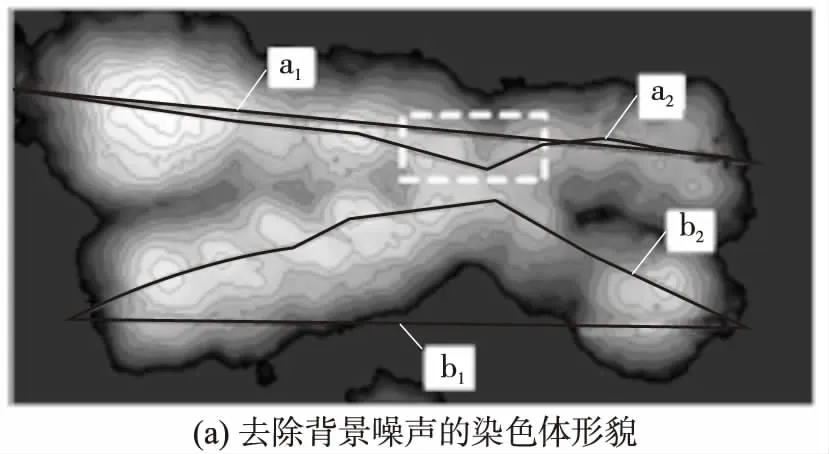
图5 染色体及其高度曲线对比
姐妹染色单体是染色体复制的结果,理论上讲,从两条染色单体上提取到的高度曲线应当存在较大的相似性.图5(b)所示的高度曲线a2与图5(c)所示高度曲线b2是利用新方法得到的两染色单体(如图5(a)所示)上的高度曲线,经对比分析发现,两曲线的高度特征存在明显差异.通过分析染色体表面的细微结构发现,染色体制备过程中样品的收缩及杂质的存在对染色体表面的高度分布情况造成了较大干扰.
空气中干燥的染色体样品表面平缓,表现出来的高度特征数目较少且不明显,因而容易受到制片条件的影响.在对较直的染色体样品进行对比分析时发现,常规方法所提取到的高度曲线与基于等高线的方法所提取到的高度曲线相似水平较高.当所分析的染色体存在较大弯曲变形时,传统的方法无法保证所有区段的取样点都在染色体中心区域,文中所提出的方法则能够保证取样点所分布的折线与染色体中心轮廓保持良好的一致性.基于等高线的高度曲线提取方法所得到的折线与染色体的轮廓重合度较好,能够更为准确地反映染色体的表面特征.
4 结语
文中提出了一种借助染色体形貌图上等高线的分布情况获取染色单体中心线上高度曲线的方法,并以空气中制备的染色体样品为研究对象,分别利用常规方法与基于等高线的方法提取目标染色体表面的高度曲线.通过对比两种方法得到的高度曲线发现,基于等高线的高度曲线能更准确地得到染色体中心区域的高度分布情况,并且得到的特征更为明显.在染色体弯曲变形较严重的情况下,文中提出的高度曲线提取方法所得到的结果更为准确.但是,目前利用实验中采用的样品制备方法所得到的染色体样品,其表面只有很少的可分辨特征,且表面受杂质及变形的干扰较大,这对高度曲线分析造成了一定干扰.如何在不影响染色体结构的前提下,尽可能地暴露出染色体内部的高度差异,是未来样品制备过程中需要解决的问题.
:
[1] NEETHIRAJAN S,HIROSE T,WAKAYAMA J,et al.Karyotype analysis of buckwheat using atomic force microscopy [J].Microscopy and Microanalysis,2011,17(4):572- 577.
[2] CHENG D H,GONG F,TAN K,et al.Karyotype determination and reproductive guidance for short stature women with a hidden Y chromosome fragment [J].Reproductive Biomedicine Online,2013,27(1):89- 95.
[3] KOLEVA V P,DRAGOEVA A P,ANDREEVA A I,et al.Comparative analysis of clastogen-induced chromosome aberrations observed with light microscopy and by means of atomic force microscopy [J].Mutation Research-Genetic Toxicology and Environmental Mutagenesis,2013,753(1):29- 35.
[4] DABAN J R.Electron microscopy and atomic force microscopy studies of chromatin and metaphase chromosome structure [J].Micron,2011,42(8):733- 750.
[5] LONERGAN N E,BRITT L D,SULLIVAN C J.Immobilizing live Escherichia coli for AFM studies of surface dynamics [J].Ultramicroscopy,2014,137:30- 39.
[6] USHIKE T,HOSHI O.Atomic force microscopy for imaging human metaphase chromosomes [J].Chromosome Research,2008,16(3):383- 396.
[7] HOSHI O,USHIKI T.Structural analysis of human chromosomes by atomic force and light microscopy in relation to the distribution of topoisomerase IIα[J].Archives of Histology and Cytology,2009,72(4/5):245- 249.
[8] TAMOYO J.Structure of human chromosomes studied by atomic force microscopy Part II:relationship between structure and cytogenetic bands [J].Journal of Structural Biology,2003,141(3):189- 197.
[9] THALHAMMER S,KOEHLER U,STARK R W,et al.GTG banding pattern on human metaphase chromosomes revealed by high resolution atomic-force microscopy [J].J Microsc,2001,202(Pt 3):464- 467.
[10] DUCKER W,SENDEN P.Direct measurement of colloidal forces using an atomic force microscopy [J].Nature,1991,353:239- 241.
[11] HOSHI O,USHIKI T.Three-dimensional structure of G-banded human metaphase chromosomes observed by atomic force microscopy [J].Archives of Histology and Cytology,2001,64(5):475- 482.
[12] KASAS S,LONGO G,DIETLER G.Mechanical properties of biological specimens explored by atomic force microscopy [J].Journal of Physics D:Applied Physics,2013,46:133001/1- 13.
[13] FORMOSA C,PILLET F,SCHIAVONE M,et al.Generation of living cell arrays for atomic force microscopy studies [J].Nature Protocols,2015,10(1):199- 204.
[14] KALLE W,STRAPPE P.Atomic force microscopy on chromosomes,chromatin and DNA:a review [J].Micron,2012,43(12):1224- 1231.
[15] HORCAS I,FERNANDEZ R,GOMEZ-RODRIGUEZ J M,et al.WSXM:a software for scanning probe microscopy and a tool for nanotechnology [J].Review of Scienti-fic Instruments,2007,78(1):013705.

