利用geNorm、NormFinder和 BestKeeper软件进行内参基因稳定性分析的方法
吴建阳+何冰+杜玉洁++李伟才+魏永赞


摘要 实时荧光定量PCR(RT-qPCR)由于具备灵敏度高、重复性好、特异性强及高通量等优点,已经成为研究基因表达分析的常用方法,但其结果的准确性取决于内参基因。在任何试验条件下都能稳定表达的内参基因几乎不存在,科研工作者为了获得精准的试验结果,首先必须从众多的内参基因中筛选出稳定的内参基因。geNorm、NormFinder和BestKeeper等是专门用于筛选稳定性内参基因的软件,但这3种软件使用方法差异很大,为了让更多的科研人员了解这些软件、节省时间,笔者结合自己的科研经验详细介绍了这3种软件的使用方法,以期为相关科研人员利用这些软件筛选内参基因提供便利。
关键词 内参基因;稳定性分析;geNorm;NormFinder;BestKeeper
中图分类号 S60 文献标识码 A 文章编号 1007-5739(2017)05-0278-04
Abstract Real-time fluorescent quantitative PCR technology(RT-qPCR) is used for gene expression analysis with high sensitivity,good repea-tability,strong specificity,high throughput,but the veracity and reliability results depend on whether select appropriate reference gene or not.However,no universally applicable reference genes with an invariant expression are available.In order to obtain accurate results,appropriate reference genes for RT-qPCR normalisation must systematically evaluate the stability of candidate reference genes prior to using in each experimental system.geNorm,NormFinder and BestKeeper are useful programs to identify appropriate reference genes for RT-qPCR analysis,but the methods for using the three softwares were different.In order to save the time to understanding the way to use them and provide convenient for new researchers,this study described the methods of application.
Key words reference gene;stability analysis;geNorm;NormFinder;BestKeeper
實时荧光定量PCR(RT-qPCR)由于具备灵敏度高、重复性好、特异性强及高通量等优点,已经成为研究基因表达分析的常用方法[1-2]。实时荧光定量PCR分为绝对定量和相对定量,在进行相对定量分析时不需要已知量的标准品,因而该方法被经常运用,但该方法需要用内参基因对目的基因进行数据校正,才能获得精确的结果[3-4]。
肌动蛋白基因(ACT)、3-磷酸甘油醛脱氢酶基因(GAPD
H)、18S rRNA、转录延伸因子基因(EF-1α)、多聚泛素酶基因(UBQ)、α微管蛋白基因(TUA)和β微管蛋白基因(TUB)等看家基因在之前的研究中常常被当作内参基因进行使用[5-8]。然而,越来越多的研究表明,在某种试验稳定表达的内参基因,在另一种试验中有可能是变化的[9-11],而在任何试验条件下都能稳定表达的内参基因几乎不存在[12-13]。如果仅仅根据文献报道使用某个常用的内参基因,不仅可能导致试验结果的不准确性,甚至可能导致结论的错误。因此,本着严谨的科学态度,科研工作者首先必须从众多的内参基因中筛选出在各自试验条件下较为稳定的内参基因。
geNorm、NormFinder 和 BestKeeper是专门用于筛选内参基因稳定性的软件,但这3种软件的使用方法和分析的侧重点各不一样,为了让相关科研人员快速了解并掌握这3种软件的使用方法,笔者结合自身使用这些软件的经历,详细介绍了这3种软件的使用要点,以期为相关科研人员分析内参基因稳定性提供便利。
1 geNorm软件
1.1 软件概述
geNorm 软件是Vandesompele等[14]在2002年编写的专门用于实时荧光定量PCR中筛选内参基因及确定最适内参基因数目的程序,该程序可以用于筛选任何试验的任意数目的内参基因,并最终挑选出2个或2个以上的内参基因组合来校正数据,可使相对定量的结果更为精确。geNorm程序通过计算出每个内参基因稳定性的M值来筛选出稳定性较好的内参基因,判定标准为M值越小内参基因稳定性越好,反之,则稳定性越差。该软件还可计算引入1个新的内参基因后标准化因子的配对变异V值,并根据Vn/Vn+1值来确定所需最适内参基因的数目。默认的V值为0.15(该值可以人为稍作调整),如果Vn/Vn+1值<0.15,则最适内参基因的数量是n个;而如果Vn/Vn+1值>0.15,则最适内参基因的数量是n+1个。
1.2 操作流程
1.2.1 修改Excel表格安全性级别。若geNorm软件打开后不能正常运行时,可能是由于Excel表格默认宏的安全性级别设置的比较高,这时需要将Excel表格安全性级别更改到最低级别。更改方法为点击Excel表格中的“工具”按钮,然后点击其下拉菜单“宏”的子菜单“安全性”选项,在弹出来的“安全性”对话框中将安全级别设置为最低。
1.2.2 计算?驻Ct值。先找到该基因在所有样品中最小的Ct(Cycle threshold)值(表达量最高),再用其他样品的Ct值减去最低Ct值,从而得到?驻Ct值,该值≥0。
1.2.3 计算2-?驻Ct值。获得每个样品每个基因的?驻Ct值后,利用Excel中的函数计算出相应样品相应基因的2-?驻Ct值,该值就是每个候选内参基因的相对定量数据,也是进行geNorm软件分析时要用到的数据。
1.2.4 制作Excel表格。在利用geNorm软件分析时是将已经处理好的各个候选内参基因的相对定量数据保存在一个新的Excel表格中,再把Excel表格导入geNorm程序中。而这个Excel表格中对于基因和样品的排列位置是有特定要求的,要求规定第一列为试验的样品(样品数量至少在2个以上,样品数量越多结论越可靠),第1行为欲筛选的内参基因,第1列第1行的单元格必须是空的,其他单元格的数据是在步骤1.2.3中计算得到的基因相对表达量的数值。
1.2.5 内参基因稳定性和合适内参基因数目的确定。关闭所有Excel程序,启动geNorm程序,点击菜单栏中的“Load input data”按钮导入已经在步骤1.2.4中制作的Excel表格,点击软件中的“Calculate”按钮,软件初步计算后再点击“Automated analysis”按钮,便可得到候選内参基因稳定性折线图,见图1(a),该图最左边基因的M值最高,是稳定性最差的基因(H3基因),右边基因的M值依次较左边基因的M值低,稳定性依次增强,最右边基因的M值最低,是稳定性最好的2个基因(CYP1和Fe-SOD2基因)。点击菜单栏中的“Print or Save report”按钮,从弹出来的对话框中选中“save output to fi”选项便可把每个候选内参基因的M值导出到一个Excel中。再次点击“Automated analysis”按钮便可得到最适数据归一化内参数目的柱形图,见图1(b),可根据每个柱形图上面的V值来确定试验所需的最适内参基因数目,判定标准为当Vn/Vn+1< 0.15时,则最合适内参基因的数量是n个;而如果Vn/Vn+1 > 0.15时,则最合适内参基因的数量是n+1个,所以该试验条件下的最适内参基因数目是2个,因为V2/3=0.135<0.15。再次点击菜单栏中的“Print or Save report”按钮,从弹出来的对话框中选中“save output to fi”选项便可把所有的Vn/Vn+1值导出到一个Excel中。
2 NormFinder软件
2.1 软件概述
NormFinder是Claus等[15]在2004年编写的用于筛选稳定性内参基因的另一种程序,该程序计算原理与geNorm 程序相似,也是先获得内参基因表达稳定值,再根据稳定值大小来筛选出最合适的内参基因,判定标准为表达稳定值最小的内参基因为最合适的内参基因。NormFinder程序不但可以比较候选内参基因的表达差异,还可以计算样品组间的变异,但该程序只能筛选出一个最合适的内参基因[16]。
2.2 操作流程
2.2.1 在Excel表格中插入NormFinder软件。先打开一个Office 2003版的Excel表格,在菜单栏中单击“工具”下拉菜单中的“加载宏”选项,通过“加载宏”对话框中的“浏览”按钮找到NormFinder软件存放的位置,点击“确定”按钮,将NormFinder软件功能加载到Excel中,加载成功后在Excel菜单栏中会多一个NormFinder按钮(图2)。
2.2.2 计算?驻Ct值。计算方法与步骤1.2.2一致。
2.2.3 计算2-?驻Ct值。计算方法与步骤1.2.3一致。
2.2.4 制作Excel表格。进行NormFinder分析是在Excel表格中进行的,但Excel表格中基因和样品的排列位置与进行geNorm软件分析时有所不同。利用NormFinder分析时,Excel表格中第1列为欲筛选的内参基因,第1行为试验中的样品,第1列第1行的单元格必须是空的,其他单元格的数据是步骤2.2.3中计算得到的基因相对表达量的数值。
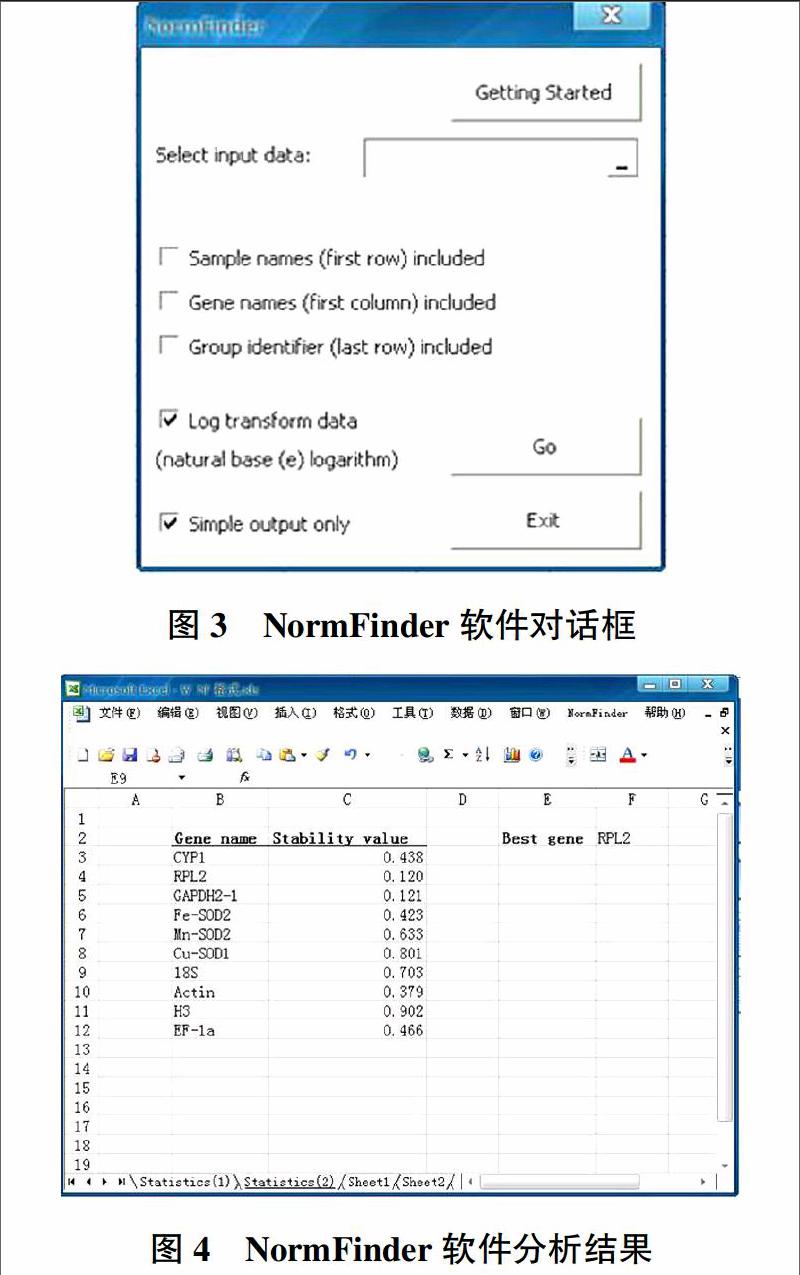
2.2.5 内参基因稳定性的确定。步骤2.2.4处理好后单击Excel菜单栏“NormFinder”按钮下拉菜单的“NormFinder”选项,便弹出对话框,如图3所示。单击“Select input date”后面的按钮,就可以利用鼠标在Excel表格中选定数据区域,选定区域后再次回到图3界面,然后单击“Go”按钮,便可获得各个基因稳定性M值,同时稳定性最好的基因(Best gene)会单独显示出来,如图4所示最稳定的基因为RPL2。
3 BestKeeper软件
3.1 软件概述
BestKeeper是Michael等[17]编写的针对内参基因和目标基因表达量分析的程序,该程序最多只能比较100个样品中10个内参基因和10个目标基因的表达水平。通过该程序计算可以获得每个基因之间产生配对的相关系数(r)、标准偏差(SD)和变异系数(CV),再通过比较各个值的大小,最终确定稳定性较好的内参基因。判定原则为相关系数越大、标准偏差和变异系数越小,内参基因稳定性越好,反之,稳定性越差;当SD>1时,则该内参基因表达不稳定。该程序不但可以用于内参基因稳定性比较,还可以分析目的基因的表达水平。
3.2 操作流程
BestKeeper程序是一个内置公式的Excel表格,在该表格中只有CP(crossing point)值输入区域(CP date input)可以进行数据的输入和修改,其他区域的单元格由于带有内置公式而不能进行任何操作。该程序操作较简单,只需要将实时荧光定量PCR所获得的CP值直接输入“CP date input”区域,输入的CP值是RT-qPCR中3次技术重复所得值的几何平均数。当CP值输入完毕后,BestKeeper程序会自动将计算好的相关系数(r)、标准偏差(SD)和变异系数(CV)等值显示在表格的下方。之后根据判定原则,确定较稳定的内参基因。
4 結语
geNorm、NormFinder和BestKeeper等软件是专门用于筛选内参基因稳定性的程序,且在多种植物中利用这些软件获得的内参基因稳定性试验成果已在较高档次的SCI期刊上发表,如在大豆[18]、马铃薯[19]、印加果[20]、香蕉[21]上的试验结果分别在BMC Molecular Biology、Journal of Experimental Botany、International Journal of Molecular Sciences、Planta期刊上发表,笔者利用这些软件分析获得的龙眼[22]内参基因稳定性结果也发表在Frontiers in Plant Science期刊。
利用geNorm和NormFinder软件分析时,需要将RT-qPCR所得到的Ct值转化为基因的相对表达量,然后按相应的格式把这些数据保存在Excel表格中,而在使用BestKeeper软件时不需要将RT-qPCR所得到的CP值进行转化,可以直接使用所得到的CP值。
geNorm分析时首先是将已经处理好的数据保存在Excel表格中,再把Excel表格导入geNorm程序中。NormF-inder分析时是在Excel表格中将NormFinder程序以宏的方式插入到Excel表格中,从而使Excel具有NormFinder分析功能。BestKeeper分析时是在内置BestKeeper公式的Excel表格中直接输入CP值进行分析。
这3种软件各具优点,geNorm软件可以用于筛选任何样品的任意数量内参基因,通过该程序分析可以筛选出合适的内参基因以及确定最适内参基因数目。NormFinder软件不但可以比较内参基因的表达差异,还可以计算样品组间的变异,但只能筛选出1个合适的内参基因。BestKeeper软件可以用于比较100个样品中10个内参基因和10个目标基因的表达水平,并最终获得较为稳定的内参基因。
5 参考文献
[1] RANSBOTYN V,REUSCH T B H.Housekeeping gene selection for qua-ntitative real-time PCR assays in the seagrass Zostera marina subjected to heat stress[J].Limnology and Oceanography Methods,2006,4(6):367-373.
[2] YOO W G,KIM T I,LI S,et al.Reference genes for quantitative analysis on Clonorchis sinensis gene expression by real-time PCR[J].Parasitology Research,2009,104(2):321-328.
[3] QUACKENBUSH J.Microarray data normalization and transformation[J].Nature Genetics,2002,32:496-501.
[4] NOLAN T,HANDS R E,BUSTIN S A.Quantification of mRNA using re-altime RT-PCR[J].Nature Protocols,2006,1(3):1559-1582.
[5] SUZUKI T,HIGGINS P J,CRAWFORD D.Control selection for RNA qu-antitation[J].Biotechniques,2000,29(2):332-337.
[6] BUSTIN S A.Quantification of mRNA using real-time reverse transc-ription PCR(RT-PCR):trends and problems[J].Journal of Molecular En-docrinology,2002,29(1):23-29.
[7] DHEDA K,HUGGETT J F,BUSTIN S A,et al.Validation of housek-eeping genes for normalizing RNA expression in real-time PCR[J].Biote-chniques,2004,37(1):112-119.
[8] KIM B R,NAM H Y,KIM S U,et al.Normalization of reverse transc-ription quantitative-PCR with housekeeping genes in rice[J].Biotechnol-ogy Letters,2003,25(21):1869-1872.
[9] TONG Z G,GAO Z H,WANg F,et al.Selection of reliable reference ge-nes for gene expression studies in peach using real-time PCR[J].BMC Molecular Biology,2009,10(1):1-13.
[10] TROND L,CATHRINE L.Reference gene selection for quantitative rea-ltime PCR normalization in tomato subjected to nitrogen,cold,and light stress[J].Analytical Biochemistry,2009,387(2):238-242.
[11] MUKESH J,AASHIMA N,AKHILESH K,et al.Validation of house-keeping genes as internal control for studying gene expression in rice by quantitative real-time PCR[J].Biochemical and Biophysical Research Communications,2006,345(2):646-651.
[12] MA R,XU S,ZHAO Y C,et al.Selection and validation of appropriate reference genes for quantitative real-time PCR analysis of gene expr-ession in Lycoris aurea[J].Frontiers in Plant Science,2016,7:651.
[13] LI M Y,WANG F,JIANG Q,et al.Validation and comparison of refer-ence genes for qPCR normalization of celery(apiumgraveolens) at diff-erent development stages[J].Frontiers in Plant Science,2016,7:313.
[14] VANDESOMPELE J,PRETER D K,PATTYN F,et al.Accurate norma-lization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J].Genome Biology,2002,3:7.
[15] CLAUS L A,JENS L J,TORBEN F R.Normalization of real-time qua-ntitative reverse transcription-PCR data:a model-based variance esti-mation approach to identify genes suited for normalization,applied to bladder and colon cancer data sets[J].Cancer Research,2004,64(15):5245-5250.
[16] PEREZ S,ROYO L J,ASTUDILLO A,et al.Identifying the most suitable endogenous control for determining gene expression in hearts from organ donors[J].BMC Molecular Biology,2007,8(1):1-23.
[17] MICHAEL W P,ALES T,CHRISTIAN P,et al.Determination of stable housekeeping genes,differentially regulated target genes and sample integrity:Best Keeper-Excel-based tool using pair-wise correlations[J].Biotechnology Letters,2004,26(6):509-515.
[18] HU R B,FAN C M,LI H Y,et al.Evaluation of putative reference genes for gene expression normalization in soybean by quantitative real-time RT-PCR[J].BMC Molecular Biology,2009,10(1):93-93.
[19] NICOT N,HAUSMAN J F,HOFFMANN L,et al.Reference gene selec-tion for RT-qPCRnormalization in potato during biotic and abiotic str-ess[J].Journal of Experimental Botany,2005,56(421):7-14.
[20] NIU L,TAO Y B,CHEN M S,et al.Selection of reliable reference genes for gene expression studies of a promising oilseed crop,plukenetia vol-ubilis,by real-time quantitative PCR[J].International Journal of Molec-ular Sciences,2014,16(6):12513-12530.
[21] CHEN L,ZHONG H Y,KUANG J F,et al.Validation of reference genes for RT-qPCR studies of gene expression in banana fruit under different experimental conditions[J].Planta,2011,234(2):377-390.
[22] WU J Y,ZHANG H N,LIU L Q,et al.Validation of reference genes for RT-qPCR studies of gene expression in preharvest and postharvest lon-gan fruits under different experimental conditions[J].Frontiers in Plant Science,2016,7:439.

