hMTERF3基因启动子区的生物信息学分析
孙美涛,王昀,李月,张晓娟,杨勇琴,杨泽芳,熊伟*
(1.大理大学基础医学院,云南大理671000;2.云南省昆虫医药研发重点实验室,云南大理671000;3.大理大学大理教学医院呼吸内科,云南大理671000)
hMTERF3基因启动子区的生物信息学分析
孙美涛1,2,王昀1,2,李月1,2,张晓娟3,杨勇琴1,杨泽芳1,熊伟1,2*
(1.大理大学基础医学院,云南大理671000;2.云南省昆虫医药研发重点实验室,云南大理671000;3.大理大学大理教学医院呼吸内科,云南大理671000)
目的:探讨人线粒体转录终止因子3基因启动子区的序列特征、转录因子及其结合位点。方法:利用Promoter 2.0、NNPP、Proscan、FirstEF软件分别预测hMTERF3基因5端上游的启动子数目及分布;利用CpG Island Searcher和CpG Plot软件预测CpG岛位置;利用P-match 1.0程序搜索TRANSFAC数据库预测与hMTERF3基因启动子结合的转录因子及其结合位点。结果:hMTERF3基因定位于8q21.2,基因全长22 216 bp,含有11个外显子和10个内含子。hMTERF3基因上游至5'侧翼共3 000 bp的核苷酸序列至少存在2个启动子区,其中1 733~2 302 bp之间可能为包含TATA盒的核心启动子区。启动子区序列中存在1个长为1 145 bp的CpG岛。hMTERF3基因启动子区存在1 055个转录因子结合位点,进化足迹法分析其保守的核心启动子区转录因子结合位点共19个。结论:hMTERF3基因启动子区的生物信息学分析能够提高基因启动子的研究效率,为后续实验构建hMTERF3基因启动子表达载体及鉴定启动子功能提供理论依据。
hMTERF3;生物信息学;启动子区;转录因子;CpG岛
人类线粒体转录终止因子3(human mitochon⁃drial transcription termination factor 3,hMTERF3)基因又称为人类线粒体转录终止因子结构域1(human mitochondrial transcription termination domain contain⁃ing 1,hMTERFD1)基因〔1〕。2007年,PARK C B等在Cell杂志上首次报道哺乳动物MTERF3基因编码的蛋白质是线粒体基因转录的负调控因子,与线粒体DNA(mitochondrial DNA,mtDNA)重链及轻链启动子区结合,抑制线粒体DNA双链的基因转录水平〔2〕。WREDENBERG A等最近的研究结果揭示哺乳动物MTERF3也能调节线粒体中核糖体大亚基的组装,并且影响线粒体中核糖体的生物合成〔3〕。hMTERF3基因的表达异常,可能与线粒体心肌病、线粒体糖尿病、神经退行性疾病、线粒体脑病、恶性肿瘤等人类疾病的发生密切相关〔4-5〕。然而,目前对于hMTERF3基因自身的转录调控及其分子机制仍不明确。
本研究利用不同的生物信息学软件对hM⁃TERF3基因序列和启动子区分别进行分析,获取该基因的启动子位置与分布、CpG岛位置及启动子区转录因子结合元件,旨在为后续实验研究中构建hMTERF3基因启动子表达载体和鉴定基因启动子功能提供基本的理论参考。
1 材料和方法
1.1hMTERF3基因组DNA序列hMTERF3基因定位于人类基因组8q22.1,该基因又被称为hCGI-12或hMTERFD1,基因全长为22 216 bp,由10个内含子和11个外显子构成〔6〕。hMTERF3基因转录的mRNA登录号为NM_015942.4,编码的蛋白质产物登陆号为NP_057026.3,但在NCBI(National Center for Biotechnology Information,NCBI)的GenBank尚未记录此基因启动子区的序列〔7〕。
1.2数据库和程序美国国立生物技术信息中心数据库:http://www.ncbi.nlm.nih.gov/genbank/。基因启动子在线分析软件:Promoter 2.0(http://www.cbs. dtu.dk/services/promoter/);NNPP(http://www.fruitfly. org/seq_tools/promoter.html/);Proscan(http://www-bi⁃mas.cit.nih.gov/molbio/proscan/);FirstEF(http://rulai. cshl.org/tools/FirstEF/)。启动子区的转录因子预测软件:P-match 1.0(http:www.gene-regulation.com/ pub/programs.html#p-match/)。CpG岛预测软件:CpG Island Searcher(http://www.uscnorris.com/cpgis⁃lands2/cpg.aspx/);CpG Plot(http://www.ebi.ac.uk/ emboss/cpgplot/index.html/)。
1.3获取hMTERF3基因序列在NCBI Gene数据库中检索hMTERF3,获得hMTERF3基因的ID为51001,采用FASTA格式获取基因信息〔8〕。
1.4hMTERF3基因启动子区序列的确定在NCBI数据库中查找,获得hMTERF3基因的转录起始位点在基因序列图上的定位,并获取转录起始位点上游-2 500 bp至转录起始点下游的+500 bp序列(包括第1外显子和第1内含子)。此段共计3 000 bp的序列包含潜在的基因启动子序列。
1.5hMTERF3基因启动子区序列的分析利用启动子在线预测软件Promoter 2.0、NNPP、Proscan、FirstEF分别在软件默认条件下对获取的hMTERF3基因上游至5'侧翼启动子区合计3 000 bp的序列进行分析。
1.6hMTERF3基因启动子区CpG岛的分析利用CpG Island Searcher和CpG Plot分别在软件默认条件下对获取的hMTERF3基因上游至5'侧翼共计3 000 bp的序列进行基因启动子区CpG岛的分析〔9〕。
1.7hMTERF3基因启动子区转录因子结合位点分析利用P-Match 1.0程序,设定矩阵序列相似性设定为0.95,核心序列相似性为0.90,输入hMTERF3基因上游至5'侧翼3 000 bp的序列,搜索TRANSFAC 5.0数据库中的脊椎动物转录因子结合元件,分别对hMTERF3基因的正、负义链转录因子结合元件进行分析〔10〕。
2 结果与分析
2.1hMTERF3基因特征GenBank中hMTERF3基因的登录号为NC_000008.11,定位于8q22.1,该基因也被命名为hCGI-12或hMTERFD1,基因组序列全长22 216 bp(Chromosome 8:96239398..96261613),由10个内含子和11个外显子构成。该基因转录的mRNA全长为1 470 bp,编码417个氨基酸构成的蛋白质,N端1~68个氨基酸为定位线粒体的导肽,紧随其后的349个氨基酸组成成熟肽。
2.2hMTERF3基因启动子区分析Promoter 2.0软件预测的结果提示,hMTERF3基因上游可能存在2个不同的启动子区,其中临界性预测位于200 bp处,1 200 bp处为最大可能预测。Neural Network Promoter Prediction(NNPP)软件预测的结果提示,hMTERF3基因上游可能存在6个不同的启动子序列,分别位于413~463 bp,467~517 bp,1 036~1 086 bp,2 048~2 098 bp,2 217~2 267 bp,2 863~2 913 bp。Proscan软件预测的结果提示,hMTERF3基因正义链有1个可能的启动子序列,位于1 663~1 883 bp;负义链则有3个可能的启动子序列,分别位于2 724~2 474 bp,1 948~1 698 bp,664~414 bp。FirstEF软件预测的结果提示,hMTERF3基因启动子在正义链位于1 733~2 302 bp之间,P值为0.998 3,在负义链位于2 564~1 995 bp之间,P值为1.000 0。见表1。
2.3hMTERF3基因CpG岛预测和分析
2.3.1 CpG Island Searcher预测结果采用在线软件CpG Island Searcher预测hMTERF3基因启动子区的CpG岛,预测标准为(G+C)%>55.00%;观察值/预期值>0.65,长度>200 bp。结果显示,hMTERF3基因CpG岛位于启动子区1 452~2 596 bp之间。CpG岛的(G+C)%=57.9%,观察值/预期值=0.832,长度= 1 145 bp,软件预测的CpG岛横跨第1启动子及转录起始位点,这与CpG岛的分布特点相符。见图1。
2.3.2 CpG Plot预测结果使用欧洲分子生物学实验室(EMBL)提供的CpG Plot在线软件,CpG岛的预测标准为(G+C)%>50.00%;观察值/预期值>0.60,长度>200 bp,在hMTERF3基因5'端上游启动子区发现3个CpG岛。第1个CpG岛位于1 448~1 770 bp之间,长度为323bp;第2个CpG岛位于1791~2313bp之间,长度为523bp;第3个CpG岛位于2337~2 732 bp之间,长度为396 bp。该软件预测的CpG岛位置与CpG Island Searcher软件预测的CpG岛位置高度重合。见图2。

表1 hMTERF3基因启动子的预测结果
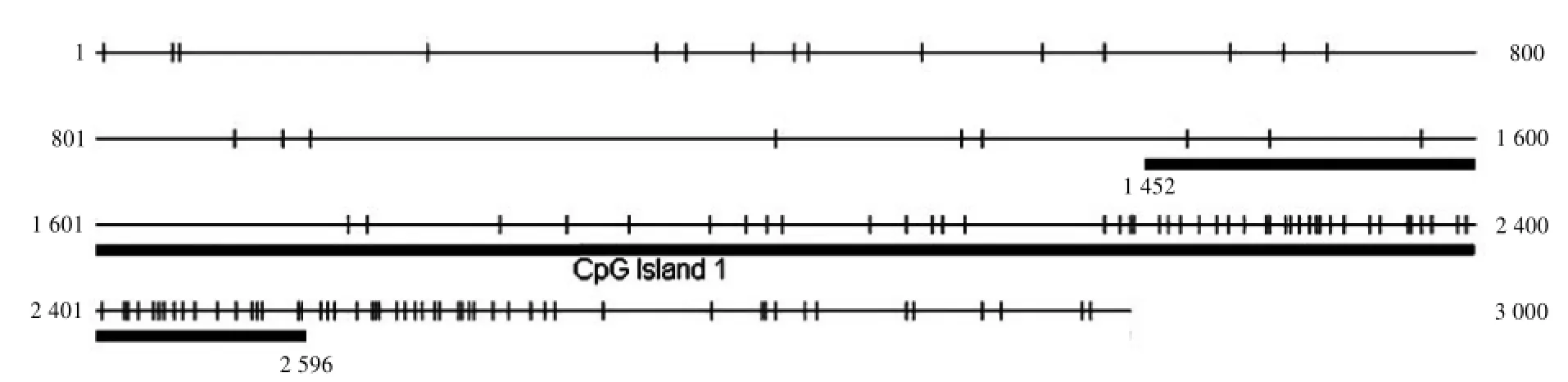
图1 CpG Island Searcher软件预测hMTERF3基因上游5'端启动子区的CpG岛(图中黑色粗线区域为CpG岛所在的位置)
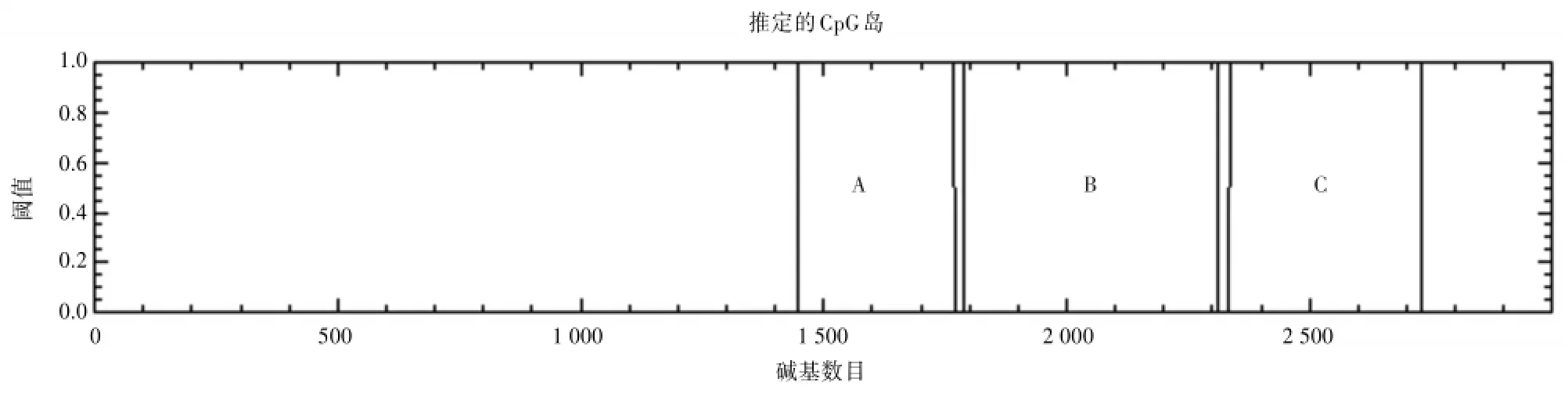
图2 CpG Plot软件预测hMTERF3基因上游5'端启动子区的CpG岛(柱状图A、B、C为CpG岛所在位置)
2.4hMTERF3基因转录因子结合位点分析利用P-Match 1.0程序检索TRANSFAC 4.0数据库,共计获得基因启动子区3 000 bp正负链转录因子结合位点1 055个,再用进化足迹法分析hMTERF3基因及小鼠同源MTERF3基因(Gene ID:66410)核心启动子区保守区域,只保留位于核心启动子保守区域内共有的转录因子结合部位19个。见表2。

表2 hMTERF3基因核心启动子区转录因子结合位点的预测结果
3 讨论
hMTERF3基因属于人类线粒体转录终止因子(mitochondrial transcription termination factor,MTERF)基因家族。该基因定位于人类第8号染色体长臂(8q22.1),由10个内含子和11个外显子构成。hMTERF3基因编码的蛋白质产物共有417个氨基酸,N端1~68个氨基酸为定位线粒体的导肽,其后349个氨基酸为成熟的hMTERF3多肽〔7〕。hMTERF3蛋白是目前为止报道的唯一一个人类线粒体基因转录的负调控因子,对人类恶性肿瘤、线粒体糖尿病、神经退行性疾病(如帕金森症、阿尔茨海默病等)等线粒体相关疾病的诊断与治疗具有良好的应用前景。
基因启动子的鉴定作为研究复杂基因调控网络的基础和前提,是细胞分子生物学领域长期以来致力研究的重要问题。使用计算方法对基因启动子进行鉴定的工作通常是基于DNA序列特征开展的,这些鉴定方法按其研究目的大致分为启动子区域搜寻和启动子调控序列鉴定两类。识别启动子区域等同于识别转录起始位点的位置,其基本原理是搜索核心序列如TATA盒,或搜索已知的转录因子结合位点等〔11〕。本研究分别采用Promoter 2.0、NNPP、Proscan、FirstEF 4种不同的启动子分析软件对hMTERF3基因启动子区进行分析,结果发现该基因至少存在着2个启动子,大致分布于500 bp和2 500 bp之内,尤其是启动子区1 733~2 302 bp的序列可能为包含TATA盒的核心启动子。预测结果与文献报道的基因启动子区存在于转录起始位点上游-2 500 bp之内是基本一致的〔12〕。
CpG双核苷酸在人类基因组中的分布很不均一,而在基因组的某些区段,CpG保持或高于正常概率,这些区段被称作CpG岛。研究表明,CpG岛主要位于基因的启动子和第1外显子区域,人类基因组中有60%以上基因的启动子含有CpG岛〔13〕。CpG岛的(G+C)%含量通常都超过50%,长度>200 bp。本研究利用CpG Island Searcher和CpG Plot两个不同软件对基因启动子区CpG岛进行分析,CpG Island Searcher软件分析出hMTERF3基因启动子区有1个CpG岛,位于启动子区的1 452~2 596 bp之间,长度为1 145 bp。CpG Plot软件则分析出hMTERF3基因启动子区有3个CpG岛,分别位于启动子区的1 448~1 770 bp、1 791~2 313 bp和2 337~2 732 bp,长度依次为323、523和396 bp,这3段序列所在的位置与CpG Island Searcher软件分析的CpG岛位置是高度重合的。
生物信息学方法鉴定基因启动子区转录因子结合位点的基本流程如下:首先,确定启动子区域的大致范围,通常取基因转录起始位点上游的1 000~3 000 bp的范围;然后,确定一类具有共同特征(诸如相似的表达谱)的基因,并获取它们的启动子区序列;接着使用多重序列比对(Multiple sequence alignment,MSA)的方法鉴定上述序列中一致性出现的模体(motif),并计算模体每个位置的碱基比例,生成相应的权重矩阵(weight matrix)。模体的权重矩阵可用以预测转录因子的结合位点,常用的转录因子数据库有JASPAR和TRANSFAC等〔14-15〕。本研究利用P-match 1.0程序搜索TRANSFAC 4.0数据库,预测hMTERF3基因启动子区共1 055个转录因子结合位点。采用进化足迹法,对人和小鼠的MTERF3基因上游5'端的保守区域进行比较分析表明,在hMTERF3基因核心启动子区1 733~2 302 bp相似性较大,对比确定同源基因启动子保守区域内共有的转录因子有19个。因此,综合结果表明这个区域可能是基因表达调控的关键部位。由于在转录因子结合位点的分析中,P-match 1.0程序只能针对已知转录因子的结合位点进行预测,对未知的或新的转录因子结合位点则无法进行分析,该预测方法存在着一定的局限性〔16〕。所以,生物信息学软件分析的结果可为后续的研究指明方向,但尚需后续的实验研究来进一步验证。
此外,随着近年来表观基因组学的迅猛发展,开发出了一大批基于基因组蛋白修饰和DNA甲基化数据预测启动子的方法,比如Segway、ChromHMM等,已经在基因组注释研究中发挥了不可替代的作用〔17-18〕。可以预见的是,利用表观基因组学的数据来鉴定启动子是未来生物信息学分析基因启动子的主要发展方向〔19〕。
综上所述,本研究首先通过NCBI GenBank数据库获取hMTERF3基因序列,并获取hMTERF3基因上游至5'侧翼3 000 bp序列,再采用不同的生物信息学软件对hMTERF3基因序列特征、启动子序列、CpG岛以及转录因子结合位点进行了预测和分析,为下一步实验构建hMTERF3基因启动子表达载体和检测启动子的活性提供基本的理论依据。
〔1〕HYVÄRINEN A K,POHJOISMÄKI J L,HOLT I J,et al. Overexpression of MTERFD1 or MTERFD3 impairs the completion of mitochondrial DNA replication〔J〕.Mol Biol Rep,2011,38(2):1321-1328.
〔2〕PARK C B,ASIN-CAYUELA J,CÁMARA Y,et al. MTERF3 is a negative regulator of mammalian mtDNA transcription〔J〕.Cell,2007,130(2):273-285.
〔3〕WREDENBERG A,LAGOUGE M,BRATIC A,et al. MTERF3 regulates mitochondrial ribosome biogenesis in invertebrates and mammals〔J〕.PLoS Genet,2013,9(1):e1003178.
〔4〕ANDERSSON D C,FAUCONNIER J,PARK C B,et al. Enhanced cardiomyocyte Ca(2+)cycling precedes terminal AV-block in mitochondrial cardiomyopathy Mterf3 KO mice〔J〕.Antioxid Redox Signal,2011,15(9):2455-2464.
〔5〕熊伟,余敏,左绍远.线粒体转录终止因子蛋白家族在线粒体基因表达中的调节作用〔J〕.中国生物化学与分子生物学报,2015,31(3):223-231.
〔6〕XIONG W,LUO Y,ZHANG C,et al.Expression,purifi⁃cation of recombinant human mitochondrial transcription termination factor 3(hMTERF3)and preparation of poly⁃clonal antibody against hMTERF3〔J〕.Appl Biochem Biotechnol,2012,167(8):2318-2329.
〔7〕熊伟,张晓娟,张海洋,等.基于生物信息学方法预测人线粒体转录终止因子3蛋白的结构与功能〔J〕.生物技术通讯,2015,26(3):367-373.
〔8〕孙勇,王良喜,孙曙光,等.人三叶因子3基因启动子区的生物信息学分析〔J〕.医学研究生学报,2013,26(4):340-342.
〔9〕唐亮,王燕,陈碧峰.人Toll样受体9基因启动子区的生物信息学分析〔J〕.生物技术通讯,2014,25(2):210-212.
〔10〕杨树德,曹奇志,冯小英,等.人LAIR-1/CD305基因启动子的生物信息学分析〔J〕.细胞与分子免疫学杂志,2010,26(8):826-828.
〔11〕KLINGENHOFF A,FRECH K,QUANDT K,et al.Func⁃tional promoter modules can be detected by formal models independent of overall nucleotide sequence similarity〔J〕. Bioinformatics,1999,15(3):180-186.
〔12〕管晓翔,陈巍魏,陈龙邦,等.p27Kip1基因启动子区的生物信息学分析〔J〕.医学研究生学报,2010,23(10):1029-1032.
〔13〕TAKAI D,JONES P A.Comprehensive analysis of CpG islands in human chromosomes 21 and 22〔J〕.Proc Natl Acad Sci USA,2002,99(6):3740-3745.
〔14〕WINGENDER E.TRANSFAC:a database on transcrip⁃tion factors and their DNA binding sites〔J〕.Nucleic Acids Research,1996,24(1):238-241.
〔15〕PORTALES-CASAMAR E,THONGJUEA S,KWON A T,et al.JASPAR 2010:the greatly expanded open-access database of transcription factor binding profiles〔J〕.Nucle⁃ic Acids Res,2010,38:105-110.
〔16〕王佐广,牛秋丽,刘雅,等.线粒体融合基因2启动子的生物信息学研究〔J〕.中国动脉硬化杂志,2013,21(2):97-104.
〔17〕HOFFMAN M M,BUSKE O J,WANG J,et al.Unsuper⁃vised pattern discovery in human chromatin structure through genomic segmentation〔J〕.Nat Methods,2012,9(5):473-476.
〔18〕ERNST J,KELLIS M.ChromHMM:automating chroma⁃tin-state discovery and characterization〔J〕.Nat Methods,2012,9(3):215-216.
〔19〕卢一鸣,屈武斌,张成岗.基于表观基因组学的DNA元件鉴定方法研究进展〔J〕.生物化学与生物物理进展,2014,41(7):640-648.
Bioinformatics Analysis of hMTERF3 Gene Promoter Region
Sun Meitao1,2,Wang Yun1,2,Li Yue1,2,Zhang Xiaojuan3,Yang Yongqin1,Yang Zefang1,Xiong Wei1,2
(1.Pre-clinical College,Dali University,Dali,Yunnan 671000,China;2.Key Laboratory of Pharmaceutical R&D of Insects in Yunnan Province,Dali,Yunnan 671000,China;3.Respiratory Department,Dali Teaching Hospital of Dali University,Dali, Yunnan 671000,China)
Objective:To investigate the characters,transcription factors and their binding sites ofhuman mitochondrial transcription termination factor 3(hMTERF3)gene promoter by different bioinformatics tools.Methods:Promoter 2.0,NNPP,Proscan,and FirstEF softwares were used to analyze the numbers and distributions ofhMTERF3gene promoter;CpG Islander and CpG Plot softwares were used to analyze the GpG island ofhMTERF3gene promoter;P-match 1.0 protocol and TRANSFAC database were used to analyze the transcription factors and their binding sites ofhMTERF3gene promoter.Results:hMTERF3gene was located on 8q21.2.The full length ofhMTERF3gene was 22 216 bp,consisting of 11 exons and 10 introns.There were at least two promoters in the 5'unconding region ofhMTERF3gene.The core promoter ofhMTERF3gene was located between 1 733-2 302 bp,containing TATA box.InhMTERF3gene promoter region,a 1 145 bp CpG island could be found.In addition,1 055 transcription factor binding sites were predicted by P-Match 1.0 protocol,and only 19 transcription factor binding sites were found in conserved core promoter region of human and mouse homologousMTERF3genes by phylogenetic foot-printing analysis.Conclusion:Gene promoter related bioinformatics analysis can improve the efficiency ofhMTERF3gene promoter research,and provide significant information for the construction of promoter expression vector,also for the further study of promoter function.
hMTERF3;bioinformatics;promoter;transcription factor;CpG island
Q71:Q811.4
A
2096-2266(2017)02-0040-06
10.3969/j.issn.2096-2266.2017.02.008
(责任编辑 李杨)
国家自然科学基金资助项目(81560458);云南省教育厅科学研究基金资助项目(2014Z126);大理大学博士科研启动基金资助项目(BSKY2012018);大理大学大学生创新创业计划资助项目(201402)
2016-01-03
2016-05-07
孙美涛,硕士研究生,主要从事病理与病理生理学研究.
*通信作者:熊伟,副教授,博士.

