基于Cytoscape的生物医学本体参数化SPARQL查询系统
,, ,
生物医学领域和关联数据的发展推动了一大批可以查询的结构化生物医学本体数据的出现。如生物通路本体[1]包括生物化学反应、复杂装配、运输和催化事件,涉及到蛋白质、DNA、RNA、小分子和复合物的物理相互作用。这些生物医学本体数据中的数据类型多样、规模庞大。虽然很多情况下,生物医学研究者通过Cytoscape[2]可视化软件把生物本体可视化出来,但这些可视化的结果是通过一定的算法归类成的节点,其中每一个节点代表一个知识分类。如Mouse genome database(MGD)这个老鼠基因组的数据源共有8 206 813个三元组,但在可视化的结果中,只表示为59个节点,184条关联弧。而当需要对某个数据源中的节点进行详细研究或对某个数据源进行重新组织,对关系数据进行检索和解析RDF数据时,就需要用到查询语言和数据获取协议 (Simple Protocol and RDF Query Language,SPARQL)[3-4]语句查询来实现。
对于普通的生物医学研究人员来说,要熟练地运用SPARQL语法并构建能满足自己需求的查询语句是一件很困难的事情。这将会使生物医学本体数据源不能得到充分的利用,很多节点内所包含的数据实体与数据关联并不能被生物医学人员发现和利用;同时由于SPARQL语法问题与数据源数据的复杂性也会提高生物医学研究人员使用的门槛,因此需要一种工具辅助生物医学研究人员完成对生物医学本体数据的查询、检索、解析与重新组织。此背景下,笔者利用参数化设计的思想研发了一个Cytoscape参数化SPARQL查询系统,该系统可以编写SPARQL查询语句通过SPARQL端点进行可视化查询,也可以导入参数化模板,然后在可视化结果中选择可视化的生物本体通路数据节点,选择相应的查询参数,系统便会自动生成SPARQL查询语句,并把查询结果可视化。
1 SPARQL查询系统研究现状
对于普通用户来说,要熟悉地掌握SPARQL语法,并在自己的机器上构建出能满足自己查询需求的SPARQL查询语句,非常困难,于是一些研究者就研究出一些半自动化的SPARQL构建器[5]辅助用户实现SPARQL查询。其中,Querymed是一个典型的基于Web表单的查询构建器,它是Seneviratne等设计出来,以便医生和生物学家集成生物学和生物医学数据源的数据库并能从中抽取具体的知识;Viziquer是一个基于可视化图形的SPARQL查询构建器;Barzdins等[6]提出了一种图形化查询语言(Graphical Query Language,GQL),它要求用户通过提供的系统工具在画布上描绘出能表达三元组模式或图模式的图形,然后系统才会将图形转换为SPARQL查询;VQB是一个基于窗口组件的SPARQL查询构建器,但它包含在Konduit工具中,用于对Nepomuk-KDE中的RDF数据建立SPARQL查询,无法对其他的RDF数据构建SPARQL查询。
参数化查询(Parameterized Query 或 Parameterized Statement)是指在设计与数据库链接并访问数据时,在需要填入数值或数据的地方,使用参数 (Parameter) 给值。参数化查询这在SQL中应用广泛,SQL的参数化查询已被视为能最有效预防SQL注入攻击[7](SQL Injection) 的攻击手法的防御方式[8]。Apache Jena中就把参数化查询的技术应用到了SPARQL中。在Apache Jena 中;用户需要用用代码来对SPARQL的查询模板进行描述,如用户需要使用ParameterizedSparqlString这个类定义一个SPARQL模板;在Apache Jena中,用户编写好模板后,通过对参数注入不同的值而得到不同的SPARQL查询语句,生成SPARQL参数化查询代码不容易查询SPARQL查询语句的字符串错误与混乱的问题,能实现远程的SPARQL参数化查询,有效地防御SPARQL注入式攻击。总的来说,参数化查询的好处在于:当频繁使用改动不大的查询语句时,能有效提高数据引擎的执行效力,避免出现查询语句语法的错误,用户可以避免重复编写繁杂的查询语句,有效防止注入式攻击。
2 SPARQL模板包的定义
为了方便SPARQL模块的共享与交流,笔者提出基于SPARQL模板包的发布共享机制。SPARQL模板包是一个由某个人或者某组织发布到网上的一个共享包,该共享包记录着它自己特有的元数据和查询参数。SPARQL模板包的RDF元数据定义见图1。
查询参数是指SPARQL查询条件的模板,而且它的这些元数据和参数是用RDF资源框架[9]描述的(如它的名字和它发布的时间点)。SPARQL模板包是利用RDF资源描述框架技术,用一个名为“query.rdf”的文件表示的。当SPARQL模板包导入成功时,它将存储在用户的电脑中。用户可以利用RDF描述规范为基础编写一个生物医学本体数据常用的参数化查询SPARQL模板包,满足自己所需要的参数化查询,进行更深层次的研究。
如图2所示,该SPARQL模板包例子使用标签:
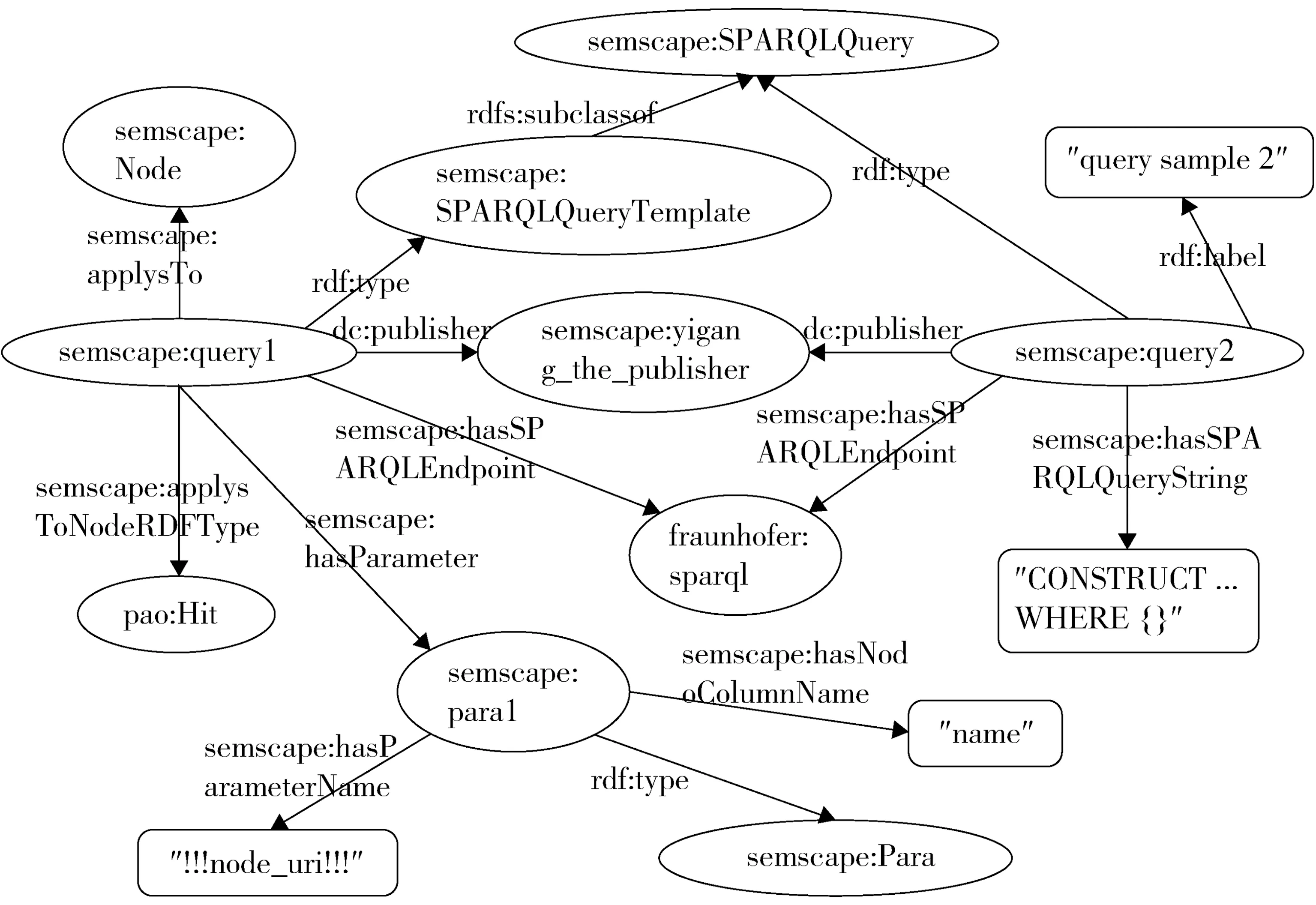
图1 SPARQL模板包的RDF元数据定义

图2 SPARQL模板包示例
笔者研发的Cytoscape插件包括2个软件模块。一是Context Synchronizer,它是SPARQL模板包的管理工具和用户界面(包括导入、检测和更新SPARQL模板包等功能),并以Cytoscape Panel面板插件形式嵌入Cytoscape中;二是Context Menu,它是SPARQL模板应用启动的工具模块(根据Cytoscape不同类型的实体,如节点、边等提供Tooltip显示和SPARQL查询功能),以Cytoscape右键菜单插件形式嵌入。
3 SPARQL模板包管理
Cytoscape插件可以让用户直接导入包含SPARQL查询语句的参数模板的模板包,只要用户提供SPARQL模板包的URL,便能快捷地导入SPARQL模板包,SPARQL模板包管理界面如图3所示。
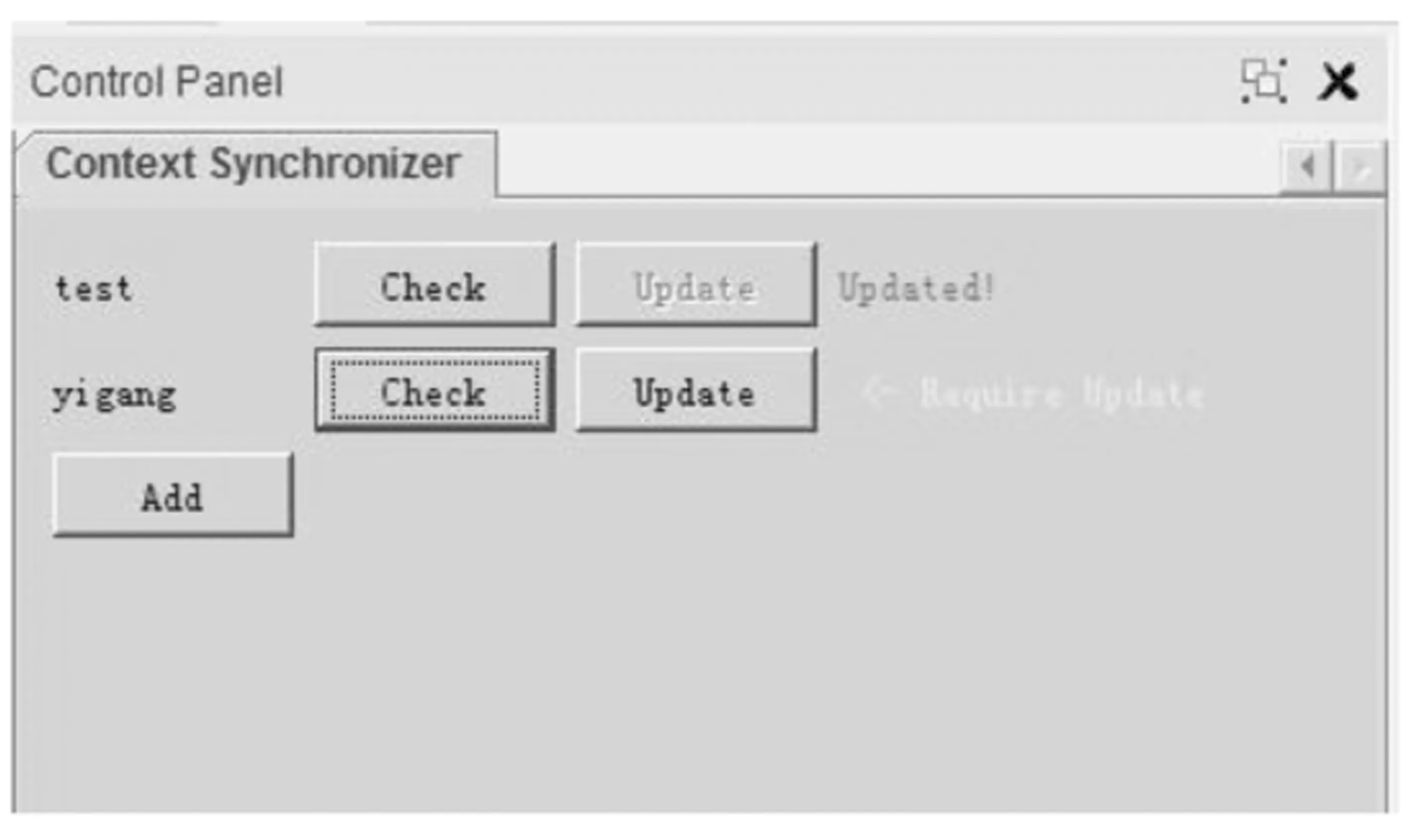
图3 SPARQL模板包管理界面
用户只要打开Context Synchronizer Pane 的UI界面,点击“Add”按钮,在弹出的对话框中输入SPARQL模板包的URL,点击“确定”即可。以我们发布的“yigang”模板包为例,点击“Add”,输入“yigang”的URL:“https://vsdlc3.googlecode.com/svn/trunk/vsdl3c/app/binary/semscape/yigang.tar.gz”点击“确定”,插件就会自动把“? yigang”这个SPARQL模板包自动下载到用户的电脑中“%user_home%/semscape/context/%context_name%的目录中”,并且插件会自动读取“yigang”模板包中的“query.rdf”文件。当导入成功时,用户可以点击“Check”按钮,插件就会自动地比较本地目录中“?node_pao…Hit_01”,SPARQL模板包中“query.rdf”中的“hasTime”时间值与当前网络URL中的“?node_pao…Hit_01”,SPARQL模板包中”query.rdf”中的“hasTime”时间值。当本地目录中“query.rdf”中的“hasTime”时间值与当前网络URL中的“query.rdf”中“hasTime”时间值一样时,它就会显示绿色的标签“Updated”,如上图X中的“test”SPARQL模板包,后面显示的绿色标签“Updated”说明这个模板包是最新的,不需要更新。当当前网络中“query.rdf”中的“hasTime”时间值比本地中“query.rdf”中的“hasTime”时间值要晚时,插件就会在模板后面显示黄色的标签“<- Require Update”,如图3中的“yigang”SPARQL模板包。
4 参数化SPARQL查询及可视化
当用户成功地通过笔者系统导入了模板包后,系统就会自动识别模板包中的信息,这时用户就可以使用导入的参数模板。用户只需要点击Cytoscape的节点或边缘,系统就会自动获取选中的Cytoscape节点或边缘信息,如选中实体的URI、RDF类型或其他属性。当系统获取到相应的信息后,就会自动地把信息与参数化模板中的配置信息作对比,并把合适的参数化查询以菜单的形式显示出来,这时用户只需要选择菜单栏中的参数化选项,系统就会自动构建SPARQL查询语句,并把可视化结果显示出来。
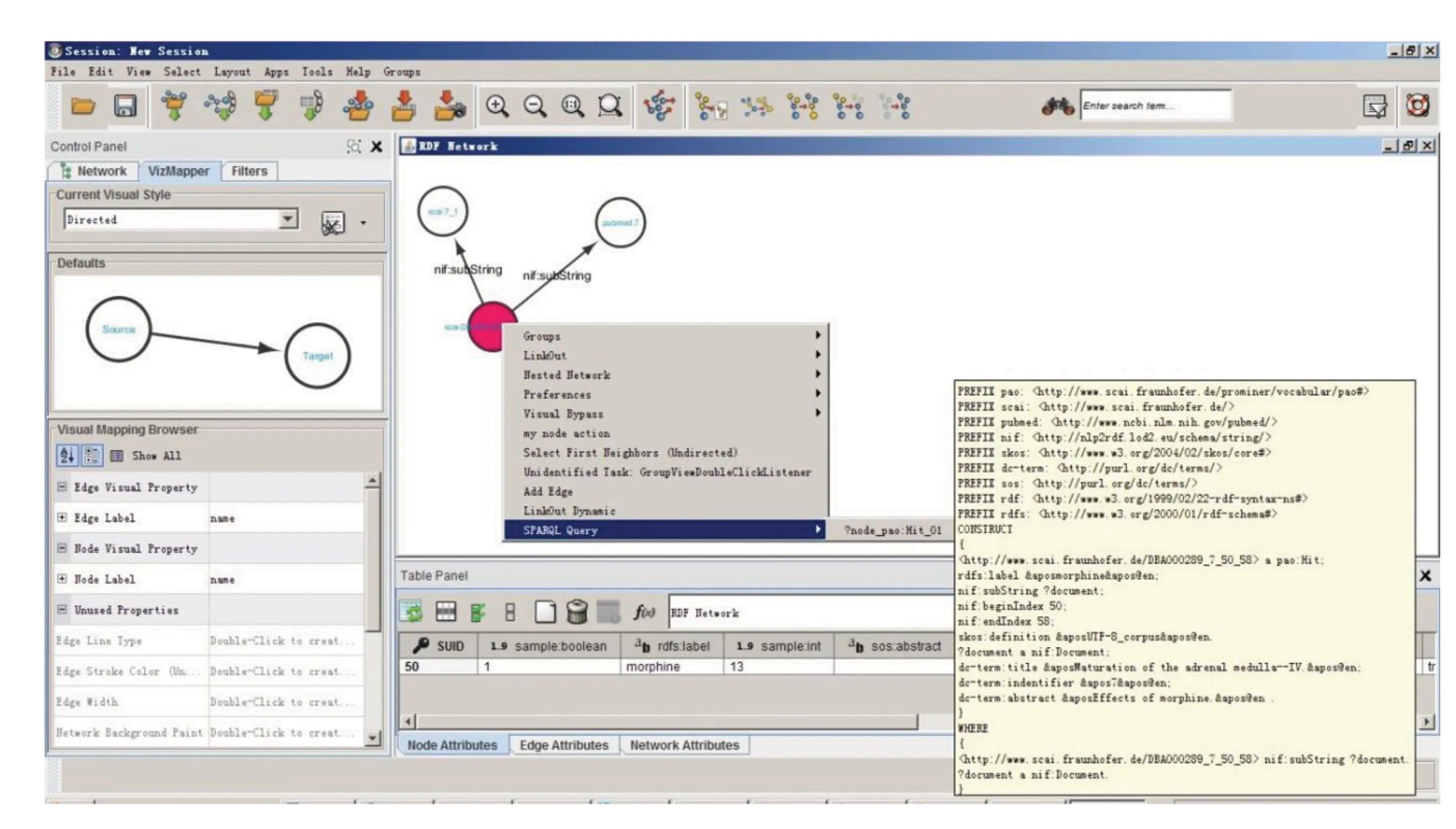
图4 参数化查询 “pao:Hit”本体知识示例
如图4所示,我们选中一个名为“scai:DBA000289_7_50_58”的一个节点,它代表一种吗啡的具体实例,这时系统就会自动地获取该节点的信息。其中该节点的RDF类型为“pao:Hit”,系统就会自动把适合RDF类型为“pao:Hit”的参数化查询模板显示出来。如图4中可以看到,系统直接把适合RDF类型为“pao:Hit”的“?node_pao…Hit_01”参数化选项显示在菜单栏中,只要用户点击选择,系统就会把名为“scai:DBA000289_7_50_58”的节点的URI“http://www.scai.fraunhofer.de/DBA000289_7_50_58”替换掉名为“?node_pao…Hit_01”的参数化模板中的“!!!node_uri!!!”,生成新的SPARQL查询语句,并把可视化结果显示出来。
其中参数化查询选项是一个用来查询RDF类型为“pao:Hit”的Cytoscape节点的一个参数化模板,其作用在于查询吗啡实体节点“scai:DBA000289_7_50_58”在文章标题为“Maturation of the adrenal medulla--IV.”(即是研究吗啡对肾上腺髓质成熟期的影响的一篇文章)之间的关联。通过该参数查询,可以有效地查询到文章标题为“Maturation of the adrenal medulla--IV.”中关于吗啡节点“scai:DBA000289_7_50_58”的内容关联,通过其中的内容可以发现吗啡实例节点“scai:DBA000289_7_50_58”对肾上腺髓质的成熟期的影响。这样用户就可以直接使用参数化查询发现吗啡节点与文章标题为“Maturation of the adrenal medulla--IV.”之间的关联,用户不需要手动构建查询语句、不需要掌握SPARQL语法、不需要编写繁琐的SPARQL查询语句,只要导入了参数化模板包,就可以通过鼠标点击选择“参数化查询选项”直接实现SPARQL查询,这是一个更快捷方便的SPARQL查询工具。
5 结语
此插件将提交到Cytoscape官方网站的插件库中,以供全球用户(特别是生物医学研究人员)下载和免费使用。在程序源代码开源的基础之上进一步吸纳Cytoscape用户和开发者的反馈意见,并在程序功能、用户界面和系统性能上进行了改进。另外,将创建和共享一系列常用的SPARQL模板包,并且在生物医学领域的高校和研究所进行推广,对有兴趣参与共享SPARQL模板包的机构或人员提供培训和指导。

