细菌对利福平耐药机制研究进展
徐凯悦(XU Kai-yue),强翠欣(QIANG Cui-xin),赵建宏(ZHAO Jiang-hong)
(河北医科大学第二医院 河北省临床检验中心,河北 石家庄 050000)
·综述·
细菌对利福平耐药机制研究进展
徐凯悦(XU Kai-yue),强翠欣(QIANG Cui-xin),赵建宏(ZHAO Jiang-hong)
(河北医科大学第二医院 河北省临床检验中心,河北 石家庄 050000)
利福平; 耐药机制; RNA聚合酶; 利福平耐药决定区; rpoB
利福霉素为袢霉素类家族成员,可用于治疗多种感染,如结核病、耐甲氧西林金黄色葡萄球菌感染、艰难梭菌引起的艰难梭菌相关性腹泻(Clostridiumdifficile-associated diarrhea, CDAD)、衣原体的持续感染[1]、旅行者腹泻[2]等。然而,利福霉素广泛用于临床感染治疗的同时,细菌耐药问题却使该杀菌“利器”变得“钝化”。结核分枝杆菌极易对利福平(rifampin, RIF)耐药,约有3.6%新增结核病和20.2%复治结核病为耐多药结核病[3]。CDAD是成人医院获得性腹泻的主要原因[4],利福霉素类中利福昔明因其口服不易被吸收,用于治疗复发性CDAD[5],然而艰难梭菌对利福霉素耐药率高达11%[6]。如何在正常发挥药物抗菌作用的同时减少细菌耐药的产生,关系临床抗感染治疗的成败。本文在介绍RIF抗菌机制的基础上,对细菌耐RIF的机制予以综述,为临床开发新的细菌转录抑制剂类药物提供帮助,以便更加有效的治疗感染。
1 RIF抗菌机制
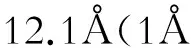
因此,细菌可通过β亚基的编码基因—rpoB突变,造成氨基酸的改变,导致对RIF亲和力降低,进而对RIF耐药。然而作用靶点突变并不是唯一的耐药机制,其他耐药机制亦有报道,如对药物的修饰灭活作用、膜通透性改变、外排泵等。
2 rpoB突变是细菌对利福平耐药(RIFr)主要机制
有关大肠埃希菌、金黄色葡萄球菌和结核分枝杆菌等细菌对RIF的耐药机制已有报道,其中最常见且可引起高水平耐药的机制为rpoB突变。
由于RNAP在细菌中较为保守,因此RIFr株的rpoB突变位点也较保守,故常将其他细菌的突变位点与大肠埃希菌相应序列进行比对。本文涉及不同细菌耐药突变位点时,除非有特别说明,一律使用大肠埃希菌β亚基氨基酸的相应位点进行描述。
2.1 大肠埃希菌RIFr尽管大肠埃希菌感染不在RIF的适应证范围,但大肠埃希菌为细菌研究的模式菌株,且该菌转录起始及终止过程研究也较为详尽。20世纪80年代,Jin等[8]对引起大肠埃希菌RIFr的rpoB位点突变进行研究,结果发现耐药突变位点位于rpoB的中部,分为三个区域:cluster I(507-533位氨基酸),cluster II(563-572位氨基酸),cluster III(687位氨基酸)。rpoB中所包含这些耐药突变的区域称为利福平耐药决定区(rifampin resistance determing region, RRDR)。上述提到的水生栖热菌RNAP中与RIF存在氢键或范德华力的12个氨基酸,均位于RRDR cluster I—II中,且除E445(水生栖热菌RpoB蛋白编码)外,其他11个氨基酸的突变均可引起RIFr[7]。此外,还有一个靠近β亚基N端的区域(N cluster),其突变亦可导致RIFr。研究[9]发现,大肠埃希菌RIFr株β亚基第146位氨基酸存在替换,而该位点位于cluster I—III外。随后, Severinov等[10]通过基因定点突变方法,证实了rpoB N cluster(143-148位氨基酸)突变可导致RIFr。
正如Jin等[8]所假设的,核心酶中β亚基各部分间似乎通过彼此合作形成RIF结合区。利用某些间接方法,我们可知大肠埃希菌RNAP活性中心及其与RIF结合位点相互作用的拓扑结构,如将聚合酶-启动子复合物与β亚基交联到一起后,进行限制性酶解和化学降解,发现RRDR中的cluster I参与活性中心的形成[11]。
2.2 结核分枝杆菌RIFr结核分枝杆菌为缓慢生长菌,其鉴定及传统药敏试验约需12周,时间长,使得患者早期不能得到个性化的治疗,增加耐药株出现的风险。而RIFr为耐多药结核的一个可靠标志,到目前为止,大部分RIFr结核分枝杆菌同时也对异烟肼耐药[12]。因此,尽可能全面地了解结核分枝杆菌中与RIFr有关的rpoB突变,有利于从分子角度快速检测耐药株。
结核分枝杆菌RIFr突变主要集中在rpoB中一个长度为81bp(β亚基第507-533位氨基酸,位于cluster I内)的热点突变区中,称为结核分枝杆菌的RIFr决定区。报道显示,约95%的结核分枝杆菌RIFr突变位于该区内[13],其中第516、526和531位氨基酸替换最多见[14]。Jamieson等[15]在2株耐多药结核分枝杆菌中发现V146F替换,而该位点位于大肠埃希菌rpoB N cluster(β亚基第143-148位氨基酸),同样引起RIFr[10]。
了解结核分枝杆菌RIFr株的rpoB突变情况,有助于利用分子方法快速检测疑似感染患者体内的结核分枝杆菌及其耐药性。现已有多种快速检测结核分枝杆菌RIFr株的方法,如高分辨率溶解曲线分析[16]以及基于锁核酸(locked nucleic acid, LNA)探针的实时PCR技术[17]等。2013年美国食品药品管理局(FDA)批准了一种商品化的基于PCR法检测痰标本中结核分枝杆菌DNA的试剂盒,可同样检测RIFr株[18]。
2.3 艰难梭菌RIFr艰难梭菌是专性厌氧革兰阳性芽孢杆菌, 被认为是引起抗生素相关性腹泻的主要病原菌之一,严重感染者可发生假膜性肠炎、肠坏死甚至死亡。近年由于艰难梭菌高产毒株(027/NAP1/BI型)在世界多个地区的暴发流行,使艰难梭菌成为医院获得性腹泻的主要病原菌[19]。利福霉素类抗生素,尤其是利福昔明可用于治疗复发性CDAD[5]。然而,艰难梭菌对利福霉素的耐药时有发生。据现有文献[6]估计,艰难梭菌对RIF的耐药率达11%,且有升高趋势。Freeman等[20]对欧洲22个国家艰难梭菌耐药监测发现,17个国家出现RIFr艰难梭菌,尤其是意大利、捷克、丹麦和匈牙利出现了高比率耐药(57%~64%)。有报道[21]指出,利福霉素暴露是患者发生耐药艰难梭菌感染的危险因素之一,甚至在用利福霉素治疗期间就可发生耐药艰难梭菌感染[22]。面对日益严峻的耐药形势,了解其耐药机制尤为重要。目前认为艰难梭菌RIFr通常与rpoB突变有关,涉及的氨基酸替换为第502、505、548、488、492和498位(艰难梭菌RpoB蛋白编码)等,大多对应于RRDR cluster I,其中第502、505位氨基酸替换最为多见[21,23-27]。
3 其他RIFr机制
除上述耐药机制之外,某些菌属本身的RNAP对RIF不敏感,如密螺旋体属、疏螺旋体属、端螺旋体属和一些土壤放线菌,原因在于其rpoB第531位的密码子为天冬酰胺而非敏感菌属中的丝氨酸[28]。
除rpoB突变外,细菌可通过其他方式对RIF产生低水平耐药,如RNAP结合蛋白的保护作用、对RIF修饰灭活、膜通透性改变和外排泵的过度表达等。
天蓝色链霉菌可耐受低浓度RIF,Newell等[29]研究显示其RNAP结合蛋白RbpA介导该菌的低水平耐药,另外,该研究发现在结核分枝杆菌、麻风分枝杆菌和白喉棒状杆菌中亦存在rbpA同源基因。近来有研究[30]显示,结核分枝杆菌RbpA通过间接的方式介导耐药,并不影响RNAP对RIF的敏感性,而是调整RNAP核心酶结构,增加核心酶对σA的亲和性,加速σA指导的转录,从而产生耐药。同时,研究发现,RbpA结合于RNAP β亚基Sandwich-Barrel Hybrid模体上,且该区域并不与RIF结合位点重合。DnaA蛋白可与RNAP结合,在一定程度上削弱RIF对RNAP的抑制作用[31]。Dey等[32]经体内及体外研究证实,耻垢分枝杆菌MsRbpA蛋白可与RNAP β亚基相互作用,提高RNAP对RIF的耐受水平,介导低水平耐药,同时该研究发现MsRbpA同源体保守存在于分枝杆菌属中。Weiss等[33]研究显示,在结核分枝杆菌中削弱CarD/RNAP β亚基的相互作用可增加该菌对RIF敏感性。
某些细菌可通过对RIF修饰使其失活从而产生耐药。常见的修饰方式为糖基化、核糖基化、磷酸化和脱色效应,其中脱色效应与单加氧酶有关[34]。在敲除皮疽诺卡菌rpoB2后,单加氧酶介导的脱色效应成为其耐药的主要方式[35]。耻垢分枝杆菌可使RIF核糖基化而对其天然耐药,该菌表达的Arr蛋白具有单ADP-核糖基转移酶作用,催化利福霉素类药物ADP-核糖基化,使其失活[36]。此外,该研究发现,arr同源基因广泛存在于环境细菌及嗜麦芽窄食单胞菌、洋葱伯克霍尔德菌、天蓝色链霉菌和谷氨酸棒状杆菌等细菌中。研究[37]显示,对RIF同系物C25位修饰后可抵抗耻垢分枝杆菌ADP-核糖基转移酶对其灭活作用。Spanogiannopoulos等[38]从环境放线菌中分离出1株RIFr放线菌—WAC1438,发现其可对RIF进行糖基化失活,rgt1438为其糖基转移酶编码基因。1994年研究[39]发现,细菌可以通过磷酸转移酶将RIF磷酸化而失活,然而,直到最近才有研究揭示了放线菌中该酶的编码基因rph[40]。该研究同时发现rph上游的一段保守序列——利福平相关元件(RAE),该序列与RIF灭活酶编码基因有关。同时研究指出,RIF敏感菌中亦存在rph同源基因,如产单核细胞李斯特菌和腊样芽孢杆菌。近来,Qi等[41]获得产单核细胞李斯特菌RIF磷酸转移酶(LmRPH)在不同催

另外,细菌还可通过降低膜通透性、调节外排泵或膜通道蛋白的表达而耐药。分枝杆菌属细菌由于其独特的富于脂质的细胞壁结构导致膜通透性低,因而对多种抗生素天然耐药。有研究[43]显示,耻垢分枝杆菌细胞壁中Wag31-AccA3与胞壁脂质的通透性和对亲脂性药物(如RIF)耐药有关,敲除Wag31后胞壁对亲脂性分子通透性增强,对RIF敏感性增加,AccA3过度表达则结果反之。许多细菌外排泵可将RIF排至细胞外,导致低水平耐药,如结核分枝杆菌[44-45]。波赛链霉菌可产生2种抗肿瘤分子——阿霉素和道诺霉素,其drrAB编码的DrrAB外排系统,属ABC结合盒类转运体,可将该2种分子排至细胞外。近有研究[46]显示,该转运体为多药转运体,RIF为其底物之一。耻垢分枝杆菌中,marRAB操纵子编码两种ABC结合盒类转运体,MarR蛋白通过调节marRAB操纵子的表达,进而影响细菌对RIF的耐药性。与其他外排泵不同的是,该菌中此2种ABC结合盒类转运体过度表达反而增加细菌对RIF的敏感性,因此,该菌中的MarR蛋白为阻遏蛋白,抑制marRAB操纵子的表达,产生耐药[47]。此外,某些细菌可通过膜表面通道蛋白的表达调节对RIF的耐药性。Danilchanka等[48]研究显示,牛分枝杆菌卡介苗膜通道蛋白CpnT的编码基因cpnT的突变,可导致RIFr,与此同时,研究人员认为该CpnT膜通道蛋白可能与临床分离的无法用已知耐药基因突变解释的结核分枝杆菌耐药有关。
4 总结与展望
rpoB RRDR碱基突变是细菌对RIF呈高水平耐药的主要机制。此外,细菌对RIF的共价修饰、RNAP结合蛋白的保护作用以及外排泵的过度表达等亦为细菌对RIFr的机制。对耐药机制的研究有助于优化治疗,减少耐药的产生,如外排泵抑制剂与利福霉素的联合应用或许可缩小单药对细菌的耐药突变选择窗,有利于减少耐药的产生;对利福霉素某些位点进行改造,抵抗灭活酶的修饰;根据细菌rpoB耐药突变情况,优化或开发新的利福霉素等。
[1] Rothstein DM, van Duzer J, Sternlicht A, et al. Rifalazil and other benzoxazinorifamycins in the treatment of chlamydia-based persistent infections[J]. Arch Pharm (Weinheim), 2007, 340(10): 517-529.
[2] Huang DB, DuPont HL. Rifaximin-a novel antimicrobial for enteric infections[J]. J Infect, 2005, 50(2): 97-106.
[3] World Health Organization. Global tuberculosis report 2013[S].WHO, 2013.
[4] Dubberke E.Clostridiumdifficileinfection: the scope of the problem[J]. J Hosp Med, 2012, 7 (Suppl 3): S1-S4.
[5] Mattila E, Arkkila P, Mattila PS, et al. Rifaximin in the treatment of recurrentClostridiumdifficileinfection[J]. Aliment Pharmacol Ther, 2013, 37(1): 122-128.
[6] Spigaglia P. Recent advances in the understanding of antibiotic resistance inClostridiumdifficileinfection[J]. Ther Adv Infect Dis, 2016, 3(1): 23-42.
[7] Campbell EA, Korzheva N, Mustaev A, et al. Structural mechanism for rifampicin inhibition of bacterial RNA polymerase[J]. Cell, 2001, 104(6): 901-912.
[8] Jin DJ, Gross CA. Mapping and sequencing of mutations in theEscherichiacolirpoB gene that lead to rifampicin resistance[J]. J Mol Biol, 1988, 202(1): 45-58.
[9] Lisitsyn NA, Sverdlov ED, Moiseyeva EP, et al. Mutation to rifampicin resistance at the beginning of the RNA polymerase beta subunit gene inEscherichiacoli[J]. Mol Gen Genet, 1984, 196(1): 173-174.
[10] Severinov K, Soushko M, Goldfarb A, et al.RifR mutations in the beginning of theEscherichiacolirpoB gene[J]. Mol Gen Genet, 1994, 244(2): 120-126.
[11] Severinov K, Mustaev A, Severinova E, et al. The beta subunit Rif-cluster I is only angstroms away from the active center ofEscherichiacoliRNA polymerase[J]. J Biol Chem, 1995, 270(49): 29428-29432.
[12] Chen X, Wang B, Yang W, et al. Rolling circle amplification for direct detection ofrpoB mutations inMycobacteriumtuberculosisisolates from clinical specimens[J]. J Clin Microbiol, 2014, 52(5): 1540-1548.
[13] Tan Y, Hu Z, Zhao Y, et al. The beginning of therpoB gene in addition to the rifampin resistance determination region might be needed for identifying rifampin/rifabutin cross-resistance in multidrug-resistantMycobacteriumtuberculosisisolates from Southern China[J]. J Clin Microbiol, 2012, 50(1): 81-85.
[14] Musser JM. Antimicrobial agent resistance in mycobacteria: molecular genetic insights [J]. Clin Microbiol Rev, 1995, 8(4): 496-514.
[15] Jamieson FB, Guthrie JL, Neemuchwala A, et al. Profiling ofrpoB mutations and MICs for rifampin and rifabutin inMycobacteriumtuberculosis[J]. J Clin Microbiol, 2014, 52(6): 2157-2162.
[16] Malhotra B, Goyal S, Bhargava S, et al. Rapid detection of rifampicin resistance inMycobacteriumtuberculosisby high-resolution melting curve analysis[J]. Int J Tuberc Lung Dis, 2015, 19(12): 1536-1541.
[17] Zhao Y, Li G, Sun C, et al. Correction: Locked nucleic acid probe-based real-time PCR assay for the rapid detection of rifampin-resistantMycobacteriumtuberculosis[J]. PLoS One, 2016, 11(6): e0157275.
[18] Centers for Disease Control and Prevention (CDC). Availability of an assay for detectingMycobacteriumtuberculosis, including rifampin-resistant strains, and considerations for its use-United States, 2013[J]. MMWR Morb Mortal Wkly Rep, 2013, 62(41): 821-827.
[19] Kelly CP, LaMont JT.Clostridiumdifficile-more difficult than ever[J]. N Engl J Med, 2008, 359(18): 1932-1940.
[20] Freeman J, Vernon J, Morris K, et al. Pan-European longitudinal surveillance of antibiotic resistance among prevalentClostridiumdifficileribotypes[J]. Clin Microbiol Infect, 2015, 21(3): 248.e9-248.e16.
[21] Curry SR, Marsh JW, Shutt KA, et al. High frequency of rifampin resistance identified in an epidemicClostridiumdifficileclone from a large teaching hospital[J]. Clin Infect Dis, 2009, 48(4): 425-429.
[22] Carman RJ, Boone JH, Grover H, et al. In vivo selection of rifamycin-resistantClostridiumdifficileduring rifaximin therapy[J]. Antimicrob Agents Chemother, 2012, 56(11): 6019-6020.
[23] O'Connor JR, Galang MA, Sambol SP, et al. Rifampin and rifaximin resistance in clinical isolates ofClostridiumdifficile[J]. Antimicrob Agents Chemother, 2008, 52(8): 2813-2817.
[24] Huang H, Weintraub A, Fang H, et al. Antimicrobial susceptibility and heteroresistance in ChineseClostridiumdifficilestrains[J]. Anaerobe, 2010, 16(6): 633-635.
[25] Spigaglia P, Barbanti F, Mastrantonio P, et al. Multidrug resistance in EuropeanClostridiumdifficileclinical isolates[J]. J Antimicrob Chemother, 2011, 66(10): 2227-2234.
[26] Miller MA, Blanchette R, Spigaglia P, et al. Divergent rifamycin susceptibilities ofClostridiumdifficilestrains in Canada and Italy and predictive accuracy of rifampin Etest for rifamycin resistance[J]. J Clin Microbiol, 2011, 49(12): 4319-4321.
[27] Nyc O, Krutova M, Liskova A, et al. The emergence ofClostridiumdifficilePCR-ribotype 001 in Slovakia[J]. Eur J Clin Microbiol Infect Dis, 2015, 34 (8): 1701-1708.
[28] Kim H, Kim SH, Ying YH, et al. Mechanism of natural rifampin resistance ofStreptomycesspp.[J]. Syst Appl Microbiol, 2005, 28(5): 398-404.
[29] Newell KV, Thomas DP, Brrekasis D, et al. The RNA polymerase-binding protein RbpA confers basal levels of rifampicin resistance onStreptomycescoelicolor[J]. Mol Microbiol, 2006, 60(3): 687-696.
[30] Hu Y, Morichaud Z, Chen S, et al.MycobacteriumtuberculosisRbpA protein is a new type of transcriptional activator that stabilizes the σ A-containing RNA polymerase holoenzyme [J]. Nucleic Acids Res, 2012, 40(14): 6547-6557.
[32] Dey A, Verma AK, Chatterji D. Role of an RNA polymerase interacting protein, MsRbpA, fromMycobacteriumsmegmatisin phenotypic tolerance to rifampicin[J]. Microbiology, 2010, 156(Pt 3): 873-883.
[33] Weiss LA, Harrison PG, Nickels BE, et al. Interaction of CarD with RNA polymerase mediatesMycobacteriumtuberculosisviability, rifampin resistance, and pathogenesis [J]. J Bacteriol, 2012, 194(20): 5621-5631.
[34] Tupin A, Gualtieri M, Roquet-Banères F, et al. Resistance to rifampicin: at the crossroads between ecological, genomic and medical concerns[J]. Int J Antimicrob Agents, 2010, 35(6): 519-523.
[35] Hoshino Y, Fujii S, Shinonaga H, et al. Monooxygenation of rifampicin catalyzed by the rox gene product ofNocardiafarcinica: structure elucidation, gene identification and role in drug resistance[J]. J Antibiot(Tokyo), 2010, 63(1): 23-28.
[36] Baysarowich J, Koteva K, Hughes DW, et al. Rifamycin antibiotic resistance by ADP-ribosylation: Structure and diversity of Arr[J]. Proc Natl Acad Sci U S A, 2008, 105(12): 4886-4891.
[37] Combrink KD, Denton DA, Harran S, et al. New C25 carbamate rifamycin derivatives are resistant to inactivation by ADP-ribosyl transferases[J]. Bioorg Med Chem Lett, 2007, 17(2): 522-526.
[38] Spanogiannopoulos P, Thaker M, Koteva K, et al. Characterization of a rifampin-inactivating glycosyltransferase from a screen of environmental actinomycetes[J]. Antimicrob Agents Chemother, 2012, 56(10): 5061-5069.
[39] Yazawa K, Mikami Y, Maeda A, et al. Phosphorylative inactivation of rifampicin byNocardiaotitidiscaviarum[J]. J Antimicrob Chemother, 1994, 33(6): 1127-1135.
[40] Spanogiannopoulos P, Waglechner N, Koteva K, et al. A rifamycin inactivating phosphotransferase family shared by environmental and pathogenic bacteria[J]. Proc Natl Acad Sci, U S A, 2014, 111(19): 7102-7107.
[41] Qi X, Lin W, Ma M, et al. Structural basis of rifampin inactivation by rifampin phosphotransferase[J]. Proc Natl Acad Sci, U S A, 2016, 113(14): 3803-3808.
[42] Stogios PJ, Cox G, Spanogiannopoulos P, et al. Rifampin phosphotransferase is an unusual antibiotic resistance kinase[J]. Nat Commun, 2016, 7: 11343.
[43] Xu WX, Zhang L, Mai JT, et al. The Wag31 protein interacts with AccA3 and coordinates cell wall lipid permeability and lipophilic drug resistance inMycobacteriumsmegmatis[J]. Biochem Biophys Res Commun, 2014, 448 (3): 255-260.
[44] Pang Y, Lu J, Wang Y, et al. Study of the rifampin monoresistance mechanism inMycobacteriumtuberculosis[J]. Antimicrob Agents Chemother, 2013, 57(2): 893-900.
[45] Li G, Zhang J, Guo Q, et al. Study of efflux pump gene expression in rifampicin-monoresistantMycobacteriumtuberculosisclinical isolates[J]. J Antibiot (Tokyo), 2015, 68(7): 431-435.
[46] Li W, Sharma M, Kaur P. The DrrAB efflux system ofStreptomycespeucetiusis a multidrug transporter of broad substrate specificity[J]. J Biol Chem, 2014, 289(18): 12633-12646.
[47] Zhang H, Gao L, Zhang J, et al. A novel marRAB operon contributes to the rifampicin resistance inMycobacteriumsmegmatis[J]. PloS One, 2014, 9(8): e106016.
[48] Danilchanka O, Pires D, Anes E, et al. TheMycobacteriumtuberculosisouter membrane channel protein CpnT confers susceptibility to toxic molecules[J]. Antimicrob Agents Chemother, 2015, 59(4): 2328-2336.
(本文编辑:熊辛睿)
Advances in rifampin resistance mechanism in bacteria
(The Second Hospital of Hebei Medical University Hebei Provincial Center for Clinical Laboratories, Shijiazhuang 050000, China)
2016-08-20
河北省自然科学基金(H2013206450);河北省科技厅基础条件平台建设项目(10966142D);河北医科大学第二医院科研基金(2h2201605)
徐凯悦 (1990-),女(汉族),河北省石家庄市人,检验师,主要从事细菌感染性病原体的检测与流行病学研究。
赵建宏 E-mail:zhaojh_2002@yahoo.com
10.3969/j.issn.1671-9638.2017.02.021
R378
A
1671-9638(2017)02-0186-05

