徐州四种市售食用菌的分子鉴定
胡扬楠 张扬 兰慧芳 殷婷婷 蒋秋改 谈思源 曾健康 温洪宇

摘 要:本文通过扩增食用菌的ITS序列对徐州市售双孢蘑菇、香菇、杏鲍菇和草菇的分类学地位进行了研究,将4种食用菌的ITS序列与Eztaxon数据库中真菌模式菌株数据进行比对,选取亲缘关系最近物种的ITS序列,采用Clustalx 1.8软件和MEGA5.0软件构建分子发育树。研究结果为双孢蘑菇、香菇、杏鲍菇和草菇的ITS序列长度分别为677bp、672bp、605bp和643bp;4种食用菌与亲缘关系最近物种构建的分子发育树表明,双孢蘑菇与模式菌株Agaricus bisporus(登录号:AJ133385)序列相似性为100%;香菇与模式菌株Lentinula edodes(登录号:JQ797414)序列相似性为100%;杏鲍菇与模式菌株Pleurotus ostreatus(登录号:AB733143)序列相似性为100%;草菇与模式菌株Volvariella volvacea(登录号:AY636049)序列相似性为99%。初步确定双孢蘑菇为蘑菇属的Agaricus bisporus;香菇为香菇属的Lentinula edodes;杏鮑菇为侧耳属的Pleurotus ostreatus;草菇为小包脚菇属的Volvariella volvacea。
关键词:食用菌;ITS序列;分子鉴定
中图分类号:S646 文献标识码:A DOI:10.11974/nyyjs.20160912001
我国食用菌种质资源丰富,而传统食用菌的分类鉴定主要是基于菌丝、孢子和子实体的形态学特征和生理生化特征。真菌形态特征易受到培养条件的影响,许多子实体类型难以获得,增加了分类鉴定的难度。真菌核糖体基因转录间隔区(Internal transcribed spacer,简称ITS)是位于真菌核糖体RNA上18S和28S基因之间的片段,其两侧分别是18S RNA基因和28S RNA基因[1],长度一般为650~750bp。White等于1990年设计了ITS1、ITS4、ITS5 3对可用于大多数担子菌和子囊菌的特异性鉴定引物[2],此后ITS序列分析技术在真菌分类和鉴定的研究中得到广泛应用。如李晶等采用形态学鉴定方法与现代分子生物学技术相结合将1种野生食用菌鉴定为金山巨菇(Tricholoma sp.)[3],雷琼等也采用同样的方法将一种大型食用菌鉴定为硫磺菌(Laetiporus sulphureus)[4]。本研究通过市售食用菌子实体的形态特征进行观察,结合ITS 序列分析初步确定分类学地位,为食用菌种质资源提供了依据。
1 材料与方法
1.1 供试材料
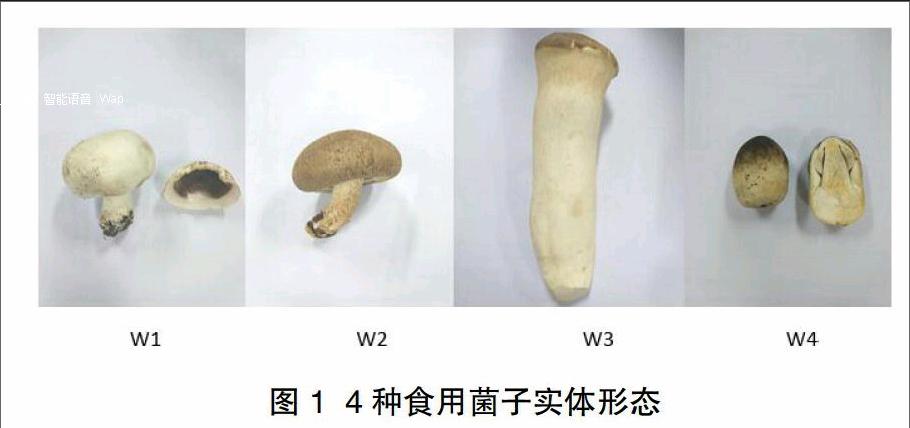
供试双孢蘑菇、香菇、杏鲍菇和草菇购自徐州市铜山新区农贸市场,编号分别为W1、W2、W3和W4。
1.2 子实体形态观察
取完整子实体,观察并描述菌盖、菌褶和菌柄等。
1.3 DNA提取
将新鲜的子实体烘干后粉碎成粉末并过筛,储存备用。称取适量样品粉末,放入研钵中加入液氮碾磨充分破碎细胞,将碾磨碎粉转入离心管中,样品DNA的提取方法参照改进的CTAB法[5]进行。提取的总DNA 经1%琼脂糖凝胶电泳检测。
1.4 ITS区段的PCR扩增
采用通用引物ITS1(5-TCCGTAGGTGAACCTGCGG -
3')与ITS4(5'- TCCTCCGCTTATTGATATGC -3')[2]扩增食用菌的ITS序列。PCR反应条件为:94℃预变性5min; 94℃变性40S,55℃退火45S,72℃延伸1min,30个循环;最后72℃延伸10 min。PCR产物4℃条件下保存。取5μL产物进行1%的琼脂糖凝胶电泳检测。
1.5 PCR产物的纯化和测序
采用UNIQ-10柱式DNA胶回收试剂盒(上海生工)从琼脂糖凝胶电泳中回收DNA。具体步骤参照试剂盒说明。经胶回收纯化后的PCR产物送至南京斯普金生物科技有限公司完成测序。
1.6 系统发育树的构建
将测序所得W1,W2,W3,W4 4种食用菌的ITS序列与Eztaxon数据库中真菌模式菌株数据进行比对,选取亲缘关系最近物种的ITS序列,采用Clustalx 1.8软件和MEGA5.0软件以Neighbor-Joining法构建分子发育树[6],重复取样1000次[7] 。
2 结果与分析
2.1 子实体形态观察
食用菌子实体形态见图1,具体特征如下:
W1:菌盖近半球形,白色,光滑;菌肉白色,厚;菌褶褐色至黑褐色,密,窄;菌柄近圆柱形,白色,光滑;具菌环,单层,膜质。
W2:菌盖扁平,边缘内卷,表面浅褐色至深褐色,边缘常有毛状或絮状鳞片;菌肉白色,稍厚;菌褶白色,密;菌柄中生,白色,弯曲。
W3:菌盖圆碟状,浅褐色;菌褶延生,密集,乳白色;菌柄偏生,中实,棒状,表面平滑,无菌环。
W4:菌盖近钟形,中部稍凸起且色深,边缘完整;菌褶着生于菌盖下方,稍密;菌柄着生于菌盖下方中央,白色稍带黄色。
2.2 系统发育分析
基于ITS基因序列,对W1,W2,W3,W4与亲缘关系近的模式菌株进行分子发育学分析,构建的系统发育树见图2。结果表明:W1与模式菌株Agaricus bisporus (登录号:AJ133385)聚为一支,置信度为100%,ITS序列相似性为100%,初步确定食用菌W1为蘑菇属的双孢蘑菇Agaricus bisporus;W2与模式菌株Lentinula edodes(登录号:JQ797414)聚为一支,置信度为100%,序列相似性为100%,初步确定食用菌W2为香菇属的Lentinula edodes;W3与模式菌株Pleurotus ostreatus(登录号:AB733143)聚为一支,置信度为100%,序列相似性为100%,初步确定食用菌W3为侧耳属的Pleurotus ostreatus;W4与模式菌株Volvariella volvacea(登录号:AY636049)聚为一支,置信度为100%,序列相似性为99%,初步确定食用菌W4为小包脚菇属的Volvariella volvacea。
3 讨论
食用菌具有较高的营养价值、药用价值和市场价值。本文4种食用菌均为徐州市场常见菌类,且具有种植技术成熟,菌体产量高等特点,有较高的食用和经济价值。食用菌采收以后,组织柔嫩,含水量高,后熟作用强,容易变质,不耐贮藏[8],徐州地处江苏北部,拥有便捷的交通运输业,使得市售食用菌的种类众多,加之徐州食用菌种植历史悠久,栽培经验丰富,发展优势明显[9]。加强食用菌种质资源的鉴定和挖掘对于发展食用菌产业具有重要意义。
采用传统形态学鉴定具有易于观察和比较优势,但其主要根据基因的表型进行研究,缺乏多态性指标,且容易受到多種因素的影响,尤其是地域分隔会造成食用菌种内的形态多样性较高的现象,给种和亲缘种的鉴定带来一定的难度和分歧[3]。本文结合ITS序列分析对常见菌进行分子鉴定,结果表明4种食用菌分别是蘑菇属的双孢蘑菇(Agaricus bisporus),香菇属的香菇(Lentinula edodes),侧耳属的杏鲍菇(Pleurotus ostreatu),小包脚菇属的草菇(Volvariella volvacea),研究结果系统完善了徐州市常见食用菌的种质资源,为进一步筛选优种、提高产量、优化母种等研究提供了理论基础。
参考文献
[1]赵欢,吴卫,郑有良,等.核糖体DNA ITS序列分析在药用植物研究中的应用[J]. 时珍国医国药,2009(4):959-962.
[2]White T J, Burns T , Lee S.Analysis of phylogenetic relationships by amplification and direct sequencing of ribosomal RNA genes[C].PCR protocols: A Guide to Methods and Applications. New York :Academic,1990,15-22.
[3]李晶,林冬梅,林占熺,等.一种野生食用菌的分子及形态学鉴定[J]. 现代农业科技,2012(8):110-111, 115.
[4]雷琼,汪俭,范晓丹,等.1株野生大型食用菌的分子鉴定与同源性分析[J]. 长江大学学报(自科版),2013(35): 7,52-55,68.
[5]臧金平,连宾,袁生.简便易行的食用菌菌丝体基因组DNA提取法[J].食品科学, 2005,26(3):66-68.
[6]Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic tree[J].Molecular Biology and Evolution,1987,4(4):406-425.
[7]Felsenstein J. Confidence limits on phylogenies:An approach using the bootstrap[J].Evolution,1985,39(4):783-791.
[8]叶自新. 蘑菇保鲜及防止其变色的研究[J]. 食品与发酵工业,1982(05):32-38.
[9]陆正丽,秦越华. 徐州市食用菌产业发展的优势与策略[J]. 现代农业科技,2008(14):93,96.
作者简介:胡扬楠(1993-),女,大学本科,研究方向:食用菌种质资源;温洪宇,博士,硕士生导师,研究方向:环境微生物学。

