生活垃圾填埋场对河流抗生素抗性基因的影响
黄福义,安新丽,2,李 力,欧阳纬莹,2,李 虎,2,苏建强*(.中国科学院城市环境研究所,城市环境与健康重点实验室,福建 厦门 602;2.中国科学院大学,北京 00049;.丽江市环境监测站,云南 丽江 67400)
生活垃圾填埋场对河流抗生素抗性基因的影响
黄福义1,安新丽1,2,李 力3,欧阳纬莹1,2,李 虎1,2,苏建强1*(1.中国科学院城市环境研究所,城市环境与健康重点实验室,福建 厦门 361021;2.中国科学院大学,北京 100049;3.丽江市环境监测站,云南 丽江 674100)
为研究生活垃圾填埋场对河流抗生素抗性基因的影响,采用高通量荧光定量 PCR技术对厦门市某垃圾填埋场附近河流水中的抗生素抗性基因的多样性和丰度进行了分析.结果表明,垃圾填埋场背景点河水检测到了57种抗性基因,下游河流水检测到了158种抗性基因,下游河流150种抗性基因显著富集(P < 0.05),其中增加倍数最大的是磺胺类抗生素抗性基因dfrA1,达2299倍,沿河而下,特别是在垃圾填埋场下游河流水中,抗生素抗性基因检出率、富集倍数都显著增加,抗性基因污染加剧;可移动元件与抗性基因显著相关;生活垃圾填埋场可能对河流水抗生素抗性基因的多样性和丰度产生了影响.
生活垃圾填埋场;河流;抗生素;抗生素抗性基因
抗生素抗性基因(ARGs)被科学家认为是一种新型的环境污染物[1],逐渐引起了世界各国和各种环境健康组织的关注.与传统的化学污染物不同,抗生素抗性基因由于其固有的生物学特性,例如可在不同细菌间转移和传播甚至是自我扩增,可表现出独特的环境行为[2].各种环境介质中,包括猪场土壤[3],制药废水[4],流经市区的河流水体[5],河流沉积物[6],施用猪粪水稻土[7],垃圾渗滤液[8],甚至在城市自来水[9]中也检测出环境抗生素抗性基因.越来越多的研究表明,致病菌耐药性与环境微生物抗性和抗生素抗性基因有关[10-13].在医疗行业、畜牧养殖行业存在着抗生素过度和使用不当现象,抗生素使用剂量不断加大和抗生素使用种类不断增加,环境中的抗生素残留以及抗生素抗性微生物不断被发现,并因此形成了一个恶性循环,并可能导致人类无药可用的境地,对人类健康造成了极大的健康风险.世界卫生组织(WHO)在2015年11月16~22日开展了首个世界提高抗生素认识周的活动,并把活动周主题定为:慎重对待抗生素.
生活垃圾填埋场是城市生活垃圾、污水厂的处置污泥、垃圾焚烧厂飞灰残渣等固体废弃物的主要的最终堆放场所.受人类生产生活的影响,这些固体垃圾中往往有抗生素残留以及含有抗生素抗性的微生物[14].同时垃圾填埋场从开始垃圾填埋到填埋场封场后一直持续地产生渗滤液.垃圾渗滤液是伴随垃圾填埋场运营整个生命周期的二次污染物,具有污染物成份复杂,污染物浓度高的特点,处理工艺复杂,垃圾浓缩液深度处置难,整体处理成本高等棘手问题,也是目前水处理研究的难题之一[15].通过研究垃圾渗滤液中四环素类抗生素、磺胺类抗生素等抗生素极其抗性基因的分布情况,表明垃圾填埋场和垃圾渗滤液中高浓度的抗生素及抗生素抗性基因可能是被低估的环境抗生素抗性的来源之一[14].垃圾填埋场填埋受短时强降雨、使用年限、管理运营不善等因素的影响,垃圾雨水混合液以及垃圾渗滤液可能会冲刷或者渗漏到周围环境水体,从而直接或间接污染河流水体,可能使得河流水环境中微生物抗生素抗性得以增强,进而造成潜在的环境健康与安全风险.
本研究采用高通量荧光定量PCR(HT-PCR)技术,对厦门市某垃圾填埋场附近河流水的抗生素抗性基因进行了研究,从环境抗生素抗性整体集合的角度出发,对河流水中抗生素抗性基因的丰度和多样性进行分析,以期增加对河流水抗生素抗性基因污染的认识,进一步提高对生活垃圾填埋场营运管制水平,为垃圾填埋场及其伴生的渗滤液潜在环境健康风险评价与管理决策提供理论支撑.
1 材料与方法
1.1 河流水样品采集
采样点位于厦门市东部固废处理中心垃圾填埋场附近的河流.本河流发源于填埋场背后山谷,并最终从厦门市同安区汇入大海.填埋场垃圾填埋库区和渗滤液污水处理区符合《生活垃圾卫生填埋技术规范》CJJ17-2004[16]中关于距离河流和湖泊 50m以外的规定.采样点位分别为区域背景点 R0,根据所处区域的地势高低和雨水汇水流向,确定填埋库区和污水处理区雨污混合液/垃圾渗滤液潜在的溢流口/渗漏区的上游 R1和下游R2采样点(图1),R1和R2点附近的土地利用方式相似,除了与垃圾填埋及处理的相关活动和少量农田外,以原生态的自然环境为主.每个点位分别采集3L河流水样,3个采样平行,所采集的河流水样品放置于低温采样箱中并迅速运回实验室的-20℃冰箱中保存,用于环境样品的DNA提取.
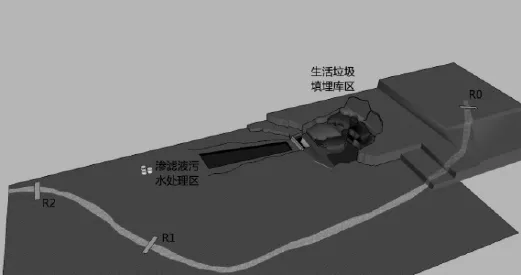
图1 河流水采样点位示意Fig.1 Sketch of sampling sites
1.2 河流水样品DNA提取
采用六联抽滤设备 JTFA0215(天津市津腾实验设备有限公司)对所采集的3个点位河流水样品进行抽滤,滤膜采用直径为50mm的0.22μm灭菌过滤膜(ADVANTEC,日本).
3个采样点位每张过滤膜上过滤的水样体积分别为1.5、0.5和0.5L.过滤好的滤膜分别用干净的镊子对折,用铝箔纸包好于-20℃冰箱中保存.取出滤膜,滤膜用灭菌过的手术剪剪成约3×5mm 的小纸块,将这些剪好的小纸块放入FastDNA® Spin Kit for Soil试剂盒(MP Biomedicals,美国)中的Lysing Matrix E tube,加入978μL试剂盒中自带的PBS缓冲液.所有经过预处理好的样品均采用FastDNA® Spin Kit for Soil试剂盒提取总DNA,提取操作步骤参照试剂盒自带说明书,最终洗脱DNA的体积为100 μL,所提取的DNA样品用QuantiFluor® dsDNA System (Promega Corporation,美国)试剂盒测定双链DNA 浓度.根据测定的样品DNA浓度,实验样品用灭菌的超纯水统一稀释至25ng/μL.最终DNA样品置于-20℃冰箱中保存.
1.3 抗生素抗性基因高通量荧光定量PCR
采用 SmartChip Real-Time PCR Systems (WaferGen Inc.,美国)高通量荧光定量反应平台.研究中所采用的 293对引物在先前的相关研究中被有效验证过[3].此外另外添加了 1对用于检定超级细菌抗生素抗性基因 blaNDM-1的引物
[13], 1对 intI1整合子基因 (class1integron)引物
[17]和1对CintI1临床医学意义上的整合子基因(clinical class 1integon)引物[18].
定量PCR扩增反应的体积为100nL.反应体系中各试剂的终浓度为:1×的 LightCycler 480SYBR®Green Master MixⅠ(Roche Inc.,美国),nuclease-free PCR-grade water,1ng/μL 的BSA,2.5ng/μL的DNA模板,1μmol/L的上下游引物.PCR反应条件为:95℃预变性10min;95℃变性30s,60℃退火延伸30s,总共40个循环;热循环仪Cycler的程序自动升温进行熔解曲线分析.高通量定量PCR每个芯片都有不添加DNA模板(用灭菌水超纯水替代)的阴性对照.qPCR反应得到的数据通过Cycler预设定的筛选条件(扩增效率介于 1.8~2.2)进行导出.根据 SmartChip Real-Time PCR Systems的灵敏度和精确度,参照DNA样品浓度,确定循环次数CT值为31时作为仪器的检测限.每个样品都进行3次技术重复实验,当3次技术重复都被扩增出来认为是阳性扩增,3个采样平行样品都是阳性扩增时,认为采样点的目的基因被有效检出.
1.4 数据分析处理
高通量荧光定量 PCR的数据采用 Excel 2010进行处理,采用R3.2.4软件[19]的vegan数据包进行PCA分析以及pheatmap数据包进行热图分析.河流水中微生物抗生素抗性基因的相对拷贝数(Gene Relative Copy Number)参照 Looft等
[20]的相关研究文献,用公式(1)进行估计计算抗生素抗性基因的丰度.根据Schmittgen等[21]关于相对定量分析方法,用Fold Change(FC)值用公式(2)来表征河流水中环境抗生素抗性的富集或者衰减状况.
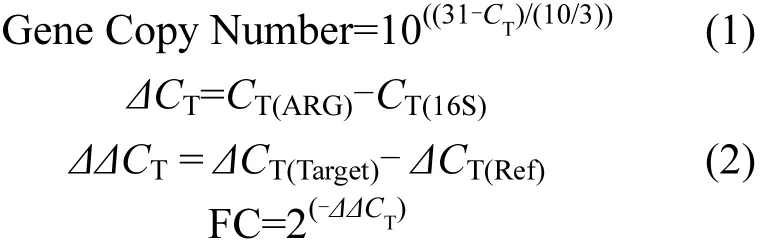
式中:CT是 PCR反应收集到特定荧光时的循环次数;ARG代表抗生素抗性基因;16S是代表16S rDNA基因;Target是环境实验样品;Ref为对照样品;FC值(Fold Change)是ARGs拷贝数的富集或衰减程度.当FC=2(-(ΔΔCT+2s))>1时,且student t检验具有显著性时,认为目标实验样品相对于对照样品显著富集.
2 结果与讨论
2.1 河流水中抗生素抗性基因的多样性
将检测的抗生素抗性基因类型分为氨基糖苷类抗性基因(Aminoglycoside)、β-内酰胺类抗性基因(Beta Lactamase)、喹诺酮类氯霉素类抗性基因(FCA)、大环内脂类-林肯酰胺类-链阳性菌素 B类抗性基因(MLSB)、磺胺类抗性基因(Sulfonamide)、四环素类抗性基因(Tetracycline)、万古霉素类抗性基因(Vancomycin)和其他类或者发挥外排泵作用(Other/efflux)八类抗生素抗性基因.河流的3个采样点检测到的抗生素抗性基因基本涵盖了目前已知的主要抗生素抗性类型(图2A),并且覆盖了3种主要的抗性机制类型——包括抗生素失活、外排泵机制和细胞保护.其中抗生素失活机制占的比例达44.88%,是河流水微生物抗生素抗性的最主要机制(图 2B).潜在的溢流口/渗漏区的下游 R2(158种)采样点检测到的抗生素抗性基因显著高于区域背景点R0(57种),并且抗性基因的多样性也显著升高(图2A),表明垃圾填埋场下游区域河流可能是潜在的抗性基因存储库.
区域背景点检测到的抗生素抗性基因都存在于潜在的溢流口/渗漏区的上游R1和下游R2采样点的水体中,这些抗性基因可能是垃圾填埋场区域“土著”的“背景抗性基因”,这些基因涵盖了主要的抗性基因类型和抗性机制,表明抗生素抗性基因在没有人类活动的区域也是广泛存在的,属于微生物的内在抗性[22].因此在 R1和 R2这 2个点检测到的其他类型的抗生素抗性基因可能是受垃圾填埋场填埋、运营过程中引入的“外源抗性基因”.进一步的分析表明,“背景抗性基因”和“外源抗性基因”都有独特的组成特征(图3),下游R2采样点β-内酰胺类抗生素抗性基因(Beta Lactamase)的比例增加,并且出现了磺胺类抗生素抗性基因(Sulfonamide)类型.β-内酰胺类抗生素是医疗行业最重要的一类抗感染药物
[23-24],也是普通居民在药店可以购买得到并常常被使用的抗生素;而磺胺类药物是一种广谱抗菌药,临床上主要用于预防和治疗感染性疾病.日常生活未被使用而且弃置的这两类抗生素往往随着生活垃圾被运送到了城市生活垃圾填埋场,垃圾填埋物中潜在的抗生素类型与检测到的抗性基因类型一致[25].因此,这两类抗性基因在垃圾填埋场检出,表明垃圾填埋等人类活动影响可能改变了河流水的抗性基因组成,可以被认为是“外源抗性基因”.
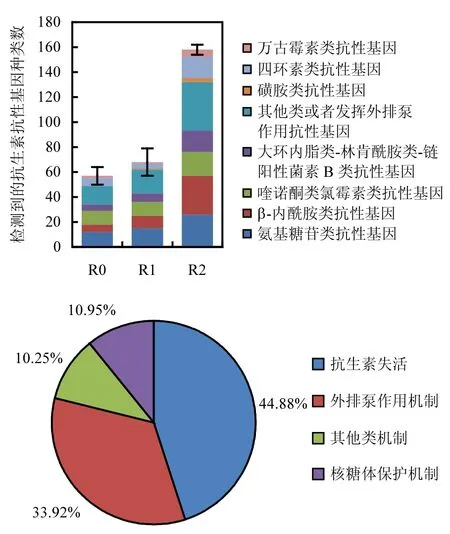
图2 河流水抗生素抗性基因检出种类特征Fig.2 Detected ARGs in river
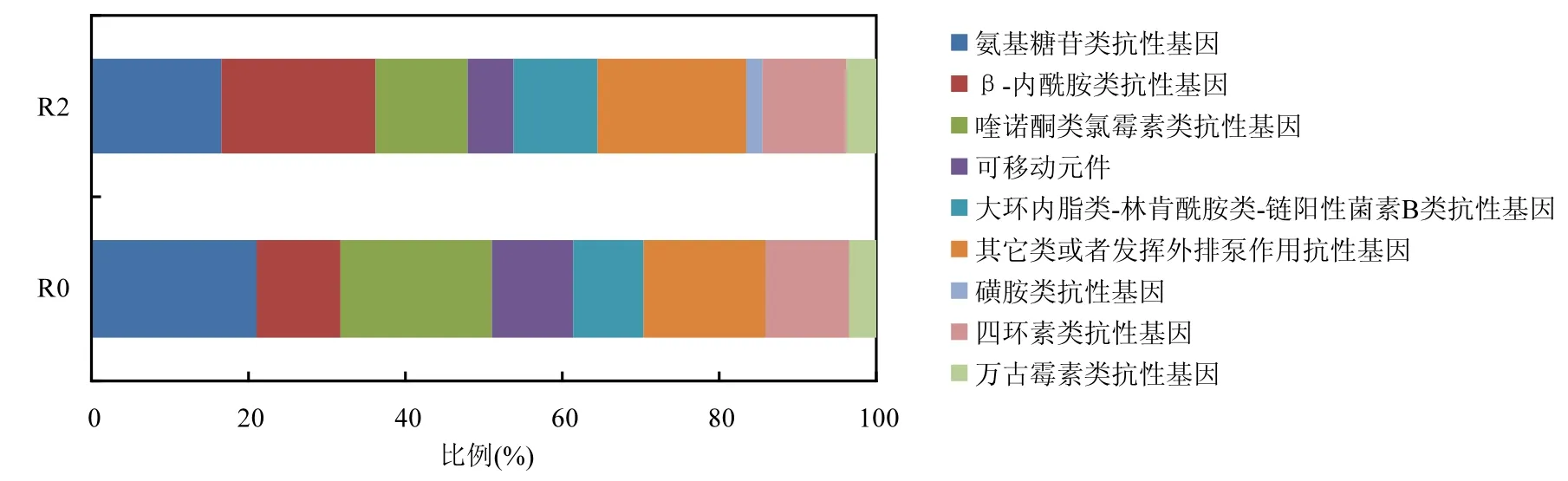
图3 河流水检出抗性基因类型比例特征Fig.3 Ratios of detected ARGs in river
2.2 河流水中抗生素抗性基因分布结构
垃圾填埋场下游河流水中抗生素抗性基因的多样性较背景点显著增加,因此用主成分分析方法来进一步评估 3个河流水采样位点的抗生素抗性基因结构差异(图 4).在 PCA1轴上(解释量为84.8%),区域背景点R0与下游R2样品形成独立两簇,明显分开,经 mrpp显著性分析检验表明垃圾填埋场下游河流水抗性的分布基因结构与背景点呈现显著差异(P<0.01).同时上游R1和背景点R0比较接近,表明垃圾填埋场对R1采样点河流水抗性基因没有显著影响.
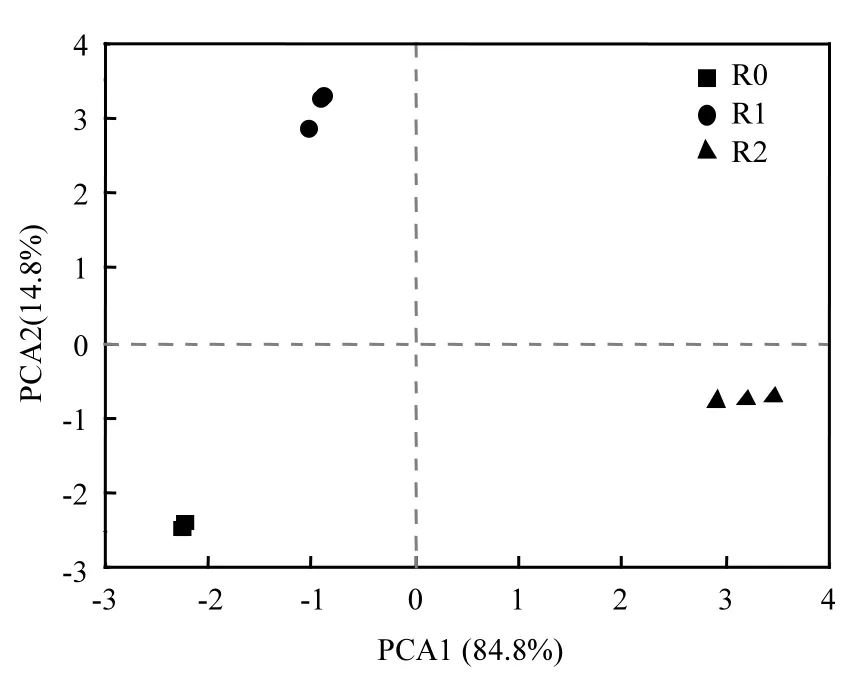
图4 河流水抗性基因主成分分析Fig.4 Principle component analysis of ARGs in river
把检测到的抗生素抗性基因用聚类热图进一步深入分析抗性基因在 3个采样位点中的分布格局和演变趋势(图 5).总体上看,样品的聚类分布结果与主成分分析结果一致,抗性基因可以聚为四类:Cluster Ⅰ,在背景点R0以极低丰度存在或者未检出,在上游R1和下游R2丰度略有增加;Cluster Ⅱ,在背景点R0和上游R1以低丰度存在或者未检出,在下游 R2以较高丰度水平存在;Cluster Ⅲ,在所有点位均有存在,并从上游到下游富集趋势依次递进;Cluster Ⅳ,所有点位均以高丰度存在,并且在下游采样点出现显著富集现象.因此,我们认为 Cluster Ⅲ和 Cluster Ⅳ是“背景抗性基因”类型,Cluster Ⅱ可以认为是“外源抗性基因”.垃圾填埋场附近河流水抗性基因与城市河流水的抗性基因的分布演变趋势格局相似[5].抗性基因热图表明,垃圾填埋活动及可能存在的雨污水冲刷和渗漏的影响下,河流水的“背景抗性基因”的丰度显著增加,而且河流水输入了“外源性抗性基因”,从上游到下游抗性基因污染加剧,改变了河流水抗性基因的分布格局.
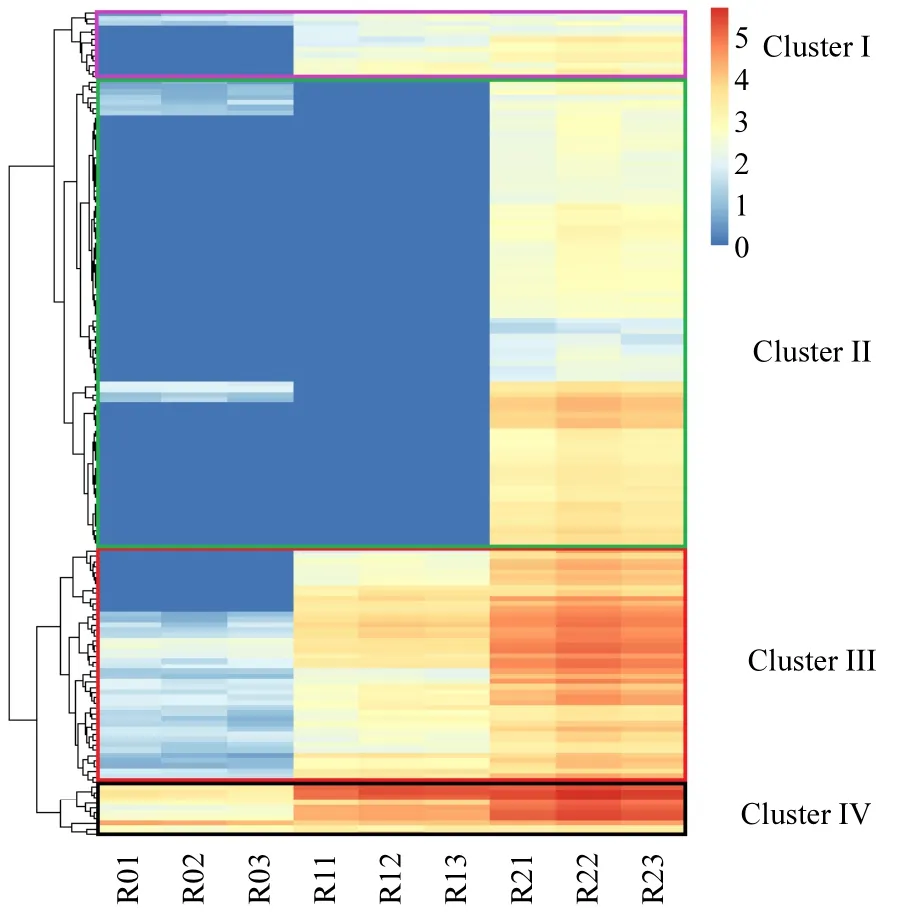
图5 河流水抗性基因相对丰度聚类热图Fig.5 Pheatmap for relative copy number of ARGs
2.3 下游河流水中富集的抗生素抗性基因
填埋场上游R1点与背景点R0抗生素抗性基因种类和丰度比较相似(没有显著性差异, P>0.05),因此我们着重分析垃圾填埋场下游河流水中抗性基因的富集情况.相对背景点 R0,垃圾填埋场下游河流水显著富集的抗性基因达到了150种,富集倍数最高的是磺胺类抗生素抗性基因dfrA1,达2299倍(图6).富集的抗性基因类类型涵盖了前述的八大类抗生素抗性类型,抗性类型种类数前三的分别是other/efflux、β-内酰胺类和氨基糖苷类抗生素抗性基因.富集结果表明,下游河流水高丰度存在的抗性基因也是富集的抗性基因,垃圾填埋场附近汇水土壤及其浅层地下水可能受到填埋场抗生素抗性基因污染.抗生素抗性基因在填埋堆体、垃圾渗滤液、附近土壤和浅层地表水的迁移、扩散和富集情况需要开展进一步的研究工作.

图6 下游河流水(R2)富集的抗生素抗性基因Fig.6 Enrichment ARGs in downstream (R2)
2.4 抗生素抗性基因与可移动元件的关系

图7 可移动元件与磺胺类类/大环内脂类-林肯酰胺类-链阳性菌素B类抗生素抗性基因丰度关系Fig.7 Corralation between MGEs and ARGs (Sulfonamide/MLSB)
Ciric等[26]证实了四环素类抗生素抗性基因与一些新型的可移动元件有关.在同一个微生物生境中,抗生素抗性基因可以随着可移动元件进行水平基因转移(HGT),从而使得抗性基因在不同种属微生物迁移传播[27-28].分析河流水中可移动元件(transposon转座子、intI1整合子基因和CintI1整合子三类)与各类抗生素抗性基因丰度关系表明,可移动元件(MGEs)与磺胺类(Sulfonamide)/大环内脂类-林肯酰胺类-链阳性菌素B类(MLSB)抗生素抗性基因均呈现极显著的线性相关(P<0.01),MGEs与 Sulfonamide和MLSB的 Pearson相关系数分别为 0.9474、0.9026(图7),可移动元件丰度高,抗性基因的丰度也高,进一步证实了垃圾填埋场附近河流水微生物中可移动元件与抗性基因的迁移传播富集有显著正相关的关系.
3 结论
3.1 抗生素抗性基因的多样性、检出率、丰度和富集程度沿着河流逐渐增加,在垃圾填埋场下游显著富集,抗性基因污染加剧;
3.2 河流的可移动元件丰度与抗性基因丰度显著相关,生活垃圾填埋场可能对河流水抗生素抗性基因产生了影响.
[1] Pruden A, Pei R, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants: studies in northern Colorado [J]. Environmental Science & Technology, 2006,40(23):7445-7450.
[2] 苏建强,黄福义,朱永官.环境抗生素抗性基因研究进展 [J]. 生物多样性, 2013,21(4):481-487.
[3] Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms [J]. Proceedings of the National Academy of Sciences, 2013,110(9): 3435-3440.
[4] 汤薪瑶,左剑恶,余 忻,等.制药废水中头孢类抗生素残留检测方法及环境风险评估 [J]. 中国环境科学, 2014,34(9):2273-2278.
[5] Ouyang W Y, Huang F Y, Zhao Y, et al. Increased levels of antibiotic resistance in urban stream of Jiulongjiang River, China [J]. Applied Microbiology and Biotechnology, 2015,99(13):5697-5707.
[6] Pei R, Kim S C, Carlson K H, et al. Effect of river landscape on the sediment concentrations of antibiotics and corresponding antibiotic resistance genes (ARG) [J]. Water Research, 2006, 40(12):2427-2435.
[7] 黄福义,李 虎,韦 蓓,等.长期施用猪粪水稻土抗生素抗性基因污染研究 [J]. 环境科学, 2014,35(10):3869-3873.
[8] 黄福义,李 虎,安新丽,等.城市生活污水和生活垃圾渗滤液抗生素抗性基因污染的比较研究 [J]. 环境科学, 2016,37(10): 3949-3954.
[9] Xu L K, Ouyang W Y, Qian Y Y, et al. High-throughput profiling of antibiotic resistance genes in drinking water treatment plants and distribution systems [J]. Environmental Pollution, 2016,213: 119-126.
[10] Hoge C W, Gambel J M, Srijan A, et al. Trends in antibiotic resistance among diarrheal pathogens isolated in Thailand over 15years [J]. Clinical Infectious Diseases, 1998,26(2):341-345.
[11] Weber D J, Raasch R, Rutala W A. Nosocomial infections in the ICU: the growing importance of antibiotic-resistant pathogens [J]. CHEST Journal, 1999,115(suppl_1):34S-41S.
[12] Fischbach M A, Walsh C T. Antibiotics for emerging pathogens [J]. Science, 2009,325(5944):1089-1093.
[13] Ahammad Z S, Sreekrishnan T, Hands C L, et al. Increased waterborne bla NDM-1resistance gene abundances associated with seasonal human pilgrimages to the Upper Ganges River [J]. Environmental Science & Technology, 2014,48(5):3014-3020.
[14] Allen S E, Boerlin P, Janecko N, et al. Antimicrobial resistance in generic Escherichia coli isolates from wild small mammals living in swine farm, residential, landfill, and natural environments in southern Ontario, Canada [J]. Applied and Environmental Microbiology, 2011,77(3):882-888.
[15] 余建恒,赵淑贤,夏耿东,等.接人垃圾渗滤液对城市污水厂运行的影响与对策 [J]. 中国给水排水, 2010,(4):95-97.
[16] CJJ17-2004 生活垃圾卫生填埋技术规范 [S].
[17] Stokes H W, Nesbø C L, Holley M, et al. Class 1integrons potentially predating the association with Tn402-like transposition genes are present in a sediment microbial community [J]. Journal of Bacteriology, 2006,188(16):5722-5730.
[18] Gillings M R, Gaze W H, Pruden A, et al. Using the class 1integron-integrase gene as a proxy for anthropogenic pollution [J]. The ISME Journal, 2015,9(6):1269-1279.
[19] R Core Team (2016). R: A language and environment for statistical computing. R Foundation for Statistical Computing, Vienna, Austria. URL https://www.R-project.org/.
[20] Looft T, Johnson T A, Allen H K, et al. In-feed antibiotic effects on the swine intestinal microbiome [J]. Proceedings of the National Academy of Sciences, 2012,109(5):1691-1696.
[21] Schmittgen T D, Livak K J. Analyzing real-time PCR data by the comparative CT method [J]. Nature Protocols, 2008,3(6):1101-1108.
[22] D'Costa V M, McGrann K M, Hughes D W, et al. Sampling the antibiotic resistome [J]. Science, 2006,311(5759):374-377.
[23] 张致平. β-内酰胺类抗生素研究的进展 [J]. 中国抗生素杂志, 2000,25(2):81-86.
[24] 杜德才,周书明,沈爱宗,等.医院抗菌药物使用强度分析 [J]. 中华医院感染学杂志, 2010,20(6):848-851.
[25] Wu D, Huang Z, Yang K, et al. Relationships between antibiotics and antibiotic resistance gene levels in municipal solid waste leachates in Shanghai, China [J]. Environmental Science & Technology, 2015,49(7):4122-4128.
[26] Ciric L, Mullany P, Roberts A P. Antibiotic and antiseptic resistance genes are linked on a novel mobile genetic element: Tn6087 [J]. Journal of Antimicrobial Chemotherapy, 2011,66(10): 2235-2239.
[27] Cruz F, Davies J. Horizontal gene transfer and the origin of species: lessons from bacteria [J]. Trends in Microbiology, 2000, 8(3):128-133.
[28] Kurland C G, Canback B, Berg O G. Horizontal gene transfer: a critical view [J]. Proceedings of the National Academy of Sciences, 2003,100(17):9658-9662.
High-throughput profiling of antibiotic resistance genes in river in the vicinity of a landfill.
HUANG Fu-yi1, AN Xin-li1,2, LI Li3, OUYANG Wei-ying1,2, LI Hu1,2, SU Jian-qiang1*
(1.Key Laboratory of Urban Environment and Health, Institute of Urban Environment, Chinese Academy of Sciences, Xiamen 361021, China;2.University of Chinese Academy of Sciences, Beijing 100049, China;3.Lijiang Environmental Monitoring Station, Lijiang 674100, China). China Environmental Science, 2017,37(1):203~209
Aquatic environment especially the river is an important reservoir of antibiotic resistance genes (ARGs). For the further insight into the ARGs in a river in the vicinity of a landfill, high-throughput qPCR technique was used to investigate the diversity and abundance of ARGs. The results showed that a total of 57ARGs was detected in control sample, while, 158ARGs was detected in the downstream river sample, in which 150ARGs in the downstream river were significantly enriched (P < 0.05). A maximum enrichment of 2299-fold was detected in dfrA1, which conferred resistance to sulfonamide. The detection number of ARGs and the enrichment of ARGs were significantly increased along the river. The abundance of mobile genetic elements was significantly correlated with the abundance of ARGs. These results implied that landfill may lead to the dissemination and enrichment of ARGs in river.
landfill;river;antibiotic;antibiotic resistance genes
X703.1
A
1000-6923(2017)01-0203-07
黄福义(1987-),男,福建连城人,硕士,助理工程师,主要研究方向为环境抗生素抗性基因污染.发表论文5篇.
2016-03-22
国家重点研发计划(2016YFD0800205);国家自然科学基金国际(地区)合作交流项目(21210008)
* 责任作者, 研究员, jqsu@iue.ac.cn

