传统中药材麦冬的DNA条形码鉴定
王广艳


摘 要:该文分析了DNA条形码序列(ITS、psbA-trnH、matK、rbcL和trnL-F)对采自10个不同产区的麦冬进行PCR扩增及测序。结果表明,ITS序列变异位点为11 bp且较稳定。叶绿体序列的变异位点较低。本研究构建了ITS及联合叶绿体片段与ITS的系统发育树,4个叶绿体片段各自构建的系统发育树结果不理想,以ITS构建的系统发育树为主。广西桂林、贵州贵定与浙江余杭聚为一支;四川什都、四川珙县与广东肇庆聚为一支;云南麻栗坡与云南石林聚为一支;江西庐山与湖南桑植聚为另一分支,位于发育树基部;以上分支均得到G+C含量和遗传距离的支持,种内具有较近的亲缘关系。ITS序列具有稳定的变异位点和鉴定位点,能够准确的鉴定不同产地的麦冬,基于ITS构建的系统发育树能够较好的区分其亲缘关系。
关键词:麦冬;ITS;叶绿体片段;遗传距离;亲缘关系
中图分类号 S567 文献标识码 A 文章编号 1007-7731(2016)24-0017-05
DNA Barcoding Technique to Identify Traditional Chinese Medicinal Materials Ophiopogon japonicas(L.f.)Ker-Gawl.
Wang Guangyan
(School of Life Sciences,the Province Key Laboratory of the Biodiversity Study and Ecology Conservation in Southwest Anhui,Anqing Normal University,Anqing 246011,China)
Abstract:DNA barcoding sequences(ITS,psbA-trnH,matK,rbcL,and trnL-F)were used to study O.japonicus from 10 different regions by PCR technique. The results showed that sequence variable sites of ITS were 11 bp and stable.Variable sites of chloroplast were low.In this study,the ITS and ITS+chloroplast phylogenetic trees were constructed,the phylogenetic trees based on chloroplast fragments were poor.ITS phylogenetic tree was dominant.O.japonicas from Guilin in Guangxi,Guiding in Guangzhou and Yuhang in Zhejiang comprised one closely clade;from Shendu in Sichuan,Gongxian in Sichuan and Zhaoqing in Guangzhou formed a closer clade;from Malipo in Yunnan and Shilin in Yunnan grouped into one closely clade;and from Lushan in Jiangxi and Sangzhi in Hunan formed another closely clade.All above clades were supported by G+C contents and genetic distances,and had closely relationships. The ITS sequence had stable variable sites and informative sites,and could accurately indentify O.japonicas from 10 different regions. The ITS phylogenetic tree could provide reliable genetic relationships.
Key words:Ophiopogon japonicas;Internal Transcribed Spacer;Chloroplast fragment;Genetic distance;Genetic relationship
麦冬为天门冬科(Asparagaceae)沿阶草属(Ophiopogon Ker-Gawl.)植物,其干燥块根具有养阴生津、润肺清心之功效,用于治疗肺燥干咳、虚劳咳嗽、津伤口渴、心烦失眠、内热消渴和咽白喉等症状,收藏于中华人民共和国药典中[1],其正品是Ophiopogon japonicus(L.f.)Ker-Gawl.。主产于浙江、四川、安徽、江西、湖南、河南等地,浙江、四川、广西等地均有栽培[2]。由于栽培环境与野生状态不同,其块根在形态上发生了部分变异,栽培品种较粗壮,野生品种较细小。随着麦冬栽培地区的扩大,流通品系增多,亟待对各品系进行质量评价研究。
DNA条形码(DNA barcoding)于2003年首次被作为物种鉴定的依据[3,4],并引起全世界科学界的关注[5-9]。DNA条形码鉴定是利用基因组中一段标准的DNA片段,对物种进行快速、准确地识别和鉴定的有效手段,具有鉴定过程快、操作简单、重复性和稳定性高等优点[10,11]。一些学者认为线粒体基因以及核基因中的单拷贝基因和内含子不可以作为物种鉴定的依据,原因是线粒体基因的变异率较低以及变异结构较少[12-16],核基因中的单拷贝基因和内含子缺乏通用引物的扩增[16]。rDNA ITS(rDNA internal transcribed spacer)区是核糖体DNA上的内转录间隔区,包括5.8S rDNA、ITS1(18S和5.8S之间)和ITS2(5.8S和16S之间),其长度比较保守,核苷酸序列变化较大,可以提供丰富的系统学信息,能反映种间差别和物种进化程度,且不易受药材加工而完全降解等特点,被广泛用于属间、种间及居群间的分子系统学及分子鉴定研究[17-19]。一些研究结果表明叶绿体基因(psbA-trnH、matK、rbcL和rpoC1)也可作为物种鉴定的依据[12,16,20-22]。
目前针对药用植物麦冬DNA条形码序列分析和鉴定的研究还较少,为此,本研究选取核基因(ITS)及叶绿体基因(psbA-trnH、matK、rbcL和trnL-F)对10个来源不同的麦冬进行序列测定,比较核基因和叶绿体基因序列,找出能准确鉴定麦冬的序列及鉴别位点,从分子水平上比较不同地区麦冬的亲缘关系。
1 材料与方法
1.1 材料 本实验选取10份不同来源地的麦冬材料。各样品采集地点见表1。凭证标本藏于中国科学院昆明植物研究所标本馆(KUN)。
1.2 DNA的提取与PCR扩增 总DNA的提取采用试剂盒法(Bioteke,Beijing,China)。PCR(聚合酶链式反应)扩增反应为25μL体系:0.5μL(10ng)DNA,0.5μL(4pmol)引物,0.5μLTaq酶(Promega),2μL2.5mM dNTP,2.5μL10×PCR Buffer,18.5μLH2O。用于PCR扩增和测序的引物以及扩增程序如下:1)ITS序列的引物用ITS4和ITS5,对于扩增不成功的样品,进一步用内含引物ITS2和ITS3[23],把ITS分为两个更短的片段来扩增:MF+ITS2和ITS3+MR,由于沿阶草属与鹿药属较为近缘,其中的MF(5' TCGAGACCCGAACGGACRAT 3')和MR(GTGCTCGGCATGGGTTCCTT)引自Meng等[24],94℃预变性3min,94℃变性30s,50℃退火30s,72℃延伸1min,32个循环,72℃继续延伸10min,保持温度为4℃;(2)psbA-trnH序列的引物用psbA-F和trnH-R[25,26],94℃预变性3min,94℃变性30s,49℃退火30s,72℃延伸1min,32个循环,72℃继续延伸10min,保持温度为4℃;(3)rbcL序列的引物用Z1和1024R[27,28],94℃预变性3min,94℃变性30s,55℃退火30s,72℃延伸1min,32个循环,72℃继续延伸10min,保持温度为4℃;(4)matK序列的引物用3F和1R[29],94℃预变性3min,94℃变性30s,50℃退火30s,72℃延伸1min,31个循环,72℃继续延伸10min,保持温度为4℃;(5)trnL-F序列的引物用Tab-c和Tab-f[30],94℃预变性3min,94℃变性30s,55℃退火30s,72℃延伸1min,31个循环,72℃继续延伸10min,保持温度为4℃。取5μLPCR产物在加有EB荧光染料的浓度为1%的琼脂糖凝胶中做电泳实验(110~230V)20min左右,在紫外光下照相,将条带清楚的PCR产物送公司测序。
1.3 数据处理 DNA序列的排序用Bioedit(ver.7.0.4.1)软件完成,排序后用MEGA(ver.7.0)软件基于Kimura2-parameter模型计算遗传距离,并采用类平均法(UPMEG)在MEGA(ver.7.0)软件中构建分子系统树,系统树各分支的置信度自举检测(bootstrap)1 000次。
2 结果与分析
2.1 碱基序列分析 10份麦冬样品序列比对后结果列于表2和表3。ITS序列的全长变化范围为600~699bp,平均G+C含量为9.96%,序列变异位点为11bp,信息位点比率为0.10%。其中,A占15.31%,T占18.08%,G占32.63%,C占33.98%。嘌呤的转换颠换率为K1=9.621,嘧啶的转换颠换率为K2=15.213。一般亲缘关系接近的物种具有更高的转换差异比例,这种差异在亲缘关系接近的类群中更为显著[31]。麦冬的ITS序列的核糖体DNA内转录间隔区ITS1和ITS2,与2个编码区5.8S和26S的界限参照Maianthemum dahuricum(Turcz.ex Fisch.et C.A.Mey.)LaFrankie[24]的序列资料确定。测定了10个产地样品的ITS1、5.8S和ITS2全序列,其中ITS1序列长度为257~281bp,平均G+C含量为27.23%;ITS2为224~230bp,平均G+C含量为30.05%。所有类群ITS序列的5.8S区长度均为143bp,平均G+C含量为36.94%。大部分序列变异发生在间隔区,5.8S区域很少发生变异。通常,G+C含量是反映亲缘关系的重要依据,亲缘关系越亲近其G+C含量相差越小[32]。从表2中可以发现,10种麦冬ITS区G+C含量为9.40%~11.17%,ITS1区G+C含量为25.96%~33.87%,ITS2区G+C含量为29.68%~30.89%,均差异较小,暗示不同产区麦冬遗传差异较小。这与碱基转换比率得出的结论一致。
psbA-trnH序列的全长变化范围为642~643bp,序列变异位点为1bp,信息位点比率为0.16%。其中,A占29.63%,T占34.10%,G占18.52%,C占17.76%。嘌呤的转换颠换率为K1=8.113,嘧啶的转换颠换率为K2=0。matK序列全长的变化范围为864~868bp,序列变异位点为42bp,信息位点比率为0%。其中,A占38.00%,T占29.94%,G占16.87%,C占15.19%。嘌呤的转换颠换率为K1=4.826,嘧啶的转换颠换率为K2=2.836。rbcL序列的全长变化范围为1 099~1 130bp,序列变异位点为10bp,信息位点比率为0.35%。其中,A占27.96%,T占29.44%,G占23.26%,C占19.34%。嘌呤的转换颠换率为K1=2.375,嘧啶的转换颠换率为K2=0。trnL-F序列全长的变化范围为927~933bp,序列变异位点为38bp,信息位点比率为0.53%。其中,A占36.01%,T占31.90%,G占16.22%,C占15.87%。嘌呤的转换颠换率为K1=24.245,嘧啶的转换颠换率为K2=52.473(表3)。
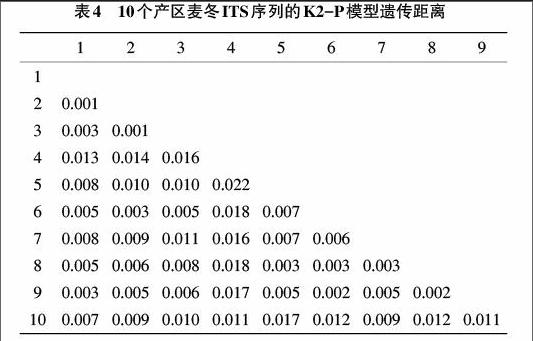
2.2 麦冬种内遗传距离分析 各产区麦冬样品的ITS序列之间的遗传距离见表4,遗传距离范围为0.001~0.022。浙江余杭与广西桂林、广西桂林与贵州贵定间的遗传距离均为0.001,表明三者间亲缘关系较近;贵州贵定与江西庐山间的遗传距离为0.022,暗示两者间亲缘关系较远。psbA-trnH遗传距离的范围为-0.000~0.003;matK遗传距离的范围为-0.000~0.048;rbcL遗传距离的范围为-0.000。-0.012;trnL-F遗传距离的范围为-0.000~0.039。基于叶绿体基因计算的遗传距离均出现负值(具体数值不再列出),说明以上叶绿体基因均不可作为不同产地麦冬鉴定或亲缘关系判断的依据。
2.3 系统发育树的构建 以类平均法(UPMEG)构建系统发育树。由于4个叶绿体片段的信息位点均较低(0~0.53%),其各自产生系统树的结果并不理想,因此系统发育树没有展示出来。联合的叶绿体片段与ITS的ILD检测表明两者并不冲突(P=0.09>0.05),因此将4个叶绿体片段与ITS联合分析构建系统发育树。ITS以及ITS与叶绿体联合构建的系统发育树见图1和图2。图1显示了基于ITS构建的系统发育树共分成分支I和分支II两大支,其中,分支I分成I1和I2两支,分支I1分成I11和I12两支,广西桂林、贵州贵定和浙江余杭聚为分支I11,四川什都、四川珙县和广东肇庆聚为分支I12;云南麻栗坡和云南石林聚为I2。江西庐山和湖南桑植聚为分支II。图2显示了基于ITS与叶绿体联合构建的系统发育树,除四川什都、云南麻栗坡及云南石林的系统位置与基于ITS构建的系统树不同外,其它分支均与基于ITS构建的系统发育树一致。由于叶绿体序列的分辨率低,本研究以ITS构建的系统树为主来讨论不同产地麦冬的亲缘关系。
3 讨论
3.1 核基因与叶绿体基因序列在中药麦冬鉴定中的价值 分子生物学的发展为中药鉴定提供了新技术和新方法,分子标记技术从诞生至今已发展至几十种,DNA条形码技术是分子鉴定的最新发展。该技术具有快速、准确、微量和通用性强等特点,不受发育阶段、供试部位、环境条件等外在条件的影响[33]。ITS序列因其基因片段短,高度变异且有长度上的保守性,在被子植物中被广泛应用[34]。越来越多的叶绿体序列也被用于药用植物鉴定和品质研究中,Cao等[35]通过测定三七的matK基因序列得出:matK基因序列可以成为三七正品基原鉴定的准确、有效手段。Kress等[16]报道了psbA-trnH序列可以作为鉴定多种被子植物的有效序列。Newmaster等[36]从GenBank上分析了1 000多条rbcL序列,证实了rbcL序列可以有效鉴定约85%的同属植物。
中药麦冬外部形态特征变异较大且栽培广泛,具有较多的野生种和栽培种。本研究表明,10个产区麦冬植物的ITS1和ITS2的碱基序列存在一定的变异,且变异位点稳定共11bp,信息位点比率为0.10%。变异主要为77位的G取代A,107位的C取代T,177位的T取代C,224位的C取代A,418位的A取代G,429位的C取代T,444位的T取代C,451位的A取代G,483位的C取代T,501位的C取代T,529位的A取代C(表5)。而5.8SrDNA在麦冬不同产地间高度保守,均为143bp,且无变异位点。虽然麦冬样品的ITS序列信息位点不是很高,但是稳定的变异位点是麦冬鉴定的依据。同时,基于ITS构建的系统发育树,10个产区的麦冬分支较为明显,将麦冬植物分成两大分支(图1)。综上所述,ITS序列可以准确的鉴定不同产区的麦冬。
叶绿体基因psbA-trnH的变异位点仅为1bp,10个产区的麦冬序列并无显著差异,且基于psbA-trnH构建的系统发育树多为平行分支;matK基因虽然具有较高的变异位点42bp,但其信息位点为0,且构建的系统发育树也多为平行分支;rbcL基因虽然变异位点为10bp,但构建的系统发育树无明显分支;trnL-F基因虽然变异位点为38bp,但构建的系统发育树均为平行支,无明显分支。同时,psbA-trnH的遗传距离范围为-0.000~0.003;matK的遗传距离范围为-0.000~0.048;rbcL的遗传距离范围为-0.000~0.012;trnL-F的遗传距离范围为-0.000~0.039,基于叶绿体基因计算的遗传距离均出现负值(具体数值不再列出)。综上所述,叶绿体基因psbA-trnH、matK、rbcL和trnL-F均不可作为鉴定麦冬的依据。
3.2 不同产区麦冬的亲缘关系 不同产区的ITS序列存在种内变异,基于ITS构建的系统发育树中,将不同产区的麦冬分成两大分支,分支I和分支II(图1)。其中,分支I分成分支I1和分支I2两支。分支I2的麦冬均产自云南,G+C含量分别为10.45%和11.17%,G+C含量较为接近,G+C含量是反映亲缘关系的重要依据,亲缘关系越亲近其G+C含量相差越小[32],且两者的遗传距离仅为0.003,以上结果均显示两者的亲缘关系较近。分支I1又分成I11和I12两支。其中广西桂林、贵州贵定和浙江余杭聚为一支,三者形态特征略有差异,可能是由于生境的差异引起的。三者的G+C含量分别为9.40%、9.41%和9.71%,广西桂林与贵州贵定的遗传距离为0.001,广西桂林与浙江余杭的遗传距离为0.001,贵州贵定与浙江余杭的遗传距离为0.003,以上均证实三者具有较近的亲缘关系。四川什都、四川珙县和广东肇庆聚为一支,三者的G+C含量分别为10.18%、10.18%和10.20%,四川什都和四川珙县的遗传距离为0.003,四川什都和广东肇庆的遗传距离为0.002,四川珙县和广东肇庆的遗传距离为0.002,三者G+C含量非常接近以及较小的遗传距离均支持三者聚为一支的观点,以上结果均表明三者具有较近的亲缘关系。分支II中的江西庐山和湖南桑植的变异尤为明显,两者自成单系。两者生境较为相似,形态上无明显分化。产自江西庐山的G+C含量为9.45%,产自湖南桑植的G+C含量为9.42%,两者G+C含量非常接近,且遗传距离仅为0.011,进一步证实了两者具有十分相近的亲缘关系。本研究中,基于联合叶绿体和ITS构建的系统发育树除四川什都、云南麻栗坡及云南石林的系统位置与基于ITS构建的系统树不同外,其它分支均与基于ITS构建的系统发育树一致。分支的不一致性可能是由于基因渐渗引起的。
参考文献
[1]国家药典委员会.中国药典2005[M].北京:化学工业出版社,2005.
[2]Chen X,Tamura M.Ophiopogon Ker-Gawl.[M].Beijing:Science Press,2000.
[3]Hebert PDN,Cywinska A,Ball SL,et al.Biological identifications through DNA barcodes[J].Proceedings of the Royal Society B-Biological Sciences,2003,270:313-321.
[4]Hebert PD,Ratnasingham S,de Waard JR.Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J].Proceedings of the Royal Society B-Biological Sciences,2003,270:96-99.
[5]Blaxter M.Molecular systematics:counting angels with DNA[J].Nature,2003,421:122-124.
[6]Gregory TR.DNA barcoding does not compete with taxonomy[J].Nature,2005,434:1067-1067.
[7]James N.Will DNA barcodes breathe life into classification[J].Science,2005,307:1307.
[8]Miller SE.DNA barcoding and the renaissance of taxonomy[J].Proceedings of the National Academy of Sciences,2007,104:4775-4776.
[9]Schindel DE,Miller SE.DNA barcoding a useful tool for taxonomists[J].Nature,2005,435:17-17.
[10]陈士林.中药 DNA 条形码分子鉴定[M].北京:人民卫生出版社,2012.
[11]陈士林,庞晓慧,罗焜,等.生物资源的DNA条形码技术[J].生命科学,2003,25:458-466.
[12]Barcodes PP.Wanted:a barcode for plants[J].Science,2007,318:190.
[13]Chase MW,Cowan RS,Hollingsworth PM,et al.Proposal for a standardised protocol to barcode all land plants[J].Taxon,2007,56:295-299.
[14]Chase MW,Salamin N,Wilkinson M,et al.Land plants and DNA barcodes:short-term and long-term goals[J].Philosophical Transactions of the Royal Society B-Biological Sciences,2005,360:1889-1895.
[15]Fazekas AJ,Burgess KS,Kesanakurti PR,et al.Multiple multilocus DNA barcodes from the plastid genome discriminate plant species equally well[J].PLoS One,2008,3:2802.
[16]Kress WJ,Wurdack KJ,Zimmer EA,et al.Use of DNA barcodes to identify flowering plants[J].Proceedings of the National Academy of Sciences of the United States of America,2005,102:8369-8374.
[17]Kim OT,Bang KH,In DS,et al.Molecular authentication of ginseng cultivars by comparison of internal transcribed spacer and 5.8S rDNA sequences[J].Plant Biotechnology Reports,2007,1:163-167.
[18]Xue HG,Zhou SD,He XJ,et al.Molecular authentication of the traditional Chinese medicinal plant Euphorbia pekinensis[J].Planta Medica,2007,73:91-93.
[19]沈洁,丁小余,张卫明,等.花椒及其混淆品的rDNA ITS 区序列分析与鉴别[J].药学学报,2005,40:80-86.
[20]Hollingsworth ML,Clark AA,Forrest LL,et al.Selecting barcoding loci for plants:evaluation of seven candidate loci with species‐level sampling in three divergent groups of land plants[J].Molecular Ecology Resources,2009,9:439-457.
[21]Hollingsworth PM.DNA barcoding plants in biodiversity hot spots:progress and outstanding questions[J].Heredity,2008,101:1-2.
[22]Lahaye R,vander Bank M,Bogarin D,et al.DNA barcoding the floras of biodiversity hotspots[J].Proceedings of the National Academy of Sciences of the United States of America,2008,105:2923-2928.
[23]White TJ,Bruns T,Lee SJWT,et al.Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics[M]// Innis MA,Gelfand DH,Sninsky JJ,eds.White In PCR protocols:a guide to methods and applications.London:Academic Press,1990.
[24]Meng Y,Wen J,Nie Z L,et al.Phylogeny and biogeographic diversification of Maianthemum(Ruscaceae:Polygonatae)[J].Molecular Phylogenetics and Evolution,2008,49:424-434.
[25]Hamilton MB.Four primer pairs for the amplification of chloroplast intergenic regions with intraspecific variation[J].Molecular Ecology,1999,8:521-523.
[26]Sang T,Crawford D,Stuessy T.Chloroplast DNA phylogeny,reticulate evolution,and biogeography of Paeonia(Paeoniaceae)[J].American Journal of Botany,1997,84:1120-1136.
[27]Olmstead RG,Bremer B,Scott K M,et al.A parsimony analysis of the Asteridae sensu lato based on rbcL sequences[J].Annals of the Missouri Botanical Garden,1993,80:700-722.
[28]Zurawski G,Perrot B,Bottomley W,et al.The structure of the gene for the large subunit of ribulose 1,5-bisphosphate carboxylase from spinach chloroplast DNA[J].Nucleic Acids Research,1981,9:3251-3270.
[29]Kilian N,Gemeinholzer B,Lack HW.Tribe Cichorieae Lam.& DC.[M]//Funk V A,Susanna A,Stuessy T,eds.Systematics,Evolution and Biogeography of the Compositae.Vienna:IAPT.
[30]Taberlet P,Gielly L,Pautou G,et al.Universal primers for amplification of three non-coding regions of chloroplast DNA[J].Plant Molecular Biology,1991,17:1105-1109.
[31]吴志刚,范传颍,包晓青,等.山药种质资源核糖体rDNA-ITS区序列分析[J].中草药,2014,8:1136-1142.
[32]Rodríguez-Trelles F,Tarrío R,Ayala FJ.Evidence for a high ancestral GC content in Drosophila[J].Molecular Biology and Evolution,2000,17:1710-1717.
[33]庞晓慧,宋经元,徐海滨,等.应用ITS2条形码鉴定中药材麻黄[J].中国中药杂志,2012,37:1118-1121.
[34]Baldwin BG,Sanderson MJ,Porter JM,et al.The ITS region of nuclear ribosomal DNA:a valuable source of evidence on angiosperm phylogeny[J].Annals of the Missouri Botanical Garden,1995,247-277.
[35]Cao H,Liu Y,Komatsu K.Identification of notoginseng(Panax notoginseng)and its adulterants using DNA sequencing[J].Journal of Chinese Medica Material,2001,24:398-402.
[36]Newmaster S,Fazekas A,Ragupathy S.DNA barcoding in land plants:evaluation of rbcL in a multigene tiered approach[J].Botany,2006,84:335-341.
(责编:张宏民)

