基于MRM的小鼠脑模板创建的研究进展
付振荣,林岚,张柏雯,宾光宇,高宏建,吴水才
北京工业大学 生命科学与生物工程学院,北京 100124
基于MRM的小鼠脑模板创建的研究进展
付振荣,林岚,张柏雯,宾光宇,高宏建,吴水才
北京工业大学 生命科学与生物工程学院,北京 100124
编者按:在最近20年中,具有高分辨率的磁共振显微成像(Magnetic Resonance Microscopy,MRM)技术被广泛应用于小动物脑神经影像学的研究之中。当前,对小动物神经影像的研究主要集中在2方面:① 通过转基因模型研究各种人类的神经系统疾病;② 建立各类小动物的脑模板和脑图谱分析平台,促进自动化分析。本期专栏首先介绍了小鼠脑模板创建的研究进展,随后通过空间协方差模型分析了高血压对大鼠大脑老化的影响,并近一步探讨了如何结合组织切片和MRM技术分析Aβ斑块在PS/APP双转基因小鼠大脑的产生与沉积。

栏目主编:林岚
北京工业大学生物医学工程系,副教授。从事运用神经影像学方法,研究与年龄相关的大脑认知退化方面的研究工作。2006年于美国亚利桑那州立大学获得生物工程博士学位。2006~2011年在美国亚利桑那州立大学和亚利桑那大学从事神经影像学方面的研究工作。运用现代神经图像处理与分析方法对健康人大脑的认知储备及阿兹海默症的发生、发展过程进行研究。2012年加入北京工业大学生物医学工程系。共发表科技论文60余篇,其中14篇文章被SCI/EI检索收录。现系【美】神经科学学会会员、【美】Mcknight大脑研究学会会员。多年来还作为课题负责人和主要参与者完成了一项北京市自然科学基金,两项亚利桑那州基金,多项NIA(美国国家老年研究院基金)项目和NIH(美国国家卫生研究院基金)项目。
高分辨率的磁共振显微成像(MRM)技术已被广泛应用于小鼠脑神经影像学的研究之中。脑模板和脑图谱是小鼠脑神经影像学研究中不可缺少的工具,但由于小鼠大脑具有体积小、微观变化细微等特点,为脑膜板和脑图谱的建立增加了很大的困难,当今国内外大量研究都致力于如何能更好的建立不同类型的小鼠脑模板。本文首先介绍了MRM的成像原理及图像的采集方式,探讨了MRM的优化措施。之后讨论了脑模板构建过程中的一些基本元素,如模板比较、配准算法等。最后介绍了小鼠脑模板在一些脑部疾病(如阿尔茨海默病、亨廷顿氏综合症等)动物模型上的基本应用。
脑图谱; 脑模板;磁共振显微成像;图像配准
0 引言
目前,由Ashburner等[1]提出的基于体素的形态学分析方法(Voxel-Based Morphometry,VBM),已经被广泛用于脑神经影像学的研究中[2-4]。VBM方法首先需要将研究的所有个体大脑的MRI结构像,空间标准化到一个“标准”空间中去,然后对标准化后的结构像进行分割,最后利用参数统计检验进行逐个体素间的统计分析。其中,空间标准化是VBM方法中最为关键的一步。由于每个被试大脑的大小、形状都不完全相同,研究人员需要一个脑模板作为参考空间来进行空间标准化,再将每幅被试的图像转换成大小、形状、空间位置等和脑模板一致的“标准”图像。目前,研究人员已经建立了多种人脑标准脑模板[5-7]。当笔者将目标图像配准到脑模板定义的“标准”空间后,通过脑图谱可以对大脑图像进行定位。大部分高精度脑图谱主要是通过人工定义解剖位置来绘制的,具有完整的脑区标记信息和空间坐标信息。通过将解剖信息进行整合可以对大脑进行三维重建,再将三维重建之后的脑图谱和脑模板进行配准就会得到标准化的数字脑图谱,见图1。近20年来,为了测量大脑的形态和功能随着个人发育、学习、疾病、年龄增长等进程发生的变化,基于MRI绘制的数字脑模板、脑图谱为神经脑科学的发展提供了重要的研究依据,已经成为脑科学研究领域的一大热点[8-10],也是脑神经科学研究必不可少的工具。
几乎每项医学领域中的重大成就都离不开动物实验,尤其是在脑科学领域。大量的科学成果都是建立在动物实验的基础上。在神经影像学的研究中,动物模型具有生命周期短、操作简单、避免对人类造成潜在风险等优势,尤其在缩短研究周期和全面认识疾病本质等方面具有不可替代的地位。磁共振显微成像(Magnetic Resonance Microscopy,MRM)[11]是在MRI的基础上发展而来的,具有较高的分辨率,帮助研究人员观察小动物脑部的细微变化的成像技术。此外,MRM与具有高分辨率的光学显微组织切面相比,具有便于重建3D可视化脑图像这一显著的特点。图像数据采集方式有离体(in vitro)和活体(in vivo)两种方式,离体数据的分辨率高于活体数据,而活体数据具有形变小等优势[11]。
随着大量小鼠疾病模型的涌现[12],利用MRM对不同类型的小鼠疾病模型进行研究已经成为了一种趋势。当我们在对小鼠的脑图像信息进行分析前,首先需要把位于不同空间位置的小鼠的脑图像转换到一个统一的“标准”空间。小鼠脑模板作为以上研究的关键环节,重要性不言而喻。目前,很多研究都致力于小鼠脑模板[13-14]和脑图谱[15]的创建。小鼠脑模板和脑图谱可以用来量化和描绘转基因小鼠的脑部解剖特征,并通过对不同转基因模型进行进一步研究来探索阿尔茨海默病(Alzheimer’s Disease,AD)等疾病的形态与功能变化[12]。本文将通过MRM图像采集、不同脑模板的介绍以及脑模板在动物疾病模型中的应用对基于MRM的小鼠脑模板展开论述(图1)。
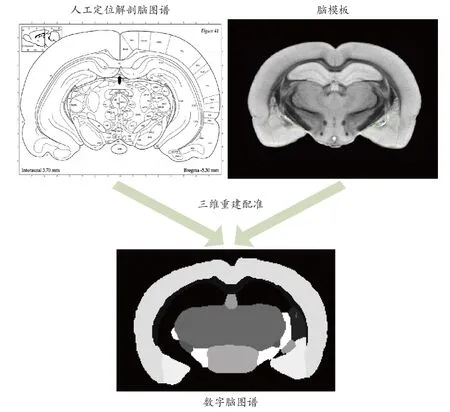
图1 数字脑图谱的创建过程
1 MRM图像采集
结构MRI是指利用核磁共振成像原理,通过外加梯度磁场检测原子核所发出的电磁波,绘制成被试样本内部的结构图像[16-18]。但是,MRI的分辨率无法满足小鼠脑图像的研究需求。随着MRI技术的发展,具有更高分辨率的MRM应运而生。MRM的图像分辨率一般为10~200 µm[19-20]。组织切片光学显微技术的分辨率为0.01~10 µm[21-22],人脑的磁共振成像的分辨率一般为500 µm~2 mm[23-24],MRM的分辨率介于这两者之间。由于其具有的3D特性,小鼠脑部MRM不仅能够观察到小鼠大脑的解剖结构,还能够避免光学组织切片导致的图像空间扭曲[25]。但是,MRM具有的一些技术性的挑战也随着该技术的发展逐渐凸显出来[26]。对于小鼠脑图像来说,单位体素至少要比人脑小3000倍以上。小的单位体素提供的回波信号会更弱,想要获得和人脑MRI图像一样清晰的解剖结构,就需要信号接收时要具有很高的灵敏度[27]。高的磁场强度可以增加信号的频率,从而达到增强信号的目的。理论上,在7 T场强下采集的图像要比在1.5 T场强下采集图像的信噪比(Signal to Noise Ratio,SNR)高出5到10倍。其具体倍数要根据被试对象的尺寸和图像噪声来确定[28]。目前,优化线圈的设计和延长扫描时间都是克服信号减弱的常用方法。在成像设备中,射频线圈能够激发驱动和接受信号,是MRM成像质量的关键因素之一[29]。一个好的射频线圈的衡量标准是:线圈体积尽可能小,能覆盖全部感兴趣的解剖区域,还能够改进信号的接收效果。随着被试对象体积的减小,线圈的灵敏度也随之增加。将线圈的尺寸从人脑大小降低到鼠脑大小,SNR可以提高20倍。进一步优化线圈的形状也可以获得灵敏度的提高。大部分对活体小鼠的研究采用鸟笼状线圈进行扫描。
在小鼠脑部MRM的研究中,样本的数据采集方式是非常重要的。目前,MRM数据的采集方式分为离体采集和活体采集两种。离体数据采集主要通过福尔马林灌注或浸没的方式对样本进行固定,也可直接扫描新鲜的大脑。Badea等[30]通过在头盖骨内进行扫描的方式来最大限度的保持鼠脑的固有形态,避免发生大的形变。离体成像数据相比于活体成像数据在实验中具有一定的优势。首先,离体样本的整体扫描时间最长可达到10~50 h[31],远大于活体样本的扫描时间(0.5~4 h)[32]。因此,相比于活体数据,离体数据能够具有更高的图像分辨率、SNR和对比度噪声比(Contrast to Noise Ratio,CNR)。对于活体数据来说,延长扫描时间具有一定的难度,因为长时间的麻醉可能会导致被试鼠的死亡。其次,在样本数据采集的过程中,离体样本是固定不动的,而活体样本由于呼吸、心跳等自然生理活动会不可避免的产生或多或少的物理位移。如果不能很好的控制这些活动带来的伪影,SNR会显著下降[33],从而影响图像的采集质量。最后,福尔马林灌注会使脑组织特性发生化学性变化,使图像对比度发生改变。毫无疑问,采集活体数据才是MRM的最大优势。与离体样本数据相比,活体数据采集也具有一定的优点。首先,进行活体样本数据采集可以最大限度地避免人工操作和脑脊液流失所产生的脑部形变。Ma等[34]将活体数据和自己之前的离体数据[35]进行对比,结果显示离体数据的脑室尺寸会发生明显的变化,有些脑室部分在离体数据中已无法确认。图2为相同年龄的C57BL/6J小鼠的MRM图像,红色框中离体数据有明显的脑室萎缩,但离体数据的SNR也明显优于活体数据。其次,活体样本可以进行多次采集,这对我们研究大脑发育以及其它脑疾病的发展过程[36-37]具有十分重要的意义。此外,活体样本还能进行脑功能图像的采集,这也是离体样本无法做到的。所以,研究人员必须根据实际情况,合理选择样本的数据采集方式,以及通过优化方法来尽可能的减小不同的数据采集方式所带来的影响。
2 小鼠脑模板
在做神经图像分析时,我们需要把不同个体的脑图像放到一起进行比较。图像配准,就是将不同时间、不同成像设备或不同条件下(光照、位置、角度等)获得的两幅或多幅图像通过空间变换到同一空间来进行比较。目前,小鼠脑图像配准一般分为2步。第一步,全局配准(仿射变换或刚体变换)来得到一副初步的配准图像。第二步,局部配准,通过一系列非线性形变进行局部的微小结构的配准,其空间变换具有更高的自由度。在配准两幅不同脑图像时,局部配准要比全局配准精确,但是需要更大的计算量。常用的小鼠脑图像非线性配准算法有很多,如B样条-FFD(Free-Form Deformation)[38]、Demons[39]、LDDMM(Large Deformation Diffeomorphic Metric Mapping)[40]、SyN(Symmetric Normalization)[41]等。
脑模板在空间标准化过程中,扮演了目标图像的角色,定义了“标准”空间,具有非常重要的作用。可以说,一个脑模板质量的好坏,对于研究人员能否正确分析脑图像起着非常关键的作用。小鼠MRM脑图谱是研究小鼠脑图像的基础,为综合不同脑成像的研究结果以及合并脑功能和解剖结构提供了一个强大的标准框架。但是,创建影像学的小鼠脑模板都是以解剖图谱为脑区分割的“金标准”来进行的,Paxions-Franklin脑图谱[42]就是其中之一,很多研究人员都将该图谱作为分割的标准[43]。Lin等[44]创建了一个活体C57BL/6J小鼠的MRM脑模板,并且将此模板和Paxions-Franklin脑图谱进行了对接。图3为小鼠脑模板的创建和脑图像统计分析流程图。

图2 相同年龄C57BL/6J小鼠MRM图像的不同采集方式
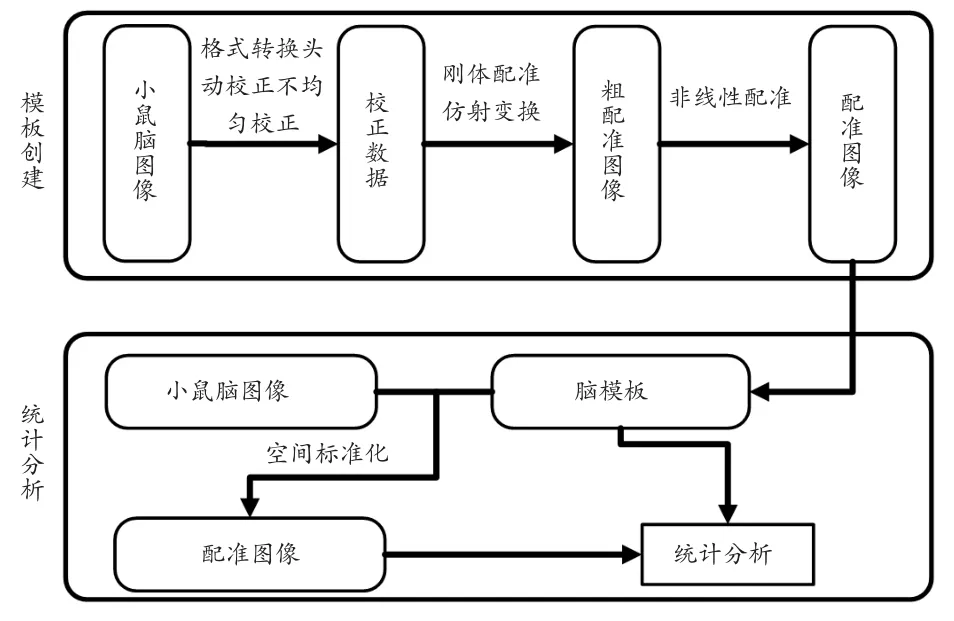
图3 小鼠脑模板创建和脑图像统计分析的流程图
Bedea等[30]对6只进行活性染色的C57BL/6J小鼠的离体大脑进行头盖骨内的MRM(9.4T)扫描,得到T1加权和T2加权2组图像。通过图像分割对提取出的脑结构的体积、面积、形状等变化进行分析,分别建立了T1和T2加权的大脑平均模板,并在此基础上创建了一个平均形状模板和一个概率性模板,这些模板都将鼠脑分割成了33个解剖结构。Ma等[35]开始了超高磁场(17.6T)的小鼠MRM的脑模板的创建。该研究利用10个离体数据的T2像建立了一系列C57BL/6J小鼠的MRM脑模板和脑图谱。通过基于图谱的分割法和人工勾描法,将整个小鼠大脑分为20个解剖结构,并且通过对以上数据的整合创建了三种类型的模板:样本的个体模板、平均最小形变模板和一个概率性模板。同样是在头盖骨内进行离体数据的MRM(7T)扫描,Dorr等[15]通过对鼠脑T2加权像进行手动分割,创建了一个拥有62个神经解剖结构的3D脑图谱。该研究扫描了40个成年C57BL/6J小鼠的离体样本并创建了一个平均T2 MRM模板。笔者认为,以上离体数据模板各有优劣,和其它2种模板对比,Dorr等[15]创建的模板具有较多的分区,而且样本基数较大,具有更高的精确度和更好的适用性。但是,Bedea等[30]能够将分割结构体积的平均形变率控制在7%,这对于离体数据的研究是十分重要的。
以上介绍的都是采用离体数据创建的模板,Ma等[34]还进行了活体样本脑模板的研究。通过对12个活体样本进行MRM(9.4T)扫描,将鼠脑划分为20个区域。该研究建立了包括12个样本的个体脑模板、一个平均脑模板、一个概率性模板和平均几何形变图。通过和之前离体数据创建的C57BL/6J小鼠脑模板[35]进行对比,可以发现形态学上的差异是非常明显的。
3 小鼠MRM脑模板和脑图谱在脑疾病模型中的应用
AD是一种起病隐匿的进行性发展的神经系统退行性疾病,是一种最常见的痴呆症。在过去几十年中,研究人员创造了种类繁多的AD转基因小鼠模型。Maherwaran等[45]通过把小鼠MRM和一个标准的数字图谱进行非线性配准,将鼠脑分为13个区域。研究发现转基因小鼠TASTPM和野生小鼠之间存在与年龄相关的结构增长差异。同时该研究团队还对比了基于图谱(Atlas-based)和基于形变的形态学测量(Deformation Based Morphometry,DBM)方法,发现两种方法都能很好的识别出横向和纵向对比中AD转基因小鼠APP(K670N,M671L)×PS1(M146V)和正常控制组小鼠C57Bl6的不同[31]。在AD转基因小鼠rTg4510和标准野生型小鼠的脑区分割中
Xie等[46]提出了基于张量的形态学测量(Tensor-based Morphometry,TBM)和基于图谱的分割方法,结果发现AD转基因小鼠rTg4510的脑室扩大,并且在海马区和皮层出现了灰质萎缩。Esteras等[47]将小鼠脑图谱的信息通过弹性变化得到校准图像,结果发现AD转基因小鼠AβPP/ PS1的脑室出现了明显的扩张,是受影响最大的区域。Lebenberg等[48]则通过联合VBM方法和3D MRM图谱来研究AD转基因小鼠APP/PS1脑部的代谢异常。该研究发现了转基因小鼠APP/PS1大脑嗅球等结构的糖代谢变化。
亨廷顿氏舞蹈症(Huntington's Disease,HD)是一种家族显性遗传性疾病。Aggarwal等[36]通过对HD转基因小鼠(R6/2和N171-82Q)的MRM图像进行纵向研究,应用建立的以MRM为基础的脑模板和基于混合效应模型的形变度量,可以得到两种HD转基因小鼠模型(R6/2和N/71-82Q)的脑萎缩的发展规律。该研究发现,和控制组相比,R6/2小鼠从第三周开始出现脑萎缩现象,主要表现为新皮层、梨状皮层、纹状体、丘脑、海马体的萎缩。N171-82Q小鼠出现脑萎缩现象的时间晚于R6/2小鼠。在第6周之后,N171-82Q小鼠的新皮层、纹状体、海马体、梨状皮层和杏仁核出现了明显的萎缩现象。Zhang等[40]最先将HD转基因小鼠R6/2的MRM影像通过基于图谱的分割之后进行纵向的形态学来研究脑萎缩的状况。活体数据研究发现,和控制组相比,R6/2小鼠的侧脑室扩张以及纹状体、皮层和海马体的萎缩是明显的。
4 结论
简而言之,由于MRM所具有的非入侵性和高分辨率的特性,目前已经成为研究各种相关疾病小鼠转基因模型过程中非常重要的工具之一。在本文中,笔者描述了小鼠脑MRM图像中的一些基本要素、小鼠MRM脑模板的创建过程以及其具体应用。它可以为标准空间中的脑图像分析提供大量支持。未来,随着各种相关疾病转基因小鼠模型技术的不断成熟化,数字脑模板必将成为分析疾病对结构区域影响的重要工具之一,并且能够作为一种重要的影响标志物标准,用来监控疾病的发展程度、治疗效果以及对疾病进行早期预测等。
[1] Ashburner J,Friston KJ.Voxel-Based Morphometry-TheMethods[J].Neuroimage,2000,11(6Pt 1):805-821.
[2] Hutton C,Draganski B,Ashburner J,et al.A comparison between voxel-based cortical thickness and voxel-based morphometry in normal aging[J].Neuroimage,2009,48(2):371-380.
[3] Good CD,Johnsrude IS,Ashburner J,et al.A Voxel-Based Morphometric Study of Ageing in 465 Normal Adult Human Brains[J].Neuroimage,2001,14(1Pt 1):21-36.
[4] Tang W,Huang X,Li B,et al.Structural brain abnormalities correlate with clinical features in patients with drug-naïveOCD:A DARTEL-enhanced voxel-based morphometry study[J].Behav Brain Res,2015,294:72-80.
[5] Ashburner J,Friston KJ.Unified segmentation[J].Neuroimage, 2005,26(3):839-851.
[6] Evans AC,Collins DL,Mills SR,et al.3D statistical neuroanatomical models from 305 MRI volumes[C].Proc IEEE-Nuclear Science Symposium and Medical Imaging Conference,1993,1813–1817.
[7] Mazziotta JA,Toga AW,Evans AC,et al.A probabilistic atlas and reference system for the human brain:International Consortium for Brain Mapping (ICBM)[J].Philos Trans R Soc Lond B Biol Sci,2001,356(1412):1293-1322.
[8] Evans AC,Janke AL,Collins DL,et al.Brain templates and atlases[J].Neuroimage,2012,62(2):911-922.
[9] Cabezas M,Oliver A,Lladó X,et al.A review of atlas-based segmentation for magnetic resonance brain images[J].ComputMethodsPrograms Biomed,2011,104(3):e158-177.
[10] Uchiyama HT,Seki A,Tanaka D,et al.A study of the standard brain in Japanese children:Morphological comparison with the MNI template[J].Brain Dev,2013,35(3):228-235.
[11] Benveniste H,Blackband S.MR microscopy and high resolution small animal MRI:Applications in neuroscience research[J].Prog Neurobiol,2002,67(5):393-420.
[12] Lin L,Fu Z,Xu X,et al.Mouse Brain Magnetic Resonance Microscopy:Applications in Alzheimer Disease[J].Microsc Res Tech,2015,78(5):416-424.
[13] Lin L,Chen K,Alexander GE,et al.Construction of mouse brain MRI templates using spm99[J].Modeling and Control in Biomedical Systems,2003,113-118.
[14] Lin L.Research on MRM template and atlas framework of mouse brain[D].Arizona:Arizona State University,2006:54-73.
[15] Dorr AE,Lerch JP,Spring S,et al.High resolution threedimensional brain atlas using an average magnetic resonance image of 40 adult C57Bl/6J mice[J].Neuroimage,2008,42(1): 60-69.
[16] Bushberg JT,Seibert JA,Leidholdt EM,et al.The Essential Physics of Medical Imaging[M].2nded.Baltimore:Williams and Wilkins,2001.
[17] Haacke EM,Brown RW,Thompson MR,et al.Magnetic Resonance Imaging Physical Principles and Sequence Design[M].New York:Wiley-Liss,1999.
[18] 俎栋林,高家红.核磁共振成像—物理原理和方法[M].北京:北京大学出版社,2014.
[19] Tyszka JM,Fraser SE,Jacobs RE.Magnetic resonance microscopy: recent advances and applications[J].Curr Opin Biotechnol,2005,16(1):93-99.
[20] Glover P,Mansfield SP.Limits to magnetic resonance microscopy[J].Rep Prog Phys,2002,65(10):1489-1511.
[21] Purger D,McNutt T,Achanta P,et al.A histology-based atlas of the C57BL/6J mouse brain deformably registered to in vivo MRI for localized radiation and surgical targeting[J].Phys Med Biol,2009,54(24):7315-7327.
[22] Graham E,Moss J,Burton N,et al.The atlas of mouse development Histology resource[J].Development,2015,142 (14):2545.
[23] Ceritoglu C,Tang X,Chow M,et al.Computational analysis of LDDMM for brain mapping[J].Front Neurosci,2013,7:151.
[24] Stucht D,Danishad KA,Schulze P,et al.Highest Resolution In Vivo Human Brain MRI Using Prospective Motion Correction[J].PLoS One,2015,10(7):e0133921.
[25] Johnson GA,Benveniste H,Black RD,et al.Histology by magnetic resonance microscopy[J].Magn Reson Q,1993,9(1):1-30.
[26] Henkelman RM.Systems Biology through Mouse Imaging Centers:Experience and New Directions[J].Annu Rev Biomed Eng,2010,12:143-166.
[27] Driehuys B,Nouls J,Badea A,et al.Small animal imaging with magnetic resonance microscopy[J].ILAR J,2008,49(1):35-53.
[28] Beuf O,Jaillon F,Saint-Jalmes H.Small-animal MRI:signalto-noise ratio comparison at 7 and 1.5 T with multiple-animal acquisition strategies[J].MAGMA,2006,19(4):202-208.
[29] Doty FD,Entzminger G,Kulkarni J,et al.Radio frequency coil technology for small-animal MRI[J].NMR Biomed,2007,20(3): 304-325.
[30] Badea A,Ali-Sharief AA,Johnson GA.Morphometric analysis of the C57BL/6J mouse brain[J].Neuroimage,2007,37(3):683-693.
[31] Maheswaran S,Barjat H,Bate ST,et al.Analysis of serial magnetic resonance images of mouse brains using image registration[J].Neuroimage,2009,44(3):692-700.
[32] Valdés-Hernández PA,Sumiyoshi A,Nonaka H,et al.An in vivo MRI template set for morphometry,tissue segmentation,and fMRI localization in rats[J].Front Neuroinform,2011,5:26.
[33] Hedlund LW,Cofer GP,Owen SJ,et al.MR-compatible ventilator for small animals: computer-controlled ventilation for proton and noble gas imaging[J].Magn Reson Imaging, 2000,18(6):753-759.
[34] Ma Y,Smith D,Hof PR,et al.In vivo 3D digital atlas database of the adult C57BL/6J mouse brain by magnetic resonance microscopy[J].Front Neuroanat,2008,2:1.
[35] Ma Y,Hof P R,Grant SC,et al.A three-dimensional digital atlas database of the adult C57BL/6J mouse brain by magnetic resonance microscopy[J].Neuroscience,2005,135(4):1203-1215.
[36] Aggarwal M,Duan W,Hou Z,et al.Spatiotemporal mapping of brain atrophy in mouse models of Huntington's disease using longitudinal in vivo magnetic resonance imaging[J].Neuroimage,2012,60(4):2086-2095.
[37] Munasinghe J,Zhang Z,Kong E,et al.Evaluation of neurodegener-ation in a mouse model of infantile batten disease by magnetic resonance imaging and magnetic resonance spectroscopy[J].Neurodegener Dis,2012,9(4):159-169.
[38] Maheswaran S,Barjat H,Bate ST,et al.Deformation based morphometry analysis of serial magnetic resonance images of mouse brains[J].Biomed Image Regist Proc,2006,58-65.
[39] Bai J,Trinh TL,Chuang KH,et al.Atlas-based automatic mouse brain image segmentation revisited: model complexity vs.image registration[J].Magn Reson Imaging,2012,30(6):789-798.
[40] Zhang J,Peng Q,Li Q,et al.Longitudinal Characterization of Brain Atrophy of a Huntington Disease Mouse Model by Automated Morphological Analyses of Magnetic Resonance Images[J].Neuroimage,2010,49(3):2340-2351.
[41] Chakravarty MM,Steadman P,Eede MC,et al.Performing label-fusion-based segmentation using multiple automatically generated templates[J].Hum Brain Mapp,2013,34(10):2635-2654.
[42] Paxinos G,Franklin KBJ.Paxinos and Franklin's the mouse brain in stereotaxic coordinates[M].Elsevier,2013.
[43] Ullmann JFP,Watson C,Janke AL,et al.A segmentation protocol and MRI atlas of the C57BL/6J mouse neocortex[J].Neuroimage,2013,78:196-203.
[44] Lin L,Chen K,Valla J,et al.MRI template and atlas toolbox for the C57BL/6J mouse brain[C].Neural Engineering,2005. Conference Proceedings.2ndInternational IEEE EMBS Conference on IEEE,2005,6-8.
[45] Maheswaran S,Barjat H,Rueckert D,et al.Longitudinal regional brain volume changes quantified in normal aging and Alzheimer’s APP x PS1 mice using MRI[J].Brain Res, 2009,1270:19-32.
[46] Xie Z,Yang D,Stephenson D,et al.Characterizing the regional structural difference of the brain between tau transgenic (rTg4510) and wild-type mice using MRI.Medical Image Computing and Computer-Assisted Intervention–MICCAI 2010[M].Springer Berlin Heidelberg,2010:308-315.
[47] Esteras N,Alquézar C,Bartolomé F,et al.Systematic evaluation of magnetic resonance imaging and spectroscopy techniques for imaging a transgenic model of Alzheimer's disease (AβPP/ PS1)[J].J Alzheimers Dis,2012,30(2):337-353.
[48] Lebenberg J,Hérard A S,Dubois A,et al.Automated indexation of metabolic changes in Alzheimer's mice using a voxelwise approach combined to an MRI-based 3D digital atlas[C].Engineering in Medicine and Biology Society (EMBC),2010 Annual International Conference of the IEEE. IEEE,2010,5636-5639.
Recent Advances in the Research of the Establishment of the MRM-based Mouse Brain Template
FU Zhen-rong, LIN Lan, ZHANG Bai-wen, BIN Guan-yu, GAO Hong-jian, WU Shui-cai
College of Life Science and Bio-engineering, Beijing University of Technology, Beijing 100124, China
The high resolution magnetic resource microcopy (MRM) has been widely used in mouse brain neuroimaging study. The mouth brain template and atlas are the tools essential to the mouse brain neuroimaging study. The mouth brain is characterized by its small volume and subtle microscopic changes, which creates difficulty for the establishment of the brain template and atlas. Nowadays many domestic and overseas’ researchers have focused on setting up different kinds of mouse brain templates. This paper first introduced the imaging principle and imaging collection methods of MRM and discussed the strategies to optimize MRM. Secondly, the paper discussed basic elements of the mouse brain template construction, such as comparison of templates, data acquisition, registration algorithms, and etc. Finally, the paper introduced the fundamental application of the mouse brain template in the research and treatment of diseases such as Alzheimer’s disease and Huntington’s disease.
R318.1
A
10.3969/j.issn.1674-1633.2016.02.006
1674-1633(2016)02-0025-06
2015-09-15
北京市自然科学基金(7143171)资助。
林岚,副教授。
通讯作者邮箱:lanlin@bjut.edu.cn
Abstract:: brain atlas; brain template; magnetic resonance microscopy; image registration

