81株铜绿假单胞菌16S rRNA基因序列测定及系统发育学分析
曾晓琮
周 露1,2
苏妙贞1,2
韩志杰1,2
陈丹霞1,2
(1. 广东省食品检验所,广东 广州 510435;2. 广东省酒类检测中心,广东 广州 510435)
81株铜绿假单胞菌16S rRNA基因序列测定及系统发育学分析
曾晓琮1,2
周 露1,2
苏妙贞1,2
韩志杰1,2
陈丹霞1,2
(1. 广东省食品检验所,广东 广州 510435;2. 广东省酒类检测中心,广东 广州 510435)
;采用16S rRNA基因序列分析法对2015年7~9月广东省食品检验所桶装水专项抽检中筛查所得的81株铜绿假单胞菌(Pseudomonasaeruginosa)进行复核鉴定。菌株经纯化培养后提取总DNA,采用细菌16S rRNA通用引物进行16S rRNA基因序列扩增,PCR扩增产物经2%琼脂糖凝胶电泳后,进行序列测定,序列经人工校对后用Clustal X进行比对分析,最后用MEGA5.1软件构建系统发育树。系统发育分析结果表明:81株铜绿假单胞菌与原鉴定结果一致。其中,编号24-3-QY、100-5-JM、106-3-JM菌株形成一个分支,28-1-WD单独为一支,其余77株野生菌和铜绿假单胞菌标准菌株ATCC27853聚为一群。该研究是2015年5月24日中国开始实施GB 19298—2014《食品安全国家标准 包装饮用水》以来,广东省首次对水源性铜绿假单胞菌进行的研究,为下一步菌种污染朔源等研究提供依据。
铜绿假单胞菌;16S rRNA基因;序列分析;系统发育树
铜绿假单胞菌(Pseudomonasaeruginosa)又称为绿脓杆菌,1872年由Schroeter命名,在分类学上属于细菌界变形菌门γ-变形菌纲假单胞菌目假单胞菌科假单胞菌属,为无芽孢端生单鞭毛革兰氏阴性小杆菌,典型嗜氧化能异养代谢。该菌在自然界分布广泛,存在于水、土壤、空气以及人和动物皮肤及肠道中[1]。该菌具有强大的环境适应性和生存能力,尤其适应潮湿的环境,甚至能适应太空微重力环境,形成生物被膜而呈现出地球上未曾见过的独特柱状冠层结构[2]。该菌是重要的食源性致病菌之一,能感染拟南芥[3]和窝苣[4-5]等较高等植物,能感染新杆状线虫[6-7]、果蝇[8]和大蜡螟[9]等无脊椎动物。更严重的是,铜绿假单胞菌也能感染免疫力下降或缺失的病人、囊性纤维化病人、烧伤和手术患者,会引发炎症和败血症,假如该菌在人体重要器官如肺部、尿道和肾脏定植,则可能会危及生命[10]。但对于健康者来说,铜绿假单胞菌是机会性致病菌。
近年来,饮水安全越来越受到人们的重视,饮用水中铜绿假单胞菌的污染情况也日渐受到关注。2012年李飞[11]在广东省矿泉水厂和山泉水厂采集了206份水样,其中43份水样检测出有铜绿假单胞菌,污染率为20.9%。徐励琴等[12]在2012~2013年间对惠州市80批次的桶装水检测中共检出8份水样含有铜绿假单胞菌,污染率为10.0%。2013年魏磊[13]对全国9个省36家厂家108份水样中筛查出36株铜绿假单胞菌。2015年,蔡双福[14]在广东市场上168份水样检测中检出2份水样含有铜绿假单胞菌,不合格率为1.2%。Silva M.E.Z等[15]从自来水、瓶装矿泉水和井水中分离得到的30株铜绿假单胞菌,并进行了药敏毒力因子等相关研究。Venier D.等[16]调查了希腊国内的瓶装矿泉水中微生物情况,从1995~2003年间共采集1 527份样品,检出90份铜绿假单胞菌,污染率为5.9%。
中国关于饮用水的标准有GB 5749—2006《生活饮用水卫生标准》、GB 8537—2008《饮用天然矿泉水》及2015年5月24日开始实施的GB 19298—2014 《食品安全国家标准 包装饮用水》。GB 19298—2014代替了GB 19298—2003《瓶(桶)装饮用水卫生标准》、GB 17324—2003《瓶(桶)装饮用纯净水卫生标准》、GB 17323—1998《瓶装饮用纯净水》,对包装饮用水中致病菌的检测设定了更严格的要求,即同一批次5份样品中不得检出铜绿假单胞菌。为保障广大人民群众饮水安全,针对GB 19298—2014《食品安全国家标准 包装饮用水》新标准的实施,广东省食品检验所在2015年7~9月开展了广东省桶装饮用水专项抽检。从中筛查出81株野生型铜绿假单胞菌,在此基础上,本研究期望通过16S rRNA基因序列分析法,在分子生物学水平上对81株野生型铜绿假单胞菌进行复核鉴定,旨在为建立具有广东特色的水源性铜绿假单胞菌菌种资源库和朔源数据库奠定基础。
1 材料与方法
1.1 原料与仪器
1.1.1 样品来源
1株铜绿假单胞标准菌株ATCC 27853:广东环凯生物技术有限公司;
81株野生型菌株(见表1):均是2015年7~9月广东省食品检验所桶装水专项抽检中依据GB/T 8538—2008《饮用天然矿泉水检验方法》、GB 19298—2014《食品安全国家标准 包装饮用水》筛查所得。
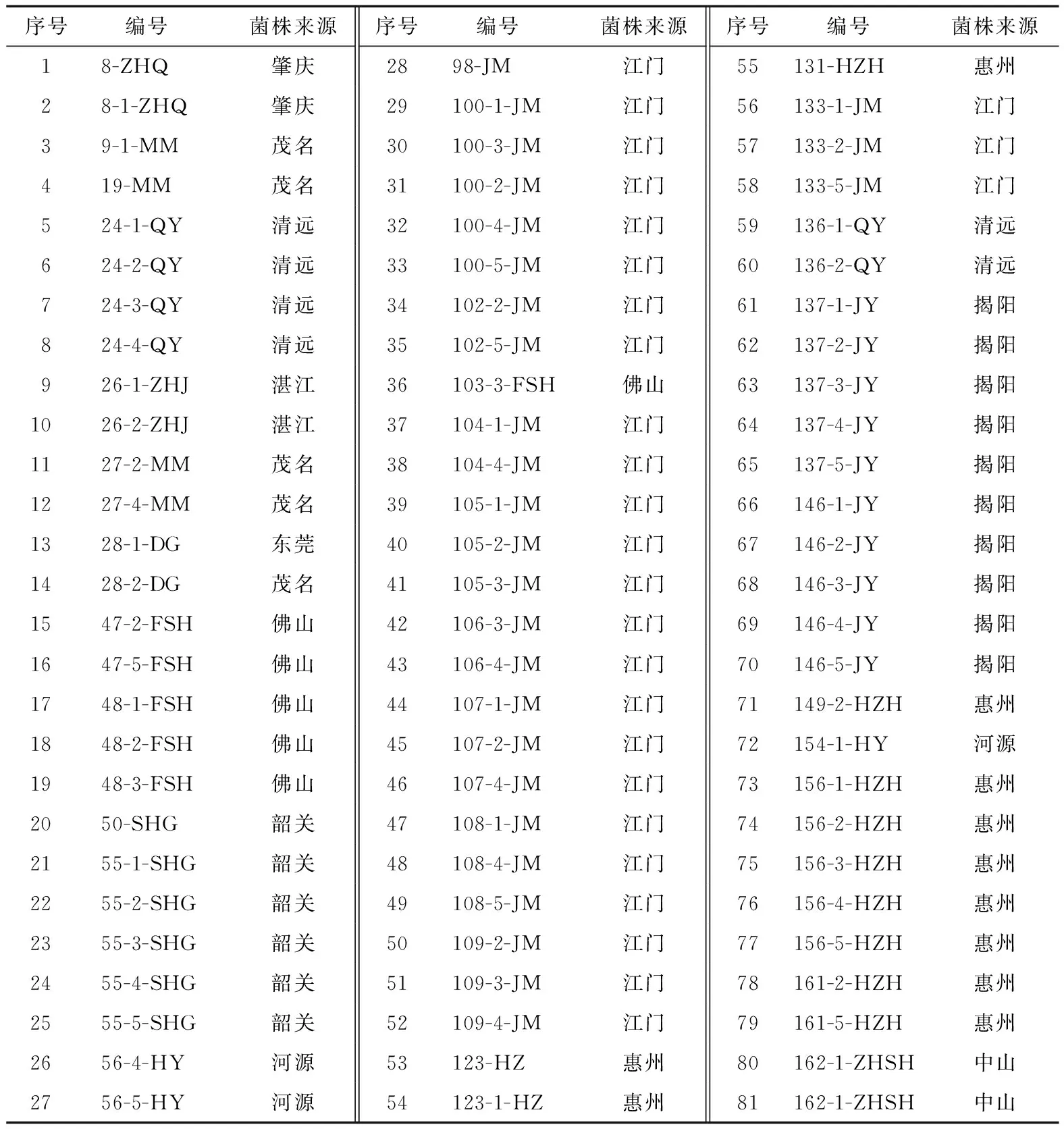
表1 试验所用野生型菌株Table 1 Wild type strains used in our research
1.1.2 培养基及主要试剂
营养肉汤:广东环凯生物技术有限公司;
DNA提取试剂盒和DNA marker 2000:日本Takara公司;
电泳级琼脂糖:西班牙Biowest公司;
超纯水、2*HsTMMix、50*TAE、Gold View:广州民塞生物科技有限公司;
PCR引物由北京华大基因研究中心合成。
1.1.3 主要仪器设备
Biometra DNA扩增循环仪:T-professional thermocycler型,德国Biometra公司;
UVP凝胶成像仪:ImageQuant 350 system型,美国GE Healthcare公司;
微量核酸测定仪:NANO型,日本岛津公司;
全自动灭菌锅:SX-700型,日本Tomy公司;
超净工作台:SW-CJ-2FD型,中国苏净安泰公司;
电热恒温培养箱:DHP-9052型,上海一恒科学仪器有限公司。
1.2 试验方法
1.2.1 菌体培养 将保存于30%甘油、-80 ℃的铜绿假单胞菌复壮培养后,在平板上划线分离,培养18 h后挑取单菌落接种到15 mL液体培养基中,36 ℃培养18 h。
1.2.2 DNA提取 按照Takara DNA Extraction Kit Ver.3.0提取81株野生型铜绿假单胞菌和1株铜绿假单胞菌标准菌株ATCC 27853总DNA,用微量核酸测定仪检测DNA的A260 nm/A280 nm值。提取的DNA保存于-20 ℃冰箱中。
1.2.3 PCR扩增及电泳检测 根据文献[17]的方法,选用27F/1492R扩增16S rRNA基因片段,正向引物序列27F:5′-AGAGTTTGATCCTGGCTCAG-3′,反向引物序列1492R:5′-GGTTACCTTGTTACGACTT-3′。扩增反应在Biometra TProfessional PCR仪上进行。反应体积为25 μL,其成分为:2 μL DNA模板(浓度25 μg/μL),2 μL引物(浓度10 μmol/L),12.5 μL 2*HsTMMix,超纯水补足25 μL。扩增程序为:首先95 ℃预变性5 min;然后94 ℃变性45 s,58 ℃退火45 s,72 ℃延伸1 min 30 s,共进行35个循环;最后 72 ℃延伸5 min。扩增结束后用2.0%琼脂糖凝胶电泳检测扩增产物(含0.005% Goldview staining)。
1.2.4 序列测定 16S rRNA基因序列测定由北京华大基因研究中心完成。
1.2.5 16S rRNA基因序列分析与构建 参考程池等[18]的方法,采用序列图谱软件Chromas,参照正、反向序列图谱,对序列人工校对。将81株野生型菌株的16S rRNA基因序列在NCBI上用BLAST进行同源性比较。从GenBank核酸序列数据库中下载假单胞属恶臭假单胞菌(Pseudomonasputida,NC 002947.3)的16S rRNA基因序列,与铜绿假单胞标准菌株ATCC 27853及81株野生型菌株序列一起,用Clustal X软件进行序列比对(alignment),然后利用MEGA 5.1生物学软件构建系统发育树,采用Neighbor-Joining法进行系统发育分析,并进行1 025次重复的Boot-straps统计学检验。
2 结果与讨论
2.1 菌种DNA PCR扩增后琼脂糖凝胶电泳检测与分析
铜绿假单胞菌标准菌株ATCC 27853和81株野生型菌株DNA提取后经琼脂糖凝胶电泳分析结果见图1,所提取片段均约为1 425 bp的特异条带,与预期大小相符。
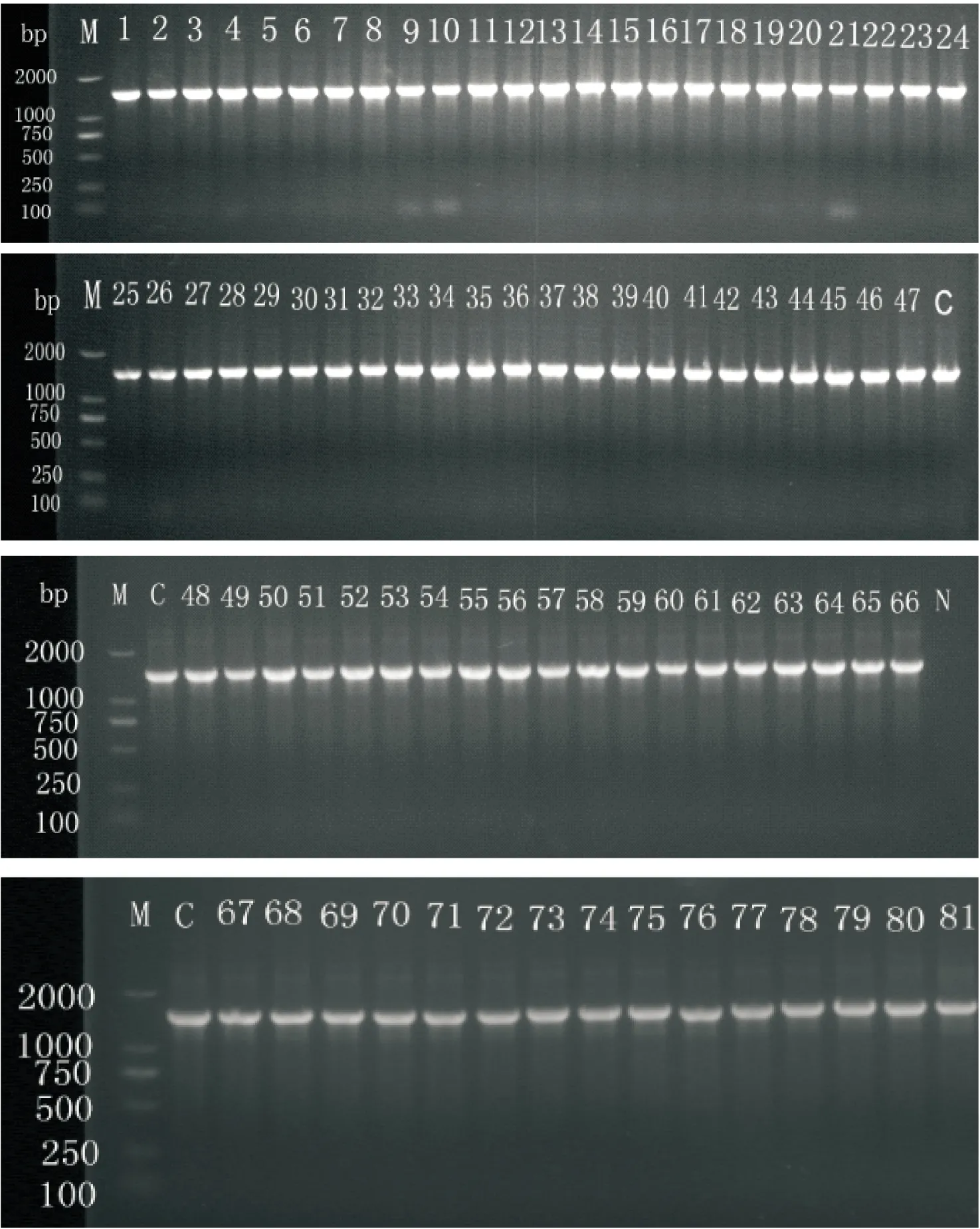
M. Marker C. 阳性对照 N. 阴性对照图1 81株铜绿假单胞菌16S rRNA 的PCR扩增产物Figure 1 16S rRNA PCR amplification products of 81Pseudomonas aeruginosa
2.2 16S rRNA基因序列分析及系统树构建
随着核酸测序技术的发展,通过16S rRNA 基因的序列测定,从分子遗传进化和分子水平上对细菌进行鉴定,已得到越来越广泛的应用,也越来越科学[19]。16S rRNA基因序列被称为“细菌化石”,因为在漫长的生物进化过程中,16S rRNA基因序列的变化非常缓慢,具有高度的保守性,可以用来标记生物的进化距离和亲缘关系,而利用16S rRNA序列中可变区序列的差异,则可以鉴定不同菌属、菌种的细菌。16S rRNA 基因序列是细菌分类鉴定的主要依据之一,研究[20]表明,16S rRNA序列同源性小于97%,属于不同的种,同源性小于93%~95%,则属于不同的属。程池等[18]采用16S rRNA基因序列分析法对中国工业微生物菌种保藏管理中心保藏的55株枯草芽孢杆菌进行复核鉴定,发现有3株菌种与原鉴定结果存在差异。张晓娟等[21]采用16S rRNA基因序列分析对西南菌种站收集保藏的23株芽孢杆菌进行了复核鉴定,发现有13株与原学名一致,有9株需要进一步核实。朱飞舟等[22]通过16S rRNA基因序列分析鉴定14种病原细菌,发现其中12种细菌可以鉴定到“种”,2种细菌可以鉴定到“属”。刘大佳等[23]采用16S rRNA基因分析技术鉴定正畸患者矫治前龈沟液的细菌种类,共鉴定出43株菌。吴至成等[24]对不同地理种群栖稻假单胞菌株进行了16S rRNA基因序列分析,发现大部分栖稻假单胞菌可根据16S rRNA基因序列鉴别不同地理种群株。
本研究将81株野生型菌株的16S rRNA基因序列在NCBI上用BLAST进行同源性比较,结果发现81株菌与铜绿假单胞杆菌模式菌(NC 002516.2)的同源性均在99%~100%,由此可以判定81株野生菌均为铜绿假单胞菌。图2是以假单胞菌属中恶臭假单胞菌(Pseudomonasputida,NC 002947.3)为外群的16S rRNA基因序列系统发育树,从系统发育树上看,81株分离株和1株标准菌株分成了3个大的分支,编号7(24-3-QY),33(100-5-JM),42(106-3-JM)形成一个分支,编号13(28-1-DG)单独为一支,其余77株野生菌和标准菌株ATCC 27853聚为一群。图2所示铜绿假单胞菌群内16S rRNA差异非常小,当这些序列在Cluster X构建系统发生树时,由于序列差异太小,系统发生树不会显示其清晰的分析结构。这也从反面说明了它们的高度相似性和一致性[25]。

标尺表示0.5%的序列差异;分支上的数据表示Bootstrap值图2 基于16S rRNA基因序列的81株铜绿假 单胞菌系统发育树Figure 2 The phylogenetic tree of 81 Pseudomonasaeruginosa based on 16S rRNA sequence
3 结论
本研究采用16S rRNA基因序列分析法对广东省食品检验所2015年7~9月桶装水专项抽检中筛查所得的81株铜绿假单胞菌进行复核鉴定。经序列比对发现,81株野生菌与GenBank中铜绿假单胞杆菌模式菌的同源性均在99%~100%,81株野生菌均为铜绿假单胞菌。从系统发育树发现,81株分离株和1株标准菌株分成了3个分支,清远24-3-QY,江门100-5-JM,江门106-3-JM形成一个分支,东莞28-1-DG菌株单独为一支,其余77株野生菌和标准菌株ATCC 27853聚为一群。本研究推测地域可能是造成菌株16S rRNA变异的主要原因[26]。而同属于一支的江门分离株100-5-JM、106-3-JM来自同一地区的不同厂家,两者是否存在联系,是否存在交叉污染,则有待进一步调查研究。
本研究是2015年5月24日中国开始实施GB 19298—2014《食品安全国家标准 包装饮用水》以来,广东对全省重要城市的水源性铜绿假单胞菌进行的研究,初步建立了具有广东特色的水源性铜绿假单胞菌菌种资源库,为饮用水中铜绿假单胞菌的风险识别及污染朔源提供了数据基础,对保障该省居民饮水安全具有重要意义。对本研究中所复核鉴定的铜绿假单胞菌进行药敏分析、毒力因子分析以及分子分型研究将会是下一步研究的重点。
[1] 闻玉梅. 现代医学微生物学[M]. 上海: 上海医科大学出版社, 1999: 383-389.
[2] KIM W, TENGRA FARAH K, YOUNG Z, et al. Spaceflight promotes Biofilm Formation byPseudomonasaeruginosa[J]. PLOS One, 2013, 8(4): 624-627.
[3] WALKER T S, BAIS H P, DEZIEL E, et al,Pseudomonasaeruginosa-plant root interactions. Pathogenicity, biofilm formation, and root exudation [J]. Plant Physiol, 2004, 134(1): 320-331.
[4] RAHME L G, STEVENS E J, WOLFORT S F, et al. Common virulence factors for bacterial pathogenicity in plants and animals[J]. Science, 1995, 268(5 219): 1 899-1 902.
[5] RAHME L G, TAN M W, LE L, et al. Use of model plant hosts to identifyPseudomonasaeruginosavirulence factors[J]. Proc Natl Acad Sci USA, 1997, 94(24): 13 245-13 250.
[6] MAHAJAN-MIKLOS S, TAN M W, RAHME L G, et al. Molecular mechanisms of bacterial virulence elucidated using aPseudomonasaeruginosa-Caenorhabditiselegant pathogenesis model [J]. Cell, 1999, 96(1): 47-56.
[7] MARTINEZ C, PONS E, PRATS G, et al. Salicylic acid regulates flowering time and links defense responses and reproductive development[J]. Plant J, 2004, 37(2): 209-217.
[8] DARGENIO D A, GALLAGHER L A, BERG C A, et al. Drosophila as a model host forPseudomonasaeruginosainfection [J]. J Bacterial, 2001, 183(4): 1 466-1 471.
[9] MIYATA S, CASEY M, FRANK D W, et al. Use of the Galleria mellondla caterpillar as a model host to study the role of the type III secretion system inPseudomonasaeruginosapathogenesis[J]. Infect Immune, 2003, 71(5): 2 404-2 413.
[10] BALTCH A L, SMITH R P.Pseudomonasaeruginosa: infections and treatment[J]. New York: M.Dekler,1994: 83-84.
[11] 李飞. 包装饮用泉水食源性致病菌分布规律和遗传多样性研究[D]. 广州: 华南理工大学, 2013: 35-36.
[12] 徐励琴, 薛计泉, 罗泽燕, 等. 桶装饮用水铜绿假单胞菌检测及耐药性分析[J]. 海峡预防医学杂志, 2015, 21(3): 56-57.
[13] 魏磊, 吴清平, 张菊梅, 等. 矿泉水和山泉水中铜绿假单胞菌污染调查及分离菌株毒力基因与耐药性分析[J]. 微生物通报, 2015, 42(1): 125-132.
[14] 蔡双福, 张琴, 黄耀雄. 食品中铜绿假单胞菌的监测分析[J]. 中国卫生检验杂志, 2015, 26(3): 875-905.
[15]SILVAME,FILHOIC,ENDOEH,etal.Characterisationofpotentialvirulencemarkersinpseudomonas aeruginosaisolatedfromdrinkingwater[J].AntonieVanLeeuwenhoek, 2008, 93(4): 323-334.
[16]VENIERID,VANTARAKISA,KOMNINOUG,etal.Microbiologicalevaluationofbottlednon-carbonated(“still”)waterfromdomesticbrandsinGreece[J].IntJFoodMicrobiol, 2006, 107(1): 68-72.
[17]LAVENIRR,JOCKTANED,LAURENTF,etal.ImprovedreliabilityofPseudomonas aeruginosaPCRdetectionbytheuseofthespecies-specificecfXgenetarget[J].JournalofMicrobiologicalMethods, 2007, 70: 20-29.
[18] 程池, 刘光全, 李金霞, 等. 55株芽孢杆菌16SrRNA基因序列测定与系统发育学分析[J]. 食品与发酵工业, 2006, 32(10): 20-24.[19]TENGLJ,HSUEHPR,HUANGYH,etal.IdentificationofBactericides thetaiotaomicrononthebasisofanunexpectedspecificapplicantofUniversal16SRibosomalDNAPCR[J].JournalofClinicalMicrobiology, 2004, 42(4): 1 727-1 730.
[20]STACKEBRANDTEB,GOEBELBM.Taxonomicnote:aplaceforDNA-RNAreassociationand16SrRNAsequenceanalysisinthepresentspeciesdefinitioninbacteriology[J].IntJSystBacteriol, 1994, 44: 842-849.
[21] 张晓娟, 宋萍, 王柱, 等. 23株芽孢杆16SrRNA基因序列扩增与系统发育分析[J]. 食品与发酵科技, 2012, 48(2): 9-12.
[22] 朱飞舟, 陈利玉, 陈汉春. 16SrRNA基因序列分析法鉴定病原细菌[J]. 中南大学学报: 医学版, 2013, 38(10): 1 035-1 041.
[23] 刘大佳, 杨明, 陈利玉, 等. 采用16SrRNA基因分析技术鉴定正畸患者矫治前龈沟液的细菌种类[J]. 中国现代医学杂志, 2016, 26(2): 43-46.
[24] 吴至成, 伍丽娴, 李丽花, 等. 不同地理种群栖稻假单胞菌株的16SrRNA基因序列分析[J]. 中国热带医学, 2012,12(12): 1 453-1 456.
[25] 邹寰, 喻子牛, 孙明. 蜡状芽胞杆菌群16SrDNA分析[J]. 华中农业大学学报, 2008, 27(4): 478-482.
[26] 赵耘, 杜昕波, 李伟杰, 等. 袋鼠源铜绿假单胞菌分离鉴定[J]. 中国预防兽医学报, 2010, 32(2): 139-141.
Identification of 81 Pseudomonas aeruginosa by phylogenetic analysis of 16S rRNA gene sequence
ZENG Xiao-cong1,2
ZHOULu1,2
SUMiao-zhen1,2
HANZhi-jie1,2
CHENDan-xia1,2
(1.GuangdongProvincialInstituteofFoodInspection,Guangzhou,Guangdong510435,China;2.GuangdongProvincialLiquorTestingCenter,Guangzhou,Guangdong510435,China)
81Pseudomonasaeruginosafrom Guangdong Provincial Institute of Food Inspection were identified by 16S rRNA sequence analysis. The DNA was isolated and the sequences of 16S rRNA gene were amplified by PCR with the bacterium universal primers, and then the PCR products were sequenced after 2% agarose gel electrophoresis. Moreover, the corrected sequences were aligned with Clustal X and the phylogenetic tree was constructed by MEGA5.1. Consequently, the identified results of the 81 strains confirmed their original identification before. On the phylogenetic tree, No. 24-3-QY strain formed a separate branch with No. 100-5-JM strain and No. 106-3-JM strain. No.2 8-1-DW formed one branch and the other 77 strains formed a separate branch withP.aeruginosaATCC 27853. This was the first research about waterborneP.aeruginosain Guangdong Province when China began to implement GB 19298—2014 “National food safety standards of packaged drinking water” since May 24, 2015. The waterborneP.aeruginosaculture collection in Guangdong was preliminarily established basing on these strains. These data will provide a powerful tool for effectively tracing source ofP.aeruginosaand controlling the water contamination in future.Keywords:Pseudomonasaeruginosa; 16S rRNA gene; gene sequence analysis; phylogenetic tree
曾晓琮,男,硕士。
周露(1982— ),女,广东省食品检验所高级工程师,博士。E-mail: zhoulu1982@sohu.com
2016—05—12
10.13652/j.issn.1003-5788.2016.11.005

