藏药材榜嘎及其混伪品的DNA条形码鉴定研究
郭豪杰 丁 钰 陈维武 刘海涛 边巴次仁 李西文
(1 中国中医科学院中药研究所,北京,100700; 2 中国医学科学院药用植物研究所,北京,100193;3 西藏奇正藏药股份有限公司,林芝,860000)
藏药材榜嘎及其混伪品的DNA条形码鉴定研究
郭豪杰1,2丁 钰1陈维武3刘海涛2边巴次仁3李西文1
(1 中国中医科学院中药研究所,北京,100700; 2 中国医学科学院药用植物研究所,北京,100193;3 西藏奇正藏药股份有限公司,林芝,860000)
目的:利用DNA条形码技术对榜嘎及其混伪品进行鉴别,为藏药榜嘎的鉴定提供准确可靠的依据。方法:通过分析榜嘎及其混伪品ITS和ITS2序列的遗传距离、种内种间变异并构建NJ系统发育树,以评价ITS2和ITS序列的鉴定效率。结果:本研究中ITS2和ITS序列扩增效率相同,在blast比对和遗传距离距离分析上,ITS序列表现出更强的鉴定能力;通过K2P距离构建NJ树,ITS2和ITS序列在榜嘎与混伪品间的种间变异无统计学意义,但ITS序列对易混物种的鉴定效率较高。结论:ITS序列可作为鉴定榜嘎及其混伪品的有效条形码。
藏药榜嘎乌头属;ITS
藏医药是中国传统医药的重要组成部分,具有悠久的历史、独特的医疗和研究价值及市场开发潜力[1]。榜嘎是藏医习用藏药材,为毛茛科乌头属植物船盔乌头(Aconitumnaviculare)或甘青乌头(A.tanguticum)的干燥全草,味苦,性凉,具有清热解毒与燥湿之功效,主要用于治疗热病等[2]。船盔乌头在我国主要分布于西藏南部3200~5000 m的山坡草地或灌木丛中,甘青乌头分布于西藏东部、云南西北部、四川西部等地3200~4800 m的山地草坡或沼泽草地[3]。《晶珠本草》将乌头属分为“白、黄、红、黑”四种,其中榜嘎属于白乌头。
乌头属药材形态相似,榜嘎有较多混用和代用品,西藏地区用叉苞乌头,青海用祁连乌头,川西用刷经寺乌头作为榜嘎使用。同时,由于地域上的差异,同名异物或同物异名的现象也是造成榜嘎混用乱用原因之一[1]。榜嘎在不同著作中曾出现多个藏语名,《蒙药正典·美丽日饰》中记载用百合科麦冬(Ophiopogonjaponicus)作为榜嘎的来源植物[4]。传统鉴别方法受地域差异、活性成分和亲缘关系等因素影响,存在鉴定局限性。乌头属植物大多具有毒性,为保障临床用药安全,寻求一种榜嘎药材快速准确的鉴定方法则显得尤为迫切。
DNA条形码技术具有通用性强、鉴定结果可靠、重复性良好等优点,被广泛用于中药材鉴定中[5]。ITS2是中国学者通过大量样本的系统研究,提出的可以作为鉴定药用植物和近缘种的通用条形码[6]。ITS/ITS2是中国植物DNA条形码研究组提出可作为种子植物核心条形码的序列[7]。目前榜嘎的鉴别主要是传统的性状和显微鉴别,我们在本文通过多种数据分析方法分析ITS和ITS2对榜嘎及其混为品的鉴别能力,为建立合适的榜嘎DNA条形码技术奠定基础。
1 材料与方法
1.1 材料与仪器 实验物种包括工布乌头(Aconitumkongboense)、铁棒锤(A.pendulum)和甘青乌头(A.tanguticum)、麦冬(Ophiopogonjaponicus)等。植物基因组DNA提取试剂盒(Tiangen Biotech Co.,中国),2×Tag PCR Master Mix(北京艾德莱生物科技有限公司,中国),引物由生工生物工程(上海)股份有限公司合成。scientz-48高通量组织研磨仪(宁波新芝生物科技股份有限公司,中国),2720PCR仪(Applied Biosystems®,USA),1-14K高速离心机(SIGMA,Germany)。
1.2 方法
1.2.1 DNA提取 取药材样品约50 mg,用高通量组织研磨仪研磨2 min(50Hz)后,利用植物基因组DNA提取试剂盒提取总DNA。
1.2.2 PCR扩增及测序 反应体系为2×Tag PCR Master Mix 12.5 μL,正反向引物(2.5 μM)各1.0 μL、模板DNA2.0 μL,加ddH2O补至25 μL。各条形码的引物序列和PCR反应程序见表1。1%琼脂糖电泳检测PCR扩增情况,由测序公司进行双向测序。

表1 引物序列和PCR反应程序

表2 Genbank数据
1.2.3 数据处理 将测序峰图用Codon Code Aligner软件进行拼接,将每个片段两端引物及低质量序列切除,得到所需片段并对ITS2进行注释剪切。同时从GenBank数据库中搜集并下载榜嘎及其混伪品ITS序列(表2),注释剪切后得到ITS2,结合实验样品序列,运用MEGA 6.0建立NJ树,计算遗传距离,分析榜嘎及其混伪品亲缘关系;采用Perl语言统计不同序列种内种间遗传差异;运用最近距离法(K2P nearest distance)和相似性搜索法(BLAST1)考察序列的鉴定效率[8]。
2 结果与分析
2.1 序列信息和鉴定效率 各序列数量、长度范围、GC含量、遗传距离、鉴定效率等信息见表3。ITS2由ITS注释而来,所以ITS2与ITS数量相等,都为148条。ITS2的长度范围为194~227bp,ITS的长度在605~777bp之间。平均GC含量,ITS2为62.15%,ITS为59.26%。ITS2和ITS的最小遗传距离相同,都为0.0000;平均遗传距离ITS较小,为0.1702,ITS2为0.2815;最大遗传距离ITS2较大,为0.8423,ITS为0.5351。从鉴定效率看,ITS表现较好,BLAST1和Nearest distance鉴定效率都为100%,ITS2的BLAST1和Nearest distance鉴定效率都为98.04%。综上所述,虽然ITS2最大遗传距离比ITS大,但是ITS最小遗传距离要小于ITS2,并且ITS的鉴定效率高于ITS2,所以从序列信息和鉴定效率方面考虑,ITS更适合作为榜嘎及其混伪品的DNA条形码。
2.2 不同序列种内种间差异 利用perl语言统计ITS2和ITS序列的6个值:种内变异(Theta)、种内最大变异(Coalescent depth)、平均种内变异(All intra-specific distance)、种间变异(Theta prime)、种间最小变异(Minimum inter-specific distance)、平均种间变异(All inter-specific distance)(表4)。ITS2和ITS序列的种间最小变异均大于种内最大变异,ITS2的种内变异、平均种内变异、种间变异和平均种间变异与ITS相似。综上所述,在种内种间差异方面,ITS2和ITS序列对榜嘎及其混伪品鉴定方面都有很好表现,无明显差别。

表3 序列信息和鉴定成功率

表4 种内种间差异分析
2.3 NJ树鉴别分析 基于K2P遗传距离建立NJ树,可以用来分析不同物种间的亲缘关系,以达到鉴别物种的目的。如图1所示,ITS2和ITS序列都可以将榜嘎及其混伪品分开,但ITS2序列对乌头属其他物种的鉴定效率相对较差。ITS序列表现出良好的鉴别能力,能够将不同的物种分到不同的分支上,更适合作为鉴定榜嘎及其混伪品的条形码序列。
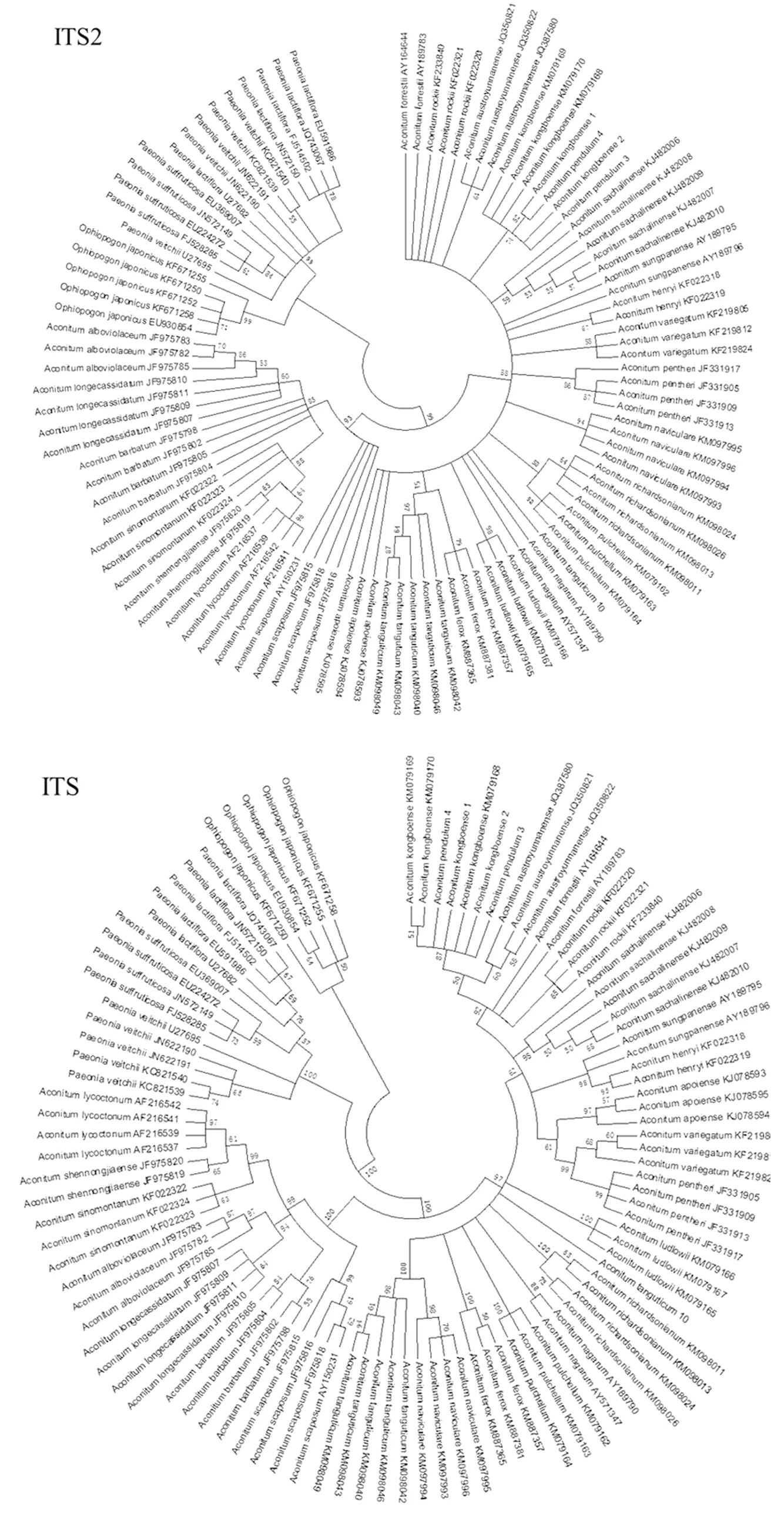
图1 NJ系统发育树
3 讨论
乌头属植物作为有毒植物及药用植物一直倍受广泛的关注,全属约有350种植物,我国约有167种,其中53种可作药用[2-3,9]。乌头属植物种间系统发育复杂,该属的分子生物学研究报道较少,多集中于生物多样性的研究,如ISSR分析、RAPD分析[10-12],鲜有生药学的分子鉴定研究。He等利用条形码技术对乌头属的19个物种134份样品进行鉴定研究,但其研究仅用到psbA-trnH序列,没有对ITS/ITS2序列进行分析[13]。榜嘎,藏名榜阿嘎保,全草均可入药,有小毒,历代藏医药书均有记载,具有清热解毒之功效,多用于各种热病的治疗,属于多基原藏药材,同时同属混伪品较多,多有毒性,传统方法鉴定困难,给临床用药带来诸多隐患。
传统藏药植物药缺乏系统分类基础研究,多采用手摸、眼看、奔闻、嘴尝、水试、火试等表观鉴定方法。藏药的使用也多依赖经验和古书记载,地区差异大,如《晶珠本草》中榜嘎为乌头属植物,《蒙药正典·美丽日饰》记载以百合科麦冬作为榜嘎的基原植物[3],因此建立一种科学快速的藏药基原物种鉴定方法迫在眉睫。DNA条形码在物种鉴定上具有快速、有效、操作简便、通用性强以及易于形成标准化等特点,已有广泛的研究和应用基础。本研究针对榜嘎及乌头属常见混伪品的ITS2和ITS序列进行比较分析,结果显示,ITS2和ITS扩增效率无统计学意义。Blast1和Nearest distance 2种分析方法上,ITS序列鉴定效率优于ITS2;通过K2P遗传距离构建NJ树,ITS序列表现出更好的鉴定能力。综合分析,ITS序列可以作为榜嘎及其混伪品鉴定的候选条形码序列。本研究为藏药材榜嘎的鉴定提供了新的分子生物学方法,可保障其临床用药安全。
[1]李敏,雷志强,钟国跃.藏医学药用毛茛科植物药材品种与标准的现状分析[J].中药新药与临床药理,2015,26(1):133-137.
[2]国家中医药管理局.中华本草[M].北京:科学出版社,1996:1709-1753.
[3]中国科学院中国植物志编辑委员会.中国植物志[M].北京:科学出版社,1996:113-326.
[4]刘治民.藏药榜嘎、榜那的资源调查和药用合理性评价[D].北京:北京中医药大学,2013.
[5]陈士林,姚辉,韩建萍,等.中药材DNA条形码分子鉴定指导原则[J].中国中药杂志,2013,38(2):141-148.
[6]Chen Shilin,Yao Hui,Han Jianping,et al.Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J].PLOS ONE,2010,5(1):1-8.
[7]Li Dezhu,Gao Lianming,Li Hongtao,et al.Comparative analysis of a large dataset indicates that internal transcribed spacer(ITS)should be incorporated into the core barcode for seed plants[J].Proceedings of the National Academy of Sciences,2011,108(49):19641-19646.
[8]郑司浩,李亚康,任伟光,等.基于DNA条形码的百合属分子鉴定[J].药学学报,2014,49(12):1730-1738.
[9]朱敏,肖培根.常用藏药榜嘎的研究[J].中药材,1989,12(10):19-21.
[10]孙一丁,李绍平,黄晓东,等.乌头干药材基因组提取及RAPD分析[J].西南农业学报,2008,21(1):138-141.
[11]田孟良,田迎秋,刘帆,等.乌头种质资源遗传多样性的RAPD分析[J].四川农业大学学报,2007,25(1):63-67.
[12]周倩,高福春,马红梅,等.新疆特产7种乌头类药材的ISSR特征分析[J].新疆医科大学学报,2014,37(12):1614-1618.
[13]He Jun,Wong Kalok,Shaw Pangchui,et al.Identification of the medicinal plants in Aconitum L.by DNA barcoding technique [J].Planta Med,2010,76(14):1622-1628.
(2016-04-12收稿 责任编辑:洪志强)
Identification of Herba Aconiti Bonga and Its Adulterants Using DNA Barcode
Guo Haojie1,2,Ding Yu1,Chen Weiwu3,Liu Haitao2,Bianba Ciren3,Li Xiwen1
(1InstituteofChineseMateriaMedica,ChinaAcademyofChineseMedicalSciences,Beijing100700,China;2InstituteofMedicinalPlantDevelopment,ChineseAcademyofMedicalSciencesandPekingUnionMedicalCollege,Beijing100193,China; 3TibetCheezhengTibetanMedicineCo.,ltd.,Linzhi860000,China)
Objective:To select an effective barcode for identifying Herba Aconiti Bonga and its adulterants using DNA barcoding technology.Methods:This study compared ITS and ITS2 sequences and analyzed genetic distances,the variations of inter-and intra-species.Neighbor-joining tree was also constructed for evaluate their identification efficiencies based on the sequences from experimentaland GenBank database.Results:The results showed that ITS and ITS2 sequences had the same amplication efficiency.ITS sequence had a better performance than ITS2 on species identification efficiency using blast and nearest distance methods.The two barcodes showed no significant difference on the identification uing NJ tree to distinguish Herba Aconiti Bonga and its adultrants.However results demonstrated that ITS can also identify all species among adultrants of Herba Aconiti Bonga.Conclusion:ITS sequence can be used as the effective barcode to identifyHerba Aconiti Bonga and its adulterants.
Tibetan medicine;Herba Aconiti Bonga;Aconitum;ITS
重大新药创制国家科技重大专项(编号:2014ZX09304307001-014;2014ZX09301308-007);国家科技支撑计划(编号:2015BAI05B02);国家科技计划港澳台合作专项(编号:2015DFM30030)
李西文,副研究员,研究方向:中药栽培与鉴定,Tel:(010)84084107,E-mail:xwli@icmm.ac.cn
R282.5
A
10.3969/j.issn.1673-7202.2016.01.003

