麻疯树油质蛋白JcOle14.3基因克隆及序列分析
宋 健,熊 宏,余进德,刘小烛,丁 勇
(西南林业大学 生命科学学院,云南 昆明 650224)
麻疯树油质蛋白JcOle14.3基因克隆及序列分析
宋 健,熊 宏,余进德,刘小烛,丁 勇
(西南林业大学 生命科学学院,云南 昆明 650224)
作为最丰富的油体结合蛋白,油质蛋白在油体的发生到分解消失过程中起着重要的生物学功能。本实验应用生物信息学和分子生物学技术分离克隆了麻疯树Jatrophacurcas全长593 bp的油质蛋白JcOle14.3基因序列,无内含子结构。JcOle14.3基因转录的mRNA包含了完整的开放阅读框,推测编码由137个氨基酸残基组成、相对分子质量为14.3 kD的稳定、两性的油质蛋白JcOle14.3。JcOle14.3理论等电点为9.65,正电荷残基(Arg+Lys)总数为10个,负电荷残基(Asp+Glu)总数为6个;不稳定系数为25.90,属于稳定性蛋白;其二级结构的主要构件为α-螺旋、随机卷曲和延伸链。根据高级结构预测结果推测JcOle14.3含有N-末端两亲性结构域、中间疏水性结构域以及C-末端两亲性结构域。C-末端两亲性结构域包含1个α-螺旋结构域,中间疏水性结构域包含由3个α-螺旋构成的跨膜结构域,并具有oleosin蛋白特征性的高度保守的脯氨酸结模体,推测在oleosin蛋白与单层磷脂层和油体锚定结合上起着重要的作用。该研究为后期JcOle14.3的异源表达和功能研究奠定了基础。
麻疯树;油体;油质蛋白;基因克隆;序列分析
植物油(Plant Oil)是现代生物质能源的理想来源之一。植物油广泛存在于开花植物的种子、花粉、根、茎和花中,但主要存在植物种子中[1-2]。植物油作为营养贮存物质积累于植物种子中,主要以甘油三酰(Triglycerides,TAGs)的形式存在。植物种子中贮存的营养物质主要有脂类、蛋白质和碳水化合物,而脂类是植物种子贮存能量最有效形式。植物体将脂类贮存于种子的亚细胞器颗粒——油体(Oil Bodies,OBs)中并为随后的生命活动以及活跃的代谢过程提供碳水化合物和能量[3-4]。油体颗粒内部主要为TAGs液态基质,外部包被磷脂单分子层(Phospholipids,PL)及嵌入其内的油体结合蛋白(Proteins associated with oil bodies)[5]。油体结合蛋白包括油质蛋白(Oleosin)、油体固醇蛋白-A(Steroleosin-A)和油体固醇蛋白-B(Steroleosin-B)、油体钙蛋白(Caleosin)[6-9]。研究表明油体结合蛋白对于植物油体的形成与稳定起着重要作用[10-12]。
麻疯树Jatrophacurcas L.又名小桐子、黑皂树、膏桐等,是大戟科Euphorbiaceae麻疯树属Jatropha多年落叶灌木植物[13]。麻疯树种子含有大量的油脂,可作为理想的现代生物质能源原料,尤其作为柴油机燃料的替代品开始凸显其重要性[13-15]。因此,为深入研究开发利用麻疯树种子的经济价值,油体结合蛋白及其基因已成为当务之急。植物种子中油体结合蛋白主要为oleosin,占90%左右。Oleosin作为最丰富的油体相关蛋白,推测油体从发生到分解及消失过程中发挥着重要的生物学功能[1,16-17]。Oleosin等油体结合蛋白在植物芝麻[18-19]、油菜[20-23]、水稻[24]、拟南芥[25]和油桐[26]等得到了一定的研究,并且oleosin蛋白所对应的基因均已被克隆并测序。作者通过NCBI网站对已经注册登录的oleosin基因信息进行搜索,结果显示麻疯树oleosin基因mRNA全长序列已有登录(GenBank序列号:EU234464.2),但目前尚无麻疯树oleosin基因DNA序列克隆和序列分析的相关报道。本实验根据NCBI网站登录的麻疯树oleosin基因的已知信息,利用生物信息学和分子生物学等相关技术设计特异性引物,克隆了与序列EU234464.2不一致的mRNA序列,命名为麻疯树油质蛋白基因JcOle14.3,同时克隆了JcOle14.3基因在染色体上的DNA序列,登录GenBank的序列号为JX073623.1,并对JcOle14.3基因和JcOle14.3蛋白特性进行了分析。
1 试验材料与方法
1.1 试验材料与试剂
试验材料为麻疯树幼叶和开花后发育6~7周的未成熟种子,采于中国科学院西双版纳植物园的3年生麻疯树植株,–80℃冰箱中保存备用。
植物总RNA提取试剂盒(RNAiso Plus)、M-MLV反转录试剂盒和pMD18-T载体购自TaKaRa公司,植物基因组DNA提取试剂盒、胶回收试剂盒购自北京天根生物技术服务有限公司,大肠杆菌DH5α菌种为实验室保存,其他试剂均购自上海生工生物工程技术有限公司。
1.2 试验方法
1.2.1 总RNA提取与检测
参照丁勇等[27]方法提取麻疯树种子的总RNA,按照北京天根生物技术有限公司植物基因组DNA提取试剂盒说明书提取麻疯树幼叶基因组DNA。采用1.2%琼脂糖凝胶糖电泳法和紫外分光光度计法分别检测总RNA样品的完整性和纯度,采用1.0%琼脂糖凝胶糖电泳法和紫外分光光度计法分别检测总DNA样品的完整性和纯度。
1.2.2 麻疯树JcOle14.3基因克隆
根据麻疯树oleosin基因mRNA已知序列(序列号EU234464.2),在其开放阅读框(ORF)区域外侧设计特异性引物 1对,上游引物为 JcOle14.3-F:5’-TATCACTGATCGTAAACCTCCA-3’,下游引物为JcOle14.3-R:5’-AAGCGAAAAAAGAAATACACCA-3’。取质量好的总DNA样品为模板,以JcOle14.3-F和JcOle14.3-R特异性引物对进行PCR,反应程序条件为:94℃预变性5 min;94℃变性30 s,53℃退火30 s,72℃延伸1 min,30个循环;72℃延伸10 min。取质量好的总RNA样品为模板,以TaKaRa公司的M-MLV反转录酶进行cDNA第一链的合成,以JcOle14.3-F和JcOle14.3-R特异性引物对进行RT-PCR,反应程序条件为:94℃预变性3 min;94℃变性 30 s,53℃退火 30s,72℃延伸1 min,30个循环;72℃延伸10 min。
1.2.3 PCR产物检测、回收、T/A克隆与测序
PCR产物和回收得到的目的片段采用浓度1%的琼脂糖凝胶电泳检测。将回收的目的片段连接到载体pMD19-T上,将重组质粒转化到大肠杆菌DH5α感受态细胞中,复苏后的菌液涂布在含有Amp、X-Gal和IPTG的LB固体培养基平板上,培养过夜后随机挑选白色菌落,将阳性克隆菌液送往北京六合华大基因科技有限公司进行测序。
1.2.4 序列的生物信息学分析
利 用NCBI中BlastX和BlastN程 序对序列mRNA进行相似性比对,Align two sequences(bl2seq)工具比对分析DNA与mRNA序列,ORF fi nder工具分析序列mRNA的开放阅读框;利用在线ExPASyProtParam(http://web.expasy.org/protparam/)程序分析对预测蛋白质的各种氨基酸含量和蛋白质的等电点、分子量和稳定性进行预测;利用NCBI BlastP程序对推导的蛋白质进行同源搜索;运用Jalview和Clustalx2软件对搜索得到同源性较高的蛋白质进行序列比对;利用MEGA5分子遗传进化分析软件通过邻位相连法(Neighborjoining)构建系统发育树,系统发育树每个统计学显著性分析以(Boot-strp)检验,重复1 000次[28]; 使 用Siganal V4.0 (http://www.cbs. dtu.dk/services/SignalP-2.0/)分析蛋白质信号肽;使用和Proscale(http://www.expasy.org/cgi-bin/protscale.pl)和TMHMM(http://www.cbs. dtu.dk/services/TMHMM)分别检测其亲水/疏水性特性和跨膜结构;利用MLRC(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_mlr.pl)程序预测蛋白质二级结构;使用NCBI提供的 Conserved Domain Search(CD-Search)服务器(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)进行蛋白功能域分析;利用Zhang Lab在线服务程序I-TASSER (http://zhanglab.ccmb.med.umich.edu/)进行蛋白质三级结构预测。
2 结果与分析
2.1 麻疯树油质蛋白JcOle14.3基因克隆
本实验获得的麻疯树种子总RNA经紫外分光光度计测定,总RNA的OD(A260/A280)值处于1.8~2.0之间,1%琼脂糖凝胶电泳结果显示28S和18S rRNA两条带型完整,且前者条带的亮度约为后者的2倍。以提取的总RNA反转录合成的第一链cDNA为模板,进行RT-PCR扩增,结果为一条特异性cDNA片断(如图1中泳道1和2),经胶回收目的条带、TA克隆、测序长度为593 bp。以提取的麻疯树叶片总DNA样品为模板,以JcOle14.3-F和JcOle14.3-R为引物进行PCR扩增,获得麻疯树油质蛋白JcOle14.3基因染色体DNA序列(如图1中泳道4和5),将目的DNA回收并克隆测序,结果得到麻疯树油质蛋白JcOle14.3基因在染色体上长593 bp的DNA序列,该序列与克隆的593 bp的mRNA序列完全一致。本实验克隆的麻疯树油质蛋白JcOle14.3基因序列登录GenBank的序列号为JX073623.1。
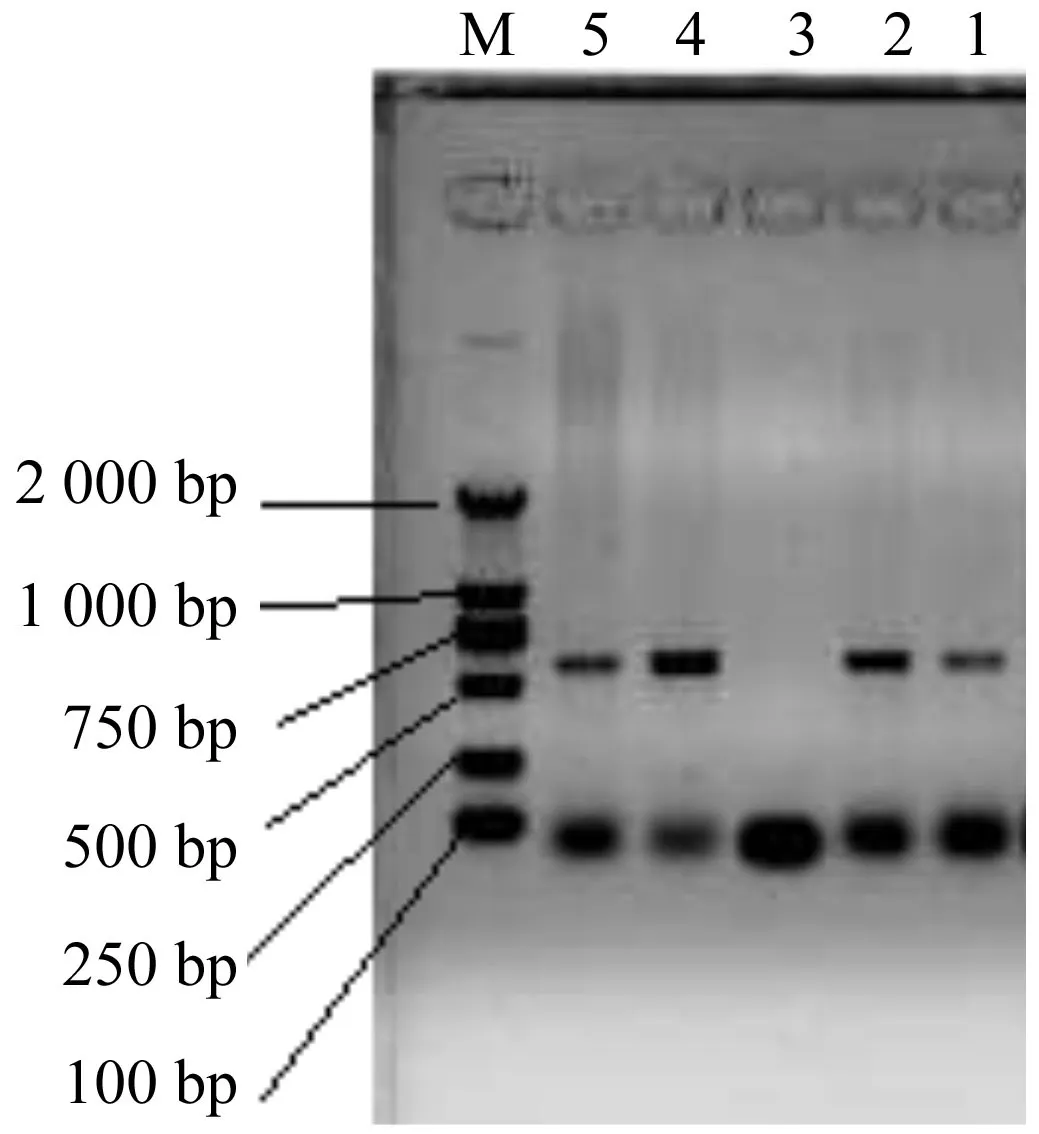
图1 麻疯树JcOle14.3基因克隆产物琼脂糖凝胶电泳图Fig.1 Agrose gel electrophoresis analysis of JcOle14.3 gene from Jatrophacurcas L.
2.2 序列分析
应用NCBI中bl2seq工具,将本实验克隆的593 bp的mRNA序列与麻疯树oleosin基因mRNA已知序列(GenBank序列号:EU234464.2)进行比对分析,结果(如图2)显示两序列在比对范围内具有97.6%的一致性,有14个碱基位点的差异。表明本实验克隆了与序列EU234464.2不一致的麻疯树油质蛋白基因序列,命名为麻疯树油质蛋白基因JcOle14.3,登录GenBank的序列号为JX073623.1。
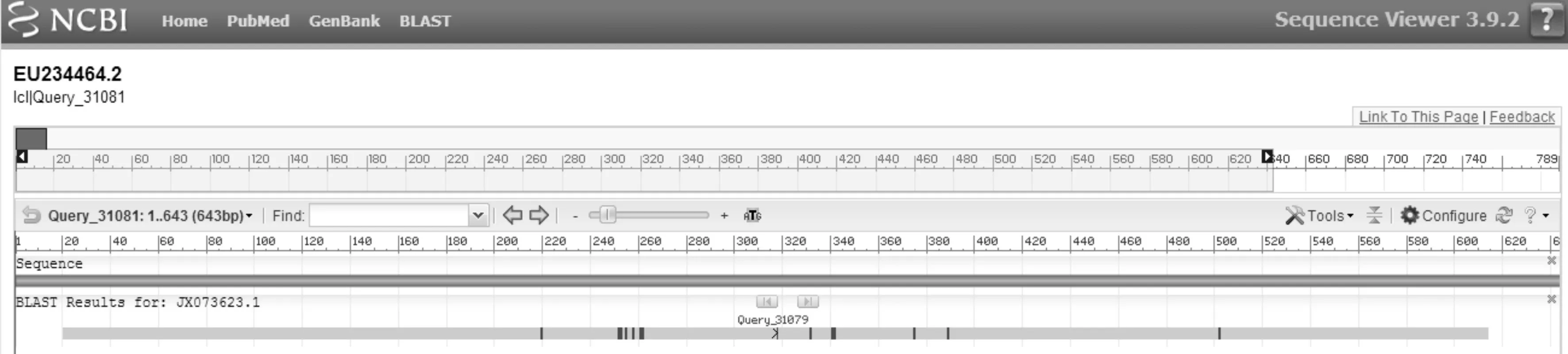
图2 序列JX073623.1和序列EU234464.2的相似性分析Fig.2 Similarlity anaylsis of nucleotides sequences between JX073623.1 and EU234464.2
本实验克隆得到的麻疯树油质蛋白基因JcOle14.3的mRNA全长593 bp,包括52 bp 5'末端非翻译区(5'UTR)、414 bp完整开放阅读框(ORF)和127 bp3'末端非翻译区(3'UTR),预测的PolA结构位置起始于545位核苷酸处。在染色体水平上,麻疯树JcOle14.3基因只含有1个外显子,没有内含子。完整的开放阅读框推测编码由137个氨基酸残基组成的14.3 kD oleosin蛋白,命名为麻疯树油质蛋白JcOle14.3,在GenBank的登录号为AFP19885.1。
通过NCBI网站Blastp工具对麻疯树JcOle14.3蛋白进行相似性搜索分析结果所示(见图3),目的蛋白包含一个潜在的保守区域“oleosindomain”,属于油质蛋白oleosin超家族蛋白之一。麻疯树JcOle14.3蛋白与麻疯树Jatrophacurcas的oleosin 1-like蛋白、桑树(川桑)Morusnotabilis的Oleosin 1、油桐Verniciafordii的oleosin I蛋白、甜橙Citrus sinensis的oleosin 1-like蛋白、蓖麻Ricinuscommunis的Oleosin 2蛋白、巨桉Eucalyptus grandis的oleosin 1-like、烟草Nicotianasylvestris的oleosin 1-like、胡杨Populuseuphratica的oleosin 1-like、油茶Camellia oleifera的OleIII蛋白和芝麻Sesamumindicum的15 kD oleosin蛋白等都具有较高的相似性,E值(E-value)从2e-64到7e-87,一致性(Identity)从76%到96%,相似度(Positives)从83%到97%(见表1)。表明本实验克隆得到的JcOle14.3基因翻译的JcOle14.3蛋白为麻疯树表达的oleosin家族蛋白之一。
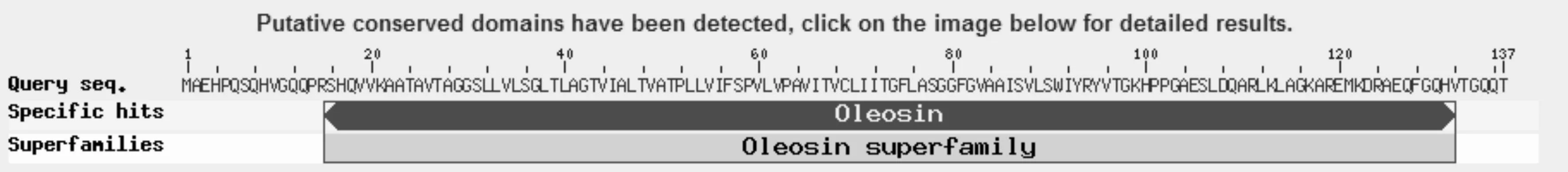
图3 麻疯树JcOle14.3蛋白氨基酸序列在NCBI中Blastp分析结果Fig.3 Blastp anaylsis result of JcOle14.3 amino acid sequence in NCBI
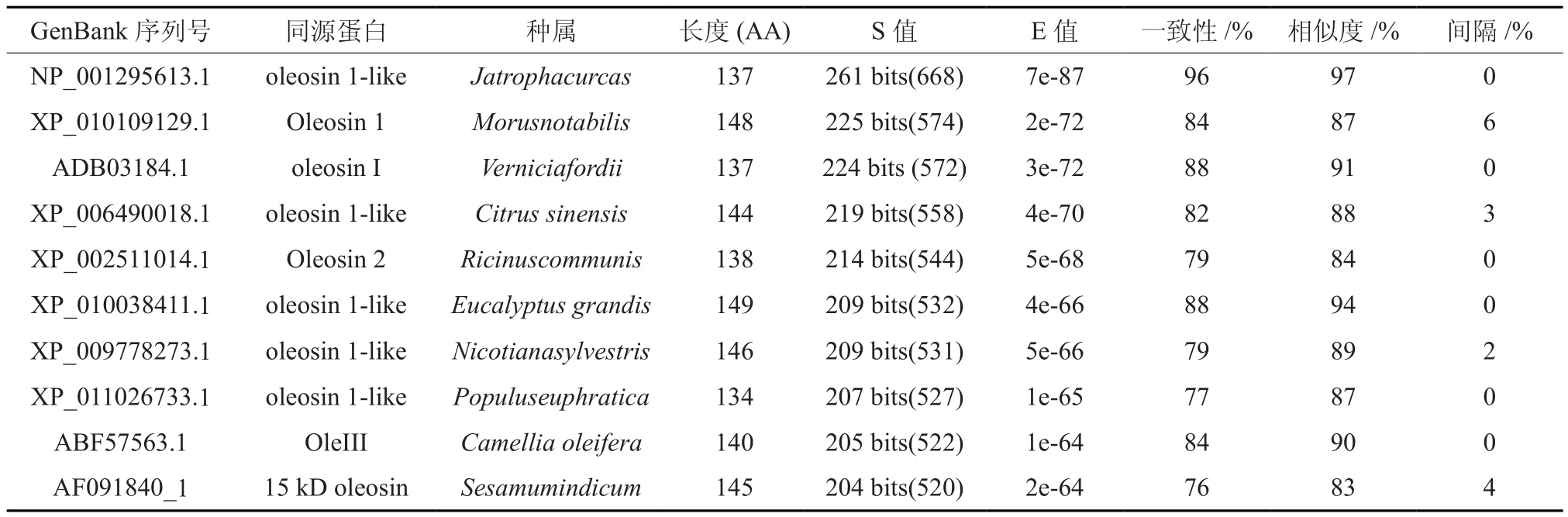
表1 麻疯树JcOle14.3蛋白与其他已知功能蛋白同源性比较Table 1 Similarlity anaylsis of JcOle14.3 with the function identidied proteins in GenBank
麻疯树JcOle14.3蛋白与其他物种中的oleosin蛋白的氨基酸序列比对结果(图4)显示,不同物种的oleosin蛋白在N端和C端具有较大的氨基酸序列差异性,而在中间氨基酸区域具有较高的保守性,尤其包含一个由3个脯氨酸和1个丝氨酸组成的脯氨酸结(Pro Knot)高度保守结构域、即ATPLLVTFSPVLVPA模体,此脯氨酸结模体是植物油质蛋白基因序列的标志性结构特征。麻疯树JcOle14.3和其他物种oleosin蛋白的进化树分析结果(图5)显示,JcOle14.3(AFP19885.1)和大戟科麻疯树oleosin 1-like(NP_001295613.1)亲缘关系最近,聚为一支,和大戟科油桐oleosin I(ADB03184.1)、大戟科蓖麻Oleosin 2(XP_002511014.1)聚为一个大支;但是与麻疯树18.2 kDa-like(NP_001295667.1)的遗传距离则较远。
蛋白质理化性质分析结果显示推导得到的蛋白JcOle14.3由137个氨基酸残基组成,相对分子质量为14.3 kD,等电点pI为9.65,可知该蛋白质在其生理条件下为碱性蛋白质;JcOle14.3蛋白质由除Asn (N)之外的19种常见的氨基酸组成,蛋白质氨基酸成分比例差异较大,其中Cys (C)和Trp (W)的含量最低为0.7%,Ala (A)的含量最高为12.4%,其它氨基酸的含量在1.5%~11.7%之间变化;正电荷氨基酸残基(Arg+Lys)总数为10个,负电荷氨基酸残基(Asp+Glu)总数为6个,因此在pH7.0的环境中它带正电荷;JcOle14.3蛋白质含有2052个总原子数,其中643个碳原子(Carbon,C)、1046个氢原子(Hydrogen,H)、178个氮原子(Nitrogen,N)、182个氧原子(Oxygen,O)、3个硫原子(Sulfur,S),分子式为C643H1046N178O182S3;摩尔消光系数(Extinction coef fi cients)为8480;不稳定系数(instability index,II)值为25.90。推测JcOle14.3属于稳定性蛋白质。

图4 麻疯树JcOle14.3蛋白与其他物种oleosin的氨基酸序列比对Fig.4 Amino acid sequences comparison between JcOle14.3 in Jatrophacurcas and oleosin-like in other species

图5 JcOle14.3与其他植物oleosin的进化树分析Fig.5 Phylogenetic analysis of JcOle14.3 and otheroleosin-like
信号肽是编码分泌性蛋白N端的一段氨基酸序列,是指导分泌性蛋白合成的关键因素,一般含有16~26个氨基酸残基,在完成功能后被切除[29]。JcOle14.3蛋白信号肽分析结果显示,最大C值(原始剪切位点的分值)为0.610,位于第44个氨基酸残基;最大Y值(综合剪切位点的分值)为0.161,位于第68个氨基酸残基;最大S值(信号肽的分值)为0.983,位于第36个氨基酸残基;平均S值为0.599,位于第1~67位氨基酸残基之间;综合计算信号肽存在的可能性值为0.018,信号锚定的可能性值为0.977,第46~47位氨基酸残基之间的剪切的值为0.006。推测JcOle14.3蛋白不存在信号肽序列,为非分泌性蛋白。
蛋白质跨膜结构域预测结果(图6)显示,JcOle14.3蛋白含有2个跨膜螺旋结构域,分别位于32~54位和64~86位氨基酸残基区域;1~31位和87~137位氨基酸残基区域构成2个亲水性区域,位于膜外胞质内;32~86位氨基酸残基区域构成疏水性区域,位于膜内。
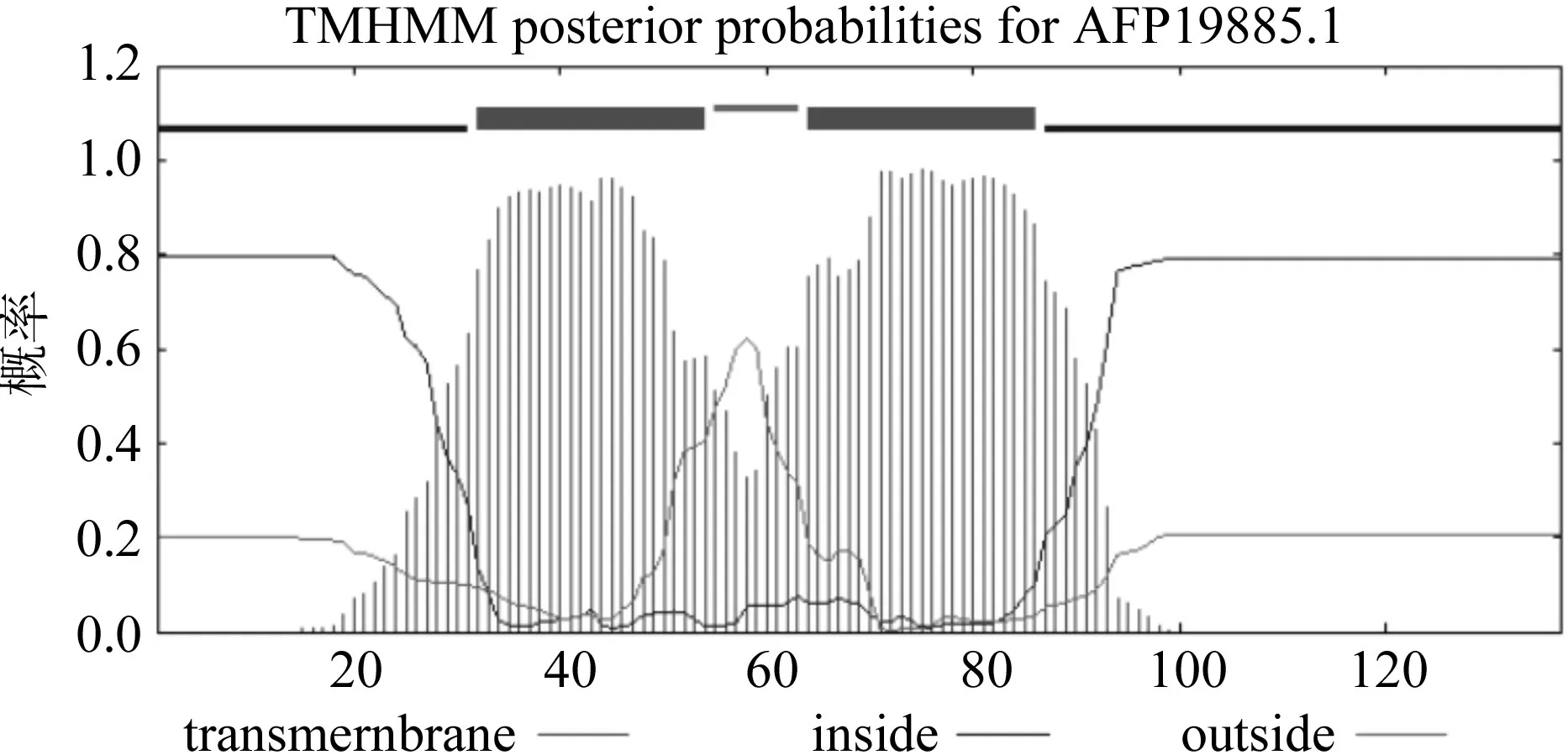
图6 JcOle14.3蛋白跨膜结构域预测Fig.6 Prediction of transmembrane regions of JcOle14.3
蛋白质疏水性分析一方面可以为二级结构预测结果提供参考,另一方面可以为结构域以及功能域的划分提供依据[22]。其中,score>0,表现为疏水性;score<0,表现为亲水性。通过疏水性分析结果显示麻疯树JcOle14.3蛋白质中疏水性最大值是3.256,位于第68位氨基酸残基处;亲水性最大值是-2.578,位于第123位氨基酸残基处。蛋白质中间区域在第21-93位氨基酸残基区域表现出较强的疏水性,在N端1-20位和C端94-133位氨基酸残基区域都表现出强亲水性(图7),这与该蛋白质性质和跨膜结构域分析结果吻合,说明麻疯树JcOle14.3蛋白质是一种两性蛋白质,即该蛋白质两末端为亲水性区域,中间为疏水性区域。
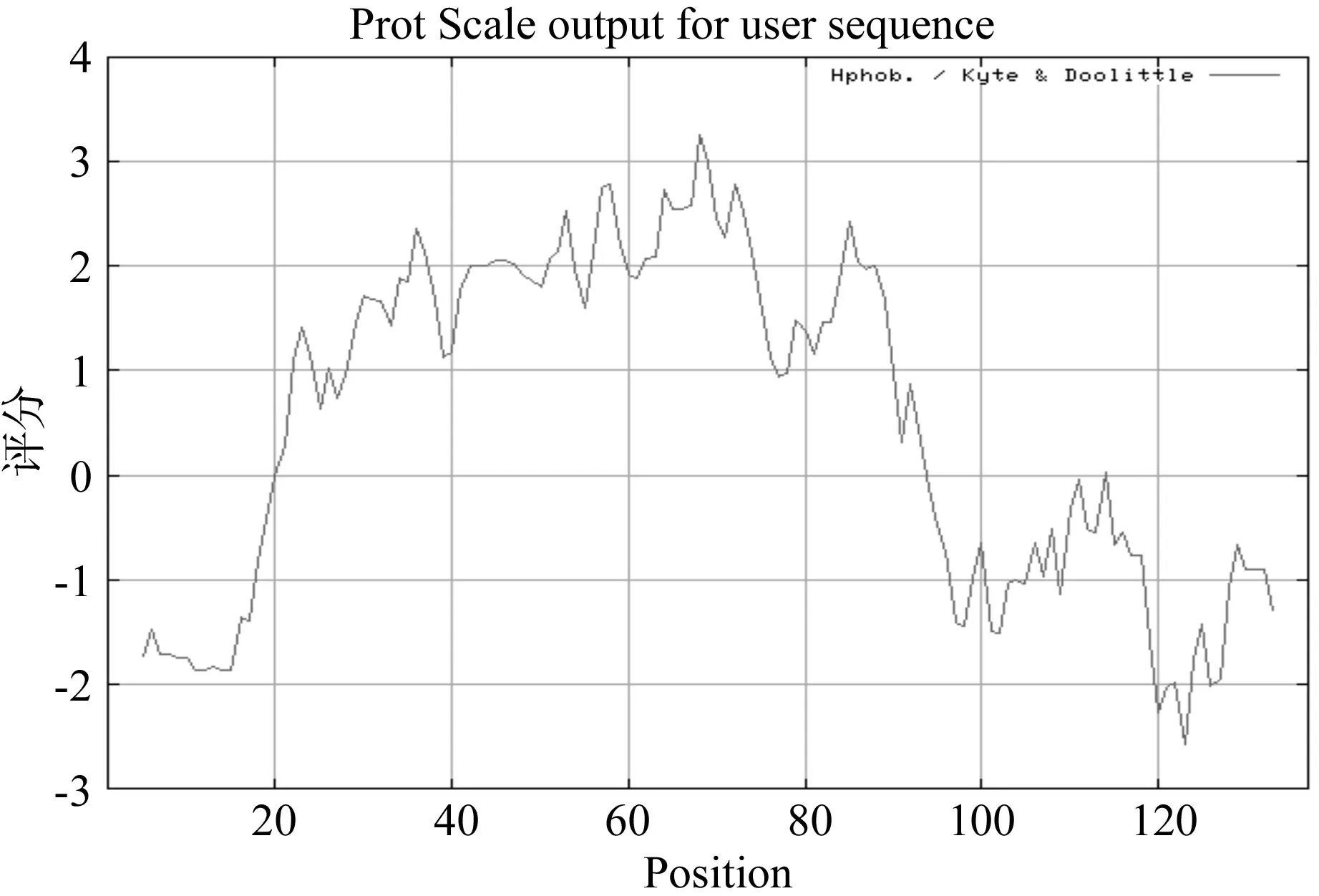
图7 JcOle14.3蛋白质疏水性和亲水性结构分析Fig.7 Prediction structure of JcOle14.3protein hydrophobicity and hydrophilicity pro fi le
蛋白质分子的多肽链通常折叠和盘曲成比较稳定的空间结构,形成比较稳定的二级结构,进一步才能完成活性功能域构象的构建,以完成特定的生命活动。根据蛋白质二级结构预测结果(图8)推测JcOle14.3蛋白二级结构由大量的α-螺旋(Alpha helix)、随机卷曲(Random coil)和少量的延伸链(Extended strand)结构组成。α-螺旋、随机卷曲和延伸链分别占49.64%、36.50%和13.87%。α-螺旋二级结构主要位于16-26位(α1)、60-76位(α2)、83-96位(α3)和105-128位(α4)氨基酸残基区域,随机卷曲二级结构主要位于1-15位、27-30位、77-82位、97-104位、129-137位氨基酸残基区域,延伸链二级结构主要位于31-36位、43-48位、54-57位氨基酸残基区域。表明α-螺旋、随机卷曲和延伸链构成了JcOle14.3蛋白二级结构的主要部分。

图8 JcOle14.3蛋白二级结构预测Fig.8 The secondary structure of the deduced JcOle14.3 protein
跨膜蛋白的三维结构预测是生物信息学研究热点之一。本实验构建的麻疯树JcOle14.3蛋白高级结构如图9所示。根据三维结构预测结果,并结合疏水性、跨膜结构和二级结构预测结果,推测麻疯树JcOle14.3蛋白由3个基本的结构域组成,包括N端30个左右氨基酸残基组成的两亲性(亲水性和亲脂性)区域、C端47个左右氨基酸残基组成的两亲性α-螺旋区域和肽段中部约60个氨基酸残基组成的高度疏水性的跨膜结构域,符合Tzen和Huang提出的oleosin蛋白结构模型[10]。JcOle14.3蛋白三维结构中(图9)显示出蛋白质二级结构预测结果(图8)中的4个α-螺旋,其中α1、α2和α3位于中心疏水性跨膜结构域,α4位于C端两亲性α-螺旋结构域。推测由3个脯氨酸和1个丝氨酸组成的脯氨酸结(Pro Knot)高度保守结构域位于α1和α2之间,可能对保证JcOle14.3蛋白向PL的正确定位和维持油体结构具有重要意义。
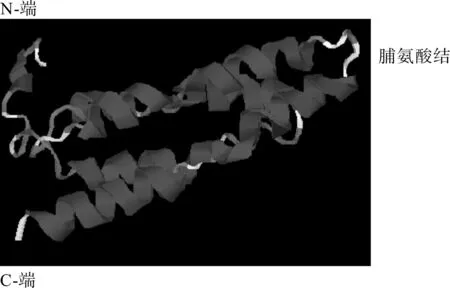
图9 麻疯树JcOle14.3蛋白质三维结构预测Fig.9 3D structure prediction of JcOle14.3 protein
3 讨 论
含油植物种子在发育期间累积大量的储藏型油脂,主要为TAGs,作为种子萌发和随后的幼苗生长所需的碳源和能量来源。TAGs被包裹在离散的、球形的特定细胞器油体中[1,3,4]。油体表面被镶嵌有油体结合蛋白质的PL组成的半单位膜所包裹着[2,7,16,19,25]。油体在植物细胞中相当稳定,是因为油体结合蛋白(主要是oleosin)提供的空间位阻(Steric Hindrance)和负电斥力(Electronegative Repulsion)效应在起作用,因此在种子成熟过程中通过互相积压形式而充满整个细胞的油体不会发生聚合[10-12]。
Oleosin为一个超家族蛋白,其基因以基因家族形式存在。1987年Vance和Huang首次运用分子克隆技术从玉米种子中成功分离16.5kD油质蛋白cDNA序列[30],之后oleosin蛋白及其基因在不同植物如芝麻[18-19]、油菜[20-23]、水稻[24]、拟南芥[25]和油桐[26]等中得到了一定的研究,并发现oleosin均以多个异构体蛋白形式存在各植物中。本实验从麻疯树种子中成功克隆了编码14.3 kDoleosin蛋白的基因JcOle14.3(GenBank序列号:JX073623.1),该基因在染色体水平上只含有1个外显子,没有内含子,JcOle14.3基因DNA复制后发生转录生成mRNA,其完整的开放阅读框推测编码由137个氨基酸残基组成的14.3 kDJcOle14.3蛋白(GenBank序列号:AFP19885.1),在mRNA序列上与已知麻疯树油质蛋白基因(GenBank序列号:EU234464.2)在比对范围内具有14个碱基位点的差异(图2),在氨基酸序列上与已知麻疯树oleosin类似蛋白(GenBank序列号:ABW90150.2)存在4%的差异性,推测oleosin蛋白在麻疯树中也是以不同的蛋白异构体形式存在,本实验获得的14.3 kD油质蛋白JcOle14.3为麻疯树oleosin蛋白家族的新成员。
已知研究表明oleosin是一种高度疏水的碱性小分子量蛋白,只存在于植物体中[31],而来自不同植物的oleosin蛋白都具有相同的结构特性,即具有3个基本的结构域:40-60个氨基酸残基组成的兼具亲水性和亲脂性N-末端区、68-74个氨基酸残基组成的中间疏水区以及33-40个氨基酸残基组成的α-螺旋两性C-末端区[10]。本实验获得的oleosin蛋白家族新成员符合Tzen和Huang提出的oleosin结构模型,推测麻疯树JcOle14.3蛋白由3个基本的结构域组成,包括N-端30个左右氨基酸残基组成的两亲性(兼具亲水性和亲脂性)区域、肽段中部约60个氨基酸残基组成的高度疏水性的跨膜结构域和C-端47个左右氨基酸残基组成的两亲性的α-螺旋结构域。预测得到的4个α-螺旋(图8)中α1、α2和α3位于中心疏水性跨膜结构域,α4位于C端两亲性α-螺旋结构域。根据中心疏水结构域中氨基酸残基的极性分布以及结构分析,推测其为反式平行的β-折叠结构,形成发夹结构并插入TAG中,其顶部为3个脯氨酸和1个丝氨酸(-PX5SPX2P-)组成的脯氨酸结(Pro-Knot),连接两个分开的α-螺旋结构(α1和α2),这与Lacey等提出的oleosin结构新模型一致[32],N-端两亲性区域和C-端两亲性α-螺旋结构域分布于油体表面朝向胞浆一面。推测麻疯树JcOle14.3蛋白类似于其他oleosin,其整个结构类似图钉一样分布在油体表面,而其中心区高度保守的由两个α-螺旋构成的脯氨酸结在其与PL和油体锚定结合过程起着重要的作用。
已有研究推测Oleosin作为最丰富的油体相关蛋白从油体的发生到分解及消失过程中发挥着重要的生物学功能。但植物体是如何响应内外源信号进行调控JcOle14.3基因表达,JcOle14.3蛋白又是如何参与到油体的形成、成熟、贮存及动员的过程,以及在麻疯树种子中JcOle14.3基因表达调控与含油量之间又存在着什么样的关系等问题还有待深入研究。
4 结 论
从麻疯树中克隆了编码14.3 kD油质蛋白(oleosin)的基因JcOle14.3。全长593 bp的JcOle14.3基因(GenBank序列号:JX073623.1)转录的mRNA包含了完整的开放阅读框,无内含子结构。麻疯树中oleosin蛋白是由多基因家族编码的。推测14.3 kD的oleosin蛋白(GenBank序列号AFP19885.1)为137个氨基酸残基组成的两性蛋白质,主要含N末端两亲性结构域、中间疏水性结构域和C-末端两亲性结构域。C-末端两亲性结构域包含一个α-螺旋结构域。中间疏水性结构域包含有一个由3个脯氨酸和1个丝氨酸组成的脯氨酸结(Proline-Knot)模体,在32-86位左右氨基酸残基组成的α-螺旋跨膜区域,推测在oleosin蛋白与单层磷脂层和油体锚定结合上起着重要的作用。
[1] Huang A H C. Oil bodies and oleosins in seeds[J]. Annual review of plant biology, 1992, 43(1): 177-200.
[2] Huang A H C. Oleosin and oil bodies in seeds and other organs[J]. Plant Physiol., 1996, 110(4): 1055-1061.
[3] Yatsu L Y, Jacks T J. Spherosome membranes: Half unitmembranes[J]. Plant Physiology, 1972, 49(6): 937-943.
[4] Murphy D J. Structure, function and biogenesis of storage lipid bodies and oleosins in plants[J]. Prog Lipid Res., 1993,32(3):247-280.
[5] Tzen J T C, Cao Y Z, Laurent P, et al. Lipids, proteins, and structure of seed oil bodies from diverse species[J]. Plant Physiol., 1993, 101 (1): 267-276.
[6] Tzen J T C, Lai Y K, Chan K L, et al. Oleosin is of or ms of high and low molecular weights are present in the oil bodies of diverse seed species[J]. Plant Physiol., 1990, 94 (3): 1282-1289.
[7] Tzen J T C, Peng C C, Cheng D J, et al. A new method for seed oil body purification and examination of oil body integrity following germination[J]. J. Biochem., 1997, 121(4): 762-768.
[8] Chen E C F, Tai S S K, Peng C C, et al. Identi fi cation of three novel unique proteins in seed oil bodies of sesame[J]. Plant Cell Physiol., 1998, 39(9): 935-941.
[9] 丁 勇,徐春雷,甘 莉.植物油体及其相关蛋白的研究进展[J].华中农业大学学报, 2008, 27(4): 558-563.
[10] Tzen J T C, Huang A H C. Surface structure and properties of plant seed oil bodies[J]. J. Cell Biol., 1992, 117(2): 327–335.
[11] Leprince O, van Aelst A C, Pritchard H W, et al. Oleosins prevent oil-body coalescence during seed imbibition as suggested by a low-temperature scanning electron microscope study of dessication-tolerant and -sensitive oilseeds[J]. Planta, 1997,204(1): 109–119.
[12] Jiang P L, Wang C S, Hsu C M, et al. Stable oil bodies sheltered by a unique oleosin in lily pollen[J]. Plant Cell Physiol., 2007,48(6): 812–821.
[13] Openshaw K. A review of Jatrophacurcas: an oil plant of unfulfilled promise[J].Biomass Bioenergy, 2000, 19(1): 1–15
[14] 胡芳名, 谭晓风, 裴 东, 等. 我国经济林学科进展[J]. 经济林研究, 2010, 28(1): 1-8.
[15] Augustus G D P S, Jayabalan M, Seiler G J. Evaluation and bioinduction of energy components of Jatrophacurcas[J].Biomass Bioenergy, 2002, 23(3): 161–164.
[16] Frandsen G I, Mundy J, Tzen J T C. Oil bodies and their associated proteins, oleosin and caleosin[J]. Physiol. Plant, 2001,112(1): 301-307.
[17] May fi eld J A, Fiebig A, Johnstone SE, et al. Gene families from the Arabidopsis thaliana pollen coat proteome[J]. Science,2001,292(5526): 2482-2485.
[18] Chen J C F, Tsai C C Y, Tzen J T C. Cloning and secondary structure analysis of caleosin, a unique calcium-binding protein in oil bodies of plant seed[J]. Plant Cell Physiol., 1999, 40(10): 1079-1086.
[19] Lin L J, Tai S S K, Peng C C, et al. Steroleosin, a sterol-binging dehydrogenase in seed oil bodies[J]. Plant Physiol., 2002, 128(4):1200-1211.
[20] Murphy D J, Cummins I, Kang A S. Synthesis of the major oilbody membrane protein in developing rapeseed embryos[J].Biochem. J., 1989, 258(1):285-293.
[21] Keddie J S, Hubner G, Slocombe S P, et al. Cloning and characterization of an oleosin gene from Brassica napus[J]. Plant MolBiol., 1992, 19(3): 443-453.
[22] 丁 勇, 陈庆波, 徐春雷, 等. 油菜油体钙蛋白基因BnClo1的克隆和表达[J].作物学报, 2008, 34(11): 1921-1928.
[23] 丁 勇, 常 玮, 刘小烛. 甘蓝型油菜BnClo1基因克隆、表达载体的构建及原核表达[J].中国农业科学, 2010, 43(2):252-258.
[24] Frandsen G I, Muller-Uri F, Nielsen M, et al. Novel plant Ca2+-binding protein expressed in response to abscisic acid and osmotic stress[J]. J. Biol Chem., 1996, 271(1): 343-348.
[25] Naested H, Frandsen G I, Jauh G Y, et al. Caleosins: Ca2+-binding proteins associated with lipid bodies[J]. Plant MolBiol., 2000,44(4): 463-476.
[26] 龙洪旭,谭晓风, 陈 洪, 等. 油桐油体蛋白基因的克隆及序列分析[J].中南林业科技大学学报, 2010, 30(4): 31-38.
[27] 丁 勇, 范红波, 张高磊, 等.麻疯树种子总RNA提取方法研究[J].中南林业科技大学学报, 2012, 32(3):158-161.
[28] Kumar S, Tamura K, Nei M. MEGA: Molecular evolutionary genetics analysis software for microcomputers[J].Comput Appl Biosci., 1994, 10 (2): 189-191
[29] 王汉屏. 不同植物防御素的生物信息学分析[J]. 植物生理学通讯, 2008, 44 (1): 25-32.
[30] Vance V B, Huang A H. The major protein from lipid bodies of maize. Characterization and structure based on cDNA cloning[J].J. BiolChem, 1987, 262(23): 11275-11279.
[31] KandpalJB,Madan M. Jatrophacurcus: a renewable source of energy for meeting future energy needs[J]. Renewable Energy,1995, 6 (2): 159-160.
[32] Lacey D J, Wellner N, Beaudoin F, et al. Secondary structure of oleosins in oil bodies isolated from seeds of safflower(Carthamustinctorius L.) and sun flower(Helianthus annuus L.)[J]. Bioche J., 1998, 334(22): 469-477.
Cloning and sequence analysis of oleosin gene JcOle14.3 in Jatrophacurcas
SONG Jian, XIONG Hong, YU Jin-de, LIU Xiao-zhu, DING Yong
(College of Life Sciences, Southwest Forestry University, Kunming 650224, Yunnan, China)
Oleosins as the most abundant oil body-associated proteins have been suggested to play an important biological function in the stability ofoil bodies, and in their synthesis and metabolism. In this study, the gene JcOle14.3 containing no intron and encoding 14.3 kD oleosin was isolated by PCR combined with bioinformatics analysis and molecular biological technique from Jatrophacurcas L..The full-length mRNAcomprised 593 nucleotides consisting of an open reading frame of 414 nucleotides encoded a putative JcOle14.3 comprising 137 amino acid residues with molecular weight of 14.3 kD. The isoelectric point of 9.65.and the total number of positively charged residues (Arg+Lys) was 10, and total number of negatively charged residues (Asp+Glu) was 6. JcOle14.3 with unstable coefficient 25.90 belongs to the stable protein. The main parts of predicted secondary structures of JcOle14.3 were α-helices, random coils and extended strand. The JcOle14.3 gene product consists of a highly hydrophobic central domain fl anked byrelatively polar N- and C-terminal domains. The results indicated that the C-terminal region forms an amphipathicα-helix. The JcOle14.3 central domain forms three α -helices and α1 and α2 were separated by the proline-knot forming a 180 turn. The oleosin central domain is highly conserved between all oleosinssequenced to date. This workalso suggested that the highly conservedproline-knotplayimportantrolesfor oil body targeting. The study results established the foundation for the heterologous expression and functional analysis of JcOle14.3.
Jatrophacurcas; oil body; oleosin;gene cloning;sequence analysis
S759.3+3
A
1673-923X(2016)06-0015-08
10.14067/j.cnki.1673-923x.2016.06.004
2015-10-20
云南省生物学优势特色重点学科建设项目(50097505);云南省高校林下生物资源保护及利用科技创新团队项目(51400605);国家自然科学基金(31460076)
宋 健,硕士研究生,熊宏为共同第一作者
丁 勇,高级实验师,硕士研究生导师;E-mail:dingyong@swfu.edu.cn
宋 健,熊 宏,余进德,等. 麻疯树油质蛋白JcOle14.3基因克隆及序列分析[J].中南林业科技大学学报,2016, 36(6):15-22.
[本文编校:吴 彬]

