5-氮杂胞苷对马氏珠母贝早期发育的影响
李耀国,孙 彤,张雅剑,巩建豪
(海南热带海洋学院 生命科学与生态学院, 海南 三亚 572022)
5-氮杂胞苷对马氏珠母贝早期发育的影响
李耀国,孙 彤,张雅剑,巩建豪
(海南热带海洋学院 生命科学与生态学院, 海南 三亚 572022)
为了解5-氮杂胞苷对马氏珠母贝早期发育的影响,该研究检测并分析了其对早期发育阶段甲基化水平和矿化基因表达的影响. 结果显示5-氮杂胞苷处理可引发基因组甲基化水平显著降低. 5-氮杂胞苷处理亦使DNMT3的表达量低于海水处理组,而矿化基因nacrein的表达量高于海水处理组.DNMT3和nacrein的表达呈显著正相关(R2=0.755,P=0.000). 基因组甲基化水平的改变可能是调控生物矿化过程的重要因素.
马氏珠母贝;去甲基化试剂;早期发育;甲基化水平;生物矿化
0 引言
生物体的表型变化主要由DNA序列的变异引起,除了常见的A、T、C、G 四个脱氧碱基的变异外,一种表观遗传DNA甲基化修饰引起了科研工作者的广泛关注.这种DNA甲基化修饰表现为甲基化转移酶将序列中胞嘧啶加上一个甲基基团,使胞嘧啶成为“5-甲基胞嘧啶”[1].5-甲基胞嘧啶主要发生在“CpG” 二核苷酸或者其它类型位点如“GpC”中的胞嘧啶上[2-3].动物中胞嘧啶甲基化在非编码DNA区域(内含子、重复元件和活性转座子元件)的失活中起到重要作用,并能通过引起染色质结构、DNA构象及DNA与蛋白质相互作用方式的改变来影响基因表达,是大多数生物生长发育过程中不可缺少的一部分[4].
甲基化敏感扩增多态性技术(MSAP,methylation sensitive amplification polymorphism)是一种可对基因组 “5′-CCGG-3′”位点胞嘧啶甲基化状态进行检测的常用方法. 其原理主要是根据HpaII和MspI这两种同裂酶对基因组“5′-CCGG-3′”双链位点中胞嘧啶甲基化状态不同而具不同酶切活性,可比较不同酶切反应对应PCR产物的电泳图谱来分析样品甲基化状况.HpaII酶可以酶切“5′-CCGG-3′”单链外部胞嘧啶甲基化序列,而MspI酶可以酶切“5′-CCGG-3′”双链内部胞嘧啶甲基化序列[5].MSAP技术已广泛应用于基因组甲基化水平检测,DNA甲基化与杂种优势的关联分析[6]以及DNA甲基化与生物抗性的关系分析等[7].
多数非脊椎动物甲基化在基因组中的分布呈一种“镶嵌模型”,即甲基化修饰的“CpG”位点散布于整个基因组中[8].其中双壳贝类DNA甲基化主要存在于基因内含子及外显子上[9],基因的DNA甲基化与其表达调节相关,其中免疫相关基因常呈现出点缀分布的甲基化状态[10].贝类甲基化的研究报道较少,主要集中于太平洋牡蛎和马氏珠母贝两个物种中. 太平洋牡蛎甲基化的基因与基因高转录丰度及组织间低表达变异存在关联,同源盒型基因的甲基化可使基因表达水平下降,DNA甲基化与基因组具有一定关联性并对早期发育产生影响[9-12].马氏珠母贝(Pinctadafucata)是广泛分布于中国广东、广西、海南省及日本沿海的具重要经济价值的珍珠贝. 其组织甲基化水平范围为11.71至14.71%,免疫相关基因galectin启动子区甲基化水平与基因的表达水平呈显著正相关,而基因组甲基化水平与galectin基因的表达水平呈负相关[13-14].目前对马氏珠母贝DNA甲基化的研究主要集中于对其基因组及基因甲基化状态的检测及分析,尚未见人工主动改变基因组甲基化水平对其早期胚胎发育及基因表达影响的研究报道.
该研究利用MSAP技术检测DNA甲基化酶抑制剂5-氮杂胞苷处理对马氏珠母贝早期发育阶段基因组甲基化水平的影响,比较处理后甲基化转移酶基因DNMT3及矿化基因nacrein的表达水平变化,并对基因表达的相关性进行了分析.目的在于获得人工主动去甲基化对马氏珠母贝早期发育影响的直接证据,并为深入探讨DNA甲基化在贝类生长发育、生物矿化中的作用提供科学基础.
1 材料与方法
1.1 样品采集和核酸提取
该研究所用2龄马氏珠母贝个体从中国科学院大亚湾海洋生物综合实验站(深圳)采集,作为育种亲贝在海水池中暂养.通过解剖观察判断亲本雌、雄性别后,设立3组马氏珠母贝雌、雄亲本单对配对的人工授精实验. 分别将每组单对配对亲本产生的精子和卵细胞进行混合以形成受精卵.每对单对配对亲本的受精卵分成两部分:对照组(采用灭菌海水浸泡处理)及实验组(采用10-5mol L-15-氮杂胞苷溶液浸泡处理). 分别对所有三组单对配对实验中受精卵、二细胞期、桑葚期胚、担轮幼虫、D型幼虫进行显微观察及样品采集(包含壳顶幼虫). 采集的样品保存于核酸保护液中.样品基因组DNA和RNA的提取按照广州美基公司的核酸提取试剂盒说明书进行操作.提取后的核酸稀释于超纯水中,核酸的完整性和浓度分别通过1.2%琼脂糖凝胶电泳和Nanodrop分光光度计OD260/280值进行分析,于-80°C保存待用.
1.2 MSAP技术检测早期发育阶段甲基化水平
利用MSAP技术对各实验组和对照组受精卵、二细胞期、桑葚期胚、担轮幼虫、D型幼虫和壳顶幼虫样品的基因组甲基化水平进行检测. 具体步骤如下:双酶切反应,利用EcoRI和HpaII,EcoRI和MspI酶组合分别对源自同一样品的两份DNA进行37 ℃下酶切6 h.酶切后经1% 琼脂糖凝胶电泳分析酶切完全性.引物接头的生成及连接:等体积的10 pmol/μL EcoRI接头F引物及10 pmol/μL EcoRI 接头R引物混合后退火形成EcoRI接头;等体积的50 pmol/μL HM 接头F引物及50 pmol/μL HM 接头R引物混合后退火形成HM 接头.其中退火程序:65 ℃,15 min;37 ℃,15 min;20 ℃,15 min;最后置于冰水混合物15 min.酶切产物与引物接头的连接:2 μL双酶切产物,50 pmol HM 接头,10 pmol EcoRI接头,T4 DNA 连接酶(350U/μL)0.2 μL,4 μL 10 × T4 DNA 连接缓冲液,加ddH2O至总体积为20 μL,16℃连接过夜.连接产物稀释10倍后作为预扩增模板.预扩增反应和选择扩增反应:预扩增反应体系设置为20 μL:包括10.5 μL Premix Ex Taq(Takara,大连),1 μL稀释10倍后的连接产物,1μL 的20 pmol/μL引物EcoR+A,1μL 的20 pmol/μL引物HM +T,加ddH2O 6.5 μL.PCR程序为94 ℃ 5 min;20个循环的94 ℃ 30 s,56 ℃ 30 s,72 ℃ 1 min;最后72 ℃ 5 min,得到预扩增产物. 选择扩增反应体系包括10.5 μL Premix Ex Taq,1 μL稀释10倍后的预扩增产物,0.5 μL的20 pmol/μL的选择扩增引物E+3,2 μL 的20 pmol/μL的选择扩增引物HM+3(3为碱基数)所用引物组合为(Eco+ATA,HM+TCT;Eco+ATA,HM+TCA;Eco+ATC,HM+TCT;Eco+ATC,HM+TCA),加ddH2O 6 μL. 反应程序分两步:第一步为13个循环的94 ℃ 30 s,65 ℃ 30 s,72 ℃ 1 min,每个循环退火温度降低0.7 ℃,直至56 ℃.第二步为27个循环的94 ℃ 30 s,56 ℃ 30 s,72 ℃ 1 min;最后72℃ 5 min.上述引物序列基于Xiong等[15]研究设计,接头引物及扩增引物序列见表1,由华大基因广州分公司合成.
1.3 变性聚丙烯酰胺凝胶电泳及银染检测
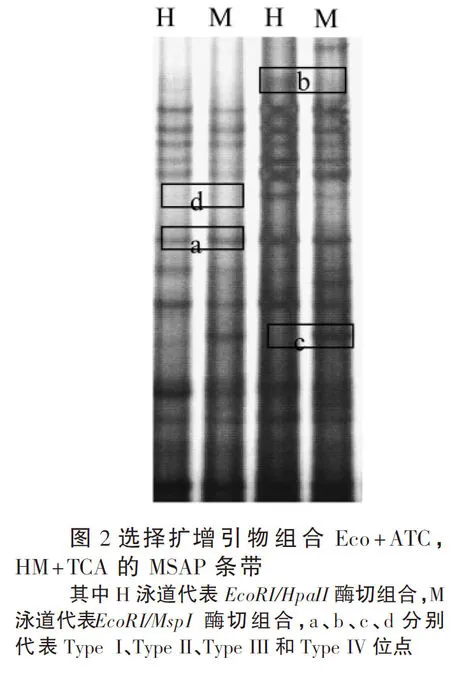
将选择扩增PCR产物与甲酰胺变性上样缓冲液按体积比3∶1混合, 95 ℃加热10 min后置冰上迅速冷却. 每管取3 μL上样于8% SDS-PAGE凝胶,200 V恒压电泳至溴酚蓝指示带到达凝胶底部. 将SDS-PAGE凝胶小心取下放入双蒸水漂洗后进行银染显色并拍照记录.根据MSAP扩增条带可分析四种甲基化情况:(1)MspI酶和HpaII酶对应的电泳泳道同一位置存在大小相同的条带,为“5′-CCGG-3′”双链序列胞嘧啶无甲基化位点(Type I 位点).(2)在HpaII酶对应泳道有一条扩增条带而在MspI酶泳道相应位置无条带,为“5'-CCGG-3'”单链序列外部胞嘧啶甲基化位点(Type II 位点).(3)在MspI酶对应泳道具有一条扩增条带而在HpaII酶泳道的相应位置无条带,为“5'-CCGG-3'”的双链序列内部胞嘧啶甲基化位点(Type III 位点)[16].(4)在MspI对应的电泳泳道及HpaII对应泳道的同一位置均无条带,而其它样品在该位置具有条带,为双链“5'-CCGG-3'”全甲基化位点(Type IV 位点)[17].DNA 甲基化水平的计算: DNA甲基化水平(%)=总甲基化位点数/(Type I + Type II+ Type III + Type IV 位点数).其中总甲基化位点数为Type II、Type III、Type IV 位点数之和.

表1 MSAP分析及荧光定量所用载体及引物序列引物及载体名称引物序列(5'-3')EcoRI接头FCTCGTAGACTGCGTACCEcoRI接头RAATTGGTACGCAGTCTACHpaII-MspI接头FGATCATGAGTCCTGCTHpaII-MspI接头RCGAGCAGGACTCATGAEco+AGACTGCGTACCAATTCAHM+TATCATGAGTCCTGCTCGGTEco+ATAGACTGCGTACCAATTCATAEco+ATCGACTGCGTACCAATTCATCHM+TCTATCATGAGTCCTGCTCGGTCTHM+TCATCATGAGTCCTGCTCGGTCADNMT3FGGACCACCTTCACAGAAACTCDNMT3RTGCGGAAAAACTCAAAAAACAnacreinFACCAGACGTAAGGGATCAGAAnacreinRATCGTTATGACCAGGGAATG18sFACACCGCCCGTCGCTACTAC18sRCGCCCTTCTTCTCGGCACAC
1.4 荧光定量检测及数据分析
利用荧光定量PCR 技术对DNA 甲基化转移酶基因DNMT3、生物矿化基因nacrein 的表达水平进行检测.步骤如下: 利用Primer Primier 5.0 软件设计两对荧光定量引物(DNMT3F和DNMT3R以及nacrein F 和nacrein R).PCR 反应在Roche LightCycler480 热循环仪中进行,体系如下: 5 μL 2 × SYBR Premix ExTaqTM,1 μL模板cDNA,荧光定量引物各0.4 μL(10 μM),加 3.2 μL去离子水至总体积为10 μL. PCR反应程序:95°C 1 min,45 个循环的95°C 5 s,55°C 15 s,72°C 60 s. 选用的参考基因为18S (Genebank登录号:AY028625.1),引物对应为18s F和18s R[14].基因的相对表达量通过Ct方法 (2-△△Ct 方法)计算.马氏珠母贝5-氮杂胞苷实验中实验组和对照组的基因相对表达量比较,各发育时期间的基因组甲基化水平比较、相关性分析均通过SPSS19.0 软件进行,显著性水平设置为P<0.05.
2 结果
2.1 早期发育胚胎观察

图1 马氏珠母贝各早期发育阶段
对马氏珠母贝各实验组、对照组早期发育状况进行了显微观察及拍照,结果如下图1所示.其中受精卵为精子与卵细胞混合后约15分钟形成,随后排出第一和第二极体. 从二细胞期至囊胚期主要体现为细胞数目的增多.当马氏珠母贝早期胚胎发育至D型幼虫期时,其外型呈较明显的“D”型. 比较对照组(灭菌海水处理)和实验组(10-5mol L-15-氮杂胞苷溶液处理)的早期发育胚胎发现两组之间并不存在明显的形态差异. 2.2 早期发育DNA甲基化水平检测
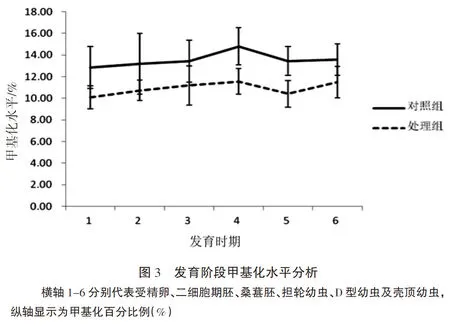
利用MSAP技术对马氏珠母贝各单对配对对照组和实验组的受精卵、二细胞期、桑葚期胚、担轮幼虫、D型幼虫及壳顶幼虫样品的DNA甲基化水平进行了检测. 共计4对选择性扩增引物被用于DNA甲基化分析(图2).结果显示总计检测到1719个“5′-CCGG-3′”位点中的胞嘧啶甲基化状态. 选择性9,平均检测位点数为35. 被检测到的位点中含211个甲基化位点,包括188个全甲基化位点和23个半甲基化位点. 对照组中受精卵、二细胞期胚、桑葚胚、担轮幼虫、D型幼虫及壳顶幼虫的DNA甲基化水平分别为12.85±1.94%,13.19±2.81%,13.41±1.94%,14.79±1.71%,13.43±1.33% 和13.55±1.45%. 处理组中受精卵、二细胞期胚、桑葚胚、担轮幼虫、D型幼虫及壳顶幼虫的DNA甲基化水平分别为10.09±1.07%,10.73±0.95%,11.19±1.82%,11.56±1.19%,10.41±1.23%和11.50±1.45%. 处理组与对照组各组内DNA甲基化水平均无显著差异. 除桑葚胚和壳顶幼虫期样品外,处理组DNA甲基化水平显著低于对照组对应样品的甲基化水平(P<0.05)(图3).早期发育各阶段中担轮幼虫的DNA甲基化水平最高, 受精卵的DNA甲基化水平最低. 随着马氏珠母贝早期发育的进行,基因组甲基化水平总体呈上升的趋势.
2.3 早期发育阶段基因表达检测
利用荧光定量PCR技术对甲基化转移酶DNMT3及生物矿化基因nacrein在对照组和实验组的表达水平进行了检测,结果如图4所示. 在对照组和处理组中,DNMT3和nacrein基因的表达量均随发育进行呈升高的趋势. 与对照组相比,经去甲基化试剂5-氮杂胞苷溶液处理后,生物矿化基因nacrein的表达量升高,但升高未达显著水平. 与对照组相比,实验组中甲基化转移酶DNMT3的表达量降低,但降低未达显著水平. 整个早期发育过程中,实验组及对照组中甲基化转移酶DNMT3和生物矿化基因nacrein的表达水平呈显著正相关(R2=0.755,P=0.000).

图4 早期发育阶段基因表达水平分析横轴1-6分别代表受精卵、二细胞期胚、桑葚胚、担轮幼虫、D型幼虫及壳顶幼虫,纵轴为相对表达水平值
3 讨论与结论
DNA甲基化修饰可从亲本遗传至子代并对许多生物代谢活动起到重要调控作用[18].如正常的基因组甲基化修饰对动物、植物的生殖细胞形成、早期胚胎发育潜能形成及发育调控具重要作用[19-20].该研究中,我们利用去甲基化试剂5-氮杂胞苷对马氏珠母贝受精卵进行浸泡处理并分析其对早期发育过程、DNA甲基化水平及甲基化转移酶、矿化基因表达的影响. 从结果可知5-氮杂胞苷处理可使马氏珠母贝早期发育受到明显影响,主要表现在三个方面:一是早期胚胎成活率降低;二是大部分早期发育阶段基因组甲基化水平显著降低(P<0.05);三是甲基化转移酶基因表达量降低和矿化基因的表达量上升. 相似的研究在同属双壳贝类的太平洋牡蛎中也开展过,5-氮杂胞苷处理可显著降低早期发育过程中的DNA甲基化水平,并引起D型幼虫等胚胎的形态异常,最终导致死亡[12].本研究中马氏珠母贝早期发育阶段经去甲基化试剂处理后,虽未发现胚胎阶段出现形态发育异常,但也出现了处理组(5-氮杂胞苷处理)存活率低于对照组(海水处理)的现象,说明早期发育过程中DNA甲基化水平的人工降低对多数个体是致死的.
动物中DNA甲基化主要发生在“CpG”二核苷酸位点上,基因组中可高达60%到90%的“CpG”二核苷酸被甲基化修饰[21].DNA甲基化转移酶基因家族成员主要为DNMT1,DNMT2,DNMT3a,DNMT3b以及DNMT3L,这些酶在DNA甲基化修饰形成中发挥重要作用. 其中DNMT3主要负责配子形成期及胚胎发育早期DNA甲基化模式的建立.目前贝类DNA甲基化转移酶基因及DNA甲基化结合蛋白的研究很少. 仅见Wang等[22]鉴定并获得了太平洋牡蛎中Dnmt1,Dnmt2,Dnmt3和MBD2/3等DNA甲基化酶核心基因.DNMT3甲基化酶表达水平的降低可能引起全基因组甲基化水平的降低[23].本研究中马氏珠母贝DNMT3甲基化转移酶基因在经去甲基化试剂5-氮杂胞苷处理后表达水平降低可能预示着全基因组甲基化水平降低,进而大规模的基因表达水平变化可能会发生.
马氏珠母贝隶属于软体动物,其外套膜分泌产物是贝壳及珍珠形成的基础.贝壳由外套膜分泌产物在外壳的内面与外套膜间沉积而成[24].马氏珠母贝外套膜nacrein蛋白是其外表皮细胞分泌基质蛋白的一种,由碳酸酐酶结构域和Gly-X-Asn结构域构成,其中Gly-X-Asn重复结构域在减弱钙化过程中起关键作用. 重组nacrein蛋白可以在含CaCl2和NaHCO3的饱和碳酸钙溶液中抑制碳酸钙的沉淀,起到负向调控贝类壳钙化过程的作用[25].该研究中5-氮杂胞苷处理可引起nacrein表达水平的升高,可能会对马氏珠母贝生物矿化过程如壳和珍珠的形成产生影响. 从研究结果还可知DNMT3基因与nacrein基因的表达量呈显著正相关,因此可以较大胆的推测,nacrein基因表达量的变化可能源于基因组甲基化水平的变化,该基因某些区域(如启动子区域) 甲基化水平的变化可能直接与其表达量变化相关.在今后的研究中,可以尝试对矿化基因启动子区、基因内部编码区等部位的甲基化水平进行检测,并分析其与基因表达的关系,为DNA甲基化在生物矿化过程中的可能作用提供证据.
该研究利用去甲基化试剂5-氮杂胞苷处理了马氏珠母贝的受精卵并研究了其对后续发育阶段的影响. 结果表明5-氮杂胞苷可引起早期发育阶段基因组甲基化水平显著降低,甲基化转移酶基因DNMT3和矿化基因nacrein的表达量均发生变化且两基因的表达呈显著正相关.研究表明基因组甲基化修饰对马氏珠母贝早期发育过程具有重要意义,通过改变基因组甲基化水平可能是调控矿化过程,影响贝壳及珍珠形成的可行途径之一.
[1]Field L M, Blackman R L. Insecticide resistance in the aphid Myzus persicae (Sulzer): chromosome location and epigenetic effects on esterase gene expression in clonal lineages [J].Biological Journal of the Linnean Society, 2003, 79(1): 107-113.
[2]Clark S J, Harrison J, Frommer M. CpNpG methylation in mammalian cells [J].Nature Genetics, 1995, 10(1): 20-27.
[3]Pontecorvo G, De Felice B, Carfagna M. Novel methylation at GpC dinucleotide in the fish Sparus aurata genome [J].Molecular Biology Reports, 2000, 27(4): 225-230.
[4]Jones P A, Takai D. The role of DNA methylation in mammalian epigenetics [J].Science, 2001, 293(5532): 1068-1070.
[5]Baurens F C, Bonnot F, Bienvenu D, et al. Using SD-AFLP and MSAP to assess CCGG methylation in the banana genome [J].Plant Molecular Biology Reporter, 2003, 21(4): 339-348.
[6]Li A, Song W Q, Chen C B, et al. DNA methylation status is associated with the formation of heterosis in Larix kaempferi intraspecific hybrids [J].Molecular Breeding, 2013, 31(2): 463-475.
[7]Sha A H, Lin X H, Huang J B, et al. Analysis of DNA methylation related to rice adult plant resistance to bacterial blight based on methylation-sensitive AFLP (MSAP) analysis [J].Molecular Genetics and Genomics, 2005, 273(6): 484-490.
[8]Suzuki M M, Kerr A R, De Sousa D, et al. CpG methylation is targeted to transcription units in an invertebrate genome [J].Genome Research, 2007, 17(5): 625-631.
[9]Gavery M R, Roberts S B. Predominant intragenic methylation is associated with gene expression characteristics in a bivalve mollusk [J].PeerJ, 2013, 1: e215.
[10]Gavery M R, Roberts S B. DNA methylation patterns provide insight into epigenetic regulation in the Pacific oyster (Crassostrea gigas) [J].BMC Genomics, 2010, 11(1): 483.
[11]Jiang Q, Li Q, Yu H, et al. Genetic and epigenetic variation in mass selection populations of Pacific oyster Crassostrea gigas [J].Genes & Genomics, 2013b, 35(5): 641-647.
[12]Riviere G, Wu G C, Fellous A, et al. DNA methylation is crucial for the early development in the oyster C. gigas [J].Marine Biotechnology, 2013, 15(6): 739-753.
[13]Li Y, Guan Y, Li Q, et al. Analysis of DNA methylation in tissues and development stages of pearl oyster Pinctada fucata [J].Genes & Genomics, 2015, 37(3): 263-270.
[14]Li Y, Huang X, Guan Y, et al. DNA methylation is associated with expression level changes of galectin gene in mantle wound healing process of pearl oyster, Pinctada fucata [J].Fish & shellfish immunology, 2015, 45(2): 912-918.
[15]Xiong L Z, Xu C G, Maroof M A S, et al. Patterns of cytosine methylation in an elite rice hybrid and its parental lines, detected by a methylation-sensitive amplification polymorphism technique [J].Molecular and General Genetics, 1999, 261(3): 439-446. [16]Lu Y, Rong T, Cao M. Analysis of DNA methylation in different maize tissues [J].Journal of Genetics and Genomics, 2008, 35(1): 41-48.
[17]Fulnecek J, KovarikA. How to interpret methylation sensitive amplified polymorphism (MSAP) profiles [J].BMC Genetics, 2014, 15(1): 2.
[18]Daxinger L, Whitelaw E. Understanding transgenerational epigenetic inheritance via the gametes in mammals [J].Nature Reviews Genetics, 2012, 13(3): 153-162.
[19]Finnegan E J, Genger R K, Peacock W J, et al. DNA methylation in plants [J].Annual Review of Plant Biology, 1998, 49(1): 223-247.
[20]Reik W, Dean W, Walter J. Epigenetic reprogramming in mammalian development [J].Science, 2001, 293(5532): 1089-1093.[21]Bird A P, Taggart M H, Nicholls R D, et al. Non-methylated CpG-rich islands at the human alpha-globin locus: implications for evolution of the alpha-globin pseudogene [J].The EMBO Journal, 1987, 6(4): 999-1004.
[22]Wang X, Li Q, Lian J, et al. Genome-wide and single-base resolution DNA methylomes of the Pacific oyster Crassostrea gigas provide insight into the evolution of invertebrate CpG methylation [J].BMC Genomics, 2014, 15(1): 1119.
[23]Qiu C, Sawada K, Zhang X, et al. The PWWP domain of mammalian DNA methyltransferaseDnmt3bdefines a new family of DNA-binding folds [J].Nature Structural & Molecular Biology, 2002, 9(3): 217-224.
[24]邓陈茂, 童银洪, 符韶. 马氏珠母贝的研究进展[J].现代农业科技, 2009,2: 204-210.
[25]Miyamoto H, Miyoshi F, Kohno J. The carbonic anhydrase domain protein nacrein is expressed in the epithelial cells of the mantle and acts as a negative regulator in calcification in the mollusc Pinctada fucata [J].Zoological Science, 2005, 22(3): 311-315.
(编校:李由明)
Effect of 5-azacytidine on Early Development ofPinctadafucata
LI Yao-guo, SUN Tong, ZHANG Ya-jian, GONG Jian-hao
(College of Life Sciences and Ecology, Hainan Tropical Ocean University, Sanya Hainan 572022,China)
To investigate the effect of 5-azacytidine on early development process of Pinctada fucata (P.fucata), genome methylation level change and expression level change of biomineralization gene were analyzed. The results showed that the treatment of 5-azacytidine resulted in a significantly decrease of the genome methylation levels of early embryos. The expression levels ofDNMT3 in the group after treatment of 5-azacytidine were lower than those in the group treated with seawater, whilst the expressions ofnacreingene were higher than those in the seawater treatment group. A significant positive correlation was observed between the expressions ofDNMT3 and nacrein genes (R2=0.755,P=0.000). The change of genome methylation level may be a key factor involved in the regulation of biomineralization process.
Pinctadafucata; demethylation agent; early development; methylation level; biomineralization
2016-10-05
海南自然科学基金面上项目(20164166);海南热带海洋学院学科带头人和博士科研启动基金项目(RHDXB201620)
李耀国(1986-),男,湖南湘乡人,海南热带海洋学院生命科学与生态学院讲师,博士,研究方向为水产动物分子育种与资源保护.
S91
A
1008-6722(2016) 05-0005-06
10.13307/j.issn.1008-6722.2016.05.02

