基于切刻内切酶信号放大的电致化学发光DNA生物传感器的研究
海洪 黄文刚 梁顺超 李建平
摘要:利用切刻内切酶的酶切作用实现信号放大,结合量子点高效的电化学发光性能,构建了一种新型电化学发光DNA生物传感器。将捕获探针DNA(cDNA)通过自组装的方式固定到金电极表面,后与目标DNA(tDNA)互补杂交形成双链DNA,利用切刻内切酶Nt.BstNBI特异性识别双链上的酶切位点(5′GAGTC3′),然后在cDNA相应的切割位点(识别序列3′端后的4个碱基处)对其进行剪切,释放出目标链,参与下一轮的杂交及酶切,通过目标物的循环利用,实现信号放大。利用N羟基琥珀酰亚胺(NHS)和1乙基33二甲基氨丙基碳化二亚胺(EDC)活化羧基化CdTe量子点表面的羧基,与电极表面残留的cDNA末端的氨基共价交联,通过测定捕获的量子点的电化学发光信号对目标DNA 进行检测。优化后的检测条件为:cDNA浓度1 μmol/L, 杂交时间60 min,Nt.BstNBI浓度0.5 U/μL,酶切反应时间4 h。在优化条件下,目标DNA浓度在2.0 × 10mol/L范围内,其对数与电化学发光强度呈线性关系,检出限为7.3×10 mol/L。人体血样加标回收率为96.4%~108.0%。
关键词 :DNA生物传感器;切刻内切酶;量子点;电致化学发光;信号放大
1 引 言
近年来,随着生命科学的不断发展,低浓度特定DNA序列的超灵敏检测在临床诊断、基因突变检测等生物学研究中显示出越来越重要的作用和意义[1~4]。与荧光法[5]、石英晶体微天平法[6]、比色法[7]等众多的DNA检测方法相比,电化学发光法因其方法简单、响应速度快、灵敏度高和选择性好而得到了广泛应用[8~10]。
目标放大和信号放大是提高DNA检测灵敏度的两种常用方法。传统的目标放大方法如聚合酶链式反应(PCR)[11]、依赖核酸序列的扩增技术(NASBA)[12]等因高的放大效率、灵敏的检测效果而被广泛应用[13]。然而这些方法一般都需要特殊的设备或昂贵的检测仪器,存在耗时、易污染、成本高、操作不便等缺点[14]。相比之下,通过目标物提高检测灵敏度的信号放大技术,快速、简便,已成为超灵敏检测DNA的研究热点之一[15~18]。切刻内切酶信号放大是近年来逐渐采用的低浓度核酸的检测方法[19]。探针DNA与目标DNA结合成双链时,形成切刻内切酶的识别位点,内切酶对探针DNA进行剪切,使得目标DNA被释放出来,并进行循环利用,实现检测信号的放大[20~24]。
电化学发光法不仅具有化学发光分析的灵敏度高、线性范围宽和仪器简单等优点,而且电化学分析控制性强,选择性好[25]。量子点作为一种新兴的纳米材料,因其独特的光学性质,如高稳定性、不易分解、荧光寿命长等[26],已被作为生物传感器的标记物和信号探针[27]。近年来,利用量子点高效及稳定的电化学发光性能研制的电化学发光生物传感器备受关注[28~31]。然而,将量子点优异的电化学发光性能与切刻内切酶的特异性相结合,进行目标物测定的研究至今未见报道。
本研究基于切刻内切酶的信号放大和量子点高效的电化学发光性能,建立了超灵敏检测DNA的方法。通过自组装方式将捕获DNA(cDNA)固定在电極表面,靶DAN分子(tDNA)与其特异性杂交形成双链,利用切刻内切酶对形成的双链中的cDNA进行识别和切割,释放出tDNA序列,参与下一轮的杂交和酶切。本传感器制作简单,灵敏度高,选择性好,具有良好的应用前景。
2 实验部分
2.1 仪器与试剂
MPIE 型电致化学发光分析系统(西安瑞迈分析仪器有限公司),采用三电极系统:工作电极为金电极(直径2 mm),参比电极为Ag/AgCl(饱和KCl)电极,对电极为铂丝电极。PGSTAT128N Autolab 电化学工作站(瑞士万通有限公司);UV2400PC紫外可见分光光度计(日本岛津公司);F7000 荧光仪(日本日立公司)。
氯化镉(CdCl2·2.5H2O)、碲粉(Te)、硼氢化钠(NaBH4)、巯基乙酸(TGA)、巯基乙醇(MCH)、N羟基琥珀酰亚胺(NHS)、1乙基33二甲基氨丙基碳化二亚胺(EDC)购于上海Aladdin公司;切刻内切酶Nt.BstNBI(New England Biolabs有限公司);其它试剂均为分析纯,实验用水为二次蒸馏水。DNA序列均购自于上海生工生物工程技术服务有限公司,使用时用TE缓冲液(10 mmol/L TrisHCl, 1 mmol/L EDTA, 100 mmol/L NaCl, pH 8.0)进行配制和稀释,碱基序列如表1所示。
2.2 水溶性量子点的制备
CdTe水溶性量子点合成方式参考文献[32]的方法并稍做改进。将250 mL 0.0025 mol/L CdCl2溶液倒入三口烧瓶中,磁力搅拌,通氮气除氧15 min,接着加入100 μL TGA,并用NaOH调节至pH≈10,继续通氮气10 min,封口。
量取3 mL二次蒸馏水倒入另一个50 mL 三口烧瓶中,同样通氮气除氧。称取0.36 g NaBH4和0.144 g Te粉加入水中,在65℃水浴和磁力搅拌下反应,直到黑色Te粉完全消失,得到紫色透明的NaHTe溶液。将得到的溶液倒入上述含有CdCl2溶液的三口烧瓶中,95℃水浴中搅拌回流2 h,得颜色透明的CdTe水溶性量子点。
2.3 电化学发光DNA生物传感器的制备
将金电极用0.05 μm Al2O3抛光至镜面,分别置于50% (V/V) HNO3、无水乙醇、水中超声清洗后,浸入含有1 μmol/L cDNA溶液中过夜,cDNA通过SAu键自组装在电极表面。将修饰电极浸入到巯基乙醇溶液中,避光孵育1 h,封闭其非特异性结合位点,分别用0.1 mol/L PBS缓冲液和0.5 mol/L NaCl溶液清洗,制得电化学发光DNA生物传感器。
3 结果与讨论
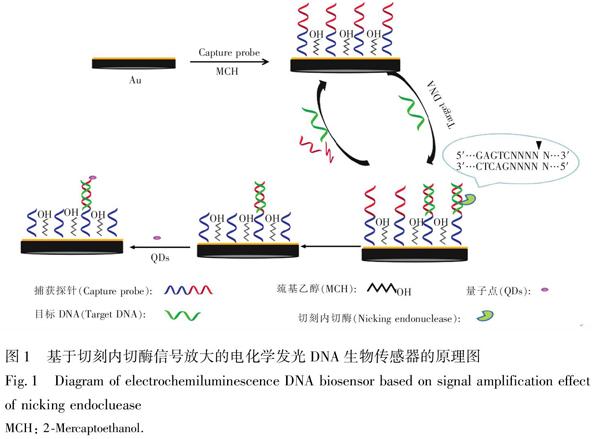
3.1 电化学发光DNA生物传感器的检测原理
如图1所示,巯基化的捕获探针cDNA通过自组装方式固定在金电极表面,tDNA与其杂交形成双链,利用切刻内切酶特异性识别酶切位点(5′GAGTC3′),在其相应的切割位点(识别序列3′末端后的4个碱基处)对cDNA进行剪切,同时也破坏了双链DNA结构,tDNA被释放出来[4,17],参与下一轮的杂交和酶切,从而循环利用目标物。采用EDC/NHS活化带有羧基的TGACdTe量子点,通过缩合反应标记在末端氨基化的cDNA链上,最后,在共反应剂K2S2O8存在下,通过测定量子点的电化学发光信号,实现DNA的超痕量检测。
3.2 量子点的表征
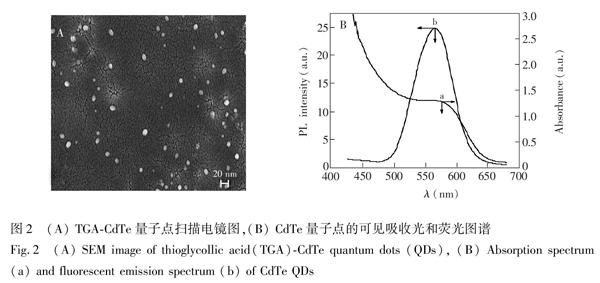
由制备的CdTe量子点的扫描电镜图(图2A)可见,量子点均匀分散,粒径约为10 nm。图2B为制备的CdTe量子点的吸收曲线和荧光曲线,量子点最大吸收波长为580 nm,当激发波长为355 nm时,量子点的最大发射波长为566 nm,表明所制備的量子点具有一定荧光性质。
3.6 实验条件优化
cDNA自组装在电极表面的量直接影响测定的灵敏度。按照2.3节步骤,在一定浓度的tDNA及内切酶作用下,考察了cDNA 浓度对实验的影响。从图6a可见,随着cDNA 浓度增大,ECL信号明显增强,大约1 μmol/L时达到最大值;浓度继续增加,信号缓慢下降。这可能是由于电极表面探针过多,密度过大,电极表面空间位阻增大[34],阻碍了量子点与cDNA缩合反应,使得ECL信号减弱;同时也影响杂交效率,不利于酶的作用。因此cDNA最佳浓度选用1 μmol/L。
DNA的杂交时间的影响如图6b所示。随着cDNA与tDNA杂交时间延长,ECL信号逐渐减弱并趋向于稳定,60 min后基本稳定。因此,本实验杂交时间选择60 min。
切刻内切酶是本实验中一个重要的影响因素,因此对酶浓度和酶切反应时间进行了优化。结果表明,在0.2~0.7 U/μL范围内,随着酶浓度增加,ECL信号逐渐减弱,在0.5 U/μL后信号基本稳定,故后续实验选择酶浓度为0.5 U/μL。在1~6 h的酶反应时间内,随着反应时间延长,ECL信号逐渐降低,4 h后,ECL信号达到基本稳定。因此,选择酶的最佳反应时间为4 h。
4 结 论
在本研究中,基于切刻内切酶进行目标物循环利用放大检测信号,结合CdTe量子点良好的电化学发光性能,成功制备了一种超灵敏检测DNA的新型生物传感器,对DNA具有较低的检出限、良好的重现性和稳定性,可应用于人血中核酸的灵敏检测。
References
1 Debouck C, Goodfellow P N. Nat. Genet., 1999, 21: 48-50
2 Hacia J G, Brody L C, Collins F S. Mol. Psychiatry, 1998, 3(6): 483-492
3 Wang H, Yeh Y S, Barbara P F. J. Am. Chem. Soc., 2009, 131(42): 15534-15543
4 Chen J H, Zhang J, Li J, Fu F F, Yang H H, Chen G N. Chem. Commun., 2010, 46(32): 5939-5941
5 SONG LuNa, ZHANG YongHua, BO HongYan, GAO Qiang. Chinese J. Anal. Chem., 2015, 43(9): 1402-1407
宋璐娜, 张永花, 薄红艳, 高 强. 分析化学, 2015, 43(9): 1402-1407
6 Wang D Z, Chen G J, Wang H M, Tang W, Pan W, Li N, Liu F. Biosens. Bioelectron., 2013, 48: 276-280
7 Ma C P, Wang W S, Mulchandani A, Shi C. Anal. Biochem., 2014, 457: 19-23
8 Jie G F, Qin Y Q, Meng Q M, Meng Q, Wang J L. Analyst, 2015, 140(1): 79-82
9 Lou J, Wang Z Y, Wang X, Bao J C, Tu W W, Dai Z H. Chem. Commun., 2015, 51(78): 14578-14581
10 Xiong H T, Zheng X W. Analyst, 2014, 139(7): 1732-1739
11 Mullis K B, Faloona F A, Scharf S J, Saiki R K, Horn G T, Erlich H. Biotech. Ser., 1992, 17-27
12 Compton J. Nature, 1991, 350(6313): 91-92
13 Lie Y S, Petropoulos C J. Curr. Opin. Biotech., 1998, 9(1): 43-48
14 Wang K, Tang Z, Yang C J, Kim Y, Fang X, Li W, WuY R, Medley C D, Cao Z H, Li J, Colon P, Lin H, Tan W H. Angew. Chem. Int. Edit., 2009, 48(5): 856-870
15 Hu Y H, Xu X Q, Liu Q H, Wang L, Lin Z Y, Chen G N. Anal. Chem., 2014, 86(17): 8785-8790
16 Lin C S, Chen Y Y, Cai Z X, Luo F, Wang Y R, Chen X. Electrochim. Acta, 2014, 147: 785-790
17 Chen J H, Zhang J, Guo Y, Li J, Fu F F, Yang H H, Chen G N. Chem. Commun., 2011, 47(28): 8004-8006
18 Zhou F L, Li B X. Anal. Chem., 2015, 87(14): 7156-7162
19 Kiesling T, Cox K, Davidson E A, Dretchen K, Grater G, Hibbard S, Lasken R S, Leshin J, Skowronski E, Danielsen M. Nucleic Acids Res., 2007, 35(18): e117
20 Zou B J, Cao X M, Wu H P, Song Q X, Wang J P, Kajiyama T, Kambara H, Zhou G H. Biosens. Bioelectron., 2015, 66: 50-54
21 Zhang K, Wang K, Zhu X, Zhang J, Xu L, Huang B, Xie M. Chem. Commun., 2014, 50(2): 180-182
22 Xu W, Xue X J, Li T H, Zeng H Q, Liu X G. Angew. Chem. Int. Edit., 2009, 48(37): 6849-6852
23 Luo F K, Xiang G M, Pu X Y, Yu J C, Chen M, Chen G H. Sensors, 2015, 15(2): 2629-2643
24 Li N, Gao Z F, Kang B H, Li N B, Luo H Q. RSC Adv., 2015, 5(26): 20020-20024
25 Blackburn G F, Shah H P, Kenten J H, Leland J, Kamin R A, Link J, Peterman J, Powell M J, Shah A, Talley D B. Clin. Chem., 1991, 37(9): 1534-1539
26 Michalet X, Pinaud F F, Bentolila L A, Tsay J M, Doose S, Li J J, Sundaresan G, Wu A M, Gambhir S S, Weiss S. Science, 2005, 307(5709): 538-544
27 Huang H P, Zhu J J. Biosens. Bioelectron., 2009, 25(4): 927-930
28 Hai H, Yang F, Li J P. RSC Adv., 2013, 3(32): 13144
29 Hai H, Yang F, Li J P. Microchimica Acta, 2014, 181: 893-901
30 WEI XiaoPing, YANG Feng, DING Fan, LI JianPing. Chinese J. Anal. Chem., 2014, 42(7): 942-947
魏小平, 楊 峰, 丁 璠, 李建平. 分析化学, 2014, 42(7): 942-947
31 HAI Hong, YANG Feng, LI JianPing. Chinese J. Anal. Chem., 2012, 40(6): 841-846
海 洪, 杨 峰, 李建平. 分析化学, 2012, 40(6): 841-846
32 Yu W W, Qu L H, Guo W Z, Peng X G. Chem. Mater., 2003, 15(14): 2854-2860
33 Jie G F, Liu B, Pan H C, Zhu J J, Chen H Y. Anal. Chem., 2007, 79(15): 5574-5581
34 Herne T M, Tarlov M J. J. Am. Chem. Soc., 1997, 119(38): 8916-8920
35 Hu R, Liu T, Zhang X B, Huan S Y, Wu C C,Fu T, Tan W H. Anal. Chem., 2014, 86(10): 5009-5016
36 Li F, Yu Y Q, Li Q, Zhou M, Cui H. Anal. Chem., 2014, 86(3): 1608-1613
37 Liu P, Yang X H, Sun S, Wang Q, Wang K M, Huang J, Liu J B, He L L. Anal. Chem., 2013, 85(16): 7689-7695
38 Cai H, Wang Y P, He P G, Fang Y Z. Anal. Chim. Acta, 2002, 469(2): 165-172
Abstract A novel electrochemiluminescence (ECL) DNA biosensor based on nicking endonuclease and the efficient ECL property of quantum dots(QDs) assisted signal amplification was developed. The capture probe DNA(cDNA) was selfassembled on gold electrode via AuS bond, and then hybridized with target DNA (tDNA) to form doublestranded DNA. Then nicking endonuclease Nt.BstNBI recognized a cutting site (5′GAGTC3′) in the double chain and cleaved cDNA strand at 4 bases away from the 3′ end of its recognition site, thus releasing the tDNA and achieving a recycle of tDNA in the next hybridization and signal amplification. Finally, carboxyl groups on the surface of CdTe QDs were activated with 1ethyl3(3dimethylaminopropyl)carbodiimide (EDC) and Nhydroxysuccinimide (NHS), and then reacted with amino groups at the terminal of residual cDNA on the electrode surface. So the QDs were fabricated and the tDNA concentration could be determined by measuring the ECL signal of the CdTe QDs. The experimental conditions were optimized and 1 μmol/L cDNA, 60 min of hybridization time, 0.5 U/μL Nt.BstNBI and 4 h of endonuclease reaction time were chosen. The experimental results showed that under optimal conditions, tDNA could be specifically assayed with a linear relationship between the ECL signal intensity and the logarithm of tDNA concentration in the range of 2.0 × 10 mol/L. The biosensor was successfully applied to determine tDNA concentration in human blood sample with recoveries of 96.4%108.0%.
Keywords DNA biosensor; Nicking endonuclease; Quantum dots; Electrochemiluminescence; Signal amplification

