白条天牛DNA条形码鉴定技术
禹海鑫,徐 梅,徐 宁,孙民琴,安榆林
(1.南通出入境检验检疫局,江苏 南通 226004; 2.江苏出入境检验检疫局,江苏 南京 210009)
白条天牛DNA条形码鉴定技术
禹海鑫1,徐 梅2,徐 宁1,孙民琴1,安榆林2
(1.南通出入境检验检疫局,江苏 南通 226004; 2.江苏出入境检验检疫局,江苏 南京 210009)
白条天牛属(鞘翅目:天牛科)昆虫是口岸植物检疫工作中多次截获的类群,为探讨线粒体细胞色素C氧化酶亚基I(mtDNA COI)基因的特定区段作为DNA条形码鉴定白条天牛种类的可行性,尝试应用该技术对国内外13种白条天牛进行分子鉴定,测定各种类mtDNA COI基因序列(其中10种检疫性白条天牛COI基因序列为国内外首次测序)并进行比对,采用MEGA 5.05构建系统进化树。结果表明:基于mtDNA COI基因的DNA条形码技术可实现对白条天牛种类快速、准确的鉴定。
白条天牛;COI基因;DNA条形码;鉴定
白条天牛属(Batocera)昆虫属于鞘翅目(Cleoptera)天牛科(Cerambycidae)沟胫天牛亚科(Laniinae)白条天牛族(Batocerini),是重要的林木害虫[1-2]。该属昆虫在全世界共有60余种,广泛分布于亚洲、非洲、澳大利亚及东欧部分地区。我国分布有云斑天牛(Batocerahorsfieldi(Hope))、橙斑白条天牛(BatoceradavidisDeyrolle)等12个种类[2-3]。除此12种外,其余种类在我国都没有发生。白条天牛(非中国种)均被列为我国检疫性昆虫[4]。此外,该属天牛全是林木业重要害虫,以幼虫在树干及枝条的木质部、韧皮部钻蛀取食,可为害柳、杨、梨、栎、女贞等多种阔叶树种[1-2]。
近年来,随着我国外向型经济的发展,木材及木包装的进出口量剧增,国内各口岸在进境木材检疫工作中也多次截获白条天牛。例如,2009年嘉兴乍浦口岸在马来西亚进口木材中首次截获托氏白条天牛(Batocerathomsoni)[5]。2012年张家港口岸在进境巴布亚新几内亚原木中截获到白条天牛非中国种(Batoceralaena)[6]。另据媒体报道,2011年和2014年,江苏吴江口岸先后在进境原木中截获了维多利亚白条天牛(Batoceravictoriana)和婆罗白条天牛(Batoceratigris)。此外,2013年张家港口岸又从来自非洲的原木中截获到一种白条天牛(Batocerawyllei)。同年,东莞口岸在菲律宾黄蕊木中截获到另一种白条天牛(Batocerasp.(non-Chinese))。
截至目前,昆虫种类鉴定主要以形态学鉴定为主,形态学鉴定往往要求虫体完整无残缺。而通常口岸实际截获到白条天牛的卵、幼虫和蛹或是肢体残破的成虫,传统的形态学鉴定方法显然并不适用[7]。因此,需要寻找新的方法来解决传统形态学鉴定中遇到的难题。基于线粒体细胞色素C氧化酶亚基I基因(mtDNA COI)的DNA条形码技术是当前比较成熟的分子鉴定技术[8]。Stauffer等[9]利用该技术,快速鉴定了欧洲7种齿小蠹。常虹等[10]应用该技术对齿小蠹属不同种类进行高效鉴定。花婧等[11]通过该技术对大小蠹属17个种类进行分类鉴定。
本研究对白条天牛属下多个种类的COI片段进行测序和对比,分析这些DNA条形码序列特征以及相互间的遗传进化关系,以找到一种能够准确高效鉴定白条天牛种类的分子方法,为研究天牛科其它天牛种类的分子鉴定方法提供参考[10]。
1 材料与方法
1.1 实验标本
本实验所用的天牛标本均由江苏出入境检验检疫局动植物与食品检测中心提供,所有标本均由国内权威天牛专家安榆林研究员鉴定,包括10种白条天牛(表1),其中8种COI序列为国内外首次测得。此外,从GenBank网站上下载了3种白条天牛的COI序列用于对比(表2)。
1.2 提取样品基因组DNA
利用GenMagBio动物细胞组织/细胞基因组DNA磁珠提取试剂盒(北京金麦格公司)提取上述样品的基因组DNA。提取方法为先用双蒸水冲洗干净用酒精浸泡过的虫体肌肉组织(少于30 mg),然后装入1.5 mL离心管并置于MM400球磨仪中震荡研磨(30次·s-1)30 s,再将磨碎组织离心10 min(12000 r·min-1)后,加入180 mL裂解缓冲液和20 mL的Proteinase K,放入55 ℃ 水中温浴10 min,以使样品组织充分裂解。加200 mL无水酒精、200 mL的缓冲液和20 mL磁珠,磁珠用来吸附基因组DNA。加入500 mL Wash Buffer去杂后,再加上20 μL Elution Buffer洗脱磁珠即得到基因组 DNA 溶液[10,12]。
1.3 COI片段扩增和测序
PCR扩增反应采用25 μL反应体系,包含DNA模板1 μL,MgCl2(25 mmol·L-1)2 μL,10×buffer 2.5 μL,d NTPs (2.5 mmol·L-1) 1 μL,rTaq DNA聚合酶(5 U·μL-1) 0.4 μL(TaKaRa公司),上、下游引物(10 μmol·L-1)各0.5 μL(南京金斯瑞合成),再加灭菌水至总体积25 μL。PCR扩增反应的条件为:94 ℃预变性持续5 min,94 ℃下变性持续40 s,55 ℃退火温度保持30 s,72 ℃下延长至1 min,设置32次循环,最后一步72 ℃下再延伸10 min即可,最后将PCR产物送至南京金斯瑞公司测序[12-13]。本实验PCR扩增采用巢氏PCR,第1轮PCR反应引物为J171和J131,第2轮PCR引物为J178和N211[14](表3)。采用巢式PCR扩增,既减少了扩出非目的基因条带的可能性,又增加了PCR检测的灵敏性和准确性[12,14]。
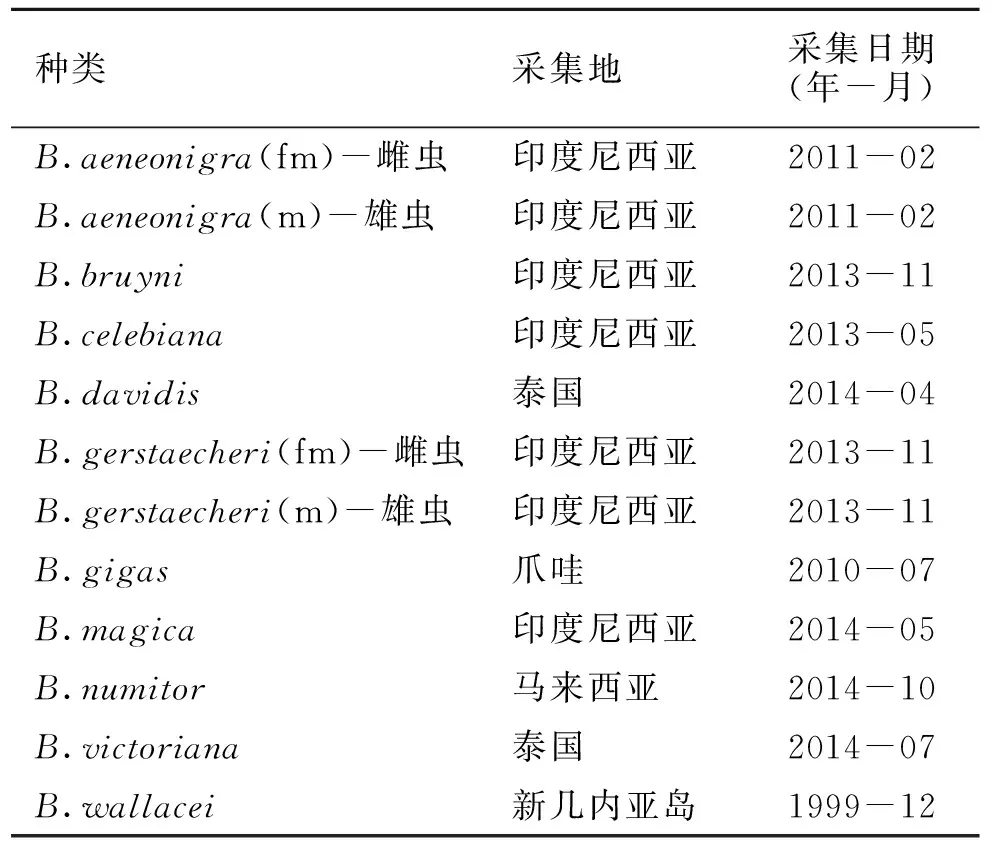
表1 标本采集地和采集日期

表2 GenBank下载的COI基因序列信息

表3 所用引物序列
1.4 序列分析
将上述测得序列导入SeqMan分析软件中[15]进行拼接和校正,并使用NCBI的“BLAST”工具确认序列的方向及可信度。然后将所测序列与GenBank上下载的白条天牛序列一同输入Clustal X 1.83软件[16]进行对比,再将序列比对结果导入Mega 5.05[17]中,算出各白条天牛种类间的转换和颠换及比值(R)、变异位点(V)和保守位点(C)等值[10,13],最后利用Mega 5.05构建系统发育树。
2 结果与分析
2.1 DNA凝胶电泳结果
本实验对12个(10种)白条天牛昆虫样本基因组DNA进行巢氏PCR扩增,电泳结果显示12个样品均在525 bp处有清晰明亮特异性好的条带,可满足后续的基因测序需要(图1)。
2.2 白条天牛DNA条形码分析
2.2.1 COI序列特征分析 将上述各条序列输入Mega 5.05后,均切为同等长度片段(434 bp)[14]。结果显示,共有保守位点、变异位点、自裔位点、简约信息位点各114、320、18、302个。此外,统计各位点的碱基平均含量发现,A为28.4%,C为16.4%,G为25.7%,T为29.5%[10,12-13]。其中,A与T含量接近,且A+T含量为57.9%,要高于G+C含量(42.1%),这也与昆虫线粒体基因各碱基组成规律相吻合[14,18]。
2.2.2 碱基替换规律 用Mega 5.05分析各位点的碱基替换规律[19]。结果发现,位点转换主要出现在A与G间,颠换主要出现在T与A间,R值为0.75。对密码子各位点研究发现,在第3位点上容易发生转换与颠换,而且转换与颠换值比较接近(R=0.80)[10]。此外,第1、2位点的R值分别为0.66、0.80(表4)。
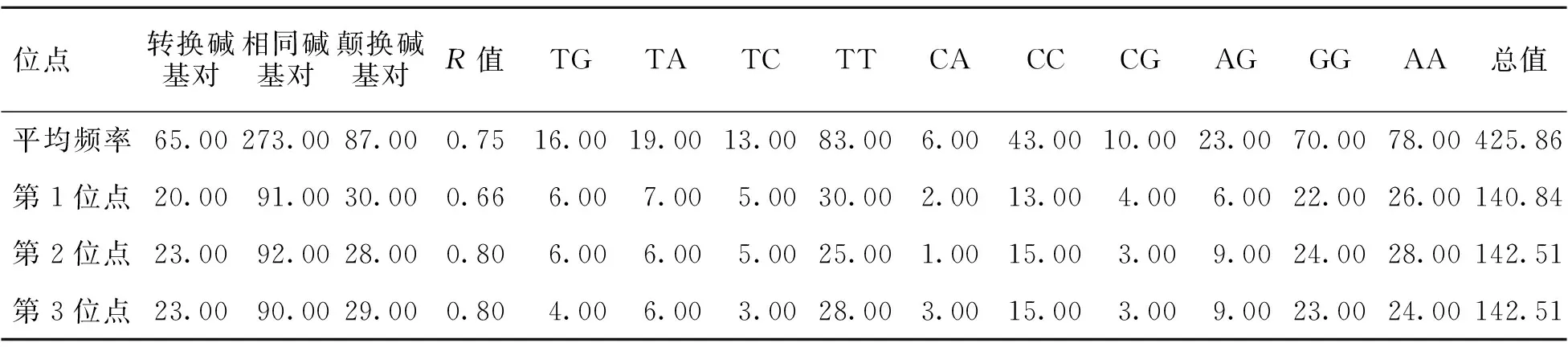
表4 核甘酸碱基替换值
2.2.3 遗传距离 据Kimure 2-parameter模型计算13种白条天牛样品遗传距离,利用Bootstrap值(1000次)检验[20]。结果显示,相同物种内的遗传距离数值在0.000~0.049内,其中B.lineolata距离最小,为0;B.horsfieldi距离最大,为0.049;而同一物种雌雄个体内的遗传距离则介于0.007~0.049之间,其中B.aeneonigra距离较小,为0.007;B.gerstaecheri距离较大,为0.026。不同种间的遗传距离介于0.002~1.423之间,平均遗传距离为0.733,其中B.gerstaecheri(m)与B.numitor之间遗传距离最小,为0.002;B.gerstaecheri(fm)与B.lineolata之间遗传距离最大,为1.423(表5)。此结果表明,白条天牛属下同一物种及同物种雌雄个体之间遗传距离稍小;而多数同属不同种类的遗传差距较大,呈现出了较为显著的遗传差异性。

表5 基于Kimure 2-parameter模型白条天牛属种内种间遗传距离
*:1为B.rubus;2为B.aeneonigra(fm);3为B.aeneonigra(m);4为B.bruyni;5为B.celebiana;6为B.davidis;7为B.gerstaecheri(fm);8为B.gerstaecheri(m);9为B.gigas;10为B.magica;11为B.numitor;12为B.victoriana;13为B.wallacei;14为B.horsfieldi1;15为B.horsfieldi2;16为B.lineolata1;17为B.lineolata2。
2.2.4 构建系统发育树 利用MEGA 5.05软件构建分子系统发育树(图2)。结果表明,B.aneonigra(fm)与B.aneonigra(m)聚为一支,说明B.aneonigra雌雄虫个体之间的亲缘关系最近,这也与形态学鉴定结果一致。B.aneonigra又与B.gerstaecheri(fm)、B.gerstaecheri(m)、B.victoriana、B.numitor、B.brutyni和B.magica聚在一起,说明它们之间的系统进化距离较近。B.celebiana与B.gigas聚为同一小支后又与B.wallace聚为一支,说明此三者间的亲缘关系较近。整体来看,上述9种(11头)天牛又同聚为一大支,除B.numitor外,余下8种都属于我国未有分布的白条天牛属(非中国种)种类。此外,在我国已有分布的白条天牛属4个种类的天牛(6头):B.rubus、B.horsfieldi、B.davidis和B.lineolata也聚为一大支,其中相同种类的白条天牛个体分别聚为一小支。上述结果表明,非中国种与多数中国种的白条天牛种类的亲缘关系较远,且不同种类的白条天牛间遗传差距较大,因此该段COI基因适合作DNA条形码,用于上述白条天牛的分子鉴定。
3 小结与讨论
近年来,DNA条形码技术在昆虫种类检疫鉴定中得到越来越广泛的运用。其中,线粒体COI基因作为一种条形码基因,其所含的遗传信息既相对保守又有足够的变异,已广泛运用于昆虫种类鉴别[21]。例如,黄丽莉等[22]利用基于线粒体COI基因的DNA条形码技术实现了6个不同地理种群的茶黄蓟马与其他4种常见蓟马的快速种类鉴定。肖永刚等[23]利用基于线粒体COI基因的DNA条形码技术完成了17种菱蜡蝉亚科的昆虫种类鉴定。另外,李京[24]通过研究天牛科66种天牛的线粒体基因序列特征,初步弄清楚了其各类群之间的亲缘关系。郑丝竹[25]对天牛科5个亚科160种天牛进行了基于COI基因序列的DNA条形码数据库的构建,并据此初步建立天牛快速可靠分子鉴定技术体系。
近年来,我国口岸截获的天牛科昆虫多以卵、幼虫及蛹虫态和肢体残缺成虫形态出现,用传统的形态鉴定方法存在着极大的困难。基于线粒体COI基因的DNA条形码技术具有准确、高效的特点,在昆虫种类鉴定中得到越来越广泛的应用,很大程度上克服了传统形态学鉴定的缺陷[26]。但是目前该技术在白条天牛属昆虫种类鉴定中的应用鲜有报道。本研究通过将最新测序的10种白条天牛COI基因与GenBank下载的白条天牛COI基因序列进行比对研究,证明了DNA条形码技术适于白条天牛属各种类的鉴定及部分非中国种与中国种类的区分鉴别。另外,本研究在国内外首次对包括B.aneonigra,B.gerstaecheri等在内的8种检疫性白条天牛COI基因序列进行测序,这不仅为白条天牛属昆虫DNA条形码数据库进行了有益的补充完善,也为下一步将DNA条形码技术广泛应用于口岸检疫性天牛的分子鉴定工作提供参考。
[1]刘莹,熊赛,任杰群,等.中国白条天牛属比较形态学研究(鞘翅目,天牛科,沟胫天牛亚科,白条天牛族)[J].动物分类学报,2012,37(4):701-711.
[2]陈乃中.中国进境植物检疫性有害生物:昆虫卷[M].北京:中国农业出版社,2009:186-194.
[3]刘莹.中国白条天牛族分类及比较形态学研究[D].重庆:西南大学,2013:1-6.
[4]农业部.中华人民共和国进境植物检疫性有害生物名录(中华人民共和国农业部公告第862号)[EB/OL].[2011-12-06].http://www.moa.gov.cn/sjzz/zzj/zcfgzzj·g-1fxwj/201112/t20111206_2424113.htm.
[5]张红英,王学,王凯,等.马来西亚原木上截获的托氏白条天牛[J].植物检疫,2009,23(3):32-34.
[6]郑庆伟.江苏口岸在国内首次截获一种检疫性白条天牛[J].农药市场信息,2012(29):1-3.
[7]刘勇,宋毓,李晓宇.基于线粒体COI基因的DNA条形码技术在昆虫分子鉴定中的应用[J].植物检疫,2010(2):46-47.
[8]Hebert P D,Cywinska A,Ball S L,et al.Biological identifications through DNA barcodes[J].Proceedings of the Royal Society B:Biological Sciences,2003,270(1512):313-321.
[9]Stauffer C.A molecular method for differentiating sibling species within the genus Ips[C]∥Gregoire JC,Liebhold AM,Stephen FM,et al.Integrating Cultural Tactics into the Management of Bark Beetle and Reforestation Pests.USDA Forest Service General Technical Report,NE-236,Radnor,PA.1997:87-91.
[10]常虹,郝德君,肖荣堂,等.基于线粒体COI基因的齿小蠹属昆虫DNA条形码研究[J].昆虫学报,2012,55(9):1075-1081.
[11]花婧,郑斯竹,安榆林,等.基于线粒体COI基因的大小蠹属昆虫DNA条形码研究[J].江苏农业科学,2014,42(3):30-32.
[12]常虹.口岸截获小蠹科昆虫DNA条形码技术研究[D].南京:南京林业大学,2013:10-26.
[13]常虹,郝德君,肖荣堂,等.基于线粒体COI基因齿小蠹属Ips昆虫DNA条形码研究[C]∥江苏省昆虫学会第十三次会员代表大会暨学术研讨会论文摘要集.扬州:2012:21-24.
[14]郑斯竹,张凯,杨晓军,等.墨天牛属昆虫COI基因比较及系统学初步研究[J].应用昆虫学报,2015(2):12.
[15]Parchman T L,Geist S K,Grahnen J A,et al.Transcriptome sequencing in an ecologically important tree species:assembly,annotation,and marker discovery[J].BMC Genomics,2010(11):180.
[16]Chenna R,Sugawara H,Koike T,et al.Multiple sequence alignment with the Clustal series of programs[J].Nucleic Acids Research,2003(31):3497-3500.
[17]Kumar S,Nei M,Dudley J,et al.Mega:abiologist-centric software for evolutionary analysis of DNA and protein sequence[J].Brief Bioinform,2008,9(4):299-306.
[18]Liu H,Beckenbach A T.Evolution of the mitochondrial cytochromeoxidase gene among 10 orders of insects[J].Molecular Phylogenetics and Evolution,1992(1):41-52.
[19]Tamura K,Peterson D,PetersonN,et al.MEGA5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Molecular Biology and Evolution,2011,28(10):2731-2739.
[20]Felsenstein J.Confidence limits on phylogenies:an approach using the bootstrap[J].Evolution,1985(39):783-791.
[21]余道坚,邓中平,陈志舜,等.昆虫分子标记基因和序列及应用[J].植物检疫,2003,17(3):156-159.
[22]黄丽莉,阙海勇,车飞.茶园茶黄蓟马及其近似种的DNA条形码鉴定[J].植物检疫,2014,28(6):68-72.
[23]肖永刚,陈祥盛.基于线粒体COI基因的17种菱蜡蝉亚科DNA条形码研究(半翅目:蜡蝉总科:菱蜡蝉科)[J].山地农业生物学报,2014,33(2):44-50.
[24]李京.天牛科线粒体基因特征与形态发育初步研究[D].郑州:河南农业大学,2013:19-53.
[25]郑丝竹.天牛科基因条形码构建及分子快速鉴定技术研究[D].南京:南京林业大学,2012:35-84.
[26]彭居俐,王绪桢,何舜平.DNA条形码技术的研究进展及其应用[J].水生生物学报,2008,32(6):916-919.
Establishment of DNA Barcode of Species ofBatocera(Coleoptera:Cerambycidae)
YU Hai-xin1,XU Mei2,XU Ning1,SUN Ming-qin1,AN Yu-lin2
(1.NantongEntry-ExitInspectionandQuatantineBureau,Nantong226004,Jiangsu,China;2.JiangsuEntry-ExitInspectionandQuatantineBureau,Nanjing210009,Jiangsu,China)
Species of the genusBatocera(Coleoptera:Cerambycidae) are often intercepted and captured in quarantine at port.To explore the feasibility of identifyingBatoceraspecies rapidly andaccurately by DNA barcode of special sequence in mitochondrial cytochrome c coxidase subunit I (mtDNA COI),we attempted to apply this technology to identify 13Batoceraspecies at home and abroad.The COI sequences of 13 species were sequenced (including 10 species were sequenced in the world for the first time)and compared.The phylogenetic tree was established by MEGA 5.05.The results indicated thatBatoceraspecies can be identified rapidly and accurately through DNA barcode based on mtDNA COI gene.
Batocera;mtDNA COI gene;DNA barcoding;identification
10.13428/j.cnki.fjlk.2016.02.017
2015-08-27;
2015-10-10
江苏出入境检验检疫局科技计划项目(2014KJ48)
禹海鑫(1984—),男,河南泌阳人,南通出入境检验检疫局农艺师,博士,从事昆虫分子及形态鉴定研究。E-mail:haixin.007@163.com。
S763
A
1002-7351(2016)02-0090-05

