纳豆激酶分子结构与潜在应用价值分析
马国兴,潘 峰,王庆波,温春光,吴雅清,许瑞安*
(1.华侨大学 分子药物研究院,福建 厦门 361021;2.分子药物教育部工程研究中心,福建 厦门361021)
纳豆激酶分子结构与潜在应用价值分析
马国兴1,2,潘峰1,2,王庆波1,2,温春光1,2,吴雅清1,2,许瑞安*1,2
(1.华侨大学 分子药物研究院,福建 厦门 361021;2.分子药物教育部工程研究中心,福建 厦门361021)
纳豆激酶因具有较强的溶血能力而著称。目前国内外大量研究人员聚焦于纳豆激酶研究,其开发利用已经获得了长足发展,但同时许多问题与不足也随之暴露出来。一方面是对纳豆激酶的开发利用大多局限于溶栓活性的利用与开发,低估了纳豆激酶应有的整体价值;其次是针对该酶开发和利用的方法过于传统,尚未形成组学大数据和生物信息学技术相结合的探索思维。因此,需要利用更为开阔的研究思路和开发手段来实现纳豆激酶开发利用的最大化。作者从纳豆激酶分子水平和家族分类层次上系统地阐述该酶分子特征,同时对纳豆激酶家族蛋白酶的应用现状进行分析,并对纳豆激酶研发前景进行了展望。
纳豆激酶;保守区域;枯草芽孢杆菌蛋白酶;Peptidase-S8-subtilian-subset蛋白酶家族
Q 815
A
1673—1689(2016)04—0342—08
纳豆激酶(Nattokinase,NK)最早是由日本学者Sumi等[1]从一种发酵食品纳豆中分离得到的,他们通过纤维平板实验发现NK具有优异的溶解纤维平板活性而名声大噪,NK也经常被称为纤溶酶(fibrinolysin)。纳豆是日本的一种传统大豆发酵食品,因此NK被人们喜闻乐道。随后,Sumi等又发起了对NK分子特性的探究,深入研究发现NK是由纳豆发酵中枯草芽孢杆菌分泌的一类丝氨酸蛋白酶(Serine protease)。NK的分子结构、分类和发掘利用广泛引起国内外其他研究人员关注,经过多年的大量研究已经基本摸清了NK的分子特性,生物信息学分析发现NK属于枯草杆菌蛋白(Subtilisin)超家族,并且与 Subtilisin E十分接近都属于Peptidase-S8-subtilian-subset家族。该家族蛋白酶在精细化工,医药和食品行业均表现出非常高的开发利用价值。同时对NK生物信息学分析必将为进一步开发利用NK提供更大的空间。
1 纳豆激酶分子结构
1.1一级结构
1982年Nakamura[2]等通过E.coli宿主-载体系统中克隆出NK基因—aprN,并利用鸟枪法测定了包括调控序列在内的1 473 bp的完整基因序列,使利用生物信息学研究NK基因成为可能。NK基因的起始密码子为GTG,随后的1 143 bp序列是由编码信号肽、前导区域、成熟肽组成的 381个的氨基酸阅读框架[3]。结构基因是TAA、TAA、TAG连续3个终止密码子,该基因终止是以非依赖ρ因子终止方式,在密码子之后一段回文序列可形成茎环结构辅助转录终止。起始密码子的上游-17 bp处为核糖体结合起点AAAGGAG序列,SD序列上游是GC含量低于28%的转录调控区域[4]。成熟纳豆激酶由长为275个氨基酸的单链多肽折叠而成。通过SDS-PAG凝胶电泳和蛋白序列预测分析NK相对分子质量约为28 000[4-5],与Sumi等用Sephadex G-100柱凝胶过滤法测得结果存在一些差异。未经修饰的NK是包含381个 氨基酸残基的蛋白前体。蛋白前体分为3部分:其中从N端第 1-29个氨基酸残基形成信号肽,第30~106个氨基酸残基形成前导区域也称前导肽(pro-peptide)。NK信号肽包含一个带3个正电荷的亲水N端以及一段不带电荷的疏水残基序列,使得NK正常分泌到胞外,其切割位点位于Ala-Glu-Ala保守序列之后。去掉信号肽的部分(含前导肽和成熟区域)为纳豆激酶酶原(pronattokinase,pro-NK),该区域对重组NK意义重大。加工修饰后会切掉N端106个氨基酸基的前导肽,剩余的275个氨基酸残基折叠成具有活性的成熟NK。
1.2保守结构域
NK 具有非常保守的结构域(conserve domains),这对NK的分类具有重要意义,根据保守区域分析NK属于枯草杆菌蛋白酶(Subtilisin)超家族的一类(Peptidases-S8-Sub-tilisin-subset),与该家族蛋白酶类似,成熟NK的活性部位存在保守性,在Asp32、His64和Ser221表现出保守性,底物结合部位保守位点位于Ser125、Leu126、Gly127处,氧离子穴是Asn155[2,6],这些位点均存在进化保守性,可以作为该家族蛋白酶身份识别位点。NK与该家族一些蛋白酶一级结构存在高度相似性,NK与枯草芽孢杆菌168(B.subtilis 168)产生的枯草杆菌蛋白酶E(Subtilisin E)基因序列存在13个碱基不同,在肽链序列组成上仅存在 2个氨基酸残基的差异,序列同源性高达99.5%。相应的,NK与枯草杆菌解淀芽孢杆菌变种 (B.subtilis var.amylosacchariticus)产生的枯草杆菌蛋白酶(subtilisin amylosacchariticus)在核酸序列和肽链序列上仅有27个核苷酸和4个氨基酸的差异,成熟肽链序列同源性为 99.3%。此外,NK与解淀粉芽孢柑橘(Bacillus amyloliquefaciens)产生的枯草杆菌蛋白酶Bpn'(Subtilisin Bpn')和地衣芽孢杆菌(Bacillus hcheniiformis)产生的枯草杆菌蛋白酶 Carlsberg (Subtilisin Carlsberg)的肽链序列同源性也高达86%和72%之多,并且催化中心不变。保守区域和序列信息为NK分类提供提供重要依据。
1.3高级结构
一些研究发现NK在结构上与其家族蛋白酶存在差异,这也是NK被看作成单独的一种蛋白酶重要依据。随着生物信息学深入研究发现NK与Subtilisin E的一级结构具有高度相似性,尤其是存在差异的突变位点的氨基酸对蛋白酶的结构活性基本不会产生什么影响[7]。因此可以推断NK与其他的枯草芽孢杆菌的蛋白折叠与结构相似性极高。Subtilisin E的结构和机理研究比较清楚,其蛋白结构与折叠方式作为NK蛋白结构研究具有非常重要的参考价值[8]。与其他的subtilisins类似,具有高级结构的NK是由一条长的氨基酸序列折叠形成,并且不需要二硫键辅助折叠[2],成熟区域的NK形成特定的空间结构域,包含催化三联体(Asp32,His64,Ser221)和富氧离子穴(N155)[9-10],保守结构位点都位于两个α螺旋和7个β折叠链形成的口袋结构域中。NK的结晶呈针状结晶,X射线晶体衍射分析NK属于C2分子空间群属,单晶格参数a,b,c分别为7.43,4.99,5.63 nm[11]。第一个模拟NK的3D结构[7]是由中国武汉大学病毒学重点实验室使用MODELLER和PROCHECK模拟程序模拟完成。这有助于我们更形象的了解NK结构,空间结构显示保守区域位点能够在碱性环境中稳定的存在于富氧离子穴中。NK是一种钙离子结合蛋白,预测纳豆激酶蛋白高级结构至少结合两个钙离子才能得以维持或者辅助NK形成特有的结构。subtilisin E存在一个与稳定相关的高亲和钙离子结合位点,其他钙离子结合位点则均表现出与折叠相关[12]。钙离子与氨基酸残基共同维持NK催化活性中心结构的准确性。目前对NK的高级结构研究还不是很透彻,但可以确定的是NK的高级结构与其他家族蛋白酶存在差异,并且活性中心保守,从结构角度可以说明了NK家族蛋白酶多样性的可能。
1.4前导肽
NK基因成熟区与信号肽之间存在一段231bp序列,可表达包含77个氨基酸残基在内前导肽。该前导肽区域广泛存在于Subtilisin家族蛋白酶的N端,非常有意思的是这一段氨基酸残基的功能类似于分子伴侣功能(molecular chaperones)。它能辅助蛋白正确折叠功能,并且辅助折叠过程中不需要ATP提供能量,因此该段区域也被称为分子内伴侣(Intramolecular Chaperone)[13]。研究发现前导肽伴随着整个蛋白的折叠过程,并且前导肽对辅助折叠的蛋白还表现出蛋白酶抑制活性,最终该前导肽也像分子伴侣那样经过自身的剪切被去掉,从而最后形成具有活性的蛋白酶,整个pro-subtilisin激活过程可分为3个步骤:(1)subtilisin结构域折叠,(2)前导肽辅助subtilisin自动折叠,(3)subtilisin的激活伴随着前导肽切割[14]。整个的折叠过程如图1所示。
没有前导肽的成熟区折叠形成一个不具活性的形式的熔融球状体结构[15]。在研究sbutilisin E中发现该区域不仅在辅助折叠过程,而且具有对变性蛋白具有很好的复性效果[13]。前导肽与枯草杆菌蛋白酶成熟活性具有密切的关系[14,16],如果该区域发生突变对蛋白的正确折叠会带来毁灭性的影响[17-18]。对前导肽的一些区域进行突变研究,发现许多氨基酸突变位置都对前导肽功能有很大影响,这也说明前导肽对蛋白酶的正确折叠具有至关重要的作用。多数NK编码框中都有前导肽序列,最近也一直争论重组的NK是否将前导肽与成熟区共表达的问题,但是随着对前导肽的深入了解,成熟肽与前导肽共表达的方法更可取。此外,已在枯草杆菌和链激酶中发现一些NK同源基因。
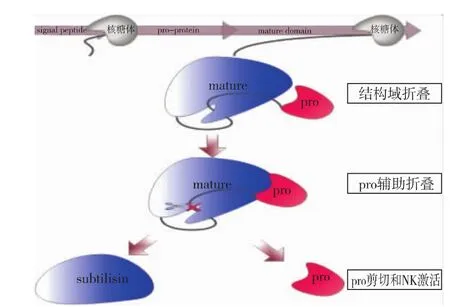
图1 NK正确折叠机制Fig.1 Foldingmechanism of NK
2 纳豆激酶家族
2.1家族分类
早在1967年Markland和Smith等就对枯草芽孢杆菌中提取的Subtilisin进行了氨基酸测序[19],此后,根据序列的保守区域分析该种Subtilisin与NK是同一家族的蛋白酶。近年来通过对NK的分子生物学特性的研究也表明其与大多数Subtilisin具有许多类似的性质,例如,NK在核苷酸序列及氨基酸组成上具有高度同源性,结构和性质上也表现出Subtilisin相似的特征。近年研究发现具有NK溶栓特性的酶不仅仅来源于纳豆发酵菌,海洋中的一些枯草芽孢杆菌也存在大量的溶栓活性酶[20]。
NK与 常 见 的SubtilisinBPN’、Subtilisin Carlsberg、Subtilisin E和Subtilisin Savinase丝氨酸蛋白酶相比较,序列惊人的相似,同源性分别为84.9%、67.8%、98.9%和 62.92%[7]。其中Subtilisin E与纳豆激酶最为相近,序列相似度达到98.9%,保守结构域完全一致,并且高级结构也达到99%的相似度。因此,NK分类可以划分到微生物丝氨酸蛋白酶超家族Peptidases-S8-Subtilisin-subset家族中,其NCBI分类:cd07477。NK与家族分类关系(如图2)。
该家族含有许多种 subtilisins,如 TK-subtilisin,SubtilisinCarlsberg,SubtilisinE和subtilisins BPN’等。其中有一些研究比较透彻的蛋白酶,如 Subtilisin BPN’,Subtilisin Carlsberg等。Subtilisin BPN’早在1965年[21]就被发现并报道,通过氨基酸测序发现 Subtilisin BPN’与 Subtilisin Carlsberg中存在83个氨基酸的差异[22]。Subtilisin BPN’很早就被广泛地应用到工业领域,例如在洗涤剂行业[23]。此家族中还一些极具工业开发利用价值的蛋白酶,例如TK-subtilisin,该酶是从一些极端环境微生物中发现的,具有非常强的热稳定性等特点[24],更适应工业生产粗泛环境。药用研究价值最相关的是Subtilisin E,它与NK结构和活性相似。这些家族蛋白酶开发利用对NK开发具有非常重要的参考价值,近10年来NK除溶栓以外,已逐渐被应用到其他应用领域(见表1)。
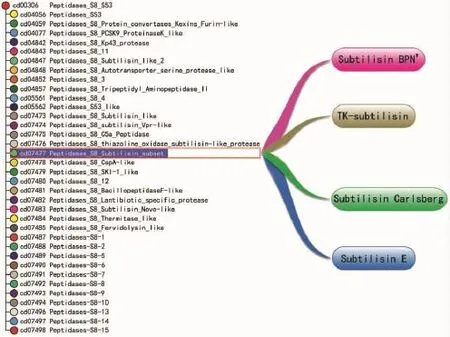
图2 NK家族蛋白酶(cd07477)分类关系简图Fig.2 A taxonom ic chart of the NK proteinase fam ily(cd07477)
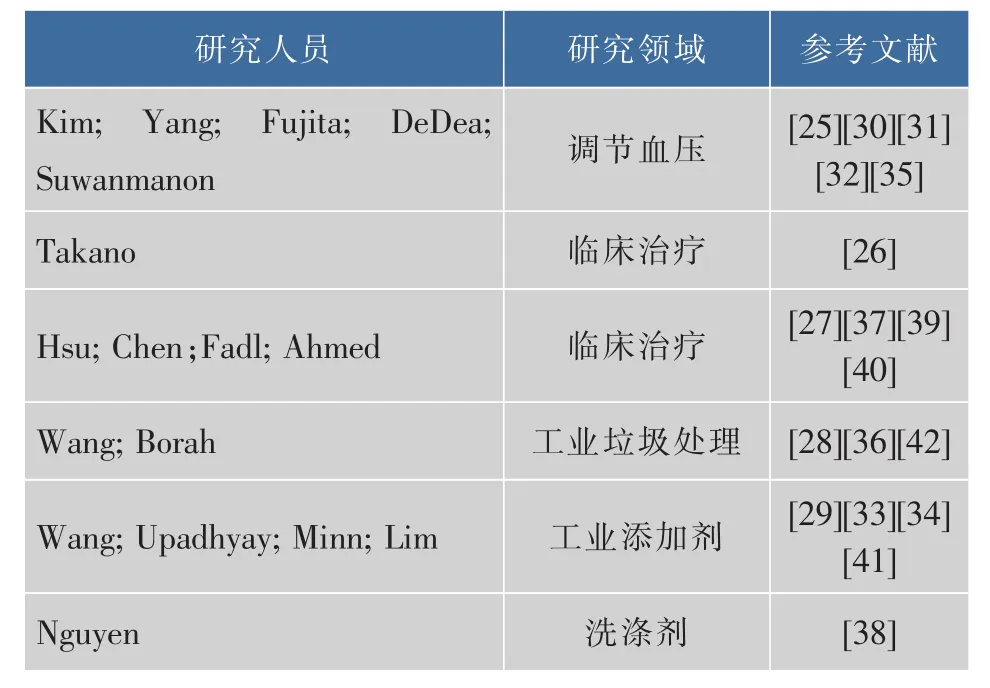
表1 近10年来年对NK非溶栓活性功能开发利用状况Table 1 Development and utilization of NK in the past decade for the non-fibrinolytic properties
2.2多样性分析
随着微生物基因组学研究不断推进和功能蛋白的深入挖掘,发现该蛋白家族存在分布广,性质多样,结构多样等特点,而且均为枯草杆菌样蛋白酶,说明该家族蛋白酶可能在枯草杆菌属基因组中十分丰富,此外,常见的芽孢杆菌属(Bacillus)的基因组也普遍存在该家族蛋白酶基因,例如目前发现的地衣芽孢杆菌、解淀粉芽孢杆菌、缓慢芽孢杆菌、嗜碱芽孢杆菌和南极嗜冷芽孢杆菌等[43]。最近随着越来越多的微生物基因组的测序分析完成,发现该家族蛋白酶不仅仅存在于芽孢杆菌属,在多种细菌,放线菌,真菌中亦有存在,表明该家族酶分布极为广泛。
结构和性质多样性主要是指蛋白分子结构多样性从而体现的性质多样,例如序列长度、蛋白晶体结构等信息。该蛋白家族除了保守区域以外,也发现存在一些其他的氨基酸残基表达序列,这可能为酶分子结构和酶学性质的改变提供了一些线索。首先,酶的相对分子质量有变化,基因组中的一些蛋白酶的预测相对分子质量已经远远超过常见28 000。该家族蛋白酶结构也存在多样性,其中发现的同种TK-subtilisin就存在4种不同的晶体结构,发现有7个可能与钙离子结合区域的不同,造成结构产生差异[12,15]。其次就是酶学性质的改变,例如嗜碱,耐高盐,耐高温,耐低温等,同时还有一些表现出一定的表面活性剂耐受能力[44]。这说明该蛋白酶家族并非是特别严紧的特异性蛋白酶,也说明长期进化可能是该酶家族出现多样性结果重要因素。
3 纳豆激酶家族蛋白酶应用价值分析
3.1皮革行业中的应用
脱毛技术是皮革工业生产中的一个重要环节,是否有效的使用脱毛剂对皮革的生产质量至关重要,碱性丝氨酸蛋白酶(Alkaline Serine Protease)溶解毛羽是一种行之有效的脱毛方法,与传统的皮革脱毛方法相比酶法脱毛不仅可以保护和软化皮革并且环保高效[45]。皮革脱毛技术可选的酶种类比较多,放线菌、细菌、霉菌中均存在脱毛相关蛋白酶[46]。NK家族蛋白酶可以破坏毛发毛根固着结缔组织让毛发松动并脱落,是一种高效脱毛蛋白酶,因此也被应用到皮革脱毛技术中。Bassem等[47]从短芽孢杆菌US575菌株(Brevibacillus brevis US575)中分离得到的29 000角蛋白酶(Peptidases-S8-Subtilisinsubset)对鸡、山羊、兔子、牛、绵羊毛皮均具有良好的脱毛效果。目前,国内也展开了脱毛相关蛋白基因的克隆表达研究,如黄庆等[48]等短小芽孢杆菌(Bacillus pumilus)发现的脱毛蛋白酶也是NK蛋白酶家族的一员。肖怀秋等[49]从芽孢杆菌BX24中获得一种中性蛋白酶,并对酶的性质进行了一系列研究。此外,毛羽处理与环境保护有关,酶法处理毛羽条件温和且不易引起环境污染[50]。因此,表明该家族蛋白酶不仅具有较好的皮革工业应用价值,还具有一定的环境垃圾处理功能[51],但目前仍缺乏NK在皮革行业脱毛工艺应用的相关研究。
3.2洗涤剂行业中的应用
洗涤剂添加酶是一个研究热点,由于目前市场上流行的洗涤剂仍存在诸多缺点,例如不同的表面活性剂的物理作用,无法同时胜任多种顽固污渍的处理。蛋白酶能将一些不易溶于水的大分子蛋白类污渍酶解成易溶于水的或易被表面活性剂乳化、增溶的小分子片段。同时,将蛋白质大分子所黏附或包裹的污垢颗粒快速释放,从而实现快速去污。当前市场上的许多洗涤剂产品都是通过添加蛋白酶来达到去蛋白质除污渍的目的,但存在洗涤剂中表面活性剂导致酶失活现象[52]。鹿桂乾等研究发现在9种液体洗涤剂添加表面活性剂中,只有AEO7和AEO9两种对酶的活性影响相对较小[53],并且洗涤环境相对比较粗放,所以对于洗涤剂添加用酶要求很高,一种对表面活性剂和洗涤环境具有很强耐受性的酶是作为洗涤剂添加酶的必要前提。
Anissa等在芽孢杆菌A21(Bacillusmojavensis A21)中发现BM1和BM2两种碱性蛋白酶均具有很好的表面活性剂耐受能力,其中BM1通关过保守区域分析属于NK蛋白酶家族,相对分子质量大约29 000,试验中表现出很好的表面活性剂耐受和环境适应能力。日前发现血红蛋白降解菌短小芽孢杆菌NJM4菌株(Bacillus pumilus NJM4)对顽固性污渍(血液污渍)具有很强的分解能力[54],可以对蛋白污渍有很好去除效果。NK家族蛋白酶基因广泛存在于微生物基因组中,因此使用基因组学筛选具有洗涤剂应用价值的基因是一种快速高效的办法。这些具有工业应用价值的功能基因可以建立基因文库,并且此家族蛋白酶的基因已经被申请为洗涤剂添加剂专利[55]。
3.3医药行中的应用
NK作为辅助溶栓的保健品药物,已在欧美和日本盛行20多年,在心脑血管疾病治疗方面前景看好。不仅如此,NK家族蛋白酶在抗菌、抗线虫、防止骨疏、阿尔茨海默病治疗和临床手术中同样存在不可估量的潜在应用价值。该家族蛋白酶能溶解菌体,具有广谱抗菌活性[56]。相关NK重组研究过程中NK表现出非常强的宿主菌毒性,重组工程菌株发酵过程常出现由NK溶菌活性所导致的菌体裂解现象,说明该家族蛋白酶具有非常好的溶菌抗菌价值。Antal等克隆得到的megA巨大芽孢杆菌抗菌肽基因中也含有NK家族蛋白酶保守区域阅读框。推测巨大芽孢杆菌抗菌肽的抗菌功能与substllin溶菌能力具有密切关系。Niu等从芽孢杆菌B16 (Bacillus sp.B16)分离的NK家族丝氨酸蛋白酶具有很好的线虫溶解活性,可以作为一种很好的线虫治疗药物,在线虫抑制碱性蛋白酶药学领域具有巨大的应用价值。该家族蛋白酶在医学领域也有非常大的开发利用价值,Takano等[26]把纳豆激酶(substilin NAT)应用到玻璃体切割手术中,最近研究发现纳豆激酶对淀粉样蛋白具有很好的溶解活性,说明有纳豆激酶可能对阿尔茨海默病也具有潜在医疗价值[27]。
3.4食品和饲料行业的应用
蛋白酶具有分解大分子蛋白形成多肽,从而提高食品或饲料营养的作用。NK本身的抗血栓和调节血脂的功效良好,因此豆豉和纳豆均可作为非常好的保健功能性食品[5]。利用蛋白酶水解能力可显著提高食品营养吸收,1999年Gesualdo等利用地衣芽孢杆菌 (Bacillus licheniformis)产生的Subtilisin Carlsberg蛋 白 酶 水 解 大 西 洋 鲱 鱼 (Clupea harengus),从而提高营养价值。蛋白酶在饲料行业的应用也是倾向于蛋白酶对大分子蛋白的降解能力,提高家畜对饲料的利用率。NK蛋白酶家族具有活性高和种类多样等特点,因此非常适合饲料行业高蛋白含量的豆粕和玉米饼的处理,可作为一种很好的饲料添加剂。
4 结语和展望
随着组学大数据时代的到来和生物信息学的发展,NK的分子相关信息逐渐被一一解释。从分子角度出发,可以把 NK定义为 Peptidases-S8-Subtilisin-subset家族的一员,由于该蛋白酶家族具有分布广、结构多样和性质多变等特点,因此该家族具有非常广泛的开发和利用价值,例如医药、精细化工、食品和饲料等行业。但是,大量针对NK的研究还是局限和停留在基因发掘和血栓治疗上,受最近的一些研究启发,发现以目前研究方法思路远远无法实现NK价值开发和利用的最大化。目前,借助生物信息学技术从基因组寻找到更多更适合血栓治疗的基因已经成为可能,生物信息对功能基因发掘和利用都具有非常重要的意义。借鉴NK家族蛋白酶的相关性质和功能来挖掘NK工业和医学的潜在价值是一种非常好的手段。
[1]Sumi H,Hamada H,Tsushima H,etal.A novel fibrinolytic enzyme(nattokinase)in the vegetable cheese Natto;a typical and popular soybean food in the Japanese diet[J].Experientia,1987,43(10):1110-1111.
[2]Nakamura T,Yamagata Y,Ichishima E.Nucleotide sequence of the subtilisin NAT gene,aprN,of Bacillus subtilis(natto)[J]. Bioscience,Biotechnology and Biochem istry,1992,56(11):1869-1871.
[3]Jain,S.C.,et al.,The crystal structure of an autoprocessed Ser221Cys-subtilisin E-propeptide complex at 2.0Åresolution[J]. Journal of Molecular Biology,1998,284(1):137-144.
[4]江晓,董明盛,江汉湖.一种食源性纤溶酶(纳豆激酶)酶学性质的研究[J].中国酿造,2002,21(1):23-25. JIANG Xiao,DONG M ingsheng,JIANG Hanhu.Study on enzyme property of Nattokinase-a kind of food fibronolytic enzyme [J].China Brew ing,2002,21(1):23-25.(in Chinese)
[5]FujitaM,Nomura K,Hong K,etal.Purification and characterization of a strong fibrinolytic enzyme(nattokinase)in the vegetable cheese natto,a popular soybean fermented food in Japan[J].Biochem ical and Biophysical Research Communications,1993,197(3):1340-1347.
[6]Bryan PN.Protein engineering of subtilisin[J].Biochim ica et Biophysica Acta(BBA)-Protein Structure and M olecular Enzymology,2000,1543(2):203-222.
[7]Zheng Z,Zuo Z,Liu Z,etal.Construction of a 3Dmodelof nattokinase,a novel fibrinolytic enzyme from Bacillusnatto:A novel nucleophilic catalyticmechanism fornattokinase[J].Journal of M olecular Graphics and M odelling,2005,23(4):373-380.
[8]Yanagisawa Y,Chatake T,Naito S,et al.X-ray structure determ ination and deuteration of nattokinase[J].Journal of Synchrotron Radiation,2013,20(6):0-0.
[9]Carter P,Wells JA.Dissecting the catalytic triad ofa serine protease[J].Nature,1988,332(6164):564-568.
[10]Bryan P,Pantoliano M W,Quill S G,et al.Site-directed mutagenesis and the role of the oxyanion hole in subtilisin[J]. Proceedings of the National Academy of Sciences,1986,83(11):3743-3745.
[11]Yanagisawa Y,Chatake T,Chiba-Kamoshida K,etal.Purification,crystallization and prelim inary X-ray diffraction experimentof nattokinase from Bacillus subtilis natto[J].Acta Crystallographica Section F:Structural Biology and Crystallization Communications,2010,66(12):1670-1673.
[12]Uehara R,Angkaw idjaja C,Koga Y,etal.Formation of the high-affinity calcium binding site in Pro-subtilisin Ew ith the insertion sequence IS1 of Pro-Tk-subtilisin[J].Biochem istry,2013,52(50):9080-9088.
[13]LiY,Hu Z,Jordan F,etal.Functionalanalysisof the propeptideof subtilisin E asan intramolecular chaperone for protein folding refolding and inhibitory abilitiesof propeptidemutants[J].Journal of Biological Chem istry,1995,270(42):25127-25132.
[14]Shinde U,LiY,Chatterjee S,etal.Folding pathwaymediated by an intramolecular chaperone[J].Proceedings of the National Academy of Sciences,1993,90(15):6924-6928.
[15]Tanaka S,Matsumura H,Koga Y,etal.Fournew crystalstructuresof Tk-subtilisin in unautoprocessed,autoprocessed andmatureforms:insight into structural changesduringmaturation[J].Journal of M olecular Biology,2007,372(4):1055-1069.
[16]Siezen R J,de VosW M,Leunissen JA M,etal.Homologymodelling and protein engineering strategy of subtilases,the family of subtilisin-like serine proteinases[J].Protein Engineering,1991,4(7):719-737.
[17]Kojima S,Iwahara A,Hisano Y,etal.Effectsof hydrophobic am ino acid substitution in Pleurotusostreatus proteinase A inhibitor 1 on its structure and functions as protease inhibitor and intramolecular chaperone[J].Protein Engineering Design and Selection,2007,20(5):211-217.
[18]Kojima S,M inagawa T,M iura K.The propeptide of subtilisin BPN'as a temporary inhibitor and effect of an am ino acid replacementon its inhibitory activity[J].FEBS Letters,1997,411(1):128-132
[19]Markland FS,Sm ith E L.Subtilisin BPN'VII.Isolation of cyanogen bromide peptides and the complete am ino acid sequence[J]. Journal of Biological Chem istry,1967,242(22):5198-5211.
[20]Agrebi R,Haddar A,Hm idet N,et al.BSF1 fibrinolytic enzyme from amarine bacterium Bacillus subtilis A26:Purification,biochemicalandmolecular characterization[J].Process Biochem istry,2009,44(11):1252-1259.
[21]Matsubara H,Kasper C B,Brown D M,et al.Subtilisin BPN'I.Physical properties and amino acid composition[J].Journal of Biological Chem istry,1965,240(3):1125-1130.
[22]Sm ith E L,Markland FS,Kasper C B,etal.The complete am ino acid sequence of two types of subtilisin,BPN'and Carlsberg[J]. Journal of Biological Chem istry,1966,241(24):5974-5976.
[23]Maurer K H.Detergentproteases[J].Current opinion in Biotechnology,2004,15(4):330-334.
[24]Uehara R,Tanaka S,Takano K,et al.Requirement of insertion sequence IS1 for thermal adaptation of Pro-Tk-subtilisin from hyperthermophilic archaeon[J].Extremophiles,2012,16(6):841-851.
[25]Kim JY,Gum SN,Paik JK,et al.Effects of nattokinase on blood pressure:a random ized,controlled trial[J].Hypertension Research,2008,31(8):1583-1588.
[26]Takano A,Hirata A,Ogasawara K,etal.Posterior vitreous detachment induced by nattokinase(subtilisin NAT):a novelenzyme for pharmacologic vitreolysis[J].Investigative Ophthalmology&Visual Science,2006,47(5):2075-2079.
[27]Hsu R L,Lee K T,Wang JH,et al.Amyloid-degrading ability of nattokinase from Bacillus subtilis natto[J].Journal of Agricultural and Food Chem istry,2008,57(2):503-508.
[28]Wang S L,Chen H J,Liang T W,et al.A novel nattokinase produced by Pseudomonas sp.TKU015 using shrimp shells as substrate[J].Process Biochem istry,2009,44(1):70-76.
[29]Wang S L,Yeh P Y.Purification and characterization of a chitosanase from a nattokinase producing strain Bacillus subtilis TKU007[J].Process Biochem istry,2008,43(2):132-138.
[30]Yang N C,Chou CW,Chen C Y,etal.Combined nattokinasew ith red yeast rice butnotnattokinase alone has potenteffects on blood lipids in human subjectsw ith hyperlipidem ia[J].Asia Pacific Journal of Clinical Nutrition,2009,18(3):310.
[31]FujitaM,OhnishiK,Takaoka S,etal.Antihypertensive effectsof continuousoraladministration of nattokinase and its fragments in spontaneously hypertensive rats[J].Biological and Pharmaceutical Bulletin,2011,34(11):1696-1701.
[32]DeDea L.The antiplateleteffectsofaspirin;nattokinaseasa blood thinner[J].Journal of the American Academy of Physician Assistants,2010,23(12):13.
[33]Upadhyay V K,Kelly A L,M cSweeney P L H.Use of nattokinase,a subtilisin-like serine proteinase,to accelerate proteolysis in Cheddar cheese during ripening[J].Le Lait,2006,86(3):227-240.
[34]Minn M,Sinha SK.The lubrication of poly(etheretherketone)by an aqueoussolution ofnattokinase[J].Wear,2012,296(1):528-535.
[35]Suwanmanon,K.and P.-C.Hsieh,Effectofγ-am inobutyric acid and nattokinase-enriched fermented beanson the blood pressure of spontaneously hypertensive and normotensiveWistar-Kyoto rats[J].Journal of Food and Drug Analysis,2014,22(4):485-491.
[36]Wang SL,Wu Y Y,Liang TW.Purification and biochem ical characterization ofa nattokinase by conversion of shrimp shellw ith Bacillussubtilis TKU007[J].New Biotechnology,2011,28(2):196-202.
[37]Chen RPY,Lee K T.Nattokinase for degrading and reducing amyloid fibrils—associated w ith alzheimer's disease,prion diseases and otheramyloidoses:U.S.Patent8,137,666[P].2012-3-20
[38]Nguyen T T,Quyen TD,Le H T.Cloning and enhancing production ofa detergent-and organic-solvent-resistantnattokinase from Bacillus subtilis VTCC-DVN-12-01 by using an eight-protease-gene-deficient Bacillus subtilis WB800[J].M icrobial CellFactories,2013,12(1):79.
[39]Fadl N N,Ahmed H H,Booles H F,et al.Serrapeptase and nattokinase intervention for relieving Alzheimer’s disease pathophysiology in ratmodel[J].Human&Experimental Toxicology,2013,32(7):721-735.
[40]Hamza,A.,M iracle enzymes serrapeptase and nattokinasemitigate neuroinflammation and apptosis associated w ith alzheimer’s disease in experimentalmodel[J].W orld Journal of Pharmacy and Pharmaceutical Sciences,2014(3):876-891
[41]Lim D H,Park J K,Kim H S,et al.Effects of feeding nattokinase as natural feed additives on m ilk production and blood metabolites in lactating dairy cows[J].Korean Journal of Organic Agriculture,2011,19(4):50-56.
[42]Borah D,Shahin L,Sangra A.Production,purification and characterization of nattokinase from bacillus subtilis,isolated from sewagewater[J].International Journal of Pharmaceutical Archive:IJPAOnline,2012,1(1).
[43]王凡强,马美荣,王正祥,等.枯草杆菌蛋白酶基因工程的研究进展[J].生物工程进展,2000,20(2):4-44. WANG Fanqiang,MA Meirong,WANG Zhengxiang,et al.Advances in gene engineering of Subtilisin [J].Progress in Biotechnology,2000,20(2):41-44.(in Chinese)
[44]Fakhfakh-Zouari N,Hm idet N,Haddar A,et al.A novel serine metallokeratinase from a new ly isolated Bacillus pum ilus A1 grown on chicken feathermeal:biochemical and molecular characterization[J].Applied Biochem istry and Biotechnology,2010,162(2):329-344
[45]陈倩,关泽文,刘彦.一株蜡状芽孢杆菌产蛋白酶脱毛性能研究[J].皮革科学与工程,2013,23(5):10-14. CHEN Qian,GUAN Zewen,LIU Yan.Study on theunhairing propertiesof proteinases produced by a Bacillus cereus[J].Leather Science and Engineering,2013,23(5):10-14.(in Chinese)
[46]金宝仲.几种制革脱毛蛋白酶性能的研究[J].皮革科技动态,1977,7(6):3-9. JING Zhongbao.Several kinds of leather depilation protease performance research[J].Leather Technology Dynam ic,1977,7 (6):3-9.(in Chinese)
[47]JaouadiN Z,Rekik H,Badis A,etal.Biochemicalandmolecular characterization of a serine keratinase from Brevibacillusbrevis US575w ith promising keratin-biodegradation and hide-dehairing activities[J].PloS one,2013,8(10):e76722.
[48]黄庆,潘皎,彭勇,等.短小芽孢杆菌(Bacillus pum ilus)脱毛蛋白酶基因的克隆与序列分析[J].高技术通讯,2004,14(2):31-35. HUANG Qing,PAN Jiao,PENG Yong,etal.Cloning and seguence analysis of dehairing protease Gene in the Bacillus pum ilus UN-31-C-42[J].Chinese High Technology Letters,2004,14(2):31-35.(in Chinese)
[49]肖怀秋,林亲录,李玉珍,等.中性蛋白酶芽孢杆菌BX24产酶条件及部分酶学性质[J].食品与生物技术学报,2005,24(2):42-56. XIAO Huaiqiu,LIN Qinlu,LIYuzhen,et al.Studies on protease production conditions and enzyme characterizations of neutral protease producing strain BacillusBX24[J].Journal of Food Science and Biotechnology,2005,24(2):42-56.(in Chinese)
[50]Odetallah N H,Wang JJ,Garlich JD,etal.Keratinase in starter diets improves grow th of broiler chicks[J].Poultry Science,2003,82(4):664-670.
[51]Fakhfakh-Zouari N,Hm idet N,Haddar A,et al.A novel serine metallokeratinase from a new ly isolated Bacillus pum ilus A1 grown on chicken feathermeal:biochemical and molecular characterization[J].Applied Biochem istry and Biotechnology,2010,162(2):329-344.
[52]Niehaus F,Gabor E,Wieland S,etal.Enzymes for the laundry industries:tapping the vastmetagenomic poolofalkaline proteases [J].M icrobial Biotechnology,2011,4(6):767-776.
[53]鹿桂乾,张利萍.蛋白酶在液体洗涤剂中的应用研究[J].现代化工,2010,30(2):124-127. LU Guiqian,ZHANG Liping.Study on application of protease in liquid detergent product[J].M odern Chem ical Industry,2010,30(2):124-127.(in Chinese)
[54]Yao D,Qu J,Chang P,et al.Production and characterization of alkaline protease from hemoglobin-degrading Bacillus pumilus NJM 4 to produce fermented bloodmeal[J].Journal of Food Science and Technology,2012,49(5):626-631.
[55]Deng A,Wu J,Zhang Y,et al.Purification and characterization of a surfactant-stable high-alkaline protease from Bacillus sp. B001[J].Bioresource Technology,2010,101(18):7100-7106.
[56]Kim W,Choi K,Kim Y,et al.Purification and characterization of a fibrinolytic enzyme produced from Bacillus sp.strain CK 11-4 screened from Chungkook-Jang[J].Applied and EnvironmentalM icrobiology,1996,62(7):2482-2488.
AReview on theM olecular Structureof Nattokinaseand ItsPotentialApplication
MA Guoxing1,2,PAN Feng1,2,WANG Qingbo1,2,WEN Chunguang1,2,WU Yaqing1,2,XU Ruian*1,2
(1.Institute of Molecular Medicine,Huaqiao University,Xiamen 361021,China;2.Engineering Research Center of Molecular Medicine,M inistry of Education,Xiamen 361021,China)
Nattokinase(NK)iswellknown for its strong thrombolytic property.However,methods for exploring the enzyme are still traditionaland its value is thus underestimated.In this review,the structure and the classification for NK were summarized.Based on the applications of NK family proteases,the potentialutilizationsof NK were predicted.
Nattokinase(NK),conserved domains,subtilisin proteinase,Peptidase-S8-fam ily-subtiliansubset-superfam ily
2015-07-08
国家自然科学基金项目(81271692);国家863计划项目(2012AA020810)。
马国兴(1987—),男,河北唐山人,生物化工博士研究生,主要从事生物化工与基因工程药物,生物信息学等研究。E-mail:mgxtongchi@163.com
许瑞安(1948—),男,福建泉州人,教授,主要从事微生物学、生物化学与分子生物学、海洋生物资源与环境、基因药物等研究。E-mail:hmmedu@hqu.edu.cn

