一个水稻根长QTLqRL4的分离鉴定
徐晓明 张迎信 王会民 任翠 王汝慈 沈希宏 占小登吴玮勋 程式华,* 曹立勇,*
(1中国水稻研究所 国家水稻改良中心, 杭州 310006;2杭州师范大学, 杭州 310036; 3浙江省超级稻研究重点实验室 杭州 310006;
一个水稻根长QTLqRL4的分离鉴定
徐晓明1,2,3,#张迎信1,3,#王会民1,4任翠5王汝慈1,3沈希宏1,3占小登1,3吴玮勋1,3程式华1,3,*曹立勇1,3,*
(1中国水稻研究所 国家水稻改良中心, 杭州 310006;2杭州师范大学, 杭州 310036;3浙江省超级稻研究重点实验室 杭州 310006;
4江西省农业科学院, 南昌 330200;5河南农业大学, 郑州 450002;*通讯联系人,E-mail:shcheng@mail.hz.zj.cn,caolycgf@mail.hz.zj.cn)
XUXiaoming,ZHANGYingxin,WANGHuimin,etal.IdentificationofqRL4,amajorquantitativetraitlocusassociatedwithricerootlength.ChinJRiceSci, 2016, 30(4): 363-370.
徐晓明, 张迎信, 王会民, 等. 一个水稻根长QTLqRL4的分离鉴定. 中国水稻科学, 2016, 30(4): 363-370.
摘要:为了分离鉴定 qRL4,以超级稻协优 9308 衍生重组自交系与轮回亲本中恢9308(R9308)回交多代的高代回交群体为材料,利用琼脂无土栽培技术,开展水稻根长QTL qRL4的分离鉴定研究,最后将 qRL4定位在第4染色体分子标记RM5687与InDel49间624.6 kb范围内。此基因的定位与分离鉴定将有助于水稻根长相关遗传机理的研究,为探究在基因水平上的水稻根系形态建成奠定了坚实基础。
关键词:水稻; 根长; 数量性状基因座
植物生长过程中,根系发挥着不可或缺的作用,包括固定,水分和养分的吸收等[1-2],如具有扎根深、健壮且分支多的根系的植物在逆境中(如缺水以及养分匮乏)具有更强的生命力。由于根系研究技术的局限性和起步较晚,人们对根系的认识深度远远不及地上部,如产量、品质、株型、叶型、抗病虫和抗非生物胁迫等性状[3]。但是研究者们已经认识到开展水稻根系及遗传育种研究的必要性。近年来,围绕水稻根系遗传研究的报道也有很多,在水稻根系QTL作图与基因克隆研究方面取得了重大突破[4]。其中,对于控制根系生长的各性状研究主要通过QTL分析,这些性状涉及根系生物量、根长和根数(以及在特殊环境下的表型)等[5-12]。由于根系生长环境复杂,而且很难获得完整的根系,导致表型鉴定准确性很低,因而精细定位根系相关性状QTLs难度较大。这可能是水稻根系相关QTLs很少被克隆的原因。
迄今为止,已克隆的根系相关基因大多是利用水稻突变体材料获得。其中,包括根冠形成相关基因[13-15]、不定根系形成相关基因[16-18]、根长基因[19-23]、水稻硝态氮吸收基因 NIA[24]、水稻穗部长出不定根突变体基因[25]、水稻根系中特异表达铵同化基因GS8[26]、水稻磷高效吸收利用的转录因子 OsPTF1[27]、硅吸收转运基因LsiL[28-29]以及二价铁转运基因OsIRT1[30]。这些根系相关基因的定位、分离、克隆研究,对人们揭示根系形态建成的遗传机理奠定了坚实基础,为开展水稻根系育种提供了有价值的参考信息。
我们在前期研究中,于第4染色体定位到一个控制根长的主效QTLqRL4,相对根长表型贡献率最高达到36.5%,成株期可以显著缩短根长8.1cm[31]。为了分离鉴定 qRL4,本研究以超级稻协优9308衍生的重组自交系(RIL)与轮回亲本中恢9308(R9308)回交多代的高代回交群体为材料,利用琼脂无土栽培技术,借助经典数量遗传学和现代分子生物学技术手段开展水稻根长QTLqRL4的分离鉴定研究。
1材料与方法
1.1试验材料
本研究小组前期利用协青早B(XB)与中恢9308(R9308)杂交构建的RILs对相关农艺性状进行了QTL定位,于第4染色体长臂上分子标记RM307与RM1205之间定位到一个QTL簇,包括不同时期的相对根长、相对根冠比、相对根干质量和相对植株干质量等性状。 qRL4被定位于标记RM3317-RM1205之间,表型贡献率达36.5%,在抽穗期可以显著缩短根长8.1cm,属于主效QTL[31]。
基于本研究小组前期利用RIL定位的结果,采用携带qRL4的RIL家系与R9308进行回交的方法,每代结合分子标记辅助选择筛选目标片段,用于在高世代回交材料中筛选含有目标QTL、背景较纯合的近等基因系材料。结果在BC4F2世代挑选得到2个目标片段为杂合型的株系X15-1和X15-2,每个株系都含有目标QTL代换区间,且代换区间为杂合状态,遗传背景尽量与轮回亲本一致,自交获得BC4F3群体。其中,在BC4F3群体中得到两个目标片段纯合的家系qRL4-NIL1和qRL4-NIL2用来验证之前定位的结果,而X15-1和X15-2自交后获得的次级分离群体共2450个单株用来缩小目标QTL的区间。为了确认分离鉴定的群体中含有目标QTL,调查了BC4F3:4群体的根系等农艺性状。
1.2种植方法
所有试验材料均采用琼脂种植。具体步骤如下:1) 将种子用70%酒精消毒1min,然后用蒸馏水清洗干净,再用5%次氯酸钠溶液消毒30min,用蒸馏水清洗干净,之后把种子均匀平铺于灭菌培养皿上,放少许蒸馏水使种子保持湿润,于35℃下催芽待用;2) 将催好芽的种子均匀播于灌好琼脂的插槽内,一个槽大约可以播16株;3) 将种植好的箱体置于培养箱内(昼夜温度分别为30℃±2℃和21℃±2℃,自然光,每天光照14h,相对湿度70%),种植水稻的插槽里面要一直保持琼脂湿润,以防琼脂干枯裂口和长菌(图1)。
琼脂的配制方法:按照国际水稻所营养液配方[32],根据需要稍作修改,用配好的营养液溶解琼脂(琼脂和营养液的质量体积比为1∶20),并煮沸使琼脂溶液呈无色透明状,然后立即将无色透明的琼脂溶液灌入插槽内,待其冷凝后则完成培养基的配制。
1.3表型考查
苗期分别对7个农艺性状进行测定,具体方法如下:1)用直尺测量每株水稻的株高和根长,然后将水稻完整根系从植株上分离下来,按单根一条条分开,统计每株的根数;2) 将分离好的茎叶和根系分别装在信封中,于烘箱中105℃下杀青1h,然后65℃下烘干至恒重,称量茎叶干质量和根系干质量,计算获得植株干质量和根冠比等数据。
1.4InDel引物开发及qRL4定位
在SSR标记RM3317与RM1205之间首先采用实验室已有的公共引物以及Gramene(http://www.gramene.org)上公布的84对公共引物和63对InDel引物,对 qRL4进行定位。利用实验室已测序的亲本协青早B和中恢9308的测序信息(北京华大基因研究中心),分别截取第4染色体RM3317和RM1205标记在两亲本上的DNA序列,利用LaserGene软件MegAlign和Editseq工具对亲本上截取的两段DNA碱基序列进行比对分析,找出两段序列之间插入缺失(Insertion/Deletion,InDel)序列位点,选取插入缺失的碱基长度为5~10bp作为设计引物序列模体,截取插入缺失位点前后各300bp序列进行序列标记开发。利用PrimerPremier5.0软件开发设计新InDel引物,引物设计的标准如下:1)长度18~24bp;2)GC含量40%~60%;3)Tm值50℃~65℃;4)没有二级结构、发夹结构、3个连续相同碱基;5)引物3′末端碱基最好为C或者G;6)设计好的引物采用RAPDB(http://rapdb.dna.affrc.go.jp/)软件进行染色体物理位置确定和标记特异性检测,特异性好的标记序列由英潍捷基生物有限公司合成(表1)。

A-催芽;B-播种;C-播种后4d;D-播种后20d。
A,Acceleratinggermination;B,Seeding;C,Fourdaysafterseeding;D,Twentydaysafterseeding.
图1琼脂种植水稻技术
Fig. 1.Technologyofricecultivationbyagargel.
2结果与分析
2.1农艺性状的遗传表现
CSSL群体与两个亲本的7个农艺性状值(表2)。株高为20.02cm,根长为5.25cm,根数均值为6.9,茎叶干质量为81.5mg,根干质量为33.8mg,植株干质量均值为0.1153g,根冠比均值为0.44,7个性状的最大值都超过了高值亲本,最小值也都小于低值亲本。亲本协青早B与中恢9308的根长、根数、茎叶干质量、根干质量、植株干质量及根冠比等6个性状均差异明显,高值亲本比低值亲本至少高10%。由表3可知,7个性状两两之间的相关性均达极显著水平。其中茎叶干质量与植株干质量的相关系数达0.98,在所有性状间表现为最大。其余各性状间相关系数均达0.1以上,大部分集中于0.2到0.6之间,只有根长与根冠比为0.75,根数与根冠比为0.78,茎叶干质量与根冠比为-0.63,根干质量与植株干质量为0.74,绝对值均大于0.6。
表1用于qRL4基因精细定位的标记
Table1.MarkersusedinfinemappingofqRL4.
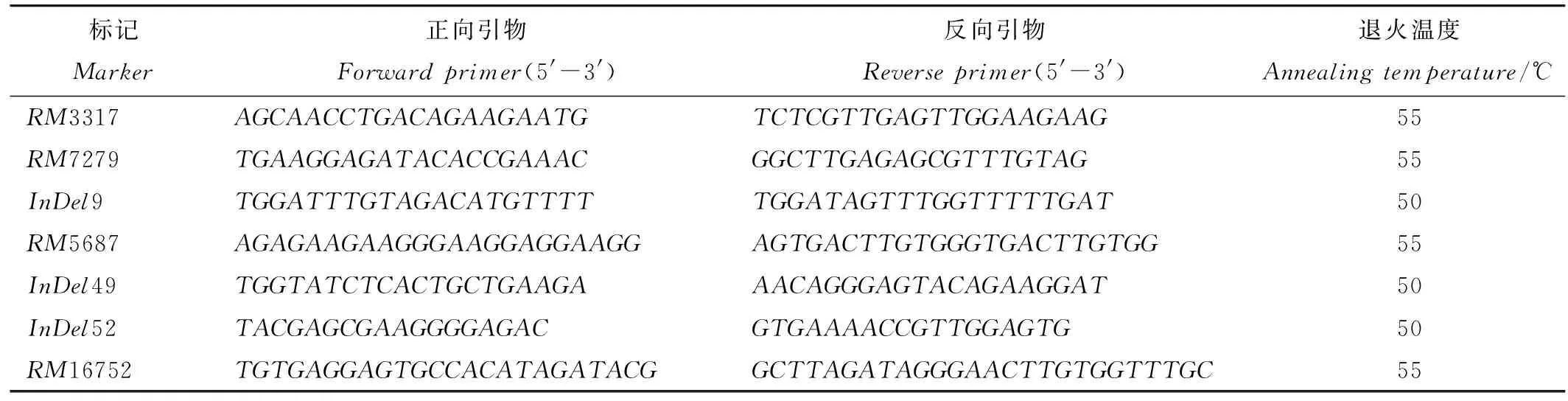
标记Marker正向引物Forwardprimer(5'-3')反向引物Reverseprimer(5'-3')退火温度Annealingtemperature/℃RM3317AGCAACCTGACAGAAGAATGTCTCGTTGAGTTGGAAGAAG55RM7279TGAAGGAGATACACCGAAACGGCTTGAGAGCGTTTGTAG55InDel9TGGATTTGTAGACATGTTTTTGGATAGTTTGGTTTTTGAT50RM5687AGAGAAGAAGGGAAGGAGGAAGGAGTGACTTGTGGGTGACTTGTGG55InDel49TGGTATCTCACTGCTGAAGAAACAGGGAGTACAGAAGGAT50InDel52TACGAGCGAAGGGGAGACGTGAAAACCGTTGGAGTG50RM16752TGTGAGGAGTGCCACATAGATACGGCTTAGATAGGGAACTTGTGGTTTGC55
2.2包含qRL4近等基因系的构建
为了验证前面定位结果的准确性,从BC4F3群体中挑选了两个包含 qRL4代换区段的株系进行近等基因系的构建(图2)。图中黑色部分代表供体亲本协青早B的基因型片段,白色部分代表轮回亲本中恢9308的基因型片段。由图3可知, qRL4-NIL1和 qRL4-NIL2根长介于两个亲本之间, qRL4-NIL1为5.12cm,偏向于低值亲本协青早B,而 qRL4-NIL2为6.33cm,偏向于高值亲本中恢9308,两者间差异达到1.21cm。从基因型上分析, qRL4-NIL1和 qRL4-NIL2只在第4染色体有一个区段的差异,虽然第5染色体也各有一个代换片段,但是因为两者的代换区段完全一致,所以引起表型差异的遗传区段不在这两个杂合片段内,只可能存在于第4染色体的代换区段内。结合分子标记信息,第4染色体上的代换区段为RM3317-RM1205,正好包含了之前定位到的 qRL4,证明两者之间的表型差异是由 qRL4引起的,也更进一步表明 qRL4位于标记RM3317和RM1205间。
2.3根长qRL4的精细定位
结合根长表型,X15-1和X15-2两个群体共筛选到289个隐性单株,利用具有多态性的9个分子标记进行分离鉴定(表4)。由表3可知,区间越大,交换单株越多,随着标记间距离的缩小,交换单株数量越来越少,最后在标记RM5687与InDel49之间没有找到交换单株,最后将 qRL4定位在标记RM5687与InDel49之间、物理距离为624.6kb范围内(图4)。
表2水稻染色体片段代换系群体7个农艺性状的表型特征
Table2.CharacterofsevenphenotypictraitsinCSSLpopulationofrice.
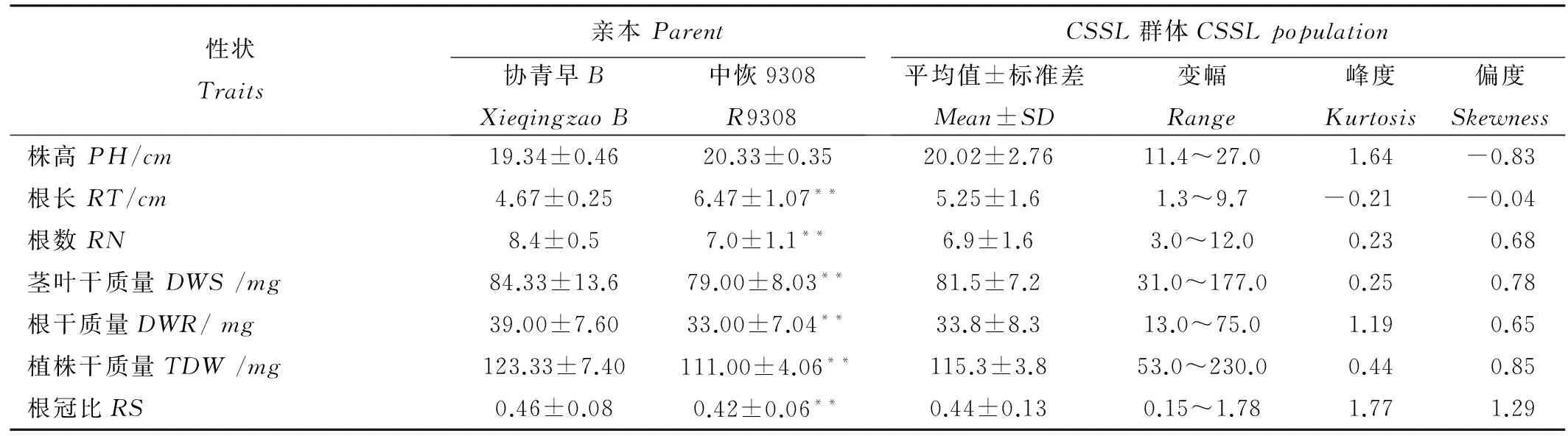
性状Traits亲本Parent协青早BXieqingzaoB中恢9308R9308CSSL群体CSSLpopulation平均值±标准差Mean±SD变幅Range峰度Kurtosis偏度Skewness株高PH/cm19.34±0.4620.33±0.3520.02±2.7611.4~27.01.64-0.83根长RT/cm4.67±0.256.47±1.07**5.25±1.61.3~9.7-0.21-0.04根数RN8.4±0.57.0±1.1**6.9±1.63.0~12.00.230.68茎叶干质量DWS/mg84.33±13.679.00±8.03**81.5±7.231.0~177.00.250.78根干质量DWR/mg39.00±7.6033.00±7.04**33.8±8.313.0~75.01.190.65植株干质量TDW/mg123.33±7.40111.00±4.06**115.3±3.853.0~230.00.440.85根冠比RS0.46±0.080.42±0.06**0.44±0.130.15~1.781.771.29
PH,Plantheight;RL,Rootlength;RN,Rootnumber;DWS,Dryweightofshoots;DWR,Dryweightofroots;TDW,Totaldryweight;RS,Root/Shoot;SD,Standarddeviation.*and**,Significantat0.05and0.01levels,respectively.Thesameasbelow.
表3水稻染色体片段代换系群体7个性状的相关性分析
Table3.CorrelationcoefficientsofseventraitsinCSSLpopulationofrice.
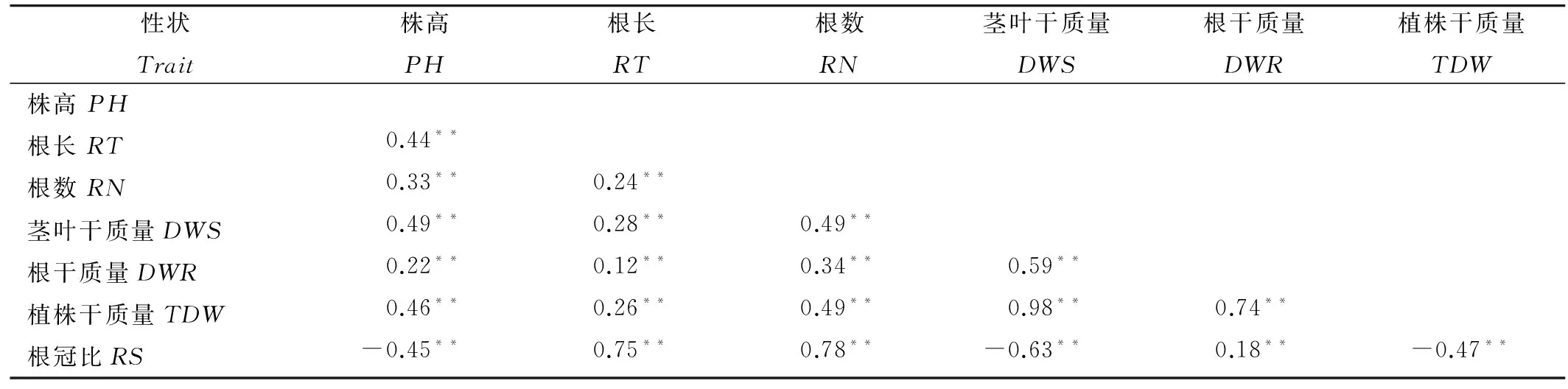
性状Trait株高PH根长RT根数RN茎叶干质量DWS根干质量DWR植株干质量TDW株高PH根长RT0.44**根数RN0.33**0.24**茎叶干质量DWS0.49**0.28**0.49**根干质量DWR0.22**0.12**0.34**0.59**植株干质量TDW0.46**0.26**0.49**0.98**0.74**根冠比RS-0.45**0.75**0.78**-0.63**0.18**-0.47**
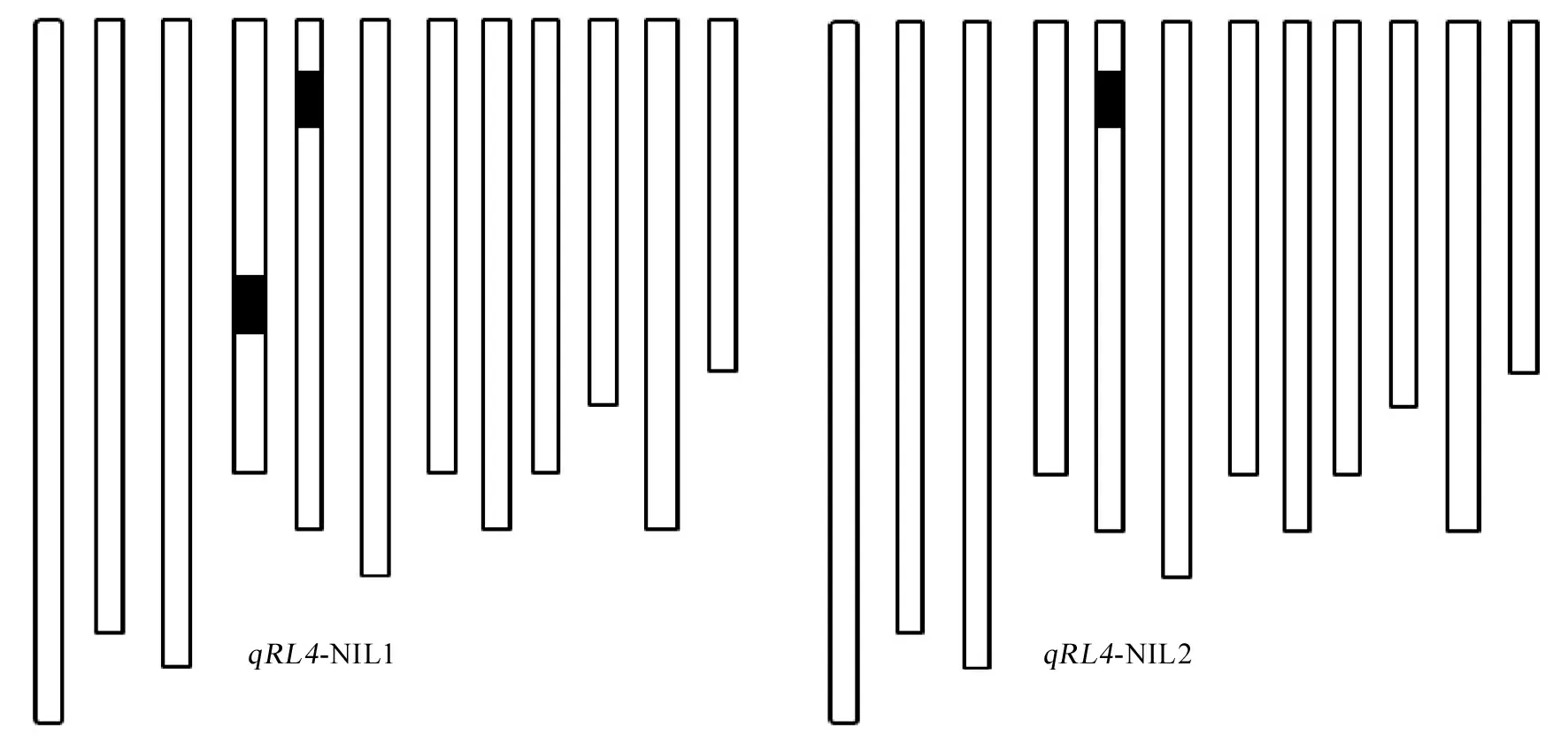
白色和黑色分别代表纯合的中恢9308和协青早B的基因型。
WhiteandblackboxesrepresenthomozygousallelesfromZhonghui9308andXieqingzaoB,respectively.
图2两个携带qRL4近等基因系的基因型
Fig. 2.GenotypesoftwoneartsogeniclinesharboringqRL4.
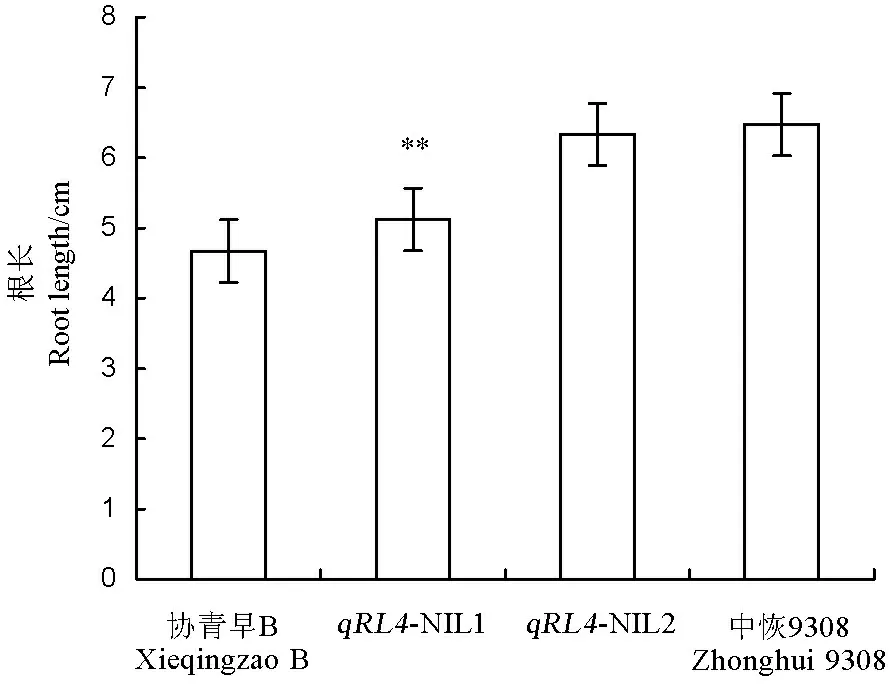
图3qRL4-NIL1和qRL4-NIL2 苗期根长比较
Fig. 3.RootlengthofqRL4-NIL1andqRL4-NIL2atseedlingstage.
3讨论
3.1琼脂种植水稻技术的优缺点
水稻根系的研究方法很多,主要有土钻法、土柱法、容器法、三维坐标容器法和虚拟根系模型等。这些方法获取的根系都会受到不同程度损伤,难以获得完整的根系。琼脂种植水稻技术是一项新技术,是本研究小组经过上百次反复试验探索出来的一项固体基质无土栽培技术。该技术采用琼脂和营养液配置培养基,把配制好的琼脂培养基灌入特制的箱体槽中,冷凝之后把水稻催芽种子种于琼脂上即可。使用此技术种植水稻可以更好地模拟水稻植株在土壤环境中生长,使根系的生长具有一定的阻力,可以更接近于土壤环境中的生长状况,比水培种植水稻技术具有明显的优点。此外,该技术还便于检测水稻苗期生长过程中各种性状,例如生理生化、胁迫、蛋白提取、根系扫描测定等,且不受时间、空间和环境影响,取材方便,管理方便。此法的缺点是操作繁琐,种子和琼脂要消毒灭菌,不消毒灭菌琼脂就容易发霉腐烂,影响水稻生长。另一缺点是琼脂表面极易干裂,要定期浇水以保证琼脂表面湿润。该技术已经申请了国家专利。
3.2控制根长的一个主效QTL
根深才能叶茂,要获得繁茂的地上部植株,地下部根系也必须生长良好。关于水稻根系特征的研究已经有很多相关报道,例如,Uga等[38]定位的一个与地表根生长有关的主效QTLqSOR1,位于第7染色体上标记RM21941与RM21976间,物理距离为812kb;Kamoshita等[39]研究了在淹水条件下一个水稻群体根系相关农艺性状的QTL。Mitsuhiro等[40]采用水培方法,在不同NH4+浓度条件下,将一个水稻苗期根长主效QTLqRL6.1,定位于C11635-P3A2标记间337kb的范围内,可解释的表型变异为13.5%~21.1%,并且可以增加产量。一些在陆稻中可以增加产量的根系性状相关QTL已有相关研究[41]。然而,有关水稻根系性状克隆的基因极少,这可能是因为大田环境下水稻根系很难获取,且性状很难考查,而普通营养液水培法又工作量太大、可操作性不高的缘故。利用构建染色体片段代换系的高代回交群体BC4F4为材料对其进行精细定位,最后将 qRL4定位在第4染色体靠近着丝粒的一个624.6kb的区域内(图4)。依据水稻注解工程数据库(RiceAnnotationProject,RAP3,http://rapdb.dna.affrc.go.jp/),在此区间 qRL4有89个候选基因,由于候选基因较多,尚不能预测 qRL4精确的候选基因。今后的工作主要是缩小 qRL4所在区间的范围,争取预测并克隆该基因,进而对其进行功能互补和分子机理探索。
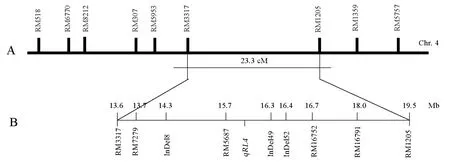
A-第4染色体遗传图;B-qRL4区间确定。分子标记对应的数字为日本晴基因组第4染色体的物理距离(Mb)。
A,LinkagemapoftheRILsderivedfromXieqingzaoB×R9308.B,IdentificationofqRL4.ThenumbersinparenthesesbesidetheDNAmarkersindicatetheirphysicalmapposition(Mb)onchromosome4ofNipponbare.
图4qRL4的精细定位
Fig. 4.LocationofqRL4onricechromosome4.
表4不同标记间重组单株数
Table4.NumberofrecombinationplantsforInDelmarkers.
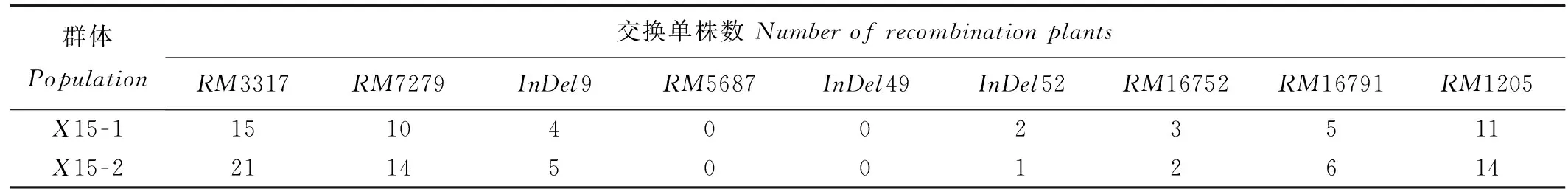
群体Population交换单株数NumberofrecombinationplantsRM3317RM7279InDel9RM5687InDel49InDel52RM16752RM16791RM1205X15-1151040023511X15-2211450012614
3.3qRL4 在水稻育种中的应用前景
生产上水分亏缺是一个限制水稻产量的重要非生物因素[42]。全世界范围内栽培水稻的区域,将近有一半的地方存在水分亏缺问题[43]。因此,改良水稻根系的吸水能力是育种上急需解决的难题。Kamoshita等[44]考查了深根比率和深根量等根系性状QTLs,将他们定位于第2、3、4、9和11染色体。然而,前人的研究都集中于水稻根系整体特征,没有对根系各性状进行细分研究,这造成了很多QTL定位结果很难继续深入。 qRL4于苗期可以明显缩短根长,在大田生产上属于劣质基因,高产水稻中最好没有该基因或者该基因不表达。在生产实践中,我们可以利用基因定点敲除技术,将该基因敲除或者沉默掉,以保证根系长度不受影响。确定候选基因并且克隆该基因,以及进行功能互补验证等,将有助于探明根系功能强大型水稻品种对水分吸收利用率高的遗传机理,为培育根系生理功能旺盛的高产品种打下坚实基础。
参考文献:
[1]川田信一郎. 水稻的根系. 北京: 农业出版社, 1984.
ShinichiroK.RiceRoots.Beijing:AgriculturePress, 1984. (inChinese)
[2]吴伟明, 程式华. 水稻根系育种的意义与前景. 中国水稻科学, 2005, 19(2): 174-180.
WuWM,ChengSH.Significanceandprospectsofbreedingforrootsysteminrice(Oryza sativa). Chin J Rice Sci, 2005, 19(2): 174-180. (inChinesewithEnglishabstract)
[3]杨建昌. 水稻根系形态生理与产量、品质形成及养分吸收利用的关系. 中国农业科学, 2011, 44(1): 36-46.
YangJC.Relationshipsofricerootmorphologyandphysiologywiththeformationofgrainyieldandqualityandthenutrientabsorptionandutilization. China Agric Sci, 2011, 44(1): 36-46. (inChinesewithEnglishabstract)
[4]李鑫, 张战, 赵一洲, 等. 水稻根系研究进展. 北方水稻, 2014, 44(2): 72-75.
LiX,ZhangZ,ZhaoYZ,etal.Researchadvanceonthericeroots. North Rice, 2014, 44(2): 72-75. (inChinesewithEnglishabstract)
[5]KamoshitaA,WadeLJ,AliL,etal.MappingQTLsforrootmorphologyofaricepopulationadaptedtorainfedlowlandconditions. Theor Appl Genet, 2002, 104(5): 880-893.
[6]KamoshitaA,ZhangJ,SiopongcoJ,etal.Effectsofphenotypingenvironmentonidentificationofquantitativetraitlociforricerootmorphologyunderanaerobicconditions. Crop Sci, 2002, 42(1): 255-265.
[7]LiZ,MuP,LiC,etal.QTLmappingofroottraitsinadoubledhaploidpopulationfromacrossbetweenuplandandlowlandjaponicariceinthreeenvironments.Theor Appl Genet, 2005, 110(7): 1244-1252.
[8]QuY,MuP,ZhangH,etal.MappingQTLsofrootmorphologicaltraitsatdifferentgrowthstagesinrice. Genetica, 2008, 133(2): 187-200.
[9]SteeleKA,PriceAH,ShashidharHE,etal.Marker-assistedselectiontointrogressriceQTLscontrollingroottraitsintoanIndianuplandricevariety. Theor Appl Genet, 2006, 112(2): 208-221.
[10]ZhengHG,BabuRC,PathanMS,etal.Quantitativetraitlociforroot-penetrationabilityandrootthicknessinrice:comparisonofgeneticbackgrounds. Genome, 2000, 43(1): 53-61.
[11]ZhengBS,YangL,ZhangWP,etal.MappingQTLsandcandidategenesforriceroottraitsunderdifferentwater-supplyconditionsandcomparativeanalysisacrossthreepopulations.Theor Appl Genet, 2003, 107(8): 1505-1515.
[12]ZhengBS,YangL,MaoCZ,etal.QTLsandcandidategenesforricerootgrowthunderfloodinganduplandconditions. J Genet Genom, 2006, 33(2): 141-151.
[13]InukaiY,MiwaM,NagatoY,etal.Characterizationofricemutantsdeficientintheformationofcrownroots. Breeding Sci, 2001, 51(2): 123-129.
[14]InukaiY,MiwaM,NagatoY,etal. RRL1, RRL2andCRL2lociregulatingrootelongationinrice. Breeding Sci, 2001, 51(4): 231-239.
[15]ZhaoY,HuYF,DaiMG,etal.TheWUSCHEL-relatedhomeoboxgeneWOX11isrequiredtoactivateshoot-bornecrownrootdevelopmentinrice. Plant Cell, 2009, 21(3): 736-748.
[16]LiuW,XuZH,LuoD,etal.RolesofOsCKH1,aricecaseinkinaseI,inrootdevelopmentandplanthormonesensitivity. Plant J, 2003, 36(2): 189-202.
[17]LiuH,WangS,YuX,etal.ARL1,aLOB-domainproteinrequiredforadventitiousrootformationinrice. Plant J, 2005, 43(1): 47-56.
[18]XuML,JiangJF,GeL,etal.FPF1transgeneleadstoalteredfloweringtimeandrootdevelopmentinrice. Plant Cell Rep, 2005, 24(2): 79-85.
[19]LiangZW,IchiiM.Morphologicalcharacterizationoftheseedlingofshort-rootmutantLM10selectedfromrice(Oryza sativaL.cv.IR8). Crop Sci, 1996, 65(3): 473-478.
[20]YaoSG,TakedaS,IchiiM.Isolationandcharacterizationofanabscisicacid-insensitivemutationthataffectsspecificallyprimaryrootelongationinrice(Oryza sativaL.). Plant Sci, 2003, 164(6): 971-978.
[21]YaoSG,MushikaJ,TakedaS,etal.Theshort-rootmutationsrt5definesasugar-mediatedrootgrowthinrice(Oryza sativaL.). Plant Sci, 2004, 167(1): 49-54.
[22]InukaiY,MiwaM,NagatoY,etal.Mechanicalstimulus-sensitivemutation,rrl3,affectsthecellproductionprocessintherootmeristematiczoneinrice. Plant Prod Sci, 2003, 6(4): 265-273.
[23]JiangH,WangS,DangL,etal.Anovelshort-rootgeneencodesaglucosamineacetyltransferaserequiredformaintainingnormalrootcellshapeinrice. Amer Soc Plant Bio, 2005, 138(1): 232-242.
[24]ChoiHK,KieinhofsA,AnG.Nucleotidesequenceofricenitratereductasegenes. Plant Mol Biol, 1989, 13(6): 731-733.
[25]TaniguchiM,FutsuharaY.Adensepaniclemutantproducingadventitiousrootsfromspikelets. Rice Gene Newsl, 1988, 5: 113-114.
[26]SakamotoA,OgawaM,MasumuraT,etal.ThreecDNAsequencescodingforglutaminesynthetasepolypeptidesinOryza sativaL. Plant Mol Biol, 1989, 13(5): 611-614.
[27]JiangH,WangS,DangL,etal.Anovelshort-rootgeneencodesaglucosamineacetyltransferaserequiredformaintainingnormalrootcellshapeinrice. Plant Physiol, 2005, 138(1): 232-242.
[28]MaJF,GotoS,TamaiK,etal.Roleofroothairsandlateralrootsinsiliconuptakebyrice. Plant Physiol, 2001, 127(4): 1773-1780.
[29]MaJF,TamaiK,YamajiN,etal.Asilicontransporterinrice. Nature, 2006, 440: 688-691.
[30]NaimatullahB,HirotakaY,NishizawaNK,etal.Cloninganiron-regulatedmetaltransporterfromrice. J Exp Bot, 2002, 53(374): 1677-1682.
[31]王汝慈. 两个生育时期水稻耐低磷胁迫相关性状的QTL定位. 北京:中国农业科学院,2009.
WangRC.QTLmappingofphosphorusdeficiencytoleranceattwodevelopmentstagesinrice(OryzasativaL.).Beijing:ChineseAcademyofAgriculturalSciences, 2009. (inChinesewithEnglishabstract)
[32]YoshidaS,FornoDA,CockJH,etal.LaboratoryManualforPhysiologicalStudiesofRice. 3rded.Manila:IRRI,1976:61- 64.
[33]吴朝晖, 周建群, 青先国. 水稻根系分布形态研究法现状及展望. 湖南农业科学, 2008(5): 11-14.
WuCH,ZhouJQ,QingXG.Advancesinmethodsofstudyingricerootsystem. Hunan Agric Sci, 2008(5): 11-14. (inChinesewithEnglishabstract)
[34]黄沆, 陈光辉. 水稻根系育种的研究现状及展望. 湖南农业大学学报:自然科学版,2009, 35(1): 35-39.
HuangH,ChenGH.Statusandprospectsofresearchonricerootbreeding. J Hunan Agric Univ: Nat Sci, 2009, 35(1): 35-39. (inChinesewithEnglishabstract)
[35]魏磊, 董华林, 武晓智, 等. 水稻根系育种研究进展. 湖北农业科学, 2012, 51(11): 2161-2163.
WeiL,DongHL,WuXZ,etal.Researchadvancesonricerootbreeding. Hubei Agric Sci, 2012, 51(11): 2161-2163. (inChinesewithEnglishabstract)
[36]魏道智. 根系的研究进展. 中国农学通报, 2009, 25(17): 105-112.
WeiDZ.Advancesofresearchonroots. Chin Agric Sci Bull, 2009, 25(17): 105-112. (inChinesewithEnglishabstract)
[37]蔡昆争. 作物根系生理生态学. 北京: 化学工业出版社, 2011, 149-162.
CaiKZ.PhysiologicalEcologyofCropRoots.Beijing:ChemicalIndustryPress, 2011: 149-162.(inChinese)
[38]UgaY,HanzawaE,NagaiS,etal.IdenticationofqSOR1,amajorriceQTLinvolvedinsoil-surfacerootinginpaddyelds. Theor Appl Genet, 2012, 124(1): 75-86.
[39]KamoshitaA,WadeJ,AliL,etal.MappingQTLsforrootmorphologyofaricepopulationadaptedtorainfedlowlandconditions. Theor Appl Genet, 2002, 104(5): 880-893.
[40]MitsuhiroO,TamuraW,EbitaniT,etal.Fine-mappingofqRL6.1,amajorQTLforrootlengthofriceseedlingsgrownunderawiderangeofNH4+concentrationsinhydroponicconditions. Theor Appl Genet, 2010, 121(3): 535-547.
[41]SteeleKA,PriceAH,WitcombeJR,etal.QTLsassociatedwithroottraitsincreaseyieldinuplandricewhentransferredthroughmarker-assistedselection. Theor Appl Genet, 2012, 126(1): 101-108.
[42]KirkGJD,GeorgeT,CourtoisB,etal.Opportunitiestoimprovephosphorusefciencyandsoilfertilityinrainfedlowlandanduplandriceecosystems. Field Crops Res, 1998, 56(1): 73-92.
[43]IsmailAM,HeuerS,MichaelJ,etal.Geneticandgenomicapproachestodevelopricegermplasmforproblemsoils. Plant Mol Biol, 2007, 65(4): 547-570.
[44]KamoshitaA,ZhangJ,SiopongcoJ,etal.Effectsofphenotypingenvironmentonidentificationofquantitativetraitlociforricerootmorphologyunderanaerobicconditions. Crop Sci, 2002, 42(1): 255-265.
收稿日期:2015-04-20; 修改稿收到日期: 2015-07-30。
基金项目:国家科技支撑计划资助项目(2011BAD35B02); 浙江省自然科学基金资助项目(LQ14C130003)。
中图分类号:Q343.1+5; S511.032
文献标识码:A
文章编号:1001-7216(2016)04-0363-08
IdentificationofqRL4,aMajorQuantitativeTraitLocusAssociatedwithRiceRootLength
XUXiao-ming1,2,3,#,ZHANGYing-xin1,3,#,WANGHui-min1,4,RENCui5,WANGRu-ci1,3,SHENXi-hong1,3,ZHANXiao-deng1,3,WUWei-xun1,3,CHENGShi-hua1,3,*,CAOLi-yong1,3,*
(1NationalRiceImprovementCenter,ChinaNationalRiceResearchInstitute,Hangzhou310006,China;2HangzhouNormalUniversity,Hangzhou310036,China;3KeyLaboratoryforZhejiangSuperRiceResearch,Hangzhou310006,China;4JiangxiAcademyofAgriculturalSciences,Nanchang330200,China;5HenanAgriculturalUniversity,Zhengzhou450002,China;*Correspondingauthors,E-mail:shcheng@mail.hz.zj.cn;caolycgf@mail.hz.zj.cn)
Abstract:To identify the qRL4, rice lines derived from the hybrid rice Xieyou 9308 with genetic backgroud of Zhonghui 9308 (R9308) and substituted segements from Xieqingzao B (XB) were agar-cultivated for phenotyping of rice seedling root length and molecular marker-based genotyping. The qRL4 was finally narrowed to a 624.6 kb interval (RM5687-InDel49) on chromosome 4. The identification of qRL4 will be helpful to clarify genetic factors controlling root architecture in rice.
Key words:rice; root length; QTL

