植物Tetraspanins蛋白的研究进展
赵倩倩, 王雨欣
(浙江师范大学化学与生命科学学院,浙江金华 321004)
植物Tetraspanins蛋白的研究进展
赵倩倩, 王雨欣
(浙江师范大学化学与生命科学学院,浙江金华 321004)
摘要Tetraspanins(TETs)是一大类进化性保守的、具有4次跨膜结构域的蛋白超家族,广泛分布于所有多细胞生物体中。对植物TETs蛋白的结构特征、系统进化,以及模式植物拟南芥的TETs蛋白超家族(AtTET1~17)和番茄Tetraspanin3蛋白的生物功能进行综述,旨在为今后更好地研究植物TETs蛋白超家族提供参考。
关键词Tetraspanins;4次跨膜结构域;AtTET1~17;Tetraspanin 3
自1990年Oren等[1]发现TAPA-1(the target of an antiproliferative antibody)以来,到1997年为止,期间发现近20个基因,均编码细胞表面蛋白。其基本结构单元均包含4个疏水的跨模区(transmenbrane domains,TM1~TM4),形成一小、一大2个细胞外环(extracelluar loop,EC1和EC2),一个小的细胞内环( intracellular loop,ICL),以及短的N-末端和C-末端尾部。由此,一个新的基因家族诞生,曾被命名为“四次跨膜超家族”(transmembrane 4,TM4或TM4SF或4TM),或“Tetraspanins超家族”。后被Maecker等[2]提议统一命名“Tetraspanins超家族”。
对TETs超家族的研究,最早起源于动物医学。研究发现,TETs蛋白位于细胞表面或细胞内的膜上,在物种间高度保守,在真核生物中是多样化的;它们在动物中的功能和作用机制也被研究地相当深入:已经证实其在精卵融合[3]、光感受器运行[4]、血管发育[5]、动物免疫应答[6]等方面起重要作用[3];一些研究理论也在医学上被用于尝试某些病毒引起的疾病防治,如人乳头癌病毒[7]等。在植物中对TETs蛋白的研究很少,目前已报道的植物TETs基因也不多。笔者对植物TETs蛋白的结构特征、系统进化,以及几个已报道的TETs基因的研究情况进行综述,旨在为今后更好地研究植物TETs蛋白超家族的生物功能及其应用提供借鉴。
1Tetraspanins的结构
TETs是一类从膜上突出3~5 nm的整合蛋白[8],类似于多细胞动物TETs的核心构造。Boavida等[9]发现植物的TETs蛋白(图1)也包含4个跨膜区(TM1~TM4):一个小的细胞外环(EC1),一个大的细胞外环(EC2),一个极小的细胞内环(ICL),一个N-末端和一个C-末端。
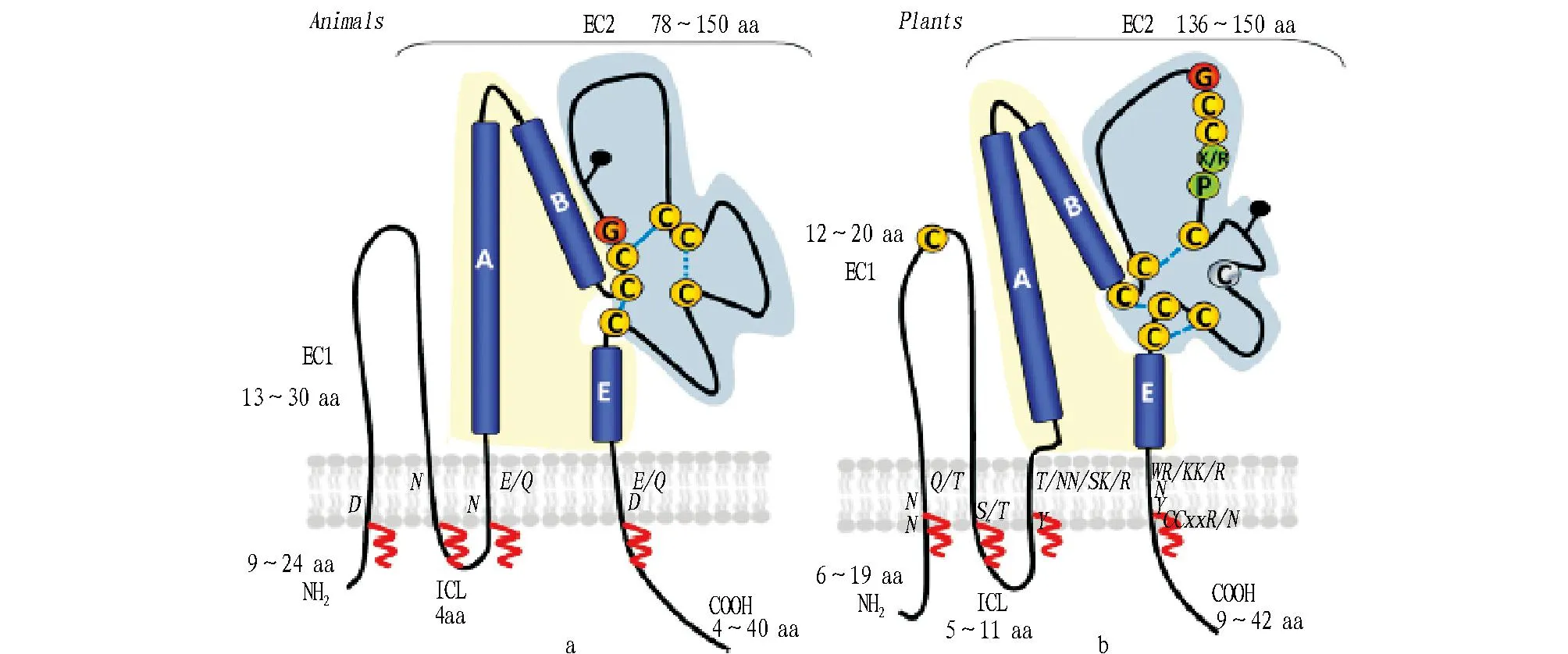
图1 动物(a)和植物(b)中Tetraspanins的拓扑结构示意[9]Fig.1 Schematic diagram of Tetraspanins topological structures in animal(a)and plant(b)
1.1跨膜区(TM1~TM4)跨膜区占TETs本身的50%,是整个分子中最保守的部分。在所有的TETs蛋白中,位于TM1、3和4部分的氨基酸残基(Asn、Gln和Glu)是高度极性化的,且这些氨基酸残基70%~90%是保守的[10],它们能够在彼此间或与其他氨基酸残基间形成强烈的氢键,从而稳定跨膜区的三级结构[11]。
1.2胞外区(EC1和EC2)与跨膜区和胞内区相比,胞外区更多地表现出序列趋异。如在人类和斑马鱼的TETs中,EC1和EC2仅显示43%~58%的一致性,而跨膜区和胞内区显示出高达72%~83%的一致性[10]。
1.2.1小细胞外环(EC1)。目前,对于EC1的了解较少。植物TETs蛋白在EC1包含一个保守性的Cys残基,而动物TETs的EC1却没有;暗示此Cys残基能够交联到EC2上的Cys残基上。此外,Masciopinto等[12]研究发现,在动物TETs蛋白成员CD81中,EC1是EC2的表面最佳表达所必需的,EC1也许可以协助折叠形成EC2[13]。
1.2.2大细胞外环(EC2)。EC2一直受到更多的关注,被研究的也比较清楚。研究推测,植物TETs蛋白的EC2也由2个亚域构成:保守性亚域和可变性亚域(图1黄色和蓝色底纹部分)。保守性亚域在近膜侧,包含3个螺旋(螺旋A、B和E);可变性亚域连续地插入保守性亚域,位于保守性亚域的顶端,在大小、二级结构和折叠方式上极其多变。这2个亚域的相对拓扑学结构,取决于核心二硫桥的出现和TETs蛋白的独特亚型数量[14]。但植物TETs的EC2包含9个Cys残基,可能形成更加复杂的二硫桥,而不是动物TETs典型的4~6个Cys残基;植物EC2的可变性亚域包含一个保守性的植物专一性“GCCK/RP”模域,在位置和序列上不同于动物TETs蛋白的“CCG”模域。
1.3胞内区(ICL,N-末端和C-末端)除EC2的可变性区域外,N-末端和C-末端是TETs蛋白中的2个可变区;但近膜侧的某些Cys残基是相对保守的、潜在的棕榈酰化位点(图1红色波浪线部分),这有助于TETs和其互作物间的作用。已经证实,在动物的TETs蛋白中,C-末端在细胞粘着[15]、膜融合[16]和溶酶体靶定位[17]过程中起重要作用[12]。截至目前,对ICL的研究较少。在ICL上,也存在棕榈酰化位点,可能有助于TETs和其伴侣蛋白(tetraspanin-partner)间的互作[18]。
2系统进化分析
研究表明,从原生动物到多细胞动物,从真菌(酵母除外)到植物和哺乳动物,TETs蛋白均存在;它们广泛存在于几乎所有的生物体内,表明TETs经历了漫长的进化历程。
目前,TETs蛋白超家族在哺乳动物中有33个成员,在果蝇中有37个成员,在秀丽隐杆线虫中有20个成员,在真菌中有3个成员和一个类似于TETs家族的成员[19]。因为酵母体内不存在TETs,在其基因组内,即使是远亲的同源物也未被找到一个[20],因此,TETs最初被认为仅存在于多细胞生物体内。
随后在原生动物变形虫Entamoebahistolytica体内鉴定出TETs蛋白,且发现在单细胞生物体变形虫D.discoideum中,TspanE蛋白包含多细胞动物典型的“CCG”模域;而其他4个Tspans却表现出一个修饰的“CCK/Y/C”模域,这类似于一些陆生植物突变体中的模域结构[21]。这表明TETs可能在单细胞到多细胞的过渡中行使功能。
另外,苔藓P.patens和石松门S.moellendorffii的TETs蛋白,也存在植物专一性的“GCCK/RP”模域,暗示“GCCK/RP”模域早在陆生植物世系之前即出现。原因可能是一个新的世系分支出现[21],或者陆生植物世系趋异分支出来前的一个遗传模域的修饰或者重组。
3植物Tetraspanins蛋白的功能
目前,对植物中TETs的研究远落后于动物。为了调查植物基因组是否编码TETs蛋白,Wang等[22]利用PLAZA对比基因组学平台的方法探究了11个植物物种,共发现155个TETs基因,并以此构建了一个系统进化树。依据拟南芥(Arabidopsisthaliana)的TETs基因,该进化树分为七大组。类似于二穗短柄草、水稻、高粱以及玉米的大多数TETs基因序列处于同一组,大豆和苜蓿也处于同一组,反映了它们的亲密关系。
研究还发现,拟南芥编码17个TETRASPANIN(AtTET1~17)基因,其中大多数的功能未知,唯一进行功能性描述的TETs成员是AtTET1基因;番茄tetraspanin3基因的功能也被报道。
3.1拟南芥AtTET1基因2000年,Cnops等[23]在研究TORNADO1(TRN1)基因时,利用EMS诱变拟南芥Col种子,获得了tornado2(trn2)突变体,描述了其总体形态表型,发现TRN1蛋白和TRN2蛋白均在初生根表皮模式建立中起重要作用,可能在同一形态建成调控途径中起作用。2003年,Olmos等[24]筛选拟南芥T-DNA插入突变体获得了ekeko突变体,鉴定、描述了EKEKO基因(At5g46700人)的结构和功能,发现该基因是植物正常发育所必需的。2004年,Peters等[25]克隆到TRN2基因,和Olmos等[24]鉴定的At5g46700一致。2006年,Cnops等[26]研究发现,TRN2蛋白包含269个氨基酸,可能是一个分泌蛋白,并由4个跨膜区域(TM1~TM4),2个胞外环(EC1和EC2),和胞质N-末端及C-末端。由此推测,TRN2蛋白是拟南芥TETs家族的成员,即拟南芥的TETRASPANIN1(TET1)蛋白,TRN2/EKEKO/TET1和TRN1基因(编码一个富亮氨酸重复蛋白)均在叶片和根的早期发育中起重要作用。2007年,Chiu等[27]对拟南芥的TRN2/TET1基因缺失突变体的研究发现,TRN2/TET1基因能够影响拟南芥顶端分生组织周边区的细胞分裂。2011年,Lieber等[28]在研究高等植物大孢子形成的遗传途径中,发现TET1/EKEKO/TTRN2和WINDHOSE1(WIH1)及WIH2基因(编码一类植物和真菌中存在,动物中却没有的小多肽)共同作用可以促进大孢子产生。
3.2拟南芥tetraspanins超家族的其他AtTETs基因除TRN2/EKEKO/TET1外,拟南芥基因组中还有16个TETs成员(TET2~TET17),但其功能尚不明确。Boavida等[9]研究拟南芥的TETs家族发现,大部分的TETs基因有一个内含子(约1 kb)和一个保守的内含子/外显子连接;TET2、TET5和TET6在编码序列的5′或3′侧多一个小的内含子,而TET10却含有10个内含子。亚细胞定位试验表明,除TET15(由于几个氨基酸残基组成的一个延伸导致其产生一个较长的胞质N-末端)外,拟南芥的其他TETs成员优先定位于质膜上。其中,TET14、TET15和TET17潜在定位于内质网上;而TET3已经明确定位在质膜上,是一个胞间连丝相关蛋白[29]。表达分析试验表明,拟南芥的TETs成员在生殖细胞或生殖组织区域呈现出独特却重叠的表达模式,这表明拟南芥的TETs成员可能存在基因冗余。
当拟南芥的TETs成员在酵母中表达时,它们之间能够形成同二聚体和异二聚体。这也表明拟南芥TETs家族存在功能冗余,且可能源自于TET-TET互作的多样性。
3.3番茄tetraspanin3基因Tetraspanin 3 蛋白是番茄TETs蛋白超家族的一员,裴延飞等[30]研究发现,番茄的tetraspanin3 基因与葡萄的同源基因在一个独立的进化分支上;其在番茄的各器官中均有表达;当正常番茄植株感染黄化曲叶病毒(TYLCV)后,tetraspanin3基因表达量上调;当敲除番茄植株的tetraspanin3基因后,叶片出现扭曲,且在接种TYLCV 后,被感染的TYLCV 病毒量降低50%以上。这表明Tetraspanin 3 蛋白与番茄叶片的生长发育有关,并对TYLCV 侵染番茄植株起正调控作用。
4展望
由于特异的病毒感染需要特别的TETs以被识别并进入细胞,一些哺乳动物的Tetraspanins蛋白成为药物作用的靶点,为预防或治疗相应疾病提供了新途径。而TETs 蛋白的高度保守性研究表明,TETs基因家族具有相似或相近的功能[31],因此,从理论上讲,植物的TETs 蛋白超家族在植物发育过程中有许多重要的作用。然而,目前对植物中TETs基因家族的研究相对较少,其作用机制和生化途径更是鲜有报道。该研究对植物TETs 蛋白超家族的研究情况进行了综述,以期为植物TETs 蛋白的深入研究和将来的实际生产应用提供参考。
参考文献
[1] OREN R,TAKAHASHI S,DOSS C,et al.TAPA-1,the target of an antiproliferative antibody,defines a new family of transmembrane proteins[J].Mol Cell Biol,1990,10(8):4007-4015.
[2] MAECKER H T,TODD S C,LEVY S.The tetraspanin superfamily:Molecular facilitators[J].FASEB J,1997,11(6):428-442.
[4] CONLEY S M,STUCK M W,NAASH M I.Structural and functional relationships between photoreceptor tetraspanins and other superfamily members[J].Cell Mol Life Sci,2012,69(7):1035-1047.
[5] BAILEY R L,HERBERT J M,KHAN K,et al.The emerging role of tetraspanin microdomains on endothelial cells[J].Biochem Soc Trans,2011,39(6):1667-1673.
[6] RUBINSTEIN E.The complexity of tetraspanins[J].Biochem Soc Trans,2011,39(2):501-505.
[7] RICHARDS K F,MUKHERJEE S,BIENKOWSKA-HABA M,et al.Human papillomavirus species-specific interaction with the basement membrane-resident non-heparan sulfate receptor[J].Viruses,2014,6:4856-4879.
[8] CHARRIN S,JOUANNET S,BOUCHEIX C.Tetraspanins at a glance[J].Journal of cell science,2014,127(Pt17):3641-3648.
[9] BOAVIDA L C,QIN P,BROZ M,et al.Arabidopsistetraspanins are confined to discrete expression domains and cell types in reproductive tissues and form homo-and heterodimers when expressed in yeast[J].Plant physiology,2013,163(2):696-712.
[10] STIPP C S,KOLESNIKOVA T V,HEMLER M E.Functional domains in tetraspanin proteins[J].Trends Biochem Sci,2003,28(2):106-112.
[11] SENES A,UBARRETXENA-BELANDIA I,ENGELMAN D M.The Cα-H…O hydrogen bond:A determinant of stability and specificity in transmembrane helix interactions[J].Proc Natl Acad Sci USA,2001,98(16):9056-9061.
[12] MASCIOPINTO F,CAMPAGNOLI S,ABRIGNANI S,et al.The small extracellular loop of CD81 is necessary for optimal surface expression of the large loop,a putative HCV receptor[J].Virus Res,2001,80(1/2):1-10.
[13] RAJESH S,SRIDHAR P,TEWS B A,et al.Structural basis of ligand interactions of the large extracellular domain of tetraspanin CD81[J].Journal of virology,2012,86(18):9606-9616.
[14] MICHEL S,ALIX D,CECILE L G,et al.Structure of the tetraspanin main extracellular domain[J].J Biol Chem,2001,276(43):40055-40064.
[15] WANG H X,KOLESNIKOVA T V,DENISON C,et al.The C-terminal tail of tetraspanin protein CD9 contributes to its function and molecular organization[J].J Cell Sci,2011,124(16):2702-2710.
[16] EDRINGTON T C,YEAGLE P L,GRETZULA C L,et al.Calcium-dependent association of calmodulin with the C-terminal domain of the tetraspanin protein peripherin/rds[J].Biochemistry,2007,46(12):3862-3871.
[17] TAKINO T,MIYAMORI H,KAWAGUCHI N,et al,Tetraspanin CD63 promotes targeting and lysosomal proteolysis of membrane-type 1 matrix metalloproteinase [J].Biochem Biophys Res Commun,2003,304(1):160-166.
[18] MAZUROV D,HEIDECKER G,DERSE D.The inner loop of tetraspanins CD82 and CD81 mediates interactions with human T cell lymphotrophic virus type 1 Gag Protein[J].J Biol Chem,2007,282(6):3896-3903.
[19] HUANG S,YUAN S,DONG M,et al.The phylogenetic analysis of tetraspanins projects the evolution of cell-cell interactions from unicellular to multicellular organisms [J].Genomics,2005,86(6):674-684.
[20] GARCIA-ESPA A A,CHUNG P J,SARKAR I N,et al.Appearance of new tetraspanin genes during vertebrate evolution[J].Genomics,2008,91(4):326-334.
[21] DESALLE R,MARES R,GARCIA-ESPAA A.Evolution of cysteine patterns in the large extracellular loop of tetraspanins from animals,fungi,plants and single-celled eukaryotes[J].Mol Phylogenet Evol,2010,56(1):486-491.
[22] WANG F,VANDEPOELE K,VAN LIJSEBETTENS M.Tetraspanin genes in plants[J].Plant Sci,2012,190:9-15.
[23] CNOPS G,WANG X,LINSTEAD P,et al.Tornado1 and Tornado2 are required for the specification of radial and circumferential pattern in theArabidopsisroot[J].Development,2000,127(15):3385-3394.
[24] OLMOS E,REISS B,DEKKER K.The ekeko mutant demonstrates a role for tetraspanin -like protein in plant development[J].Biochem Biophys Res Commun,2003,310(4):1054-1061.
[25] PETERS J L,CNOPS G,NEYT P,et al.An AFLP-based genome-wide mapping strategy[J].Theor Appl Genet,2004,108(2):321-327.
[26] CNOPS G,NEYT P,RAES J,et al.The TORNADO1 and TORNADO2 genes function in several patterning processes during early leaf development inArabidopsisthaliana[J].Plant cell,2006,18(4):852-866.
[27] CHIU W H,CHANDLER J,CNOPS G,et al.Mutations in theTORNADO2 gene affect cellular decisions in the peripheral zone of the shoot apical meristem ofArabidopsisthaliana[J].Plant Mol Biol,2007,63(6):731-744.
[28] LIEBER D,LORA J,SCHREMPP S,et al.ArabidopsisWIH1 andWIH2 genes act in the transition from somatic to reproductive cell fate[J].Curr Biol,2011,21(12):1009-1017.
[29] FERNANDEZ-CALVINO L,FAULKNER C,WALSHAW J,et al.Arabidopsisplasmodesmal proteome[J].PLoS ONE,2011,6(4):18880.
[30] 裴延飞,刘廷利,连梓伊,等.番茄中受TYLCV 诱导上调表达基因tetraspanin3 的功能分析[J].南京农业大学学报,2015,38(6):896-900.
[31] HUANG S,TIAN H,CHEN Z,et al.The evolution of vertebrate tetraspanins:Gene loss,retention,and massive positive selection after whole genome duplications[J].BMC evolution biology,2012,10:306.
作者简介赵倩倩(1989- ),女,河南郏县人,硕士研究生,研究方向:水稻抗病基因的克隆和功能。
收稿日期2016-04-10
中图分类号Q 946
文献标识码A
文章编号0517-6611(2016)14-146-03
Research Progress of Tetraspanins Protein in Plant
ZHAO Qian-qian,WANG Yu-xin
(College of Chemistry and Life Science, Zhejiang Normal University, Jinhua, Zhejiang 321004)
AbstractTetraspanins (TETs) is a large class of evolutionary conserved proteins with four transmembrane domains, which are widely distributed in all multicellular organisms. In this research, we reviewed the structural characteristics, system evolution of the plant TETs protein, as well as the biological function of model plant Arabidopsis thaliana TETs protein super family and tomato Tetraspanin3 protein, so as to provide references for better research of plant TETs protein super family.
Key wordsTetraspanins; Four transmembrane domain; AtTET1-17; Tetraspanin3

