基于Android的代谢网络可视化和生化反应搜索的实现
张浩然 张兆祥 刘 娟* 胡黔楠
1(武汉大学计算机学院 湖北 武汉 430072)2(天津工业生物技术研究所 天津 300308)
基于Android的代谢网络可视化和生化反应搜索的实现
张浩然1张兆祥1刘娟1*胡黔楠2
1(武汉大学计算机学院湖北 武汉 430072)2(天津工业生物技术研究所天津 300308)
摘要代谢网络和生化反应代谢路径的研究是合成生物领域的重要内容,目前亟需相关计算机软件来辅助分析。针对这种情况以及目前移动互联网发展趋势,开发了一种基于Android的代谢网络可视化和生化反应搜索引擎系统。该系统将Android作为展示平台,显示代谢网络的三维模型,提供生化反应的搜索,这些都为生物合成和药物发现领域的研究人员提供了参考。系统用美观的三维模型显示代谢网络,同时提供简单的便捷的操作界面供用户查询生化反应。
关键词Android系统代谢网络可视化生化反应
0引言
合成生物学是21世纪初新出现的一门交叉学科,在通过阐明并模拟生物合成的基本规律基础上,将工程学科的理念应用到生物学方面,达到人工设计并构造旧的、拥有特殊生理功能的生物系统,从而开辟廉价生产药物、化学品等的生物制造方法。与传统的生物学研究方式完全相反,合成生物学是从最基本的要素开始逐步构造生物体的零部件直到人工生命系统。
研究基因组规模的代谢网络和不同的生化反应代谢路径是合成生物学的一个重要内容。通过计算机辅助模拟方式研究代谢网络逐渐成为生物学家的一个重要研究手段。而代谢网络的可视化则是研究复杂代谢网络的重要环节,传统的代谢网络可视化工具多以二维方式展示,但是三维可视化可以更加生动地描述代谢网络中的元素,这样既能在宏观层面上直观展示以代谢途径为单位的功能模块,也能微观层面上探索网络元素之间的关联,为生物合成和药物发现领域的研究人员提供了参考[1]。生化反应在维持生命和促进细胞生长的过程中起着非常重要的作用,因此本文整合了RxnFinder搜索引擎来方便研究人员搜索生化反应,RxnFinder是一个专业的生化反应搜索引擎,用于从KEGG反应数据库中搜索反应,生物信息学数据库KEGG是由日本京都大学生物信息学中心的Kanehisa实验室建立,它包含了超过8000条生化反应[2]。
互联网的快速发展带动了其他行业的快速发展,未来互联网也在逐渐从传统互联网向移动互联网转变,移动互联网的高速发展也为生物技术发展带来契机。本文开发一款基于Android操作系统的代谢网络的三维可视化,同时整合了生化反应的搜索引擎,充分利用移动客户端的优势,方便研究人员研究代谢网络和查询生化反应。
1系统结构设计
1.1系统总体设计
新一代的互联网程序朝着面向服务,面向应用,面向资源的方向发展。同时在目前移动互联网终端用户数量爆发性增长的大趋势下,针对具体需求分析,本文采用基于C/S的系统架构,充分利用服务器和Android客户端硬件环境的优势,将任务合理分配到客户端和服务器端分别实现,既充分利用了客户端的计算能力减轻了服务器端运行负荷,又降低了服务器与客户端通讯所产生的流量开销。本文的整个系统架构如图1所示,用户通过Android客户端来进行交互,Android客户端将用户的请求发送到Web服务器,Web服务器负责接收客户端的请求分析处理后进一步请求后台数据库来提供查询与存储支持。在后台服务器选择上,由于需要与武汉大学组合生物合成和新药发现教育部重点实验室平台进行整合,从兼容性和可维护性方面考虑,决定使用由Python语言实现的开源框架Django,也方便了将来进行扩展。服务器端数据库则采用加州大学伯克利分校计算机系开发的PostgreSQL数据库,PostgreSQL不仅提供了良好的可移植性方便了数据的转移,同时其丰富的引擎选择为应用提供了良好的性能保证。
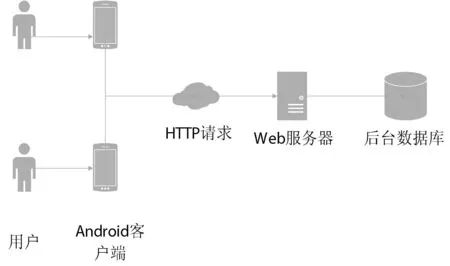
图1 系统总体架构图
1.2Android客户端设计
本文的Android系统的总体结构如图2所示。Android程序整体就是一种典型MVC结构,其中M指逻辑模型,V指视图模型,C指控制器。视图层一般采用层次化的XML配置文件来描述,控制层一般通过众多的Activity协同处理来实现,模型层则是处理数据库和网络等耗时的IO操作。MVC模式将应用程序的逻辑控制与界面展示完全分开,互不干扰。这种模式既方便了与他人进行合作开发,每人只需要开发自己的功能模块而不会相互影响,也方便了以后修改业务流程或业务规则改变时,只需要改动MVC的模型层,而不需要对整个系统进行太大的改动[3]。
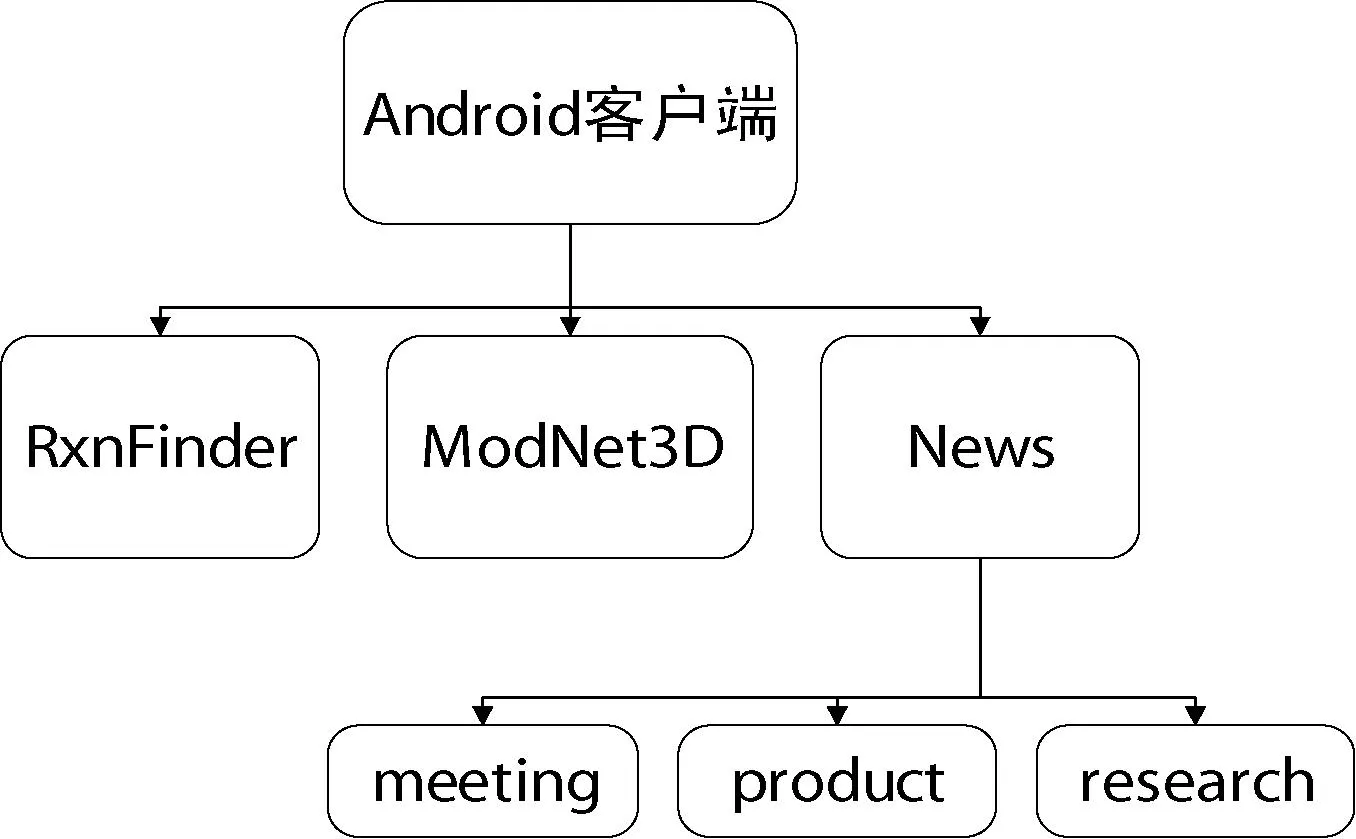
图2 Android软件结构图
本文的Android软件主要分为三个部分:生化反应的搜索(RxnFinder),代谢网络的三维可视化(ModNet3D),生物合成领域的新闻显示(News)。
(1) 本文目前的代谢网络主要是基于大肠杆菌的代谢网络,通过专门针对手机等嵌入式设备而设计的OpenGLES进行渲染,最终绘制成球棍状三维模型。根据代谢网络的定义,代谢网络可以划分为三个层次:代谢途径,反应以及化合物。因此,在代谢网络三维模型中,球体代表化合物,棍代表化合物之间的反应,而每一个连通的图则是一个代谢途径(Pathway)。用户既可以通过PathwayID、PathwayName来搜索指定的Pathway,也可以通过CompoundID、CompoundName来搜索包含指定Compound的Pathway。
(2) 生化反应的搜索数据主要来源于KEGG反应数据库,用户能够通过MoleculeName(分子名称)、ECNumber(反应酶的代号)、PathwayName(途径名称)来搜索相关的生化反应。用户可以查看反应的详细信息,包括反应两端的分子结构,反应的催化酶的详细信息。
(3) 新闻模块则显示一些生物合成相关的科研、会议、产品等新闻。
1.3服务器端设计
服务器端的主要功能是处理客户端KEGG反应数据库的信息和新闻信息的请求,服务器前端使用了基于异步IO并发的Nginx服务器,并通过多重机制保证服务效率如Web响应缓存机制,基于Apache的多路负载均衡,GZIP压缩等并支持用户身份验证操作。将需要频繁更新的数据放在服务器端方便以后对数据进行增删改,而Android客户端本地不需要改任何代码就可以获取最新数据。当客户端的HTTP请求到来时,服务器端会对客户端的请求进行解析,查询WEB缓存,缓存命中直接将结果返回到客户端,否则将访问后台数据库获取数据后返回到客户端。本文主要通过JSON(JavaScriptObjectNotation)来传递服务器和客户端之间的数据。JSON是一种轻量级的数据交换格式,主要使用键值对形式保存数据,并支持与许多数据类型的原生转换,另外与常规使用的XML格式相比,减少了大量的冗余信息,既可以加快客户端与服务器端的通信速度,还可以节约手机客户端的流量。
2主要算法实现
为了能够将代谢网络较好地展示成三维模型,本文使用受力驱动布局中的KamadaKawai(KK)算法。这也是代谢网络可视化的核心算法。KK布局算法将节点之间的边看成弹簧,无论弹簧是被压缩还是被拉伸,弹簧都会存在能量。如果两个节点相距过近,则弹簧会被压缩,算法会自动将两个节点距离分开,相反,如果两个节点相距过远,则弹簧被被拉伸,算法就会将两个节点距离拉近。KK布局算法会自动调整所有顶点,直到整个代谢网络达到能量最小值[4]。
算法输入:图G= (V,E)
算法输出:V中所有顶点坐标值
算法评价函数如下所示:
首先初始化顶点集V的坐标,(xi,yi,zi)和(xj,yj,zj)分别表示顶点(vi,vj)的三维坐标,ki,j表示弹簧的弹性系数,li,j表示弹簧间初始长度,算法的目标就是寻找出点集V中所有点的坐标位置,使E最小。由于寻找最优解是一个NP完全问题,因此本文采取了模拟退火算法来求出趋于全局最优解的最优解。其基本思想如下:
(1) 初始化顶点V的坐标,初始化解状态S,初始化温度T。
(2) 随机选取一个顶点在空间内随机移动一小段距离,得到新解状态S′,计算E(S′)。
(3) 如果E(S′) (4) 如满足终止条件,则将当前解作为最优解,结束算法,否则,将T减少,转第(2)步。 3Android软件实现与测试 3.1Android软件实现 在开发环境的选择上,虽然Google推出了自己的Android开发环境AndroidStudio,但是由于最近才发布,目前还存在不少bug待解决,因此最终采用Eclipse+ADT开发环境,同时采用AndroidSDK4.4.2+JavaJDK6开发平台,程序编译完成后就会生成apk文件,拷贝到移动终端就可以编译运行[5]。 本地数据存入SQLite数据库中,软件采用模块化开发思想,提供详细的对外接口,做到高内聚,内耦合方便后续修改和增加新功能,并通过github进行版本控制,可以很方便地与他人一起开发。 Github上有众多开源Android项目可以供我们学习使用,同时我们也可以修改别人的开源代码运用到自己的项目上。本文主要使用了JPCT-AE,async-http-client等开源库。 JPCT-AE是一款封装了OpenGLES的3D游戏引擎,开发人员只需要调用它提供的API就可以实现一些很酷的三维效果,而且可以在Android平台上实现快速渲染。async-http-client是一个封装了Apache的HttpClient开源库,但是它所有的Http请求都会在主UI线程之外执行,而且回调函数会在Android主UI线程执行,因此你不需要担心常见的ANR(ApplicationNotResponding)问题。 3.2Android软件测试 3.2.1RxnFinder模块 RxnFinder模块通过MoleculeName,ECNumber,PathwayName从后台数据库中查询生化反应。例如,用户可以选择PathwayName,然后输入methioninebiosynthesis(甲硫氨酸生物合成),就可以搜索到这个pathway相关的一些反应,可以进一步点击反应列表来查看反应详细信息,如图3所示。 图3 基于Android的生化反应搜索效果图 3.2.2ModNet3D模块 ModNet3D模块通过OpenGLES库来展示代谢网络的三维模型[6]。用户可以直接选择Pathway来搜索Pathway的三维模型,也可以通过选择compound来搜索包含这个compound的pathway三维模型。例如,用户选择polymyxinresistance(多粘菌素抗性途径)这个Pathway,手机屏幕上就会显示这个pathway的代谢网络。如图4所示,用户可以滑动屏幕来旋转三维模型,还可以通过手指来放大和缩小三维模型。当用户点击一个球体(化合物)时,软件会显示化合物的详细结构和名称。因为绝大部分的反应是在酶的作用下发生的,所以当用户点击一条边(生化反应)时,软件会自动跳转到Expasy网站查询作用于这个反应的酶的详细信息。Expasy是由瑞士生物信息学研究所维护的一个专业的蛋白质分析系统,是一个生物信息的门户。 图4 基于Android的代谢网络三维图 3.2.3News模块 News模块主要提供生物合成领域科研,会议,产品等新闻信息。当用户点击某个新闻模块时,服务器端的新闻信息列表以Json格式传递给客户端,客户端只需要进行解析并显示详细新闻列表。当用户点击某个具体新闻时,就会链接到该新闻的网站,显示详细新闻信息。所有的新闻数据都存放在服务器端,更新时只需要更新服务器端新闻信息,而已经浏览过的新闻数据则存储在本地sqlite数据库中,以免再次更新新闻损耗流量。 4讨论 本文开发的Android软件实现了代谢网络可视化,整合了生化反应搜索引擎RxnFinder。针对基因组规模的代谢网络,使用经典布局算法,设计了一个三维可视化系统ModNet3D。与电脑相比,手机屏幕上显示三维模型更易操作,用户只需要使用一些简单的手势就可以操作这个三维模型。客户端目前提供了以大肠杆菌代谢网络为例的可视化分析,为生物合成和药物研发领域的研究人员提供了使用案例。同时,以后制作了新的代谢网络,只需要在服务器端更新,然后在Android本地端进行同步后就可以通过客户端来观察新的代谢网络。生化反应搜索引擎可以通过分子名称,反应酶代号,途径名称来搜索相关反应的详细信息,用户可以很快捷地查询反应表达式,分子结构,反应酶信息等,极大方便了生物合成领域研究人员检索KEGG反应[7,8]。 本系统目前仍然存在一些不足之处,这也是下一步改进的方向。由于手机屏幕的限制,无法像电脑屏幕显示大量信息,所以只能显示用户选择的具体代谢途径,无法把整个大肠杆菌的代谢网络显示在手机屏幕上。另外受服务器端系统规模限制对 移动端用户数量支持有限,当用户达到一定规模后,会出现反应迟钝的假死现象,后续通过分布式架构改进它。 5结语 本文针对基因组规模的代谢网络,设计了一个基于Android的三维可视化系统ModNet3D,同时为了方便研究人员对生化反应进行分析,整合KEGG反应数据库,提供了一系列方式供用户搜索生化反应。随着移动互联网的快速发展,未来合成生物领域与移动互联网之间会产生更多的交集,会有越来越多的开发人员开发生物领域相关的软件。因此,目前亟需一个开放的平台,可以供互联网研究人员与生物研究人员交流的平台,相互促进发展,使移动互联技术在生物领域产生更大的影响。 参考文献 [1] 许晓彤.基因组规模代谢网络的三维可视化系统的设计与实现[D].武汉:武汉大学计算机学院,2013. [2]HuQN,DengZ,HuHN,etal.RxnFinder:biochemicalreactionsearchenginesusingmolecularstructures,molecularfragmentsandreactionsimilarity[J].Bioinformatics,2011,27(17):2465-7. [3]RetoMeier.Android4高级编程[M].北京:清华大学出版社,2013. [4]BeckerMY,RojasI.Agraphlayoutalgorithmfordrawingmetabolicpathways[J].Bioinformatics,2001,17(5):461-467. [5]WeimengLee.Android编程入门经典[M].北京:清华大学出版社,2012. [6]KevinBrothaler.OpenGLES应用开发实践指南:Android卷[M].北京:机械工业出版社,2014. [7]CroftD,O’KellyG,WuG,etal.Reactome:adatabaseofreactions,pathwaysandbiologicalprocesses[J].NucleicAcidsRes,2011,39(Databaseissue):D691-7. [8]KanehisaM,GotoS,FurumichiM,etal.KEGGforrepresentationandanalysisofmolecularnetworksinvolvingdiseasesanddrugs[J].NucleicAcidsRes,2010,38(Databaseissue):D355-60. IMPLEMENTATION OF ANDROID-BASED METABOLIC NETWORKS VISUALISATIONANDBIOCHEMICALREACTIONSEARCH Zhang Haoran1Zhang Zhaoxiang1Liu Juan1*Hu Qiannan2 1(School of Computer,Wuhan University,Wuhan 430072,Hubei,China)2(Tianjin Institute of Industrial Biotechnology,Tianjin 300308,China) AbstractResearch on metabolic networks and metabolic pathway of biochemical reaction is important for biosynthesis, and the related computer software is urgently needed at present to carry out assistant analysis. In light of this situation and based on current development trend of mobile Internet, we developed an Android-based system for metabolic networks visualisation and biochemical reaction search engine. The system regards the Android as the display platform, it demonstrates the 3D model of metabolic networks and provides biochemical reaction searches, both of these provide the references for researchers in biosynthesis and drug discovery fields. The system displays the metabolic networks with beautiful 3D model, and meanwhile presents simple and convenient operation interface for users to search the biochemical reactions. KeywordsAndroid systemMetabolic networkVisualisationBiochemical reaction 收稿日期:2014-12-26。张浩然,硕士生,主研领域:数据挖掘。张兆祥,硕士生。刘娟,教授。胡黔楠,教授。 中图分类号TP311 文献标识码A DOI:10.3969/j.issn.1000-386x.2016.06.054


