鹿科6属MHC-DRA第二外显子变异特点及与Cytb系统进化比较
由玉岩, 张成林, 张金国, 阮向东, 张林源, 钟震宇, 郑常明, 刘学锋, 王伟, 曹长雷
(1. 圈养野生动物技术北京市重点实验室 北京动物园, 北京 100044; 2. 国家林业局调查规划设计院,北京 100714; 3. 北京麋鹿生态实验中心, 北京 100076; 4. 长江师范学院, 重庆 408100)
鹿科6属MHC-DRA第二外显子变异特点及与Cytb系统进化比较
由玉岩1, 张成林1, 张金国1, 阮向东2, 张林源3, 钟震宇3, 郑常明1, 刘学锋1, 王伟1, 曹长雷4
(1. 圈养野生动物技术北京市重点实验室 北京动物园, 北京 100044; 2. 国家林业局调查规划设计院,北京 100714; 3. 北京麋鹿生态实验中心, 北京 100076; 4. 长江师范学院, 重庆 408100)
摘 要探讨了鹿科6属动物主要组织相容性复合体dra(mhc-dra)第二外显子的变异特点,揭示其分子水平上的进化选择模式,并与cyt b进行了比较。采用测序法,获得鹿科6属动物dra第二外显子246 bp的序列。研究结果表明,多态性位点数18个,独立变异位点13个。mhc-dra第二外显子显示出较低的核苷酸变异,且6属有各自独特的变异位点。氨基酸序列中抗原结合位点共计20个。其中,鹿科6属动物的氨基酸位点63,驼鹿属氨基酸位点18,是不同于马属和人属的抗原结合位点,推测鹿科动物可能有着不同的抗原结合模式。根据非同义替代率(Ka)和同义替代率(Ks)比值的计算结果,表明鹿属mhc-dra第二外显子为正向选择,其余5属均为纯化选择。对mhc-dra第二外显子及cyt b分别运用邻接法(NJ),最大似然法(ML)和贝叶斯推论(BI)进行系统发育重建得到相似结果,形成了旧世界鹿类与新世界鹿类的姐妹类群。麂亚科cyt b和mhc-dra第二外显子的基因型可能是鹿亚科与其它亚科之间的过渡型,而气候变化和环境变迁可能是推动mhc-dra第二外显子分化的潜在原因。
关键词mhc-dra;分子进化;系统进化;选择压力
主要组织相容性复合体(Major histocompatibility complex,mhc)位于动物染色体的特定区域,是编码主要组织相容性抗原的一组紧密连锁的基因群。在脊椎动物中,mhc是最具多态性的基因区域,其核苷酸多样性水平是基因组平均水平的2倍,是探讨遗传多样性变化模式较为理想的基因[1,2]。同时,mhc在动物免疫系统中也发挥着非常重要的作用[3,4]。当动物暴露于病原体或寄生虫等的环境中时,可驱动并保持其天然种群mhc等位基因的多样性,并随时间的累积,等位基因多样性逐步升高,以此来增加免疫基因应对外环境中多样病原体的能力[5]。而mhc等位基因的跨物种多样性已经在很多中性选择系统中被证实,其中包括鱼类[6]、啮齿动物[7]、有蹄动物[8]、食肉动物[9]和灵长类动物[10]等。mhc家族里大部分基因都呈高度多态,而mhc-dra相对来说则很保守[11]。dra属于mhc-Ⅱ,编码Ⅱ类抗原分子的a链,在抗原识别和呈递、免疫应答与调控等方面起着重要的作用,是疾病抗性与易感性的候选标记基因[12]。目前已证实mhc-dra分子的变异与家畜的一些疾病存在着相关性[13]。
线粒体DNA(Mitochondrial DNA, mtDNA)为核外遗传物质,是共价闭合的双链,其结构简单,呈母性遗传,进化速度快且不发生重组,是一种应用广泛的分子标记[14]。其中,细胞色素b(Cytochromeb,cytb)是构成线粒体氧化磷酸化系统复合体III的蛋白质之一,也是其中唯一由线粒体基因组编码的蛋白质。基因在线粒体基因组中进化速度适中,能包含从种、属水平的系统发育信息[15]。而且在一定的进化尺度内,基因不受饱和效应的影响[16],目前已被广泛应用于动物种内或近缘种间系统发育[17-20]、遗传多样性[21]、系统进化和分类等方面的研究[22, 23]。
鹿科动物多为野生种群,它们分布于世界各地,在维持自然界生态环境的稳定性中占有重要地位[24]。早在21世纪90年代初,已应用不同遗传标记对鹿科动物开展了研究,阐明物种间的现行关系和历史渊源,如通过12S rRNA、16S rRNA和D-Loop区来探讨鹿亚科、麂亚科、獐亚科以及空齿鹿亚科系统发生关系等方面的研究[25-27],但针对鹿科动物mhc的进化机制还有待深入研究。鹿科(Cervidae)mhc与人类mhc相类似,均由多个高度多态性的基因座组成I类、Ⅱ类和Ⅲ类基因组成,且结构和功能皆较保守[28]。随着对野生动物mhc研究的不断深入,针对鹿科动物mhc的研究也逐步开展,如对白尾鹿(Odocoileusvirginianus)mhc-drb等位基因多样性的研究[29],白尾鹿、马鹿(Cervuselaphus)、狍(Capreoluscapreolus)及驼鹿(Alcesalces)间mhc-drb系统进化关系的研究[29],驯鹿、马鹿与圈养羊、牛等跨物种mhc-dra系统进化关系的研究[30],以及对林麝(Moschusmoschus)dra和drb特点的研究[31]。由于mhc作为抗病研究的主要候选基因,研究其基因家族具有较好的应用价值[12]。因此,本研究以mhc-dra这一疾病抗性与易感性的候选基因为遗传标记,对鹿科中的獐属(Hydropotes)、驼鹿属(Alces)、驯鹿属(Rangifer)、麂属(Muntiacus)、麋鹿属(Elaphurus)和鹿属(Cervus)mhc-dra的变异特点,系统进化关系及分子水平上的进化选择模式进行探讨,为我国鹿科6属动物的疾病预防,遗传保护等提供分子生物学依据。同时,通过与进化速度适中且能包含种、属水平系统发育信息的cytb的进化情况进行比较,进一步揭示mhc-dra的进化历程,探讨其起源与分化关系。
1材料与方法
1.1 样本采集与基因数据的获得
本研究中的鹿科4属5种的组织样本来自于北京动物园的历史存留的样本,另外2属3种的mhc-dra第二外显子序列来自Genbank;cytb序列均来源于GenBank(表1)。待测样本保存于-80℃超低温冰箱。

表1 鹿科6属动物mhc-dra第二外显子及cyt b序列来源
括号内的数字表示个体数量
1.2基因组提取及目的基因获得
全基因组使用组织与血液基因组提取试剂盒(Qiagen, Valencia, CA)进行提取。使用引物对dra(F:5′-CAACTCATCCCTACTCTCCATCAC-3′;R:5′-AGGCGAAAGTCTCAGTAACAAAAA-3′)对mhc-dra片段进行扩增。PCR反应总体积为25 μL,模板浓度30~50 ng,75 mmol/L Tris-HCl(pH9.0),50 mmol/L KCl,2.5 mmol/L MgCl2,20 mmol/L (NH4)2SO4,0.01% (v/v) Tween-20,0.2 mmol/L dNTP,引物各10 μmol/L,1 U 的TaKaRaTaqDNA聚合酶。反应条件为:95℃预变性3 min;95℃变性15 s,56.5°C退火30 s, 72°C延伸30 s,40个循环; 72℃再延伸10 min。扩增产物由上海生工进行测序。
1.3数据分析
结合从GenBank中下载的序列,使用软件BioEdit 7.0.5.3 和Clustal X 1.81对所得序列进行序列编辑、比对和剪切。使用MEGA4.1软件计算核苷酸和氨基酸的多态位点数(polymorphic sites),独立变异位点数(singleton variable sites),平均碱基组成(average nucleotide composition),属间遗传距离(基于Kimura 2-parameter替换模型)进行计算。使用软件DNAsp4.0对Ka(非同义替代率)和Ks(同义替代率)的值进行计算[32]。目前,Ka/Ks被广泛地应用于检测基因的选择压力,当二者比值大于1时表明突变导致了氨基酸的改变,即为正向选择(positive selection),若小于1则表明选择为纯化选择(purifying selection)。
利用Modeltest3.06[33]进行系统进化重建模型选择,计算相关参数。本研究针对鹿科7属DRA和cytb序列分别运用邻接法(Neighbor-joining method, NJ),最大似然法(Maximum likelihood method, ML)和贝叶斯法(Bayesion inference,BI)进行系统发育重建。运用PAUP4.0b10软件进行系统进化树NJ和ML的重建。ML树的节点支持率通过100次重抽样的方式进行估计。BI树通过使用软件MrBayes 3.1进行重建;对两个并行的马尔科夫链(Monte carlo markov chains)运行1 000 000代,每100代抽样1次,直至两个并行的马尔科夫链平均标准偏差低于0.01。放弃前10%的抽样树,其余的保留用以构建BI树。所有构建的系统进化树均以亚洲野牛(Bosgaurus)dra第二外显子和cytb序列作为外群(基因库登录号分别为AF385486和AB077316)。
通过MDIV软件对属间分歧时间进行计算。MDIV软件使用HKY模型,马尔科夫链 (Markov chain) 运行2 000 000次,放弃前10%的分析结果。Mmax和Tmax分别设定为10,并以最高的后验概率值对T和θ似然值进行计算[34]。MDIV计算分歧时间t= Tpop*(θ/2μk)。其中,Tpop是种群分歧时间,θ是种群核苷酸突变率,μ是每个核苷酸的进化速率,k是核苷酸序列的碱基数。种群分歧时间根据鹿科动物线粒体cytb的进化速率约为每百万年5.14%[35]和哺乳动物mhc进化速率为每百万年1.56%[36]进行计算。
2结果
2.1鹿科6属mhc-dra序列组成及变异
通过对所得鹿科6属动物dra第二外显子序列的比对和剪切,获得246 bp的序列。其中,多态性位点数18个,独立变异位点13个。其中,多态性位点占总位点数的3.252%。属间平均遗传距离为0.018(表2)。鹿科6属dra序列比对后显示出较低的核苷酸变异(图1)。在所有6属中,dra序列的属间遗传距离范围在0.004 ~ 0.041(表2)。鹿科6属动物核苷酸A、T、C、G所占比例分别为 0.261,0.246,0.214和0.279(表3)。

图1 鹿科6属mhc-dra核苷酸组成比较
黑色和灰色*共同表示多态性位点数;灰色*表示独立变异位点;属的单倍型以序号表示
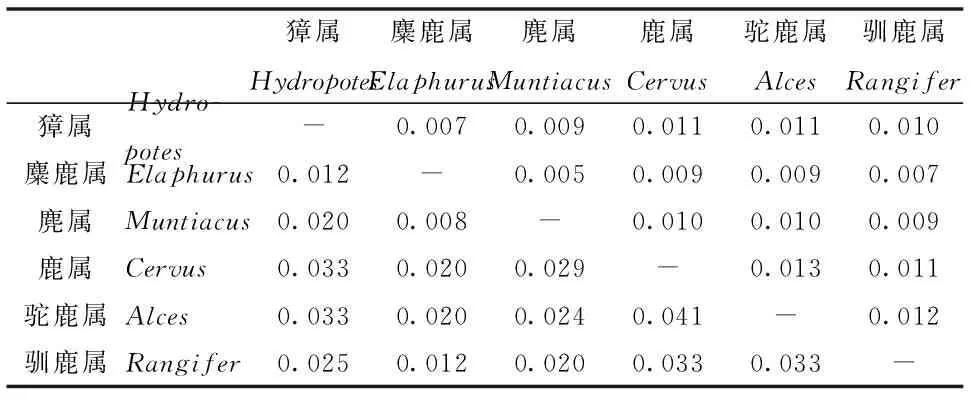
表2 鹿科6属动物间mhc-dra第二外显子序列的遗传距离
下三角数据为配对遗传距离;上三角数据为配对遗传距离的标准误
鹿科6属的分析均以各属的单倍型进行分析。在鹿科动物中,除麋鹿属外,其它5属的mhc-dra序列均有各自独特的变异位点。如,獐属位点68、236,麂属位点33、179、245,鹿属位点5、80、131、140、160、244,驼鹿属位点3、20、58、228,以及驯鹿属位点31、51均为其各自的特有变异位点。
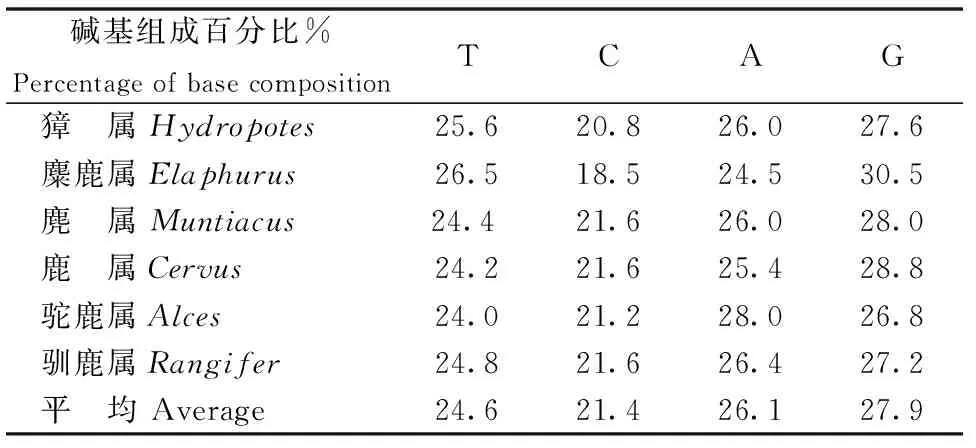
表3 鹿科6属的平均碱基组成
2.2鹿科6属mhc-dra氨基酸序列组成及变异
研究结果表明,鹿科6属mhc-dra氨基酸序列的多态位点共计7个,占分析总氨基酸数的8.536%。鹿科6属氨基酸的抗原结合位点是基于人属[24]和马属动物[25]进行推测得出(图2)。獐属的氨基酸位点63,以及驼鹿属氨基酸位点19,是完全不同于马属和人属的一个抗原结合位点。在与多肽侧翼区域相互作用的残基中,氨基酸位点47、76在鹿属与马属中均出现了变异。除上述3个氨基酸位点外,其余鹿科5属动物的变异位点均在多肽结合位点及其相互作用残基区域之外(图2)。根据人属和马属动物的氨基酸结构特点,推测黑色倒三角标记的位点为初级的抗原结合位点;白色倒三角处是与多肽侧翼区域相互作用的残基;蓝色字体标注的氨基酸表示鹿科6属、马属与人属间的氨基酸变异,红色字体标注的氨基酸表示鹿科动物独有氨基酸变异,绿色标注的氨基酸表示鹿科6属、马属和人属共有的氨基酸位点(图2)。

图2 鹿科mhc-dra与其它物种的氨基酸差异
“.”表示与獐属序列比对相一致;“*”为多态性位点,属的单倍型以序号表示
2.3鹿科6属mhc-dra序列的进化选择
mhc-dra核苷酸共编码82个氨基酸(图2)。通过对氨基酸序列比对,发现4个同义位点变异和15个非同义位点变异。整个dra编码区的Ka/Ks值表明鹿属为正向选择(Ka/Ks>1,表4),其余5属均为纯化选择(Ka/Ks<1,表4)。
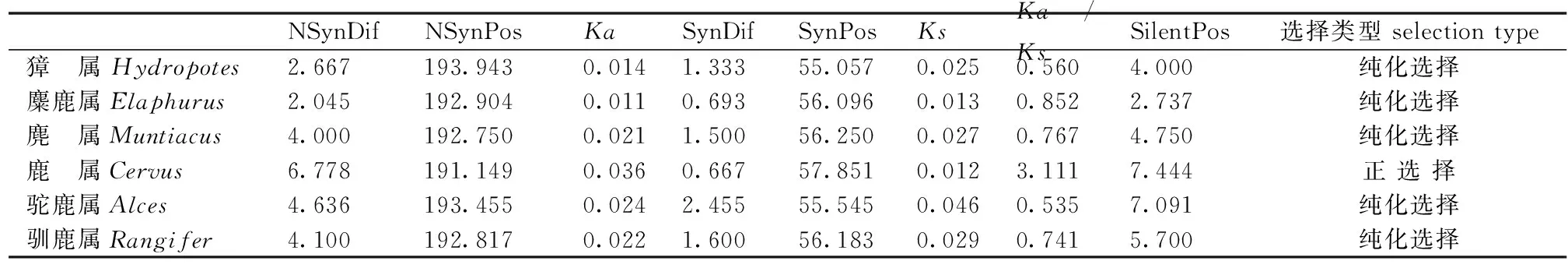
表4 鹿科6属mhc-dra第二外显子进化选择
NsynDif为非同义替换变异数;NSynPos为非同义位点数;SynDif为同义替换变异数;SynPos为同义位点数;Ka为非同义替代率;Ks为每个同义位点的同义替代率;SilentPos为沉默位点数
2.4mhc-dra第二外显子和cytb系统发育重建
通过Modeltest 3.7软件确定mhc-dra第二外显子序列系统发育重建模型为F81+G+I。单倍型的碱基频率A、T、C、G频率分别为 0.261、0.246、0.214和0.279;转换颠换比(transition/transversion,R)为3.476;不变位点比率(Pinvar)为0.09,γ分布型参数为2.42,shape=0.50,-lnL=573.78。
确定cytb的系统发育重建模型为GTR+G+I。单倍型的碱基频率A、C、G和T分别为0.311、0.288、0.131和0.292;转换颠换比为12.03;不变位点比率为0.39,γ分布型参数为25.03,shape=0.49,-lnL=4 807.46。
基于mhc-dra第二外显子以及cytb序列构建的NJ、ML和BI系统发育树具有相似的结构。因此,本研究仅列出了ML树(图3),而ML、NJ和BI树的节点支持率以及各分支的分歧年代详见表5。基于cytb的系统发育分析结果表明,旧世界鹿的种间分化时间大约发生在上新世早期至上新世晚期,新世界鹿的种间分歧大约发生在上新世的中、晚期(表5)。然而,基于mhc-dra第二外显子构建的系统发育树分析结果表明,该基因在进化历史中的分歧时间更晚,且最早的分歧发生在上新世晚期(表5)。

图3 鹿科6属动物mhc-dra第二外显子(A)和cyt b(B)的ML系统进化树
图中依次标注了种名、属名和亚科名;灰色粗体实线表示属,黑色粗体实线表示科,黑色虚线表示新世界鹿类和旧世界鹿类;黑色线段表示单位分支长度;各分支序号代表各结点支持率和分歧年代,详见表5
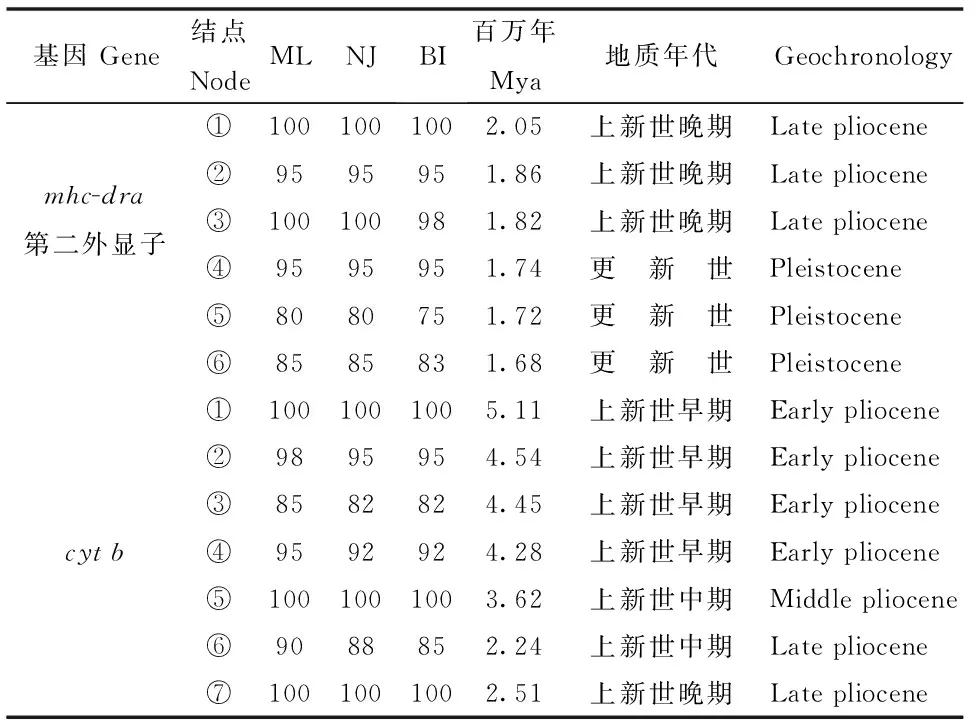
表5 mhc-dra第二外显子和cyt b系统进化树各结点支持率及分歧年代
3讨论
3.1鹿科6属mhc-dra第二外显子核苷酸及氨基酸序列变异
mhc的多样性和选择模式对于理解免疫功能适应性意义是必不可少的[37]。mhc处于机体特异免疫反应的中心地位,各类生物mhc的生物学功能相同[38]。它们参与机体免疫应答、免疫调节,并与某些病理状态有着密切的关系。经典的mhc分子的主要功能是通过外源和自身肽呈递给T-细胞的宿主免疫应答[39]。mhcII则控制着体液免疫应答中的T、B及巨噬细胞间的相互作用,同时也参与细胞免疫[40]。mhc-dra组成了mhcⅡ类分子复合体的a肽链,对于mhcⅡ在细胞表面的稳定表达和抗原肽的有效呈递起到了重要的作用[38]。mhcII遗传背景的差异和多样性水平的高低都会导致个体或不同物种对疾病抗性和易感性的差异[41]。而等位基因多样性是通过物种分化事件中的平衡选择来维持[42]。鹿科动物mhc-dra氨基酸和核苷酸序列的变异远较非人灵长类和马科动物少(图2),却高于犬、牛、羊、猪等经济动物[37];鹿科6属动物mhc-dra编码的氨基酸多态性位点比例则要高于核苷酸的多态性位点比率。通过对鹿科6属动物mhc-dra多态性的研究,还促进了鹿科动物中新的dra等位基因和抗原结合位点的发现。鹿科动物的核苷酸和氨基酸组成均表现出与马属、人属不同的变异特点,其中很多位点均表现出鹿科特有的核苷酸和氨基酸变异(图1和图2)。氨基酸中有一些氨基酸位点是特殊的,如鹿科动物氨基酸位点14、15、33、42、60、63、68完全不同于马属和人属。其中鹿科的抗原结合位点63(N),是完全不同于马属或人属的抗原结合位点(D/E)[37, 39]。有研究表明,mhcII类分子与许多疾病有着很强的相关性[43],例如,现已证实暴发过恶性卡他热的美洲野牛mhc个体变异的存在,使其对恶性卡他热病毒的抗性存在差异[44]。因此,该研究也可能暗示了鹿科动物有着不同的抗原结合模式,而抗原结合位点的变异则有可能导致氨基酸功能、疾病易感性或抗性的差异,这一推测尚有待今后的进一步深入研究。此外,有研究结果表明,维持mhc多样性的主要机制在于潜在病原体的驱动[45],而在鹿属mhc-dra中抗原结合位点相对保守,即非同义突变相对较少。因此,本研究进一步推测mhc-dra可识别外界病原体的范围也相对较小[46]。
3.2鹿科6属动物mhc-dra第二外显子的进化选择模式
鹿科动物常处于比较强的病原体的选择压力之下[47, 48],因此,进一步研究mhc-dra第二外显子的适应性进化将促进我们对鹿科动物免疫基因的了解。通常,选择压力是外界施于生物体进化过程的压力,以改变进化过程的前进方向,从而给予能够适应自然环境者得以存活和繁衍的力量;而正向选择则可以维持物种特定的等位基因跨越很长的时间尺度[42]。目前,科学家所公认的是mhc的多态性最可能通过正向选择来维持,而该选择是密码子的非同义突变高于同义突变所形成[49]。相对而言,dra在鹿科6属动物中非同义突变并不多,基于mhc-dra第二外显子序列Ka/Ks的研究结果,揭示了鹿科6属动物是以纯化选择为主导,以正选择为辅。其中,鹿属为正向选择,暗示其进化处于更高的选择压力之下,而使其更趋向于更多的异义突变。然而,同属于鹿亚科的麋鹿属则为纯化选择,暗示其被选择的压力相对较小。其它4属也同样处于纯化选择之下,该结果暗示了在进化过程中鹿科动物mhc-dra相对保守,也暗示了该基因可能具有比较重要的生物学功能[50]。
3.3鹿科动物mhc-dra第二外显子cytb系统进化比较
曾有学者利用线粒体12S rRNA、16S rRNA和cytb序列探讨了鹿亚科、麂亚科、獐亚科以及空齿鹿亚科的系统发生关系,暗示了鹿亚科为其它三个亚科的姐妹群[25],认为现行分类系统中,麋鹿属与鹿属有较近的进化关系,中国马鹿在系统发生上是一单系群[27]。而本研究通过对鹿科6属mhc-dra第二外显子序列和cytb序列的进一步研究,也得到了相一致的结果。与马属动物的dra第二外显子基因不同[37],鹿科6属动物dra第二外显子和cytb的系统进化分析结果相似,均具有较明显的系统进化分支结构。mhc-dra第二外显子与cytb的系统进化结果均形成了旧世界鹿类(麂属、麋鹿属、鹿属)与新世界鹿类(獐属、驼鹿属、驯鹿属)的两大姐妹类群,且分化时间分别处于上新世晚期和上新世早期。根据cytb全序列的分析结果,暗示麂亚科是由上新世中期向上新世晚期的过渡时期分化的类群,而麂亚科的mhc-dra第二外显子的分化则是上新世晚期向更新世过渡时期出现的。因此,本研究推测麂亚科cytb和mhc-dra第二外显子的基因型可能是鹿亚科与其它亚科之间的过渡型。
根据已知最古老有角鹿类化石的资料可以将出现时间追溯到19 ~ 21百万年以前[25]。然而,鹿亚科和麂亚科最早发现其化石的时间大约在6 ~ 8百万年前晚中新世时期的亚洲或欧洲[51],而空齿鹿亚科的祖先可能会被追溯到更早的时间[52]。然而,本研究中根据现有序列推算得到的历史分歧时间较化石出现时间更晚,鹿亚科和麂亚科cytb序列的分歧时间大约在4.28百万年前的上新世早期,而mhc-dra序列的分歧时间约在1.74百万年前的更新世时期。空齿鹿亚科和獐亚科的cytb和mhc-dra第二外显子序列的分歧时间分别约在5.11百万年前的上新世早期和2.05百万年前的上新世晚期。根据已有对鹿科cytb序列进化历史的研究结果,麇鹿属与鹿属的分化时间在2.15 ~ 2.50百万年前[35]。然而,本研究得到的麇鹿属与鹿属分歧时间大约在3.62百万年前,这可能是由于研究所涉及的鹿属动物物种差异和地域性差异所致。虽然有研究表明,哺乳动物mhc等位基因的分歧时间往往早于物种的分歧时间[53],但本研究中所涉及的鹿科6属动物mhc-dra分化时间却晚于物种分化的时间,这可能与样本数较小有一定关系。鹿科6属动物的mhc-dra第二外显子的分化时间大约在2.05 ~ 1.68百万年前的上新世晚期至更新世时期。两百万年前,恰逢第四纪大冰期,第四纪气候变化引起整个自然环境的变化,脊椎动物也表现出一定的适应性进化[54]。而mhc-dra第二外显子的分化正处于第四纪大冰期气候变化最为剧烈的时期,这可能也是推动mhc-dra第二外显子的分化的原因之一。
参考文献:
[1]GAUDIERI S, DAWKINS R L, HABARA K, et al. SNP profile within the human major histocompatibility complex reveals an extreme and interrupted level of nucleotide diversity[J]. Genome Research, 2000, 10(10):1579-1586.
[2]MEYER D, THOMSON G. How selection shapes variation of the human major histocompatibility complex: a review[J]. Annals of Human Genetics, 2001, 65(1):1-26.
[3]POTTS W K, SLEV P R. Pathogen-based models favoring MHC genetic diversity[J]. Immunological Reviews, 1995, 143(1):181-197.
[4]DITCHKOFF S S, HOOFER S R, Lochmiller R L, et al. MHC-DRB evolution provides insight into parasite resistance in white-tailed deer[J]. The Southwestern Naturalist, 2005, 50(1):57-64.
[5]WAKELAND E K, BOEHME S, SHE J X, et al. Ancestral polymorphisms of MHC class-II genes divergent allele advantage[J]. Immunological Reviews, 1990, 9(2):115-122.
[6]OTTOVA E, SIMKOVA A, MARTIN J F, et al. Evolution and trans-species polymorphism of MHC class II beta genes in cyprinid fish[J]. Fish Shellfish Immunological, 2005, 18(3):199-222.
[7]CUTRERA A P, LACEY E A. Trans-species polymorphism and evidence of selection on class II MHC loci in tuco-tucos (Rodentia: Ctenomyidae) [J]. Immunogenetics, 2007, 59(12):937-948.
[8]HEDRICK P W, PARKER K M, GUTIERREZ-ESPELETA G A, et al. Major histocompatibility complex variation in theArabianoryx[J]. Evolution, 2000, 54(6):2145-2151.
[9]SEDDON J M, ELLEGREN H. MHC class II genes in European wolves: a comparison with dogs[J]. Immunogenetics, 2002, 54(7):490 500.
[10]OTTING N, DE GROOT N G, DOXIADIS G G, et al. Extensive MHC-DQB variation in humans and non-human primate species[J]. Immunogenetics, 2002, 54(4):230-239.
[11]SENA L, SCHNEIDER M P, BRENIG B, et al. Potymorphism in MHC DRA and DRB alleles of water buffalo(Bubalusbubalis) reveal different features from cattle DR alleles[J].Animal Genetic, 2003, 34(1):1-10.
[12]蒲兰屏, 席冬梅, 樊月园, 等. 犏牛主要组织相容性复合物DRA基因(MHC-Class II-DRA)的分子特征分析[J].中国畜牧兽医, 2012, 39(9):12-17.
[13]YANG J, HUSTON L, BERGER D, et al.Expression of HLA-DP0401 molecules for identification of DP040 1 restricted antigen specific T cells[J].J Clin Immunol, 2005, 25(5):428-436.
[14]杨柯, 马春艳, 马凌波, 等. 凡纳滨对虾引进群体和养殖群体的PCR-RFLP分析[J]. 海洋渔业, 2010, 32(1):16-23.
[15]李金莲,石有斐,布仁其其格,等. 三大不同品种马mtDNA Cytb基因PCR-RFLP分析[J].遗传,2006, 28(8):933-938.
[16]陈建兴, 乌尼尔夫, 杨丽华, 等. 以Cytb基因探讨家马的母系起源[J]. 内蒙古农业大学学报, 2011, 32(4):1-5.
[17]曹丽荣,王小明,饶 刚,等. 从细胞色素b基因全序列分析岩羊和山羊、绵羊的系统发生关系[J].兽类学报, 2004, 242(2):109-114.
[18]章 群,任 岗,钱开诚,等. 鳜类系统发育线粒体Cytb基因全序列分析[J]. 生态科学, 2006, 25(5):430-432.
[19]潘巧娃,雷富民,杨淑娟,等. 基于细胞色素b的鸫亚科部分鸟类的系统进化[J]. 动物学报, 2006, 52(1):87-98.
[20]代金霞,郑哲民. 蝽科部分昆虫细胞色素b基因序列及其系统发育关系的探讨[J]. 动物学研究, 2004, 25(5): 397-402.
[21]周慧, 李迪强, 张于光,等. 藏羚羊mtDNA D-Loop区遗传多样性研究[J]. 遗传, 2006, 28(3):299-305.
[22]周继亮, 张亚平, 黄美华,等. 蝮亚科蛇线粒体细胞色素b基因序列分析及其系统发育[J]. 动物学报, 2001, 47 (4):361-366.
[23]HELM-BYCHOWSKI K, CRACRAFT J. Recovering phylogenetic signal from DNA sequences: relationships within the corvine assemblage (Class Aves) as inferred from complete sequences of the mitochondrial DNA cytochrome-b gene[J]. Molecular Biology and Evolution 1993, 10(6):1196-1214.
[24]孙红梅, 丛波, 邢秀梅, 等. 鹿科动物主要组织相容性复合体(MHC)的研究进展[J]. 经济动物学报, 2006, 10(4):234-237.
[25]MICHAEL M M, KRAUS F, RYDER O A. Phylogeny and evolution of antlered deer determined from mitochondrial DNA sequences[J]. Proceedings of the National Academy of Sciences, 1990, 87(16):6127-6131.
[26]RANDI E, NADIA M, FRANCOISE C, et al. A mitochondriai DNA control region phylogeny of the Cervinae:Speciation in Cervus and implications for conservation[J]. Animal Conservation, 2001, 4(1):1-11.
[27]刘向华, 王义权, 刘忠权, 等. 从Cyt b基因序列探讨鹿亚科动物的系统发生关系[J]. 动物学研究, 2003, 24(1): 27-33.
[28]GUSTAFSON A L, TALLMADGE R L, RAMLACHAN N, et al. An ordered BAC contig map of the equine major histocompatibility complex[J]. Cytogenet Genome Research, 2003, 102(1-4):189-195.
[29]VAN DEN BUSSCHE R A, HOOFER S R, LOCHMILLER R L. Characterization of Mhc-DRB allelic diversity in white-tailed deer (Odocoileusvirginianus) provides insight into Mhc-DRB allelic evolution within Cervidae[J]. Inmmunogenetics, 1999, 49(5):429-437.
[30]BALLINGALL K T, ROCCHI M S, MCKEEVER D J, et al. Trans-species polymorphism and selection in the MHC class II DRA genes of domestic sheep[J]. Plos One, 2010, 5(6), e11402.
[31]LI L, ZHU Y, GE Y F, et al. Characterization of major histocompatibility complex DRA and DRB genes of the forest musk deer (Moschusberezovskii) [J]. Chinese Science Bulletin, 2013, 58(18): 2191-2197.
[32]NEI M, GOJOBORI T. Simple methods for estimating the numbers of synonymous and nonsynonymous nucleotide substitutions[J]. Molecular Biology Evolution, 1986, 3(5): 418-426.
[33]POSADA D, CRANDALL K A. Modeltest: testing the model of DNA substitution[J]. Bioinformatics, 1998, 14(9): 817-818.
[34]ZHANG H, YAN J, ZHANG G, et al. Phylogeography and demographic history of Chinese black-spotted frog populations (Pelophylaxnigromaculata): evidence for independent refugia expansion and secondary contact[J]. BMC Evolutionary Biology, 2008, 8:21.
[35]SKOG A, ZACHOS F E, RUENESS E K, et al. Phylogeography of red deer (Cervuselaphus) in Europe[J]. Journal of Biogeography, 2009, 36(1):66-77.
[36]MAYER W E, JONKER M, KLEIN D, et al. Nucleotide sequences of chimpanzee MHC class I alleles: evidence for trans-species mode of evolution[J]. The EMBO Journal, 1988, 7(9):2765-2774.
[37]KAMATH P L, GETZ W M. Adaptive molecular evolution of the major histocompatibility complex genes, DRA and DQA, in the genus Equus[J]. BMC Evolutionary Biology, 2011, 11:128.
[38]楚秋霞, 徐照学, 施巧婷, 等. 南阳牛BoLA-DRA基因的克隆及其在大肠埃希菌中的表达[J]. 动物医学进展, 2010, 31(6):5-10.
[39]RECHE P A, REINHERZ E L. Sequence variability analysis of human class I and class II MHC molecules: functional and structural correlates of amino acid polymorphisms[J]. Journal of Molecular Biology, 2003, 331(3):623-641.
[40]ANDERSSON L, BOHME J, RASK L, et al.Genomie hybridization of bovine class II MHC genes: Extensive polymorphisms of DQB and DQB genes[J].Animal Genetics, 1986, 17(2):95-112.
[41]AMORENA B, STONE W H.Serologically defined (SD) locus in cattle[J].Science, 1978, 201(4351):159-161.
[42]KLEIN J. Origin of major histocompatibility complex polymorphism: the trans-species hypothesis[J]. Human Immunology, 1987, 19(3):155-162.
[43]DIAZ S, ECHEVERRIA M G, IT V, et al.Development of an ELA-DRA gene typing method based on pyrosequencing technolog[J].Tissue Antigens, 2008, 72(5): 464-468.
[44]TRAUL D L, LI H, DASGUPTA N, et al.Resistance to malignant catarrhal fever in America bison(Bisonbison) is associated with MHC class Iia polymorphisms[J].Animal Genetics, 2007, 38(2):141-146.
[45]HEDRICK P, KIM T. Genetics of complex polymorphisms: parasites and maintenanceof MHC variation[M]. Cambridge: Harvard University Press, 1998.
[46]HUGHES A L, NEI M. Pattern of nucleotide substitution at major histocompatibility complex class-I loci reveals overdominant selection[J]. Nature, 1988, 335(6186):167-170.
[47]杨明凡, 崔保安, 魏战勇, 等. 麋鹿魏氏梭菌病和巴氏杆菌病混合感染[J]. 中国兽医杂志, 2004, 40(3): 51-52.
[48]刘永张, 王荣琼, 杨玉钊, 等. 麋鹿感染放线菌病例[J]. 经济动物学报, 2011, 15(2): 80-81.
[49]PIERTNEY S B, OLIVER M K. The evolutionary ecology of the major histocompatibility complex[J]. Heredity, 2006, 96(1):7-21.
[50]姬广超,王明辉,高会江,等. 哺乳动物MT基因的进化选择与功能分歧[J]. 东北农业大学学报, 2010, 41(9):82-88.
[51]SOLOUNIAS N. The Turolian fauna from the island of Samos, Greece with special emphasis on the Hyaenids and the Bovids[J]. Contribute Vertebrata Evolution, 1981, 6(1):99-232.
[52]VISLOBOKOVA I A. Fossil deer of Eurasia[J]. Tr Paleontol Inst Akad Nauk SSSR, 1990, 240:1-208.
[53]FIGUEROA F, GUNTHER E, KLEIN J. MHC polymorphism predating speciation[J]. Nature, 1988, 335(6187):265-267.
[54]谢宇平. 第四纪地质学及地貌学[M]. 北京: 地质出版社, 1994.
Variation characteristic of MHC-DRA exon 2 in six deer genera of family Cervidae and phylogenetic evolution compared with Cyt b
YOU Yu-yan1, ZHANG Cheng-lin1, ZHANGJin-guo1, RUAN Xiang-dong2, ZHANG Lin-yuan3,ZHONG Zhen-yu3, ZHENG Chang-ming1, LIU Xue-feng1, WANG Wei1, CAO Chang-lei4
(1. Beijing Key Laboratory of Captive Wildlife Technologies, Beijing Zoo, Beijing 100044; 2. National Forest Inventory and Design Institute, Beijing 100714; 3. Beijing Milu Ecological Research Center, Beijing 100076;4. Yangtze Normal University, Chongqing 408100, China)
AbstractIn this study, the variation characteristics and revealed evolutionary selection mode at the molecular level of the major histocompatibility complex dra (mhc-dra) exon 2 in the six deer genera of family Cervidae were investigated and compared with cyt b gene. 246 bp of mhc-dra exon 2 sequences of six deer genera in family Cervidae were obtained based on sequencing method. The results showed that there were 18 polymorphic sites and 13 singleton variable sites. The nucleotide variations of mhc-dra exon 2 were low and each genus had its own unique variation loci. There were 20 putative antigen-binding sites in this region, in which position 63 and 18 were different from genus Equus and Homo, which suggested the six deer genera might have different antigen-binding model. According to the ratio of non-synonymous substitution rates (Ka) and synonymous substitution rates (Ks), the mhc-dra exon 2 is positive selection for Cervus, and is purifying selection for the other five genera. The Neighbor-joining method (NJ),Maximum likelihood method (ML) and Bayesion inference (BI) of the mhc-dra exon 2 and cyt b were in a similar result, and all formed sister groups between the old world deer and the new one. The gene type of cyt b and mhc-dra exon 2 clearly showed subfamily Muntiacinae is a transition group between subfamily Cervinae and other subfamilies. Climate and environmental changes might be potential reasons to promote the differentiation of mhc-dra exon 2.
Key wordsmhc-dra; molecular evolution; phylogenetic evolution; selective pressure
收稿日期:2015-09-16;修回日期:2015-09-28
基金项目:国家自然科学基金项目(No.31200145)资助
作者简介:由玉岩,博士,高级工程师,研究方向为保护生物学,E-mail: youyy351@163.com。
中图分类号Q953
文献标识码A
文章编号2095-1736(2016)03-0034-07
doi∶10.3969/j.issn.2095-1736.2016.03.034

