环介导等温扩增联合横向流动试纸条可视化检测哈维氏弧菌的研究
程蝶 柴方超 蔡怡 周前进 陈炯
(宁波大学生物与海洋科学系,宁波 315211)
环介导等温扩增联合横向流动试纸条可视化检测哈维氏弧菌的研究
程蝶 柴方超 蔡怡 周前进 陈炯
(宁波大学生物与海洋科学系,宁波 315211)
以哈维氏弧菌(Vibrio harveyi)为材料,利用环介导等温扩增技术(LAMP)进行核酸扩增,借助横向流动试纸条(LFD)完成产物检测,旨在建立一种可用于哈维氏弧菌快速检测的LAMP-LFD新技术。以哈维氏弧菌的溶血素基因(vhhA)为检测靶标设计了3对特异性引物(其中,上游内引物vhhA-FIP由生物素标记),进行由生物素标记的LAMP反应;同时设计1条异硫氰酸荧光素(FITC)标记的探针,与获得的LAMP产物进行特异性杂交,杂交产物经LFD完成检测。经优化,LAMP的反应条件为63℃反应40 min,由LFD完成结果判读共需50 min。结果表明,LAMP-LFD方法能特异性地检出哈维氏弧菌,对创伤弧菌等其他9种水产养殖重要病原菌的检测结果呈阴性。利用该方法,针对细菌纯培养物的检测灵敏度为1.0×102CFU/mL或2 CFU/反应,针对污染有该菌的大黄鱼组织的检测灵敏度为5×102CFU/mL或20 CFU/反应,均是以LAMP外引物vhhA-F3/vhhA-B3的常规PCR方法的100倍。因此,该方法能够快速、准确地检出哈维氏弧菌,有望在海水养殖过程哈维氏弧菌的监测和即时检测中普及使用。
哈维氏弧菌;溶血素基因;环介导等温扩增技术;横向流动试纸条;检测
哈维氏弧菌(Vibrio harveyi)是一种革兰氏阴性的嗜盐弧菌,普遍分布于近岸的海水环境中,是海洋水体和许多海生动物的正常菌群[1,2]。同时,哈维氏弧菌又是一种重要的机会致病菌,可感染鱼、虾、贝类等多种海洋动物,引发死亡率较高的弧菌病[3]。在东南亚地区,哈维氏弧菌是虾养殖产业的主要细菌性病害之一[4,5];该弧菌也曾给大洋洲各国斑节对虾的养殖带来毁灭性的打击[6,7]。哈维氏弧菌可使鲍鱼养殖大面积减产,该菌感染被认为是自1998年以来法国养殖和野生的欧洲鲍螺出现季节性大面积死亡的主要原因[8]。在我国,虾的早期死亡综合征(early mortality syndome,EMS)已成为虾养殖产业的重要掣肘[9],而研究表明哈维氏弧菌即是该新发病的主要细菌性病原之一[9,10],同时也是对虾白尾病的重要诱因[11]。此外,该病原也是我国多种养殖鱼类、贝类和蟹类的重要病原[12-15]。
哈维氏弧菌与其他多种致病弧菌(如副溶血弧菌、溶藻弧菌等)在表型和基因型非常相似[16],这给种型水平上细菌的鉴定带来了一定的困难。例如,利用传统的细菌分离培养和生化鉴定或借助鉴别培养基不能将哈维氏弧菌与其他弧菌进行有效区分,而16S rRNA的序列分析也只能将其鉴定至科属水平[17,18]。基于多位点序列分析(MLSA)或基因组测序的方法可有效区分不同弧菌,但仅限于实验室的弧菌分类学研究[18,19]。因此,许多研究致力于哈维氏弧菌其他标志性基因的鉴定和筛选,并在此基础上建立了多种快速检测方法,如聚合酶链式反应技术(PCR)[20,21]、多重PCR技术[22,23]、环介导等温扩增技术(loop-mediated isothermal amplification,LAMP)等[24,25]。这些方法因其高灵敏度和特异性、检测快速等特点,在哈维氏弧菌的检测领域获得了广泛应用。但是,在对这些方法获得的产物进行检测分析时,要么通过琼脂糖凝胶电泳的方式完成,需要接触EB等有毒试剂,要么借助于荧光染料,容易造成结果的假阳性。LAMP-LFD技术是将LAMP的反应产物与特异性的探针杂交,在横向流动试纸条(lateral flow dipstick,LFD)以显色的方式呈现实验结果,通过肉眼即可快速完成结果判读。该方法完全摒弃了EB等有毒试剂,也摆脱了对电泳装置和凝胶成像系统等的依赖,整个检测时程也仅在LAMP反应的基础上增加了8-10 min,而且由于特异性探针的引入,使得结果更加特异,因此,在微生物的基层检测和现场快速检测中展现了重大潜力。目前,该技术已成功运用于传染性脾肾坏死病毒(infectious spleen and kidney necrosis virus,ISKNV)、传染性肌肉坏死病毒(infectious myonecrosis virus,IMNV),以及创伤弧菌(Vibrio vulnificus)等水产病原微生物的检测[26-28]。本研究根据哈维氏弧菌溶血素基因vhhA的保守区设计多条引物和1条异硫氰酸荧光素标记的探针,条件优化后建立LAMP-LFD检测方法,并通过少量的样本检测,分析该方法在实际检测应用中的可行性,为哈维氏弧菌引发的弧菌病的快速诊断和早期检测提供更加可靠、便捷的技术手段。
1 材料与方法
1.1 材料
迟缓爱德华菌(Edwardsiella tarda MCCC235)中国海洋微生物菌种保藏管理中心;创伤弧菌(V. vulnificus ATCC 27562)、哈维氏弧菌(V. harveyi ATCC 33866)、河流弧菌(V. fluvialis ATCC 33809)、嗜水气单胞菌(Aeromonas hydrophila ATCC 7966)购自中国普通微生物菌种保藏中心;单增李斯特菌(Listeria monocytogenes ATCC 19115)购自广东省微生物菌种保藏中心;副溶血弧菌(V. parahaemolyticus ATCC 33847)由浙江省疾控中心张严峻研究员惠赠;海豚链球菌(Streptococcus iniae ATCC 29178)、 溶 藻 弧 菌(V. alginolyticus ATCC 17749)购自美国菌种保藏中心;鳗弧菌香鱼分离株(V. anguillarum ayu-H080701)由本实验室保存。
1.2 方法
1.2.1 细菌培养和基因组DNA的制备 研究涉及的弧菌划线接种于TCBS固体培养基,28-30℃培养12-24 h后,挑取单菌落于碱性蛋白胨水中震荡培养至所需浓度;迟缓爱德华菌、嗜水气单胞菌、海豚链球菌,以及单增李斯特菌划线接种于LB或BHI固体培养基,30或37℃培养后,挑取单菌落于LB或BHI液体培养基中震荡培养至所需浓度。
细菌基因组DNA的制备参考王耀焕等[28]的方法,具体操作:取新鲜培养的细菌液体培养悬液1mL 12 000 r/min离心2 min,弃上清;菌体沉淀使用100 μL的细菌裂解液(Tris-HCl 0.1 mol/L,蛋白酶K 0.2 μg/μL,Triton X-100 2.0%)充分悬浮,沸水浴10 min,立即冰浴7 min;重复上一步操作1次;12 000 r/min离心2 min,取上清定容至100 μL,-30℃贮存,用作LAMP和PCR扩增的模板。
1.2.2 引物设计 选择NCBI上公布的哈维氏弧菌溶血素(vhhA,登录号:AF293430)基因,通过序列比对分析后,依次选择8个高度保守的基因区段设计3对特异性引物(外引物vhhA-F3和vhhA-B3、内引物vhhA-FIP和vhhA-BIP、环引物vhhA-LF和vhhA-LB)用于LAMP反应;同时设计1条探针vhhA-HP,可与LAMP产物进行特异性杂交,用于LFD检测(表1,图1)。其中,内引物vhhA-FIP的5'端进行生物素标记,探针vhhA-HP的5'端进行异硫氰酸荧光素标记。上述引物和探针由英潍捷基(上海)贸易有限公司合成。另外,将外引物vhhA-F3和vhhA-B3也作为PCR扩增的特异性引物,扩增的预期片段大小约为310 bp。

表1 根据哈维氏弧菌vhha基因序列设计的LAMP-LFD扩增引物序列和探针

图1 哈维氏弧菌溶血素基因vhhA LAMP-LFD引物序列和探针设计示意图
1.2.3 LAMP反应条件的优化 LAMP反应的体系参考Ding和王耀焕等[26,28]的方法,具体组成如下(25 μL):MgSO46.5 mmol/L,KCl 10 mmol/L,(NH4)2SO410 mmol/L,Tris-HCl 20 mmol/L(pH 8.8),Triton X-100 0.1%,甜菜碱 1.6 mol/L,dNTPs 1.4 mmol/L,外引物vhhA-F3和vhhA-B3各0.2 μmol/L,内引物vhhA-FIP和vhhA-BIP各1.6 μmol/L,环引物vhhALF和vhhA-LB各0.4 μmol/L,Bst DNA聚合酶(New England BioLabs,美国)8 U,哈维氏弧菌基因组DNA模板 2 μL。阴性对照不加任何模板DNA。
取新培养的哈维氏弧菌,经平板计数法获得菌液浓度为2.4×108CFU/mL,使用液体培养基(碱性蛋白胨水)将该菌液浓度调整为1.0×108CFU/mL,并定为起始浓度。将该原始菌液进行连续10倍浓度梯度稀释,获得1.0×108CFU/mL、1.0×107CFU/mL、1.0×106CFU/mL、1.0×105CFU/mL、1.0×104CFU/mL、1.0×103CFU/mL、1.0×102CFU/mL和1.0×101CFU/mL等8个浓度的菌液,按1.2.1所述的方法分别提取其基因组DNA。选择较低模板浓度(相当于1.0×104CFU/mL),分别在61、63和65℃条件下温育 60 min,每个温度下,平行制备3个样品,然后经80℃热激5 min终止反应,利用1.5%的琼脂糖凝胶电泳检测产物,确定LAMP的最佳反应温度。选择2个浓度的菌液(较高模板浓度相当于1.0×108CFU/mL,较低模板浓度相当于1.0×104CFU/mL)作为反应时间优化的模板,在优化的温度下进行温育,温育时间分别为0、10、15、20、25、30、40和50 min,通过1.5%的琼脂糖凝胶电泳检测产物,确定最佳反应时间。
1.2.4 利用横向流动试纸条(LFD)检测 本研究使用的横向流动试纸条(LFD)购自Milennia Biotec GmbH(Milenia GenLine HybriDetect by Milenia Biotec GmbH,Germany,http://www.milenia-biotec.de/), 其检测线位置标记有生物素抗体,可特异性地与生物素标记的LAMP产物结合而在检测线上呈现颜色变化。内引物vhhA-FIP经生物素标记后,利用优化后的LAMP反应体系进行LAMP反应;反应结束后不使用高温致使反应终止,而是向反应体系中20 pmol的经FITC标记的探针vhhA-HP,继续反应5 min;取5 μL反应液加入到100 μL 检测buffer中混匀;将LFD试纸条的检测端竖直浸入buffer溶液中,静置3-5 min,肉眼判断结果。
1.2.5 LAMP-LFD的特异性实验 用于LAMP-LFD特异性实验的细菌种类主要包括弧菌(创伤弧菌、河流弧菌、副溶血弧菌、溶藻弧菌和鳗弧菌)、迟缓爱德华菌、单增李斯特菌、海豚链球菌,以及嗜水气单胞菌等9种水产养殖或水产品中的常见病原菌。将各菌株的新培养物调整浓度至1×105CFU/mL,按
1.2.1 所述提基因组DNA,并以此为模板,利用优化的反应条件,进行有生物素标记的LAMP反应,产物经LFD检测;同时利用1.5%的琼脂糖凝胶电泳的方法判断实验结果。
1.2.6 LAMP-LFD的灵敏度实验 分别取1.2.3所述的不同稀释浓度的哈维氏弧菌菌液提取的基因组DNA 2 μL为模板,每个浓度平行制备3个样品。利用优化的反应条件进行有生物素标记的LAMP反应,产物经LFD检测;同时利用1.5%的琼脂糖凝胶电泳的方法判断实验结果。
同样,取上述各稀释度的基因组DNA为模板,以外引物vhhA-F3和vhhA-B3作为引物,进行PCR反应。PCR反应体系(25 μL),包括:10× PCR buffer 2.5 μL,Taq DNA 聚合酶(5 U/μL)(TaKaRa,日本)0.15 μL,dNTPs(2.5 mmol/μL)2 μL,vhhA-F3(10 pmol/μL)1 μL,vhhA-B3(10 pmol/μL)1 μL,基因组DNA模板 2 μL。PCR反应程序:94℃预变性3 min;94℃变性30 s,52℃退火30 s,72℃延伸30 s,30个循环;最后,72℃延伸10 min。反应产物利用1.5%琼脂糖凝胶电泳检测。
1.2.7 LAMP-LFD的重复性实验 取1 mL新鲜培养的哈维氏弧菌菌液,计数并调整浓度至1.0×108CFU/mL,连续10倍浓度梯度稀释至LAMP-LFD检测的最低浓度,平行制备3个样品。按1.2.1所述提取基因组DNA,并以此为模板,采用优化的LAMP反应体系进行扩增,验证方法的可重复性。
1.2.8 哈维氏弧菌人工污染大黄鱼组织实验及田间样本的检测 取1 mL新鲜培养的哈维氏弧菌菌液,计数并调整浓度至1.0×108CFU/mL,10倍浓度梯度稀释后,选取浓度1.0×105、1.0×104、1.0×103、1.0×102及1.0×101CFU/mL的菌液各1 mL与等体积的大黄鱼肌肉组织匀浆液混匀。每个浓度平行制备3个样品。取1 mL细菌污染的肌肉组织样品,采用动物组织基因组DNA提取试剂盒(宝生物工程(大连)有限公司,中国)提取细菌污染样品的基因组DNA,溶解于50 μL的ddH2O中,用作LAMP和PCR测试的模板。健康大黄鱼的肝脏组织匀浆液作阴性对照。
收集具有一定临床症状的养殖动物22份,包括大黄鱼样品8份,南美白对虾14份。首先对各样品进行细菌分离,通过鉴别培养基筛选结合生化实验的方法对病原进行分离、鉴定;同时分别取适量动物组织,利用动物组织基因组DNA提取试剂盒(宝生物工程(大连)有限公司,中国)提取基因组DNA,采用LAMP-LFD和PCR方法进行检测。
2 结果
2.1 LAMP扩增条件的确定
为确定最佳反应温度,以较低浓度的基因组DNA为模板,分别选择61、63及65℃进行LAMP反应60 min。结果表明,利用上述3个温度获得的LAMP产物均能够检测到明显的梯状条带,未添加DNA模板的反应管未检测到明显的梯状条带。其中,在相同的反应体系下,当反应温度为63℃时,扩增产物的条带更加清晰,所以选择63℃作为LAMP反应的最适温度(图2-A)。
当模板浓度较高时(1.0×108CFU/mL),反应15 min即可出现明显的梯状条带;反应至30 min时,扩增产物的浓度几乎不再增加(图2-B)。当选用较低模板浓度时(1.0×104CFU/mL),反应至20 min时,利用琼脂糖凝胶电泳才可明显地检测到梯状条带;继续延长反应时间,扩增产物的浓度逐渐增加;反应时间延长至40 min时,产物浓度不再明显变化。为保证实际检测过程中,当遇到较低模板浓度时,仍可有效地检测出病原,我们将LAMP反应的最佳时间确定为40 min(图2-C)。
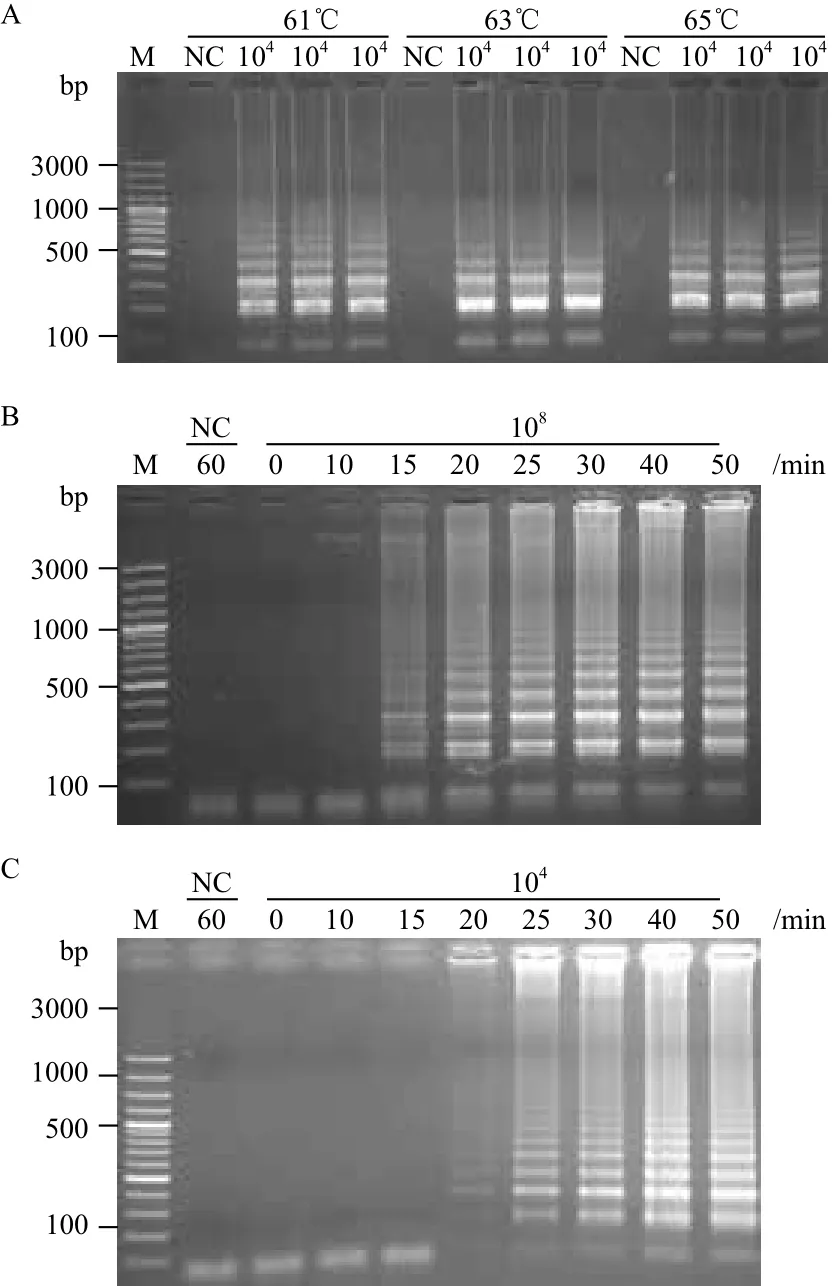
图2 LAMP反应条件的优化
2.2 LAMP-LFD的特异性
选择常见的水产致病菌为模板进行LAMP-LFD方法的特异性分析,按优化的LAMP反应条件63℃温育40 min。产物经LFD检测发现,以哈维氏弧菌基因组DNA为模板的反应呈阳性,而以其他供试弧菌、海豚链球菌、单增李斯特菌等致病菌的基因组DNA为模板时,反应皆呈阴性,以蒸馏水为模板的反应也呈阴性(图3)。
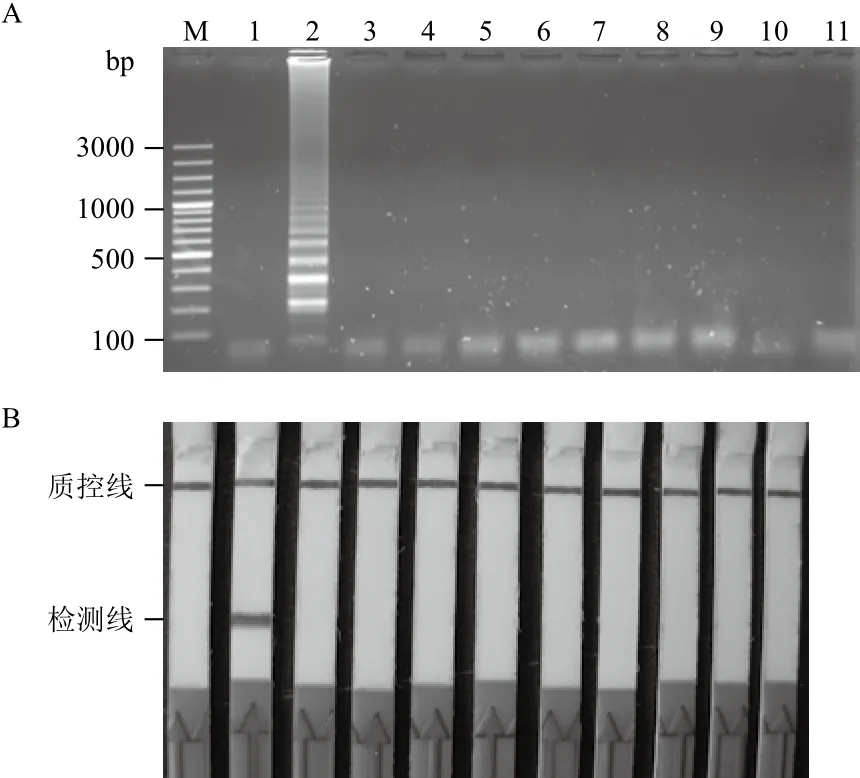
图3 LAMP(A)和LAMP-LFD(B)特异性分析
2.3 LAMP-LFD的灵敏度验证
选 用 1.0×107、1.0×106、1.0×105、1.0×104、1.0×103、1.0×102及1.0×101CFU/mL等7个浓度的基因组DNA作为模板,进行有生物素标记的LAMP反应。实验结果表明,经LFD检测的最低模板浓度为1.0×102CFU/mL或2 CFU/反应(图4-E);利用1.5%琼脂糖凝胶电泳同样可检测到的最低模板浓度为1.0×102CFU/mL(图4-A、4-B、4-C);而以vhhA-F3和vhhA-B3为特异性引物的PCR方法能够检测到的最低模板浓度为1.0×104CFU/mL或200 CFU/反应(图4-D)。
2.4 LAMP-LFD重复性分析
重新取哈维氏弧菌的新鲜培养悬液,调整至可检测的最低模板浓度(1.0×102CFU/mL),提取基因组DNA后,以优化的反应体系63℃温育40 min。结果表明,以1.0×102CFU/mL浓度的哈维氏弧菌基因组DNA为模板时,LFD检测结果呈阳性;以蒸馏水为模板时,检测结果均呈阴性,具有良好的重复性。
2.5 LAMP-LFD检测哈维氏弧菌人工污染大黄鱼组织灵敏度及适用性分析
不同浓度的哈维氏弧菌等体积污染大黄鱼组织发现,LAMP-LFD可从1.0×103CFU/mL污染的大黄鱼肌肉组织中稳定检测到病原,检测灵敏度为5×102CFU/mL或20 CFU/反应。PCR方法只能从1.0×105CFU/mL污染的大黄鱼肌肉组织中稳定检测到病原(表2)。LAMP-LFD对健康大黄鱼肌肉组织的检测结果为阴性,特异性良好。
利用传统的细菌分离培养方法对收集的病样检测表明,8份大黄鱼样品中有2份样品中可成功分离出哈维氏弧菌,而14份南美白对虾中有4份样品分离出哈维氏弧菌。利用LAMP-LFD方法从传统方法鉴定阳性的样品中均可检测出哈维氏弧菌,而且从传统方法鉴定阴性的2份病虾样品中检测到哈维氏弧菌的存在;PCR方法也能够从传统方法鉴定阳性的样品中检测出哈维氏弧菌,而传统方法检测阴性的1份病虾样品中检测到该病原菌(表3)。
3 讨论
哈维氏弧菌能够感染多种海洋脊椎动物和无脊椎动物,而在我国,该病原已成为对虾养殖中的主要病原菌,也是鱼类、贝类及蟹类养殖的重要细菌性病原。因此,建立快速、准确的检测技术,实现病原菌的早期诊断和基层的即时检测,对于该病的有效预防和监测具有重要意义。本研究选择哈维氏弧菌的溶血素基因作为检测靶标,建立了便捷的LAMP-LFD技术,可特异性的从细菌培养物或动物组织样品中检测到哈维氏弧菌病原。
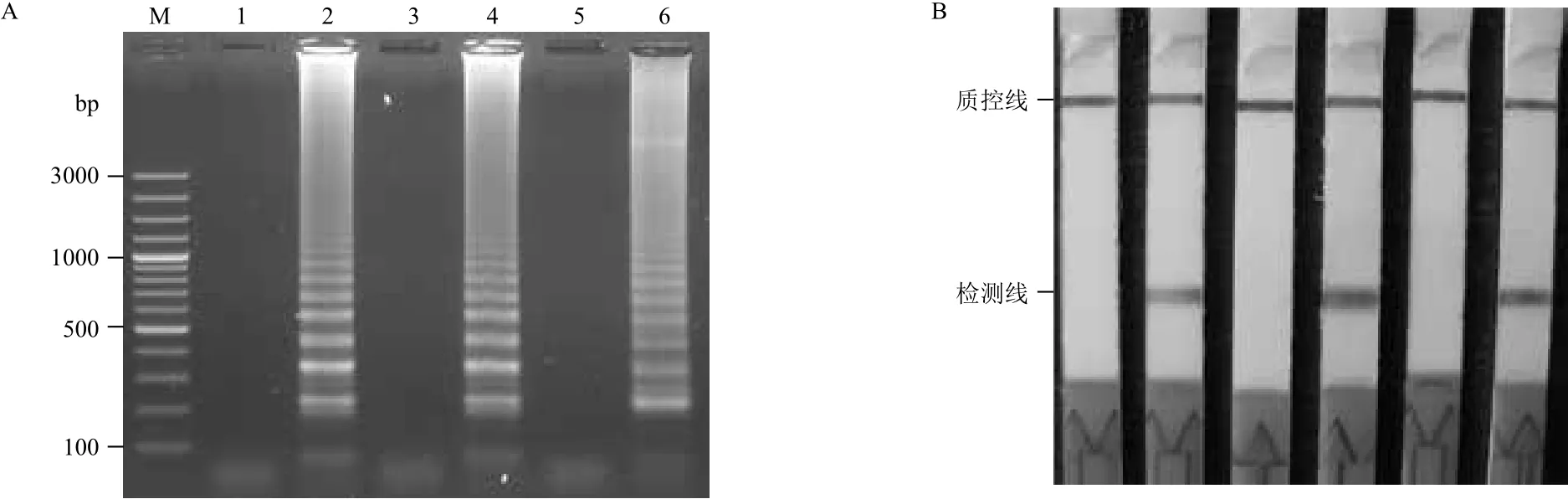
图5 LAMP(A)和LAMP-LFD(B)的重复性试验

表2 哈维氏弧菌人工污染健康大黄鱼肌肉组织的检测结果
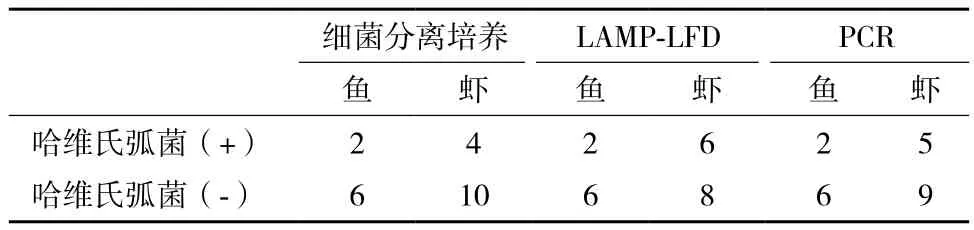
表3 细菌分离培养、LAMP-LFD和PCR对野外病样的检测结果
灵敏度是衡量核酸检测类方法的重要技术指标。Pang等[29]选择toxR作为分类标记,建立的PCR方法,可从4.0×103细菌/mL的哈维氏弧菌纯培养物中检测到细菌的存在;Caipang等[21]以热激蛋白家族dnaJ基因为检测靶标,建立了可特异性检测亚洲鲈鱼体内哈维氏弧菌感染的PCR方法,检测灵敏度达到了100 fg的细菌基因组DNA。而Schikorski等[20]基于TaqMan探针技术建立了实时荧光定量PCR技术,应用于欧洲鲍鱼哈维氏弧菌的检测,灵敏度可达18 个细菌。多重PCR技术在哈维氏弧菌和弧菌菌群其他弧菌的同步检测和鉴别中取得了一定程度的应用。Cano-Gomez等[22]建立的多重PCR技术,应用于哈维氏弧菌与相似弧菌坎氏弧菌(V. campbellii)、藻弧菌(V. owensii)、轮虫弧菌(V. rotiferianus)的区别鉴定,检测灵敏度可达到30 pg的细菌基因组DNA。而Haldar等[23]选择弧菌的溶血素基因建立的多重PCR技术,可分别特异性地检出哈维氏弧菌、坎氏弧菌,以及副溶血弧菌,最低检测下限在10-100个细菌。环介导等温扩增技术被认为具有比PCR更加优越的灵敏度[30]。Cao等[24]将毒素调控基因(toxin-positive regulatorygene,toxR)作为检测靶标,建立了针对哈维氏弧菌的环介导等温扩增技术,灵敏度为17.2 个细菌,与基于TaqMan探针的实时荧光定量PCR技术的检测灵敏度相当。而Thongkao等[25]基于vhhP2基因建立的环介导等温扩增技术,进一步将该方法的灵敏度提高到了0.6 CFU。本研究建立的LAMP-LFD方法,针对哈维氏弧菌纯培养物的检测灵敏度为1.0×102CFU/mL或2 CFU/反应;而利用该方法能够从污染强度达到5×102CFU/mL的大黄鱼组织中稳定检测到哈维氏弧菌的存在,具有良好的灵敏度。
操作便捷,检测快速是目前核酸检测类方法不断改进的重要目标。环介导等温扩增技术因其核酸扩增过程不再需要类似PCR的多个温度模块,在1个温度下即可完成扩增过程,极大的降低了反应仪器构造的复杂度,在操作便捷程度上具有比PCR明显的优势[31]。针对LAMP产物的检测,常用的检测手段包括琼脂糖凝胶电泳检测,利用荧光染料,或通过利用浊度计检测,同样具有与PCR产物检测相似的弊端,如检测仪器昂贵、假阳性率高、易接触有毒试剂等[32,33]。LAMP-LFD技术利用荧光素标记的探针特异性地与LAMP产物结合,在很大程度上降低了结果的假阳性,同时LAMP产物通过LFD试纸条检测,完全摒弃了对凝胶成像系统、浊度计、荧光定量PCR仪等设备的依赖,检测更加适应于基层的即时检测[28]。在检测耗时方面,Cao和Thongkao等[24,25]利用LAMP进行核酸扩增的时间分别是60 min和90 min,而本研究建立的LAMPLFD技术,核酸扩增仅需40 min,加上LFD判断结果的时间也只需要50 min。
4 结论
本研究以哈维氏弧菌的溶血素基因为检测靶标,建立了LAMP-LFD方法。该方法可特异性检出哈维氏弧菌,针对创伤弧菌等其他弧菌,迟缓爱德华菌、单增李斯特菌、海豚链球菌及嗜水气单胞菌等常见水产病原菌均无交叉反应,自模板加入后,整个检测过程仅需50 min即可完成,针对细菌纯培养物的检测灵敏度达到1.0×102CFU/mL或2 CFU/反应,重复性良好,仪器需求简单,可应用于哈维氏弧菌的基层即时检测。
[1]Pedersen K, Verdonck L, Austin B, et al. Taxonomic evidence that Vibrio carchariae Grimes et al. 1985 is a junior synonym of Vibrio harveyi(Johnson and Shunk 1936)Baumann et al. 1981[J]. Int J Syst Bacteriology, 1998, 48(3):749-758.
[2]Vattanaviboon P, Mongkolsuk S. Unusual adaptive, cross protection responses and growth phase resistance against peroxide killing in a bacterial shrimp pathogen, Vibrio harveyi[J]. FEMS Microbiology Letters, 2001, 200(1):111-116.
[3]Austin B, Zhang XH. Vibrio harveyi:a significant pathogen of marine vertebrates and invertebrates[J]. Letters in Applied Microbiology, 2006, 43(2):119-124.
[4]Gräslund S, Bengtsson BE. Chemicals and biological products used in south-east Asian shrimp farming, and their potential impact on the environment—a review[J]. Science of the Total Environment, 2001, 280(1):93-131.
[5]Liu PC, Lee KK, Yii KC, et al. Isolation of Vibrio harveyi from diseased kuruma prawns Penaeus japonicus[J]. Current Microbiology, 1996, 33(2):129-132.
[6]Ruangpan L, Danayadol Y, Direkbusarakom S, et al. Lethal toxicity of Vibrio harveyi to cultivated Penaeus monodon induced by bacteriophage[J]. Diseases of Aquatic Organisms, 1999, 35:195-201.
[7]Harris L, Owens L, Smith S. A selective and differential medium for Vibrio harveyi[J]. Applied and Environmental Microbiology, 1996, 62(9):3548-3550.
[8]Nicolas JL, Basuyaux O, Mazurie J, et al. Vibrio carchariae, a pathogen of the abalone Haliotis tuberculata[J]. Diseases of Aquatic Organisms, 2002, 50(1):35-43.
[9]Lightner DV, Redman RM, Pantoja CR, et al. Early mortality syndrome affects shrimp in Asia[J]. Glob Aquacult Advocate, 2012, 1(15):40.
[10]Tran L, Nunan L, Redman RM, et al. Determination of the infectious nature of the agent of acute hepatopancreatic necrosis syndrome affecting penaeid shrimp[J]. Diseases of Aquatic Organisms, 2013, 105(45):e55.
[11]Zhou J, Fang W, Yang X, et al. A nonluminescent and highly virulent Vibrio harveyi strain is associated with “bacterial white tail disease” of Litopenaeus vannamei shrimp[J]. PLoS One, 2012, 7(2):e29961.
[12]Chen C, Cheng B, Yu H, et al. Isolation and identification of a pathogenic bacterium from Epinephelus septemfasciatus with exophthalmic disease[J]. Prog Polym Sci, 2010, 31:25-33.
[13]Zhang FP, Peng ZL, Zhang J, et al. Isolation and identification of the pathogenic strain of Vibrio harveyi from Miichthys miiuy[J]. Acta Microbiologica Sinica, 2010, 50:304-309.
[14]王江勇, 孙秀秀, 王瑞旋, 等. 杂色鲍肌肉萎缩症病原菌的分离鉴定及系统发育分析[J]. 南方水产科学, 2010, 6(5):21-26.
[15]Zhang XJ, Bai XS, Yan BL, et al. Vibrio harveyi as a causative agent of mass mortalities of megalopa in the seed production of swimming crab Portunus trituberculatus[J]. Aquaculture International, 2014, 22(2):661-672.
[16]Thompson FL, Gevers D, Thompson CC, et al. Phylogeny and molecular identification of vibrios on the basis of multilocus sequence analysis[J]. Applied and Environmental Microbiology, 2005, 71(9):5107-5115.
[17]Cano-Gomez A, Bourne DG, Hall MR, et al. Molecular identification, typing and tracking of Vibrio harveyi in aquaculture systems:current methods and future prospects[J]. Aquaculture, 2009, 287(1):1-10.
[18]Thompson CC, Vicente AC, Souza RC, et al. Genomic taxonomy of vibrios[J]. BMC Evolutionary Biology, 2009, 9:258.
[19]Gurtler V, Mayall BC. Genomic approaches to typing, taxonomy and evolution of bacterial isolates[J]. International Journal of Systematic and Evolutionary Microbiology, 2001, 51:3-16.
[20]Schikorski D, Renault T, Paillard C, et al. Development of TaqMan real-time PCR assays for monitoring Vibrio harveyi infection and a plasmid harboured by virulent strains in European abalone Haliotis tuberculata aquaculture[J]. Aquaculture, 2013, 392:106-112.
[21] Caipang CMA, Pakingking JrRV, Apines-Amar MJS, et al. Development of a polymerase chain reaction(PCR)assay targeted to the dnaJ gene of Vibrio harveyi, a bacterial pathogen in Asian seabass, Lates calcarifer[J]. AACL Bioflux, 2011, 4:447-454.
[22]Cano-Gomez A, Høj L, Owens L, et al. A multiplex PCR-based protocol for identification and quantification of Vibrio harveyirelated species[J]. Aquaculture, 2015, 437:195-200.
[23]Haldar S, Neogi SB, Kogure K, et al. Development of a haemolysin gene-based multiplex PCR for simultaneous detection of Vibrio campbellii, Vibrio harveyi and Vibrio parahaemolyticus[J]. Letters in Applied Microbiology, 2010, 50(2):146-152.
[24]Cao YT, Wu ZH, Jian JC, et al. Evaluation of a loop-mediated isothermal amplification method for the rapid detection of Vibrio harveyi in cultured marine shellfish[J]. Letters in Applied Microbiology, 2010, 51(1):24-29.
[25]Thongkao K, Longyant S, Silprasit K, et al. Rapid and sensitive detection of Vibrio harveyi by loop-mediated isothermal amplification combined with lateral flow dipstick targeted to vhhP2 gene[J]. Aquaculture Research, 2013, 46(5):1122-1131.
[26]Ding WC, Chen J, Shi YH, et al. Rapid and sensitive detection of infectious spleen and kidney necrosis virus by loop-mediated isothermal amplification combined with a lateral flow dipstick[J]. Archives of Virology, 2010, 155(3):385-389.
[27]Puthawibool T, Senapin S, Kiatpathomchai W, et al. Detection of shrimp infectious myonecrosis virus by reverse transcription loopmediated isothermal amplification combined with a lateral flow dipstick[J]. J Virol Methods, 2009, 156(1):27-31.
[28]王耀焕, 王瑞娜, 周前进, 等. 环介导等温扩增联合横向流动试纸条快速检测创伤弧菌检测方法的建立[J]. 生物技术通报, 2014(6):81-87.
[29]Pang L, Zhang XH, Zhong Y, et al. Identification of Vibrio harveyi using PCR amplification of the toxR gene[J]. Letters in Applied Microbiology, 2006, 43(3):249-255.
[30]Imai M, Ninomiya A, Minekawa H, et al. Development of H5-RTLAMP(loop-mediated isothermal amplification)system for rapid diagnosis of H5 avian influenza virus infection[J]. Vaccine, 2006, 24(44):6679-6682.
[31] Mori Y, Notomi T. Loop-mediated isothermal amplification(LAMP):a rapid, accurate, and cost-effective diagnostic method for infectious diseases[J]. J Infec Chemother, 2009, 15(2):62-69.
[32]Borst A, Box ATA, Fluit AC. False-positive results and contamination in nucleic acid amplification assays:suggestions for a prevent and destroy strategy[J]. European Journal of Clinical Microbiology and Infectious Diseases, 2004, 23(4):289-299.
[33]Wong ML, Medrano JF. Real-time PCR for mRNA quantitation[J]. Biotechniques, 2005, 39(1):75-85.
(责任编辑 狄艳红)
Visual Detection of Vibrio harveyi Based on Loop-mediated Isothermal Amplification Combined with a Lateral Flow Dipstick
CHENG Die CHAI Fang-chao CAI Yi ZHOU Qian-jin CHEN Jiong
(Department of Biology and Marine Science,Ningbo University,Ningbo 315211)
Based on nucleotide enrichment by a loop-mediated isothermal amplification(LAMP)and chromatographic visualization by a lateral flow dipstick(LFD)assay,this work aims to develop a novel LAMP-LFD method for the rapid detection of Vibrio harveyi. Three pairs of primers were designed using the hemolysin gene(vhhA)of V. harveyi as detection target,and used in LAMP reaction,among which the forward inner primer vhhA-FIP was biotinylated. Similarly,a fluorescein isothiocyanate(FITC)-labeled probe vhhA-HP was designed to specifically hybridize with LAMP products. And then the hybridized LAMP products were visually detected by LFD. The optimized LAMP was performed at 63℃ for 40 min;and visual detection via LFD took 50 min. The results indicated that LAMP-LFD was able to specifically identify V. harveyi from other 9 pathogenic bacteria commonly existing in the aquatic animals,such as V. vulnificus. The detection limit of LAMP-LFD was 1.0×102CFU/mL for V. harveyi pure cultures(equivalent to 2 CFU per reaction),and 5×102CFU/mL for V. harveyi contaminated tissues of large yellow croaker(equivalent to 20 CFU per reaction),both of which were 100 times lower than that of the conventional PCR method using both outer primers vhhA-F3/vhhA-B3. Therefore,this rapid and accurate LAMP-LFD method is a promising alternative in the surveillance and point-of-care test of V. harveyi in sea farming.
Vibrio harveyi;hemolysin;loop-mediated isothermal amplification;lateral flow dipstick;assay detection
10.13560/j.cnki.biotech.bull.1985.2016.06.010
2015-08-31
国家高技术研究发展计划(“863”计划)(2012AA020101),国家星火计划(2014GA701007),浙江省重大科技专项重点社会发展项目(2013C03045-1),宁波市科技创新团队项目(2015C110018),宁波大学学科项目(XKL14D2083)
程蝶,女,研究方向:生物技术;E-mail:17855822426@163.com
周前进,男,博士,讲师,研究方向:动物疫病诊断技术;E-mail:mumu2325@163.com

