家养禽类肠道可培养细菌抗生素抗性的种类、数量和分布
周俊雄, 马荣琴, 李冬松, 田容川, 李敏昱, 罗艺彬, 刘平平, 田宝玉
(1.福建师范大学生命科学学院工业微生物教育部工程研究中心,福建 福州 350108;2.福州时代中学,福建 福州 350007)
家养禽类肠道可培养细菌抗生素抗性的种类、数量和分布
周俊雄1, 马荣琴1, 李冬松1, 田容川2, 李敏昱1, 罗艺彬1, 刘平平1, 田宝玉1
(1.福建师范大学生命科学学院工业微生物教育部工程研究中心,福建 福州 350108;2.福州时代中学,福建 福州 350007)
摘要:采用微生物培养和影印法对家养禽类鸡、鸭和肉鸽肠道可培养细菌抗生素抗性的种类、数量和分布进行了调查.结果表明:在调查的鸡、鸭和肉鸽3种禽类中,肠道可培养细菌抗生素抗性的分布非常普遍,所有测试细菌至少可以抗一种抗生素,抗5种以上抗生素的细菌占测试细菌的比例分别为75.0%、58.9%和97.4%;个别细菌对测试的10种抗生素均具有抗性.在抗生素方面,肠道可培养细菌对萘啶酮酸、盐酸四环素、盐酸克林霉素、磺胺嘧啶和红霉素的抗性分布比较广泛.提取细菌基因组并扩增16S rRNA基因,并对这些细菌进行分子鉴定的结果表明,分离的肠道可培养细菌为埃希氏大肠杆菌.在美国国家生物技术信息中心数据库进一步搜索的结果显示:与肠道可培养细菌同源性大于99%的细菌广泛分布于不同的环境、宿主和病原菌中,表明这些肠道可培养细菌抗生素抗性是一个潜在的环境微生物以及人体病原菌抗生素抗性的传播来源.
关键词:抗生素; 家养禽类; 肠道可培养细菌; 抗药性; 大肠杆菌
抗生素自上世纪40年代被发现并进入临床医疗以来,一直作为抗感染治疗的最主要手段,拯救了千百万人的生命,被誉为人类20世纪最伟大的发现之一[1].在过去的几十年间,新的抗生素不断被发现并进入临床;抗生素的应用范围也由原来的人畜感染控制发展到水产养殖等,还被作为饲料添加剂和促生长剂广泛应用于养殖业并进入环境中,导致环境中抗生素抗性细菌和抗性基因日益增加,从而影响生态环境和人类健康[2- 3].中国是抗生素生产大国,也是抗生素使用大国.根据化学工业学会和制药工业学会2005年的统计数据,我国每年抗生素原料生产量约为21万t,其中很大一部分,约有9.7万t(占46.1%)用于畜牧业[4].研究表明,长期广泛使用抗生素的直接后果很可能引起动物体内抗性基因通过排泄物对养殖区域周边环境造成基因污染,导致环境中病原菌抗生素抗性普遍增强,抗生素的治疗效果日益下降,对人和动物的健康产生严重威胁[2-5],临床感染性疾病的治疗也将面临巨大的挑战.随着抗生素的使用,抗性细菌在数量、多样性以及抗性强度上也都将显著增大.
目前,几乎临床上使用的所有抗生素都有其抗性细菌,多重耐药菌株的检出也相当普遍,甚至出现了能抵抗绝大多数抗生素的“超级细菌”.广泛存在且不断增长的细菌抗生素抗性或将成为我国食品安全和人类健康领域的最主要威胁之一[5].2010年出现的“超级细菌”就是细菌多重抗性的典型实例,其携带有耐药基因NDM-1,能够编码金属β-内酰胺酶,能够水解大多数抗生素,导致由该类细菌引起的感染性疾病难以医治[6-7].广泛存在的抗生素抗性细菌和由此导致的严重危害提出了这样的疑问:如此多样且广泛存在的病原菌抗性从哪里来?为什么短短几十年的抗生素应用会导致环境中病原菌抗性的爆发?
本试验采微生物培养、影印法和细菌16S rDNA分子鉴定等方法,对市场上出售的鸡、鸭和肉鸽3种禽类肠道可培养细菌的抗生素抗性的种类、多样性和生态分布进行了调查,通过搜索美国国家生物技术信息中心(NCBI)数据库同源细菌的环境分布,探索家养禽类肠道细菌的抗生素抗性与环境微生物以及人体病原菌抗性起源和相互传播的相关性.
1材料与方法
1.1材料
1.1.1样品采集试验所用新鲜鸡、鸭和肉鸽粪便均来自当地农贸市场,对于每一种调查的家禽种类,均保证收集的粪便来自5个或5个以上不同的个体.采集的新鲜粪便分别用密封袋密封,用记号笔标记采集地点、采集日期和采集人,置4 ℃的冰箱保存备用.采集的样品在24 h内处理完毕.
固体培养基为LB固体培养基,含10 g胰蛋白胨、5 g酵母浸出粉、10 g NaCl和15 g琼脂粉,以蒸馏水定容至1 L,pH为7.0,于1×105Pa灭菌20 min,备用.
1.2方法
1.2.1样品处理称取新鲜鸡、鸭和肉鸽的粪便各5 g,分别放入盛有25 mL无菌生理盐水并带有3~5粒玻璃珠的三角烧瓶中,旋转振荡使粪便与无菌生理盐水充分混匀,静置5 min.移取1.0 mL粪便上清液进行梯度稀释,分别吸取不同稀释梯度的溶液100 μL,涂布于LB固体平板培养基上,于37 ℃培养24 h.以平板上150~200个菌落为标准,确定稀释梯度.按预试验确定的稀释梯度将不同粪便上清液分别稀释后再次涂布在至少5个LB固体培养基上,于37 ℃培养24 h,观察菌落形态.
1.2.2可培养细菌的分离纯化分别从每个样品稀释平板上随机挑取200~250个单菌落,在平板上划线挑取单克隆.选取的单克隆再次划线,经过两次纯化,保证得到单一纯化的菌株.纯化的菌株分别进行显微观察,按菌落形态、颜色和大小分别归类,并分别接种到LB液体培养基上,于37 ℃、220 r·min-1培养24 h,按20%的比例加入适量甘油,置-20 ℃冰箱保种.
1.2.3抗生素储备液的制备按要求将10种抗生素(表1)制成储备液[8],过滤除菌后置-20 ℃的冰箱中保存备用.
1.2.4影印平板试验分别移取50 μL单菌落菌液于对应的96孔板,备用.分别配置表1中所需工作浓度的抗生素LB固体培养基方形平板,冷却备用.用带有96针的影印工具蘸取适量菌液,分别点于含有10种抗生素的方形LB固体平板上,将平板置于37 ℃培养12~16 h.观察平板菌落的生长情况并计数,统计各菌株对每种抗生素是否具有抗药性,并据此计算各个样品分离的细菌对各种抗生素的抗性比例.抗性比例/%=具有抗性的菌株数/总测试菌株数×100.
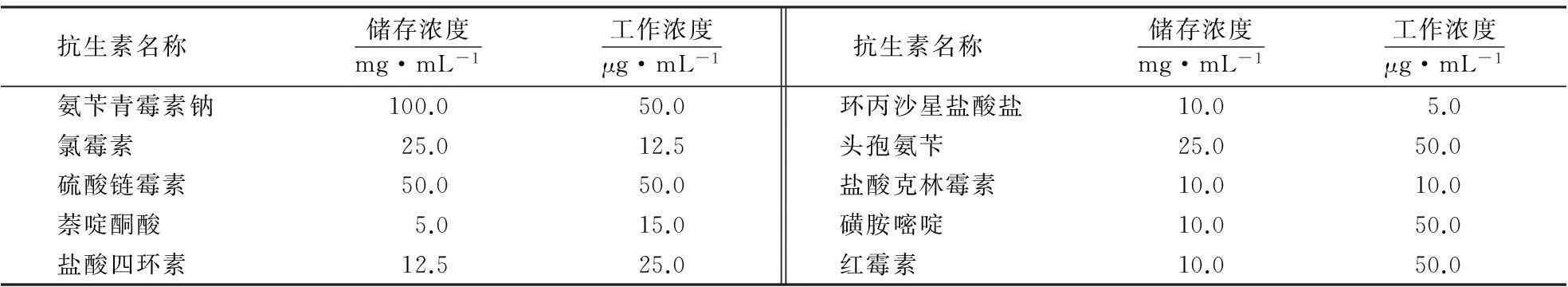
表1 10种抗生素的工作浓度
1.2.5肠道可培养细菌基因组DNA的提取为了确定分离纯化的3种禽类肠道可培养细菌的种类,纯化的细菌菌落根据菌落大小分为2~3类.在每一类中随机挑选5株菌株作为代表性菌株进行基因组提取、16S rRNA基因的PCR扩增以及测序.
比喻要生动形象,才能触动心灵。《生命的“电量”》一课中,丁老师用图片“你的生命还剩多少‘电量’”来比喻:中国人口平均寿命,男性74岁,女性77岁。每个人生命“电量”的余量:“10后”剩 69年,“00后”剩 59年,“90 后”剩 49年,“80后”还有 39年,“70后”还有 29 年,“60后”还有 19年,“50后”还有9年。这个比喻借助图片表现,特别生动形象,让学生有一种紧迫感。而不同年龄段生命“电量”的展示,让孩子产生联想,有更多的思考。
将挑取的菌株接种于LB液体培养基中,于37 ℃、220 r·min-1培养过夜.取1.5 mL菌液,于8 000 r·min-1离心5 min,去上清液.基因组的提取采取改良的酚氯仿法抽提,基本过程如下:在细胞沉淀中加入750 μL裂解缓冲液,枪头吹打混匀后加入少许石英砂和400 μL酚/氯仿,涡旋振荡3 min,于11 000 r·min-1离心5 min,弃沉淀.将上清液转入另一干净的1.5 mL管中,加入等体积的酚/氯仿,翻转5次,静置5 min,于11 000 r·min-1离心5 min.将上清液转入另一干净的1.5 mL管中,按50.0 μg·mL-1的工作浓度加入适量RNase,于37 ℃水浴1 h.之后按80.0 μg·mL-1的工作浓度加入适量蛋白酶K,于55 ℃水浴30 min,再次加入等体积的氯仿翻转5次,静置5 min,于11 000 r·min-1离心5 min.将上清液转入另一干净的1.5 mL管中,加入等体积的异丙醇,在-20 ℃静置1 h,于13 000 r·min-1离心15 min.弃上清液,沉淀用1 mL 70%乙醇洗两次,加入50 μL TE缓冲液(pH 7.5)溶解,置-20 ℃的冰箱中保存备用.提取的基因组DNA采用1%琼脂糖凝胶电泳检测并用超微量分光光度计进行定量.
1.2.6肠道可培养细菌16S rRNA基因的PCR扩增及系统进化树的构建以提取的肠道可培养细菌基因组为模板,采用通用引物27f/1492r进行PCR扩增细菌16S rRNA基因[9].PCR反应条件:95 ℃预变性5 min;94 ℃变性 45 s,56 ℃退火45 s,72 ℃延伸90 s,循环25次;72 ℃延伸10 min.扩增产物纯化后送至生工生物工程(上海)股份有限公司双向测序,获得的序列递交至RDP数据库确定其分类地位.同时在NCBI数据库中对各个序列进行BLAST序列比对,筛选同源序列.将分离纯化的16S rRNA基因序列与下载的同源序列通过SeaView软件的ClustalX程序进行完全比对.编辑后用SeaView软件的Tree程序构建PhyML系统进化树[10].
2结果与分析
2.1鸡肠道可培养细菌抗生素抗性
2.1.1鸡肠道可培养细菌对抗生素的抗性比例研究表明,尽管动物肠道微生物主要由严格或兼性厌氧微生物构成,好氧条件下的细菌在抗生素抗性的获得和传播中扮演了更为重要的作用[11-12].对分离纯化的192株细菌的抗生素抗性进行调查的结果(图1)表明:在192株细菌中,对萘啶酮酸、盐酸四环素、磺胺嘧啶和红霉素表现出较高的抗性比例,分别为70.8%、77.1%、81.3%和71.9%,对磺胺嘧啶的抗性比例最高;对硫酸链霉素和头孢氨苄的抗性比例较低,只有20.3%的细菌对头孢氨苄具有抗性.在鸡肠道可培养细菌中,对抗生素具有抗性的细菌比例与该抗生素在市场中的应用时间相关,一些较早引入市场以及畜牧养殖的抗生素,如磺胺嘧啶和盐酸四环素具有较高的抗性比例;一些新抗生素,如头孢氨苄则抗性比例较低.
2.1.2鸡肠道可培养细菌携带抗生素抗性的数目具有多重抗性的细菌,即“超级细菌”是医疗实践中造成抗生素治疗失败的主要原因.图2表明,192株鸡肠道可培养细菌均对一种或一种以上的抗生素产生抗性,大多数细菌携带抗生素抗性的数目在4~7个之间,携带5个以上抗生素抗性的细菌一共有139株,占总检测细菌数目的72.4%,说明多重耐药细菌在鸡肠道可培养细菌中比较普遍.
2.2鸭肠道可培养细菌抗生素抗性
2.2.1鸭肠道可培养细菌对抗生素的抗性比例对分离纯化的192株细菌的抗生素抗性进行调查的结果(图3)表明:对抗生素具有抗性的细菌在鸭肠道可培养细菌中的分布也较为广泛,但抗性比例明显低于鸡肠道可培养细菌,其中对氯霉素、红霉素、氨苄青霉素钠、磺胺嘧啶和硫酸链霉素的抗性比例相对较高,在50%以上;对头孢氨苄的抗性比例较低,只有29.2%.鸭肠道可培养细菌的抗性比例普遍低于鸡肠道可培养细菌的主要原因可能与鸭的饲养环境相对开放有关,流动的水生环境避免了抗性细菌在鸭饲养环境中的进一步富集;另一方面,这些抗性细菌也可能在更为广泛的环境中进行扩散和传播.
2.2.2鸭肠道可培养细菌携带抗生素抗性的数目图4表明:192株鸭肠道可培养细菌均对一种或一种以上的抗生素产生抗性,大多数细菌携带抗生素抗性的数目在4~6个之间;携带4个以上抗生素抗性的细菌一共有150株,占总检测细菌数目的78.1%,携带5个以上抗生素抗性的细菌占总检测细菌数目的58.9%,有3株细菌对9种抗生素表现出明显的抗性.
2.3肉鸽肠道可培养细菌抗生素抗性
2.3.1肉鸽肠道可培养细菌对抗生素的抗性比例从分离纯化的肉鸽肠道可培养细菌中随机挑取240株菌株用于抗生素抗性调查.结果(图5)表明:在调查的3种禽类中,肉鸽肠道可培养细菌对抗生素具有抗性的细菌比例最高,对10种抗生素的抗性比例均在80%以上,其中对磺胺嘧啶和盐酸克林霉素的抗性比例最高,分别达到了90.8%和93.3%;同样,对一些目前临床上尚在应用一线的新抗生素,如头孢类抗生素,抗性比例也达到了82.1%,这与其他动物肠道中绝大多数细菌对头孢类抗生素较为敏感不一致.肉鸽肠道中高抗性细菌比例可能主要与其较短的饲养周期有关.肉鸽的饲养周期一般在45 d左右,而乳鸽只需25~30 d哺喂即可上市.较短的饲养周期可能导致较高的抗生素残留以及较高的抗性比例.这个结果与先前对蜜蜂肠道可培养细菌抗生素抗性的调查结果是一致的,新购自于种蜂场的蜂群肠道细菌对抗生素具有较高的抗性比例[9].
2.3.2肉鸽肠道可培养细菌携带抗生素抗性的数目图6表明:与高的抗性细菌比例一致,肉鸽肠道可培养细菌也携带有较多的抗生素抗性种类,多种抗性更为普遍;240株细菌均对3种或3种以上的抗生素产生抗性,携带5种及5种以上抗生素抗性的细菌占总检测细菌数目的97.4%,携带7种以上抗生素抗性的细菌占总检测细菌数目的85%,说明多重抗性细菌在肉鸽肠道中分布非常普遍,共有66株细菌对10种抗生素均表现出抗性.
2.4肠道可培养细菌的分子鉴定及系统进化树的构建
3种禽类肠道可培养细菌根据其菌落形状、颜色和大小分为2~3类,在每一类中随机挑选5株细菌进行基因组提取,每个样品挑取10~15株细菌作为代表性菌株.以提取的基因组DNA为模板,采用通用引物27f/1492r扩增细菌16S rRNA基因.纯化回收后的16S rRNA基因经测序,最终有21个样品双向测序成功并得到全长序列,测序成功的序列递交RDP数据库进行分子鉴定.鉴定结果表明,3种禽类肠道可培养细菌均属于肠杆菌科埃希氏大肠杆菌.序列比对结果表明,大部分同一来源的细菌16S序列完全一致.在随后的分析中将序列完全一致的细菌合并,最终确定8株作为代表性菌株,包括5株鸡肠道可培养细菌(鸡_C20、 鸡_J29、 鸡_J79、 鸡_C4、 鸡_C161)、2株鸭肠道可培养细菌(鸭_110、鸭_10)和1株鸽肠道可培养细菌(鸽_G2).8株代表性菌株的16S rRAN基因全长序列用于下一步构建系统进化树.
将8株代表性菌株的16S rRNA基因序列在NCBI nt数据库进行比对,选取并下载序列同源以及肠杆菌科主要属典型菌株的16S rRNA基因序列,采用SeaView软件的ClustalX程序将目的序列与已知的肠杆菌科16S rRNA基因序列进行比对,编辑后用SeaView软件的Tree程序构建PhyML系统进化树(图7).从图7可以看出,3种禽类可培养细菌都聚集在肠杆菌科的埃希氏菌属—志贺氏菌属分枝上,显示了这些来源于不同家禽并携带有多种耐药性的细菌高度同源,彼此间具有很近的亲缘关系.
2.5肠道可培养细菌在环境中的分布及其抗性的潜在传播途径
为了调查3种禽类肠道可培养细菌在环境中可能的分布及其抗性的潜在传播途径,本试验收集和统计了NCBI数据库中与肠道可培养大肠杆菌序列具有高度同源性的细菌16S rRNA基因序列(序列同源性>99%),并统计了这些序列的来源与分布信息.结果表明,NCBI数据库中报道了大量的与肠道可培养细菌16S rRNA基因序列同源性大于99%的细菌,这些细菌广泛分布于世界各地,以亚洲、欧洲和北美洲分布最多,且分布在不同环境中,从沿海到内陆,从表层海水到海底热泉沉积物,从热带到寒带均有分布.根据这些细菌所分布的环境以及这些环境之间的相互关联,构建了细菌在不同环境之间可能存在的交流和传播网络(图8).
前期研究表明,病原菌主要是通过基因水平转移从同一或亲缘关系较近的携带有抗性基因的细菌类群中获得抗性基因.动物肠杆菌类细菌很可能是抗性基因传播以及人体病原菌获得抗性基因的重要载体.
一方面,大肠杆菌具有非常广泛的分布范围,能够适应从动物肠道到自然的土壤、水等多样性的环境,而频繁的人口流动为这些细菌的广泛传播提供了便利条件;其次,Sommer et al[13]研究表明,病原菌携带的抗性基因与肠道可培养细菌(通常为兼性厌氧细菌,或者是机会致病菌)以及环境中的同类细菌具有高度的同源性,说明病原菌可能从肠道可培养细菌获得抗性基因以及这些抗性基因的环境来源.因此,环境中不同来源的大肠杆菌相互传播和交流的网络也很有可能成为耐药性基因在不同环境微生物和人类病原菌相互传播的潜在途径[3].
3讨论
长期以来,畜禽养殖过程中抗生素的使用一直被认为是除医疗应用之外,病原菌和环境微生物耐药性增加的另一个重要原因[2-3,12-14].近年来随着畜牧养殖集约化发展,抗生素滥用情况日益严重,直接后果很可能导致在动物肠道微生物中产生丰富的抗性耐药菌,这些抗性菌株通过排泄物及畜牧活动扩散到环境中,并在环境中传播扩散[2,5,13-15].
本试验结果表明:3种禽类肠道存在普遍而种类繁多的抗抗生素细菌;所有测试细菌至少对一种或一种以上的抗生素具有抗性,不同来源的动物肠道细菌对不同种类抗生素的抗性具有明显差异.鸡肠道的大多数细菌对萘啶酮酸、盐酸四环素、盐酸克林霉素、磺胺嘧啶和红霉素具有较高的抗性,对头孢氨苄比较敏感;鸭肠道的大多数细菌对氨苄青霉素钠、氯霉素、硫酸链霉素、磺胺嘧啶和红霉素具有抗性,对环丙沙星盐酸盐比较敏感;而肉鸽肠道的大多数细菌对测试的10种抗生素均具有明显的抗性.3种禽类肠道中抗抗生素细菌的比例以及这些细菌对不同抗生素抗性的差异可能与家禽的饲养方式以及抗生素的使用时间有关.在3种禽类中,鸭的饲养环境相对较为开放,饲养周期长,避免了抗性细菌在一个较为狭小环境下的富集,因此抗性比例和抗抗生素的数目都较低;而肉鸽饲养环境狭小,大肠杆菌在环境、饮用水、饲料和灰尘中高度富集,且肉鸽饲养周期短,只有25~30 d,在饲养过程中一旦使用抗生素会有较高的残留而导致抗性细菌的富集[16-17].
从3种禽类粪便中分离得到的细菌菌株经分子鉴定均为大肠杆菌,这些菌株的16S rRNA基因序列相互之间的同源性大于99%.前期研究表明,尽管大肠杆菌以及其他肠杆菌科细菌在肠道微生物中所占的比例不到1%,但长期以来这些兼性厌氧细菌一直被认为是抗生素抗性细菌的一个主要来源和传播介质[18-19].一个重要的原因是这些细菌在环境中广泛分布,从土壤、水生环境到肠道环境都可以检测到这些细菌的存在,因此,大肠杆菌也被广泛用作检测食品和饮用水是否污染的一个重要指标[19];另一个原因是这些细菌携带大量种类丰富的抗生素抗性基因[15,19].本试验中,鸡、鸭和肉鸽肠道可培养细菌大多数具有多重耐药性,携带5种或5种以上抗生素抗性的细菌分别占总检测细菌数目的72.4%、58.8%和97.5%.前人对不同来源大肠杆菌耐药性的调查也证实了这一点.如宋立等[20]对我国1970~1990年和2000年收集的326株食品动物大肠杆菌的抗性调查表明,食品动物大肠杆菌对青霉素类、四环素类、氨基糖甙类、氟喹诺酮类、磺胺类和氯霉素类抗生素都有较高的耐药率.而据国家细菌耐药性监测中心对1986年分离的71株和2001年分离的86株正常猪粪便大肠杆菌抗生素耐药性的调查表明,这些菌株对环丙沙星、氯霉素、四环素、复方磺胺嘧啶、链霉素和庆大霉素等都有明显的耐药性[21].多抗耐药细菌的出现可能与不同的抗生素抗性基因之间存在连锁现象有关.研究表明,环境中存在种类繁多且浓度较高的抗生素时,一种抗生素对其他种类的抗生素抗性基因可能具有协同诱导作用[22].Brenciani et al[23]发现,在125株携带大环内脂类抗生素抗性基因erm(B)的酿脓葡萄球菌中,有117株也携带四环素抗性基因tet(M),并证明基因erm(B)和tet(M)是连锁的.Levy et al[15]发现,用添加亚治疗剂量土霉素的饲料喂养的鸡的排泄物中,大肠杆菌不仅具有土霉素抗性,还具有链霉素、磺胺嘧啶和氨苄青霉素抗性.因此,分布普遍而又携带广泛耐药性的肠杆类细菌已经成为近年来研究细菌耐药性进化和传播的主要关注热点.
微生物耐药性可以通过垂直和水平转移两种机制在微生物间传播.垂直转移指抗性基因由亲代传给子代,具有典型的种属特异性;水平转移则是指抗性基因通过转导、接合、转座和转化作用在亲缘关系较近的微生物之间传播.研究表明,即使在几乎没有人为影响的环境中也存在多种抗生素抗性细菌,其所携带的抗性基因与临床上发现的致病菌携带的抗性基因具有高度的一致性,这主要就是抗性基因之间发生水平转移的结果[12,15,24-25].然而,研究表明抗性基因的水平转移主要发生在亲缘关系较近的种内或种间.如尽管在人类肠道微生物占绝对优势的未培养厌氧细菌(如拟杆菌等)也携带许多抗性基因.序列分析表明,病原菌的抗性基因与那些可培养的兼性厌氧菌所携带的抗性基因序列更相似.Sommer et al[13]通过分析人体肠道微生物中可培养细菌携带的抗性基因序列(主要是肠杆菌)以及通过功能宏基因组学手段得到的未培养细菌的抗性基因序列发现,病原菌(如肺炎克雷伯氏菌、大肠杆菌和沙门氏菌)与可培养细菌携带的抗性基因序列具有高度的一致性,而与未培养细菌的抗性基因序列的同源性很低,说明病原菌主要通过水平转移的方式从可培养的亲缘关系较近的微生物中获得抗性基因.在本试验中,不仅从3种不同饲养环境的禽类肠道细菌中分离得到的大肠杆菌菌株高度同源,而且这些细菌广泛分布于不同的环境,包括猪、牛、鸡等各种动物粪便,土壤,水生环境中,表明微生物在不同动物以及不同环境之间相互传播,而且其所携带的抗性基因也可能随着这些传播途径和传播网络进行扩散和传播.
参考文献
[1] AMINOV R I. A brief history of the antibiotic era: lessons learned and challenges for the future [J]. Frontiers in Microbiology, 2010,1(1):107-113.
[2] COLOMER-LLUCH M, IMAMOVIC L, JOFRE J, et al. Bacteriophages carrying antibiotic resistance genes in fecal waste from cattle, pigs, and poultry [J]. Antimicrobial Agents and Chemotherapy, 2011,55(10):4908-4911.
[3] Frank M, AARESTRUP F M. Veterinary drug usage and antimicrobial resistance in bacteria of animal origin [J]. Basic & Clinical Pharmacology & Toxicology, 2005,96:271-281.
[4] 任成山,赵晓晏.“超级细菌”的由来和未来[J].中华肺部疾病杂志(电子版),2010,3(5):54-58.
[5] 罗义,周启星.抗生素抗性基因(ARGs)——一种新型环境污染物[J].环境科学学报,2008,28(8):1499-1505.
[6] YONG D, TOLEMAN M A, GISKE C G, et al. Characterization of a new metallo-β-lactamase gene, blaNDM-1, and a novel erythromycin esterase gene carried on a unique genetic structure inKlebsiellapneumoniaesequence type 14 from India [J]. Antimicrobial Agents and Chemotherapy, 2009,53(12):5046-5054.
[7] ADDISON J B. Antibiotics in sediments and run-off waters from feedlots [J]. Residue Reviews, 1984,92:1-28.
[8] ALLEN H K, MOE L A, RODBUMRER J, et al. Functional metagenomics reveals diverse-lactamases in a remote Alaskan soil [J]. ISME Journal, 2009,3(2):243-251.
[9] TIAN B Y, FADHIL N H, POWELL J E, et al. Long-term exposure to antibiotics has caused accumulation of resistance determinants in the gut microbiota of honeybees [J]. mBio, 2012,3(6):e00377-12.
[10] GOUY M, GUINDON S, GASCUEL O. SeaView version 4: a multiplatform graphical user interface for sequence alignment and phylogenetic tree building [J]. Molecular Biology and Evolution, 2010,27(2):221-224.
[11] KEMPER N. Veterinary antibiotics in the aquatic and terrestrial environment [J]. Ecological Indicators, 2008,8(1):1-13.
[12] HEUER H, SCHMITT H, SMALLA K. Antibiotic resistance gene spread due to manure application on agricultural fields [J]. Current Opinion in Microbiology, 2011,14(3):236-243.
[13] SOMMER M O, DANTAS G, CHURCH G M. Functional characterization of the antibiotic resistance reservoir in the human microflora [J]. Science, 2009,325:1128-31.
[14] CAPRIOLI A, BUSANI L, MARTEL J L, et al. Monitoring of antibiotic resistance in bacteria of animal origin: epidemiological and microbiological methodologies [J]. International Journal of Antimicrobial Agents, 2000,14:295-301.
[15] LEVY S B, FITZGERALD G B, MACONE A B. Changes in intestinal flora of farm personnel after introduction of a tetracycline-supplemented feed on a farm [J]. New England Journal of Medicine, 1976,295:583-588.
[16] 赵宝华,卜柱,徐步,等.肉鸽大肠杆菌的分离与鉴定[J].经济动物学报,2010,14(4):226-227.
[17] 柏文芳,伍清林,金兰梅,等.肉鸽大肠杆菌的分离鉴定与耐药性的研究[J].家畜生态学报,2013,34(3):58-61.
[18] BAILEY J K, PINYON J L, ANANTHAM S, et al. CommensalEscherichiacoliof healthy humans: a reservoir for antibiotic-resistance determinants [J]. Journal of Medical Microbiology, 2010,59:1331-1339.
[19] MARSHALL B M, OCHIENG D J, LEVY S B. Commensals: underappreciated reservoir of antibiotic resistance [J]. Microbe, 2009,5(4):231-238.
[20] 宋立,宁宜宝,沈建忠,等.中国不同年代食品动物大肠杆菌耐药性调查研究[J].中国科学C辑:生命科学,2009,39(7):692-698.
[21] 金少鸿,马越.国内细菌耐药性监测及研究进展[J].中国新药杂志,2005,14(3):265-268.
[22] 杨凤霞,毛大庆,罗义,等.环境中抗生素抗性基因的水平传播扩散[J].应用生态学报,2013,24(10):2 993-3 002.
[23] BRENCIANI A, BACCIAGLIA A, VECCHI M, et al. Genetic elements carrying erm (B) inStreptococcuspyogenesand association with tet (M) tetracycline resistance gene [J]. Antimicrobial Agents and Chemotherapy, 2007,51:1 209-1 226.
[24] FORSBERG K J, REYES A, WANG B, et al. The shared antibiotic resistome of soil bacteria and human pathogens [J]. Science, 2012,337:1 107-1 111.
[25] DAVIES J, DAVIES D. Origins and evolution of antibiotic resistance [J]. Microbiology and Molecular Biology Reviews, 2010,74:417-433.
(责任编辑:施晓棠)
Diversity and distribution of antibiotic resistance for gut culturable bacteria from domestic poultry
ZHOU Junxiong1, MA Rongqin1, LI Dongsong1, TIAN Rongchuan2,LI Minyi1, LUO Yibin1, LIU Pingping1, TIAN Baoyu1
(1.Engineering Research Center of Industrial Microbiology of Ministry of Education, College of Life Science, Fujian Normal University, Fuzhou, Fujian 350108, China; 2.Fuzhou Era Middle School, Fuzhou, Fujian 350007, China)
Abstract:The wide-use, misuse and even abuse of antibiotics in recent years have led to an increase in the resistance of environmental and pathogenic microorganisms to antibiotics. In this study, diversity and ecological distribution of antibiotic resistances for culturable intestinal bacteria from domestic poultry, including chicken, duck and pigeon, were investigated using traditional microculture and replica plating techniques. Results showed that all the isolated bacteria were resistant to at least 1 of the 10 tested antibiotics. Proportions of bacteria which were resistant to greater than or equal to 5 antibiotics were 75%, 58.9% and 97.4% for chicken, duck and pigeon, respectively. And 66 out of 192 (34%) isolates were resistant to all the antibiotics. Moreover, a variety of bacteria, with the highest overall proportion in pigeon and lowest in duck, showed resistance to nalidixic acid, tetracyyline, clindamycin, sulfadiazine and erythromycin. In order to identify bacteria that presentd high and multi-drug antibiotic resistance, 16S rRNA genes of 8 representative strains were amplified and followed by phylogenetic analysis. It turned out that the bacteria were grouped into the branch of Escherichia coli under the family of Enterobacterium. Referring to database from National Center of Biotechnology Information (NCBI), sequences with 99% similarity with the 8 strains were widely distributed in a variety of environment, including soil, animal host and pathogenic bacteria. Results indicated that culturable gut bacteria from domestic poultry was a potential source of antibiotic resistance for environmental microbiota and human pathogenic bacteria.
Key words:antibiotics; domestic poultry; culturable intestinal bacteria; resistance; Escherichia coli
DOI:10.13323/j.cnki.j.fafu(nat.sci.).2016.01.010
中图分类号:X171
文献标识码:A
文章编号:1671-5470(2016)01-0056-09
作者简介:周俊雄(1989-),男,硕士研究生.研究方向:应用与环境微生物.Email:zhoujunxiong@sohu.com.通讯作者田宝玉(1973-),男,副教授.研究方向:环境微生物.Email:tianby@fjnu.edu.cn.
基金项目:国家自然科学基金资助项目(31170108);福建省杰出青年科学基金项目(2009J06014).
收稿日期:2015-01-23修回日期:2015-09-16

