烟草青枯菌FQY_4宿主特异性候选基因分析
蔡刘体,陆 宁,沈子霞,石俊雄
(1.贵州省烟草科学研究院,贵州贵阳550081;2.贵州省烟草公司遵义县公司,贵州遵义563003)
烟草青枯菌FQY_4宿主特异性候选基因分析
蔡刘体1,陆 宁1,沈子霞2,石俊雄1
(1.贵州省烟草科学研究院,贵州贵阳550081;2.贵州省烟草公司遵义县公司,贵州遵义563003)
为青枯菌与烟草的相互作用研究提供宿主特性候选基因,在烟草青枯菌FQY_4基因组测序的基础上,采用基因组及同源基因比较,分析青枯菌株系GMI1000、Po82、CFBP2957、CMR15、PSI07和FQY_4的核心基因及FQY_4宿主特异性候选基因。结果表明:FQY_4基因组中有632个宿主特异性候选基因,其中染色体、巨大质粒分别有365个和267个,包括跨膜蛋白、信号蛋白和细胞壁水解相关的基因,移动元件蛋白和许多未知功能的蛋白基因,以及宿主特异性候选三型分泌系统的42个效应蛋白因子(T3Es)。
烟草;青枯菌;青枯病;宿主;基因
青枯菌〔Ralstonia solanacearum(Pseudomonas solanacearum)〕是一种土传性的细菌性病原菌,属于革兰氏阴性菌。青枯菌种性非常复杂[12],按照种系型(Phylotype)划分框架及对应的地理起源信息,青枯菌可分为4个种系型:种系型Ⅰ和Ⅱ分别由亚洲和美国菌株组成,种系型Ⅲ的成员由非洲菌株组成,种系型Ⅳ包括来自印度尼西亚和澳大利亚的R.syzygii和BDB[23]。
青枯菌是一个复杂的种,遗传多样性丰富[45],青枯菌宿主众多,包含50多个科中的200多种植物,其中包括重要的农作物烤烟、马铃薯、西红柿、辣椒和香蕉等[67]。由青枯菌侵染导致的烟草青枯病是危害烟草的主要病害之一[8],在与宿主相互作用的长期进化中,形成了宿主适应性的一些特异性基因。
在病原菌-宿主相互作用的长期进化过程中,青枯菌复杂种获得了一些宿主特异性基因。Genin[9]综述了青枯菌株系适应植物寄主的分子特征,包括青枯菌适应宿主进化过程中获得或丢失的三型分泌系统的效应蛋白因子。Guidot等[10]通过多宿主演化试验研究了青枯菌适应植物宿主的相关基因,包括青枯菌在适应的宿主起作用的efpR基因,Ailloud等[11]通过基因组比较方法分析了宿主分别为香蕉和马铃薯青枯菌株系的宿主特异性候选基因。
随着生物技术和测序技术的发展,近年来已经报道青枯菌多个株系的全基因组的测序,包括青枯菌株系GMI 1000[12]、Po 82[13]和CFBP 2957[14]等,这些青枯菌株系全基因组测序信息的解析和积累,有助于分析青枯菌复杂种中株系宿主的特异性基因,有助于青枯菌与其宿主相互作用的研究。为给青枯菌与烟草的相互作用研究提供宿主特性候选基因,在完成FQY_4基因组测序的基础上,通过基因组比较分析了FQY_4基因组中宿主特异性候选基因。
1 材料与方法
1.1 材料
烟草青枯菌FQY_4,全基因组序列通过Hiseq 2 000高通量测序完成,FQY_4基因组在GenBank的登录号分别是CP004012和CP004013[15]。其他青枯菌株系的种系型及基因组信息从GenBank数据库中获取(表1)。

表1 青枯菌株系种系型及宿主信息Table1 The phylotypes and hosts information of R.solanacearumstrains
1.2 基因组比较分析
基因组比较及烟草青枯菌FQY_4基因组和巨大质粒上宿主特异性基因分析采用OMA algorithm和honology blast比较法;烟草青枯菌FQY_4三型分泌系统的宿主特异性效应蛋白因子分析采用ScanYourGenome(网址:http://iant.toulouse.inra.fr/T3E)和T3Es数据库分析比对法。
2 结果与分析
2.1 FQY_4基因组染色体上宿主特异性候选基因
从图1可看出,不同演化型的青枯菌6个株系具有核心基因2 932个,但是也有菌株自身特有的基因,特异性基因数目因青枯菌株系的不同而存在差异,其中菌株PSI07有自身特异性基因407个。这些特异性基因可能是在病原菌-宿主相互作用的长期进化过程中形成,也是宿主特异的候选基因。由表2可知,烟草青枯菌FQY_4染色体上具有365个宿主特异性候选基因,其中包括与青枯菌侵染宿主相关的跨膜蛋白、信号蛋白和细胞壁水解相关的酶,还有许多功能未知的蛋白。
2.2 FQY_4巨大质粒上宿主特异性候选基因
从图2看出,5个青枯菌株系的巨大质粒共有的核心基因数有928个,特异性基因数目因青枯菌株系的不同有差异,烟草青枯菌FQY_4有自身特异性基因267个,与同是种系型I的GMI 1000株系的巨大质粒自身特有基因数目(261个)相差较小。由表3可知,烟草青枯菌FQY_4巨大质粒上宿主特异性候选基因包括与侵染宿主相关的多糖降解酶、效应蛋白因子及许多未知功能的蛋白。

图1 青枯菌株系(6个)染色体基因组核心基因Fig.1 The core genes on chromosome of six R.solanacearumstrains
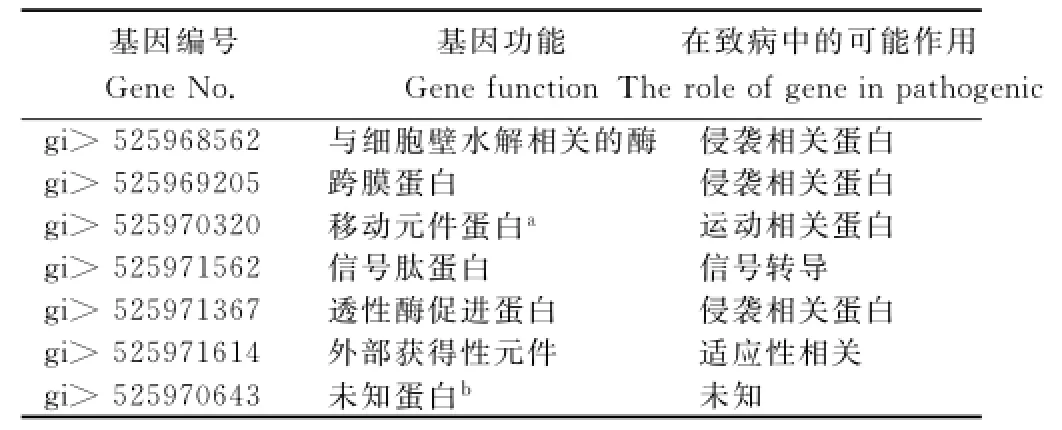
表2 部分青枯菌FQY_4基因组上部分宿主特异性候选基因信息Table2 Part of candidate genes on chromosome of FQY_4strains for host specificity
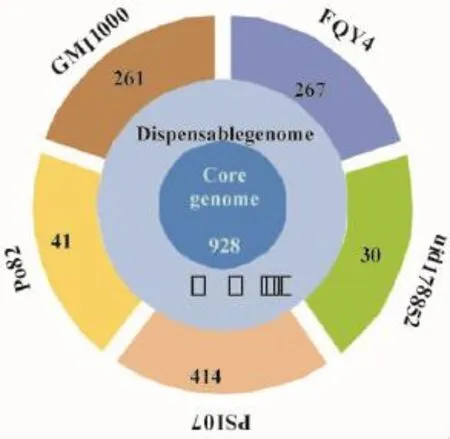
图2 青枯菌株系(5个)巨大质粒基因组核心基因示意图Fig.2 The core genes on huge plasmid of five R.solanacearumstrains

表3 部分青枯菌FQY_4巨大质粒上宿主特异性候选基因信息Table3 Part of candidate genes on huge plasmid of FQY_4strains for host specificity

表4 青枯菌株系的T3Es数目比较Table4 The comparison of T3Es number in different R.solanacearumstrains

表5 烟草青枯菌FQY_4的宿主特异性候选T3Es信息Table5 The information of candidates T3Es in R.solanacearumFQY_4for host specificity
2.3 三型分泌系统中宿主特异性效应蛋白因子
青枯菌三型分泌系统中有丰富的效应蛋白因子(T3Es)[17],不同种系型的青枯菌株系有T3Es 60~90个,其中核心T3Es有32个[1718]。从表4可知,烟草青枯菌FQY_4有宿主特异性候选T3Es 42个。由表5可知,烟草青枯菌FQY_4的宿主名称、位置信息和部分功能信息。从功能角度看,部分T3Es具有酶的功能,如乙酰转移酶、泛素连接酶和果胶裂解酶等,由此推测较多的核心T3Es功能可能与青枯菌复杂种的进化过程有关,但大部分T3Es的功能尚有待于进一步研究验证,尤其是其中的假基因拷贝是否有其他生物学功能或作用,目前仍不清楚。
3 结论与讨论
试验结果显示,不同宿主的青枯菌株系的宿主特异性候选基因数有较大差异,FQY_4染色体上有632个候选基因(染色体上有365个,巨大质粒上有267个),而同是种系型I但宿主不同的GMI 1000株系宿主特异性候选基因为286个(染色体上25个,巨大质粒上261个);宿主同为西红柿的青枯菌株系GMI 1000、CFBP 2957、CMR 15和PSI 07的宿主特异性候选基因数也有较大差异,说明青枯菌对其宿主有其自身一些分子特征[9]。
青枯菌的三型分泌系统(the type III secretion system,T3SS)及效应蛋白因子(effector proteins,T3Es)在青枯菌侵染致病过程中起重要作用[1921]。Genin等[20]预测青枯菌能产生70~80个T3Es,其对宿主的适应性有较大贡献,Ailloud等[11]分析了青枯菌核心T3Es中的14个效应蛋白因子RipG5、RipB、RipW、RipAC、RipAB、RipR、RipE1、RipAM、RipAN、RipAY、RipAJ、RipF1、RipAI和PopC-like,说明在青枯菌复杂种与其宿主的相互作用中,可能出现了数百次效应蛋白因子的获得和丢失事件。烟草青枯菌FQY_4宿主特异性候选效应蛋白因子是否在青枯菌与烟草相互作用的进化过程中出现获得和丢失事件有待于进一步分析和研究。
青枯菌宿主特异性基因的确定及其在于宿主相互作用中所起的作用,需要更多的不同种系型、不同地里起源及不同最适宿主的青枯菌株系的全基因组信息和数据提供支持。另外,青枯菌中致病基因复杂网络调控的机理及功能,在培养基中已经有大量的报道和阐述,但是在致病过程中,这些网络调控在机理中的功能知之甚少[2223]。随着越来越多不同种系型青枯菌株系测序的完成[2425]和青枯菌众多致病因子及其调控途径和网络的阐明[2627],预计能更有效地获得青枯菌这个复杂种中的核心基因和各自的宿主特异性基因,为青枯菌与其宿主相互作用的研究提供更多的基因信息。
[1]HAYWARD A C.Characteristics of Pseudomonas solanacearum[J].Journal of Applied Microbiology,1964,27(2):265-277.
[2]FEGAN M,PRIOR P.How complex is the Ralstonia solanacearumspecies complex[M]//ALLEN C,PRIOR P.Bacterial wilt disease anDThe Ralstonia solanacearumspecies complex.St.Paul,MN:American Phytopathological Society Press,2005:449-461.
[3]DENNY T P.Plant pathogenic Ralstonia species[M]//Gnanamanickam SS.Plant-Associated Bacteria.Dordrecht,The Netherlands:Springer Publishing,2006:573-644.
[4]HAYWARD A C.Biology and epidemiology of bacterial wilt caused by Pseudomonas solanacearum[J].Annual Review of Phytopathology,1991,29:65-87.
[5]GENIN S,DENNY T P.Pathogenomics of the Ralstonia solanacearumspecies complex[J].Annual review of phytopathology,2012(50):67-89.
[6]GENIN S.Molecular Traits Controlling Host Range And Adaptation To Plants In Ralstonia Solanacearum[J].New Phytologist,2010,187(4):920-928.
[7]GUIDOT A,WEI J,FERDY J B,et al.Multihost experimental evolution of the pathogen Ralstonia solanacearum unveils genes involved in adaptation to plants[J].Molecular Biology &Evolution,2014,31(11):2913-2928.
[8]AILLOUD F,LOWE T,CELLIER G,et al.Comparative genomic analysis of Ralstonia solanacearum reveals candidate genes for host specificity[J].Bmc Genomics,2015,16(1):1-11.
[9]SALANOUBAT M,GENIN S,ARTIGUENAVE F,et al.Genome sequence of the plant pathogen Ralstonia solanacearum[J].Nature.2002,415:497-502.
[10]XU J,ZHENG H J,LIU L,et al.Complete genome sequence of the plant pathogen Ralstonia solanacearumstrain Po82[J].J Bacteriol,2011,193(16):4261-4262.
[11]REMENANT B,COUPAT-GOUTALAND B,GUIDOT A,et al.Genomes of three tomato pathogens within the Ralstonia solanacearumspecies complex reveal significant evolutionary divergence[J].BMC Genomics,2010,11(1):379.
[12]CAO Yi,TIAN Baoyu,LIU Yanxia,et al.Genome sequencing of Ralstonia solanacearum FQY_4,isolated from a bacterial wilt nursery used for breeding crop resistance[J].Genome Announcements,2013,1(3):13.
[13]REMENANT B,DE CAMBIAIRE J C,CELLIER G,et al.Ralstonia syzygii,the blood disease bacterium and some Asian R.solanacearumstrains form a single genomic species despite divergent lifestyles[J/ OL].PloS One,2011,6(9):e24356[2016-03-25].http://www.plosone.org/article/info%3Adoi% 2F10.1371%2Fjournal.pone.0024356#pone-0024356-g004.
[14]PEETERS N,CARR RE S,ANISIMOVA M,et al.Repertoire,unified nomenclature and evolution of the Type III effector gene set in the Ralstoniaso lanacearumspecies complex[J].BMC genomics,2013,14(1):859.
[15]蔡刘体,刘艳霞,孟琳,等.烟草青枯菌FQY_4基因组中原噬菌体生物信息学分析[J].中国烟草学报,2015,21(1):82-88.
[16]朱贤朝,王彦亭,王智发.中国烟草病虫害防治手册[M].北京:中国农业版社,2001.
[17]徐进,冯洁.植物青枯菌遗传多样性及致病基因组学研究进展[J].中国农业科学,2013,46(14):2902-2909.
[18]周训军,杨玉文,王静,等.烟草青枯菌遗传多样性分析[J].中国烟草学报,2014,20(4):69-74.
[19]CORNELIS G R,VAN Gijsegem F.Assembly and function of type III secretory systems[J].Annu Rev Microbiol,2000,54(1):735-774.
[20]GENIN S,BOUCHER C.LessonSLearned from the genome analysis of Ralstonia solanacearum[J].Annu Rev Phytopathol,2004,42(9):107-134.
[21]BüTTNERTTNER D,BONAS U.Getting acrossbacterial type III effector proteins on their way to the plant cell[J].EMBO J,2002,21(20):5313-5322.
[22]GENIN S,BRITO B,DENNY T P,et al.Control of the Ralstonia solanacearumType III secretion system(Hrp)genes by the global virulence regulator PhcA[J].FEBSLett,2005(579):2077-2081.
[23]YOSHIMOCHI T,HIKICHI Y,KIBA A,et al.The global virulence regulator PhcA negatively controls the Ralstonia solanacearumhrp regulatory cascade by repressing expression of the PrhIR signaling proteins[J].J Bacteriol,2009,191(10):3424-3428.
[24]LI Zefeng,WU Sanling,BAI Xuefei,et al.Genome sequence of the tobacco bacterial wilt pathogen Ralstonia solanacearum[J].Journal of Bacteriology,2011,193(21):6088-6089.
[25]RAMESH R,GAITONDE S,ACHARI G,et al.Genome Sequencing of Ralstonia solanacearum Biovar 3,Phylotype I,Strains Rs-09-161and Rs-10-244,I-solated from Eggplant and Chili in India[J/OL].Genome Announcements,2014,2(3):e00323-14[2016-03-25].http://europepmc.org/abstract/MED/24874667.
[26]ZULUAGA A P,SOL M,LU H,et al.Transcriptome responses to Ralstonia solanacearum infection in the roots of the wild potato Solanum commersonii.[J].BMC Genomics,2015,16(1):246-262.
[27]CAI H,SHENG Y,YAN Y,et al.CaWRKY6transcriptionally activates CaWRKY40,regulates Ralstonia solanacearum resistance,and confers high-temperature and high-humidity tolerance in pepper[J].Journal of Experimental Botany,2015,66(11):3163-3174.
(责任编辑:孙小岚)
Analysis of Candidate Genes of Ralstonia solanacearum FQY_4for Host Specificity
CAI Liuti1,LU Ning1,SHEN Zixia2,SHI Junxiong1
(1.Guizhou Tobacco Research Institute,Guiyang,Guizhou550081;2.Zunyi Branch of Guizhou Tobacco Company,Zunyi,Guizhou563003,China)
In order to obtain the candidate genes of tobacco R.solanacearumfor host specificity,based on the sequenced genome of FQY_4,the core genes and special genes of GMI1000,Po82,CFBP2957,CMR15,PSI07and FQY_4strains were analyzeDThrough the methods of genome comparative and homology blast.The results showeDThat there are 632candidate genes for its host specificity in the FQY_4genome.Among them 365and 267candidate genes on its chromosome and huge plasmid respectively,including the genes relateDTo trans-membrane proteins,signal proteins and cell wall hydrolysis,the genes relateDTo mobile element proteins,many unknown functional proteins and 42effector proteins(T3Es).
tobacco;Ralstonia solanacearum;bacterial wilt;host;gene
S572
A
1001-3601(2016)10-0411-0005-04
2016-04-25;2016-09-27修回
中国烟草总公司项目“烟株根际土壤微生物施肥响应及调控技术研究”(11020142009);中国烟草总公司贵州省分公司项目“烟草青枯菌致病基因家族分析”(黔烟合201203),“青枯菌噬菌体的筛选及其防控烟草青枯病的探索”(黔烟合201203)
蔡刘体(1974-),男,副研究员,博士,从事土肥植保工作。E-mail:cailiuti01@163.com

